G群轮状病毒鸽群分离株的全基因分析
王楷宬,王素春,庄青叶,邱 源,侯广宇
(中国动物卫生与流行病学中心,山东青岛 266032)
G群轮状病毒鸽群分离株的全基因分析
王楷宬,王素春,庄青叶,邱 源,侯广宇
(中国动物卫生与流行病学中心,山东青岛 266032)
为分析鸽轮状病毒中国分离株的特性,采用高通量测序方法,测定从山东省青岛市某活禽交易市场采集的肉鸽粪便样品中分离的轮状病毒基因组,并对其进行特性分析与比较基因组研究。结果显示,G群轮状病毒鸽群分离株全基因包含11个节段,总长度为17 803 bp,具有与其他轮状病毒相似的结构和基因顺序。与24株轮状病毒代表性毒株的VP6氨基酸序列进化分析结果,以及其他5个结构蛋白氨基酸序列遗传进化分析结果均证实,该病毒属G群轮状病毒。该毒株与2011年从香港鸽群中分离的G群轮状病毒同源性最高,但衣壳糖蛋白VP7的氨基酸序列同源性仅为71.7%,抗原性存在较大差异。
鸽;轮状病毒;基因组;高通量测序
轮状病毒(Rotavirus)是动物腹泻病的重要病原之一,1969年首先在腹泻的犊牛粪便中被发现,此后在人、绵羊、猪、马、犬、猫、兔、鼠、猴和禽中均有发现,因病毒粒子的电镜观察形态与车轮相似而得名。该病毒颗粒直径为70 nm,由3层构成。完整的病毒颗粒表面光滑,称光滑型颗粒。外壳自然脱落或经化学处理脱落后,变成直径50 nm的粗糙型颗粒。粗糙型颗粒进一步降解后,辐条脱落而留下约37 nm的病毒核心结构。核心结构呈六角形[1]。轮状病毒基因组为分节段的双链RNA,含11个片段,分别编码病毒蛋白VP1-4、VP6、VP7和非结构蛋白NSP1-5。根据中和试验,该病毒分为A、B、C、D、E、F、G和H群。
禽腹泻是严重影响禽存活率与生产力的一种常见疾病,对养禽业危害较严重。研究证实禽类因腹泻而死亡的数量占总死亡数的50%~70%以上。轮状病毒、腺病毒、疱疹病毒、细小病毒、类细小病毒和副粘病毒,以及大肠杆菌等均可引起禽类腹泻。其中,禽轮状病毒是引起禽类病毒性腹泻的最重要病原之一,临床上引起禽类拉稀、消瘦、生长发育受阻等[2]。至今轮状病毒A群[3]、D群[4]、G群[5]和F群[6]均能感染禽类。1981年Vindevogel[3]从鸽子粪便中检测到A群轮状病毒。之后又有研究者从鸽子中分离到轮状病毒,并对其特性进行研究[7-9]。2013年Tung等[10]从鸽群中首次检测到G群轮状病毒,并解析其基因组。2017年Pauly等[11]从尼日利亚的鸽群中分离到D群轮状病毒。
我国对禽群轮状病毒的研究主要针对鸡群的感染状况和病原特性。马洪超等[12]于1989年首次在我国鸡群中分离到A群轮状病毒。之后,有研究人员对鸡轮状病毒的特性等进行研究[13]。但是我国对其他禽类感染的轮状病毒研究极少,也未见鸽群中检测到轮状病毒的报道。为了解我国感染鸽群的轮状病毒特性,本研究采用高通量测序方法,对首次在我国肉鸽中分离的一株轮状病毒进行测序,分析该病毒的基因组特性。
1 材料和方法
1.1 材料和仪器
Ion PGM Template OT2 400 Kit、Ion PGM Sequencing 400 Kit V2、E-Gel SizeSelect 2%Agarose、Ion 316 Chip Kit V2、Dynabeads MyOne Streptavidin C1 Beads、Qubit核酸浓度测定仪及配套试剂均购自Life technologies公司;超净工作台(美国Forma Scientific);移液器(Eppendorf);孵化器(德州诚信孵化设备有限公司);高速台式离心机(德国Heraeus Biofuge primoR);PCR扩增仪(Perkin Elmeter Gen Amp PCR System 9600)。高性能计算平台为Dell T630塔式服务器,具有2颗Intel(R)Xeon(R)CPU E5-2620 v3 @2.40GHz,内存264 G,存储23 T,操作系统版本CentOS Linux release 7.1.1503(Core)。
1.2 核酸提取
2014年秋季自山东省青岛市某活禽交易市场采集的肉鸽粪便样品中检测到鸽轮状病毒(pigeon/China/10/2014)[14];将该份样品充分混匀后,经高速离心和过滤,去除大分子物质和细菌,使用QIAamp Viral RNA Mini Kit(Qiagen,德国)提取病毒核酸。
1.3 高通量测序(NGS)文库构建
对提取的病毒核酸,采用中国动物卫生与流行病学中心报道的方法[14]进行NGS文库构建。具体 方 法:8 μL 病 毒 RNA 中 加 入 1 μL 100 μmol/L的引物A15N6和2 μL无核酸酶的水进行混合,72 ℃ 5 min;然后将RNA引物混合物放在冰上孵育至少3 min;在混合物中加入4 μL 5×first-strand buffer、1 μL dNTP(100 μmol/L)、2 μL DTT(0.1 mol/L)、1 μL RNaseOUT ™ Recombinant Ribonuclease Inhibitor(40 U/μL) 和 1 μL SuperScript® III Reverse Transcriptase(200 U/μL)(Invitrogen,USA),25 ℃反应 15 min,42 ℃反应 30 min,70 ℃ 15 min,终止反应;反应产物中再加入1 μL RNase H(TaKaRa,Japan),37 ℃ 反应20 min;采用DynaMag™-2 Magnet 和Agencourt® AMPure® XP Reagent(Beckman Coulter,USA)纯化合成第一链cDNA;在纯化的第一链cDNA中加入引物B15N6,70 ℃ 5 min;再加入 1 μL Klenow fragment(5 U)(NEB,USA)、5 μL 10×NEBuffer 2、2 μL dNTP(100 μmol/L) 和1 μL DTT(0.1 mol/L),37 ℃反应 30 min;最后采用 1×Phusion High-Fidelity Buffer,10 μmol/L 引 物A30 和B30(表1),0.5 U Phusion High-Fidelity DNA Polymerase(NEB,USA),进行PCR扩增。将PCR产物采用E-Gel® SizeSelect™ Agarose Gel进行电泳;将筛选和回收的约450 bp扩增产物作为NGS文库。

表1 引物与引物序列
1.4 NGS测序
将NGS文库稀释为26p mol/L,应用Ion PGM Template OT2 400 Kit对DNA文库进行测序前的样品处理。处理后的样品加样至Ion 316 芯片,置于PGM测序仪进行测序。
1.5 测序数据分析
NGS测序数据经质控后,采用CLC genomics workbench 8.5.1(Qiagen,Germany), 将 测 序得到的reads参考GenBank中已发表的鸽轮状病毒Pigeon/HK18全基因序列[10](GenBank登录号:NC_019712)进行mapping 拼接,并分析拼接完成的基因组中的ORF,注释后的序列提交GenBank。
1.6 基因组特性分析
已报道的文献[15]和病毒分类委员会第九次分类报告中均以VP6氨基酸的相似性来确定轮状病毒的分群,若相似性大于60%则判为同一个群。本文采用MEGA 6.0[16]将Pigeon/China/10/2014的VP6氨基酸序列分别与GenBank中轮状病毒科的代表毒株(表2)的VP6氨基酸序列进行比对和分析,分析该毒株的分类地位。代表毒株中包含鸡、火鸡、鸽子、人、鼠、牛、羊、猪、兔、马、犬、猫、猕猴和骆马等动物的轮状病毒分离株。同时,将所有禽类感染的已报道全基因的毒株与部分群的人源毒株的基因进行下载(参与VP6分析的毒株只有部分报道了全基因),应用MEGA 6.0[16]分别与Pigeon/China/10/2014其余5个结构蛋白(VP1-4和VP7)的氨基酸序列进行比对和分析。将此毒株的所有节段氨基酸序列与已报到的所有3株鸽源轮状病毒进行同源性分析。

表2 基因比较分析所使用的轮状病毒毒株
2 结果与分析
2.1 基因组解析
芯片上样率为73%;测序文库得到质量较高的序列数据测序数据。CLC genomics workbench 8.5.1拼接得到鸽轮状病毒所有的11个节段,总长度为17 803 bp;GenBank登录号分别为MF768259~MF768269。Pigeon/China/10/2014 基 因组各基因节段详细信息见表3。
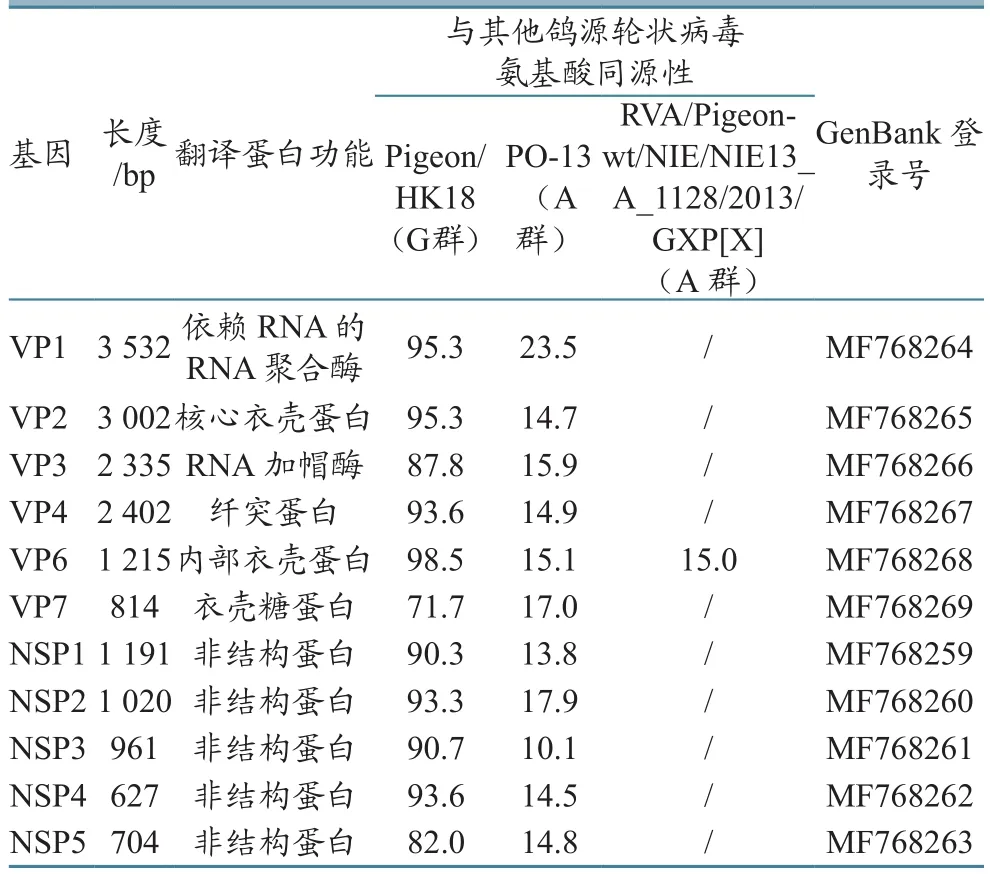
表3 Pigeon/China/10/2014基因组各基因节段的详细信息
2.2 基因组比较分析
VP6氨基酸序列分析显示,该毒株属于G群轮状病毒(图1),且各结构蛋白氨基酸遗传进化关系(图2、图3、图4、图5、图6)基本一致,均与2011年自香港分离的鸽源轮状病毒同源性最高。本研究分离的Pigeon/China/10/2014属G群轮状病毒,与Pigeon/HK18同源性最高,各基因氨基酸同源性为71.7%~99.0%;而与鸽群分离的A群轮状病毒PO-13同源性较低,11个基因氨基酸序列同源性均低于25%(表3)。
3 讨论与结论
轮状病毒可感染多种宿主。感染禽类宿主的报道主要集中于鸡,其次是火鸡和鸽。文中对所有已报道全基因的禽源轮状病毒进行基因进化分析,综合已发表的文献[11]发现,鸡可感染A、D、F、G群轮状病毒,鸭、鹅、火鸡和鹌鹑可感染A群轮状病毒,鸽子能感染A、D和G群轮状病毒。本研究之前共有12株鸽源轮状病毒被报道,分别为日本分离的A群轮状病毒、香港分离的G群轮状病毒、尼日利亚分离的9株A群和1株D群轮状病毒(未公布基因序列)。我国未见其他从鸽群中分离到轮状病毒的报道。
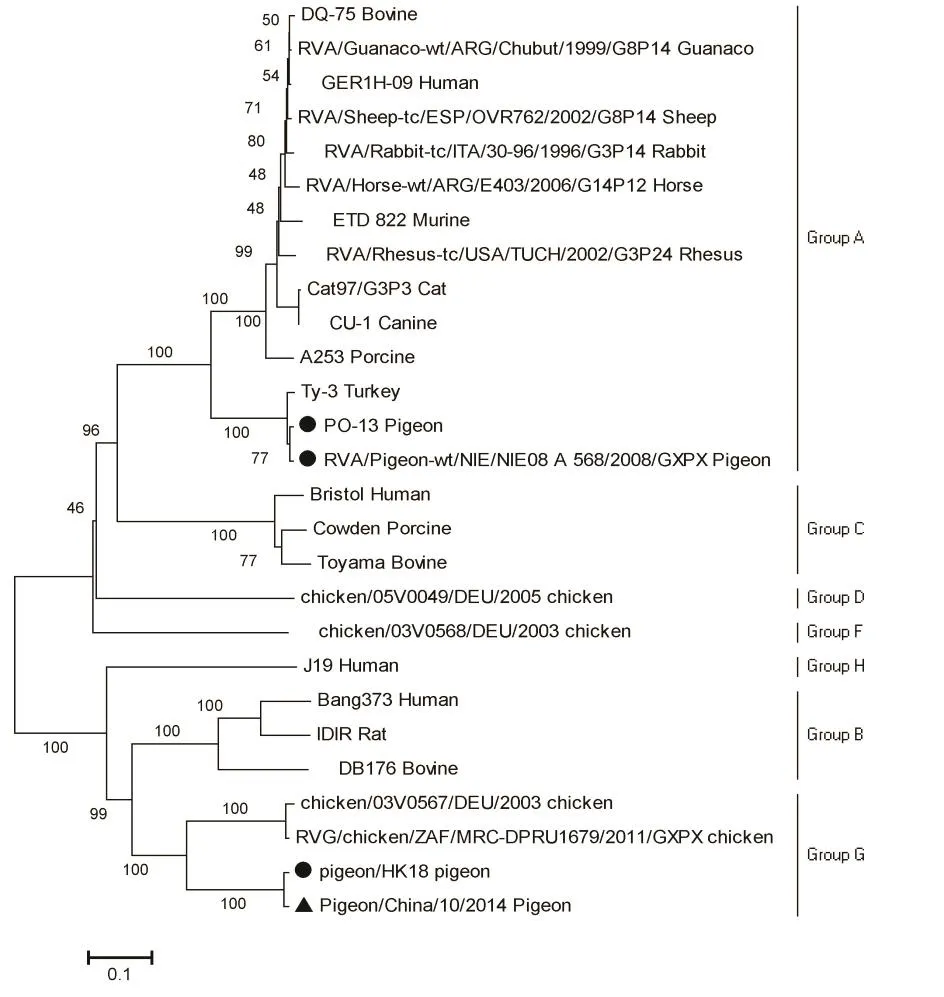
图1 轮状病毒VP6氨基酸序列进化分析

图2 轮状病毒VP1氨基酸序列进化分析

图3 轮状病毒VP2氨基酸序列进化分析
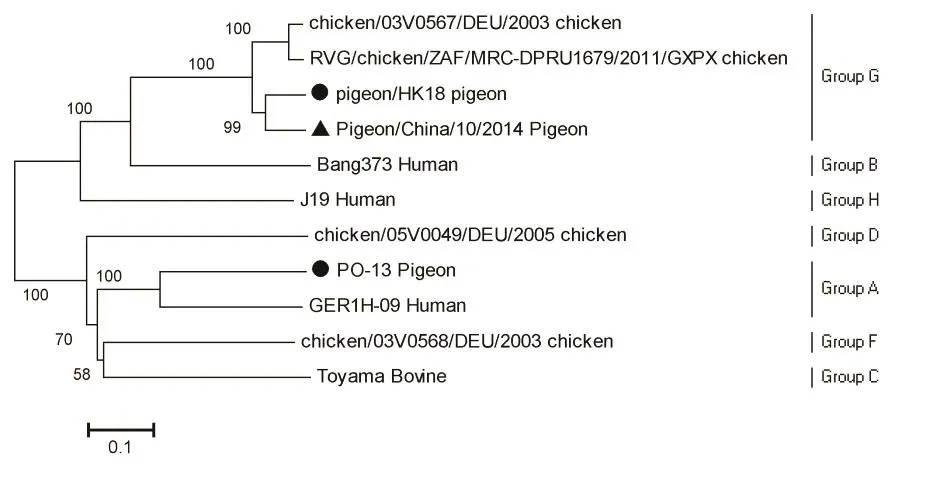
图4 轮状病毒VP3氨基酸序列进化分析

图5 轮状病毒VP4氨基酸序列进化分析

图6 轮状病毒VP7氨基酸序列进化分析
本研究采用高通量测序方法对一株鸽群分离的轮状病毒全基因进行测序和分析,通过VP6的氨基酸序列进化树和同源性分析,确定该病毒为G群轮状病毒。对此毒株其余结构蛋白进行分析,也证实该病毒应属G群。轮状病毒基因组分为11个节段,其中结构蛋白VP1具有依赖RNA的RNA聚合酶作用,VP2构成病毒的核心衣壳,VP3作为病毒RNA的加帽酶发挥作用。另外非结构蛋白主要参与病毒的复制(NSP2-NSP3)与形态发生(NSP4-NSP5),与宿主蛋白相互作用,共同影响发病机理及免疫反应(NSP1)。VP7和VP4是病毒主要的交叉中和抗原,自然感染时能够刺激机体产生中和抗体,并且在 RV 的感染过程中,VP7和VP4能够相互作用,在病毒进入宿主细胞过程中发挥重要作用[17]。
从该病毒与轮状病毒属其他毒株的遗传进化关系来看,本研究测序的毒株与2011年香港分离的鸽源轮状病毒最近,除VP7基因外,其余10个基因的氨基酸序列同源性均达到90%以上。VP7基因编码轮状病毒的衣壳糖蛋白。该蛋白存在于轮状病毒表面,是病毒中和抗体的主要结合位点。这说明此次分离的病毒虽与2011年香港分离的鸽源轮状病毒同属G群,但其抗原性存在较大差异。香港分离的G群轮状病毒来源于野鸽,而青岛市分离的此株G群轮状病毒分离自活禽市场售卖的肉鸽。VP7的差异是否与宿主不同有关,还有待进一步研究部。
综上所述,本文报道了世界上第2株分离自鸽子的G群轮状病毒全基因,同时对禽源的各群轮状病毒进行进化分析。研究结果有利于了解我国鸽轮状病毒特性,进一步明确其与其他轮状病毒的进化关系。
[1] 陆承平.兽医微生物学[M]. 5版. 北京:中国农业出版社,2013.
[2] 陈剑杰,李巨银. 禽轮状病毒研究进展[J]. 畜牧 兽 医 杂 志,2007,26(1). Doi:10.3969/j.issn.1004-6704.2007.01.011
[3] VINDEVOGEL H U,DAGENAIS L,LANSIVAL B,et al. Incidence of rotavirus,adenovirus and herpesvirus infection in pigeons [J]. Veterinary record,1981,109(13):285-286.
[4] BEZERRA D A,DA SILVA R R,KAIANO J H,et al.Detection of avian group D rotavirus using the polymerase chain reaction for the VP6 gene [J]. Journal of virological methods,2012,185(2):189-192.
[5] STUCKER K M,STOCKWELL T B,NYAGA M M,et al. Complete genomic sequence for an avian group G rotavirus from South Africa [J]. Genome announcement,2015,3(2):107-115.
[6] MASCARENHAS J D,BEZERRA D A,SILVA R R,et al. Detection of the VP6 gene of group F and G rotaviruses in broiler chicken fecal samples from the Amazon region of Brazil [J]. Archives of virology,2016,161(8):2263-2268.
[7] GOUGH R E,COX W J,DEVOY J. Isolation and identification of rotavirus from racing pigeons [J].Veterinary record,1992,130(13):273.
[8] MINAMOTO N,OKI K,TOMITA M,et al. Isolation and characterization of rotavirus from feral pigeon in mammalian cell cultures [J]. Epidemiology and infection,1988,100(3):481-492.
[9] ROHWEDDER A,SCHUTZ K I,MINAMOTO N,et al. Sequence analysis of pigeon,turkey,and chicken rotavirus VP8* identifies rotavirus 993/83,isolated from calf feces,as a pigeon rotavirus [J]. Virology,1995,210(1):231-235.
[10] PHAN T G,VO N P,BOROS A,et al. The viruses of wild pigeon droppings [J]. Plos one,2013,8(9):e72787.
[11] PAULY M,ONI OO,SAUSY A,et al. Molecular epidemiology of avian rotaviruses group A and D shed by different bird species in Nigeria [J]. Virology Journal,2017,14(1):111.
[12] 马洪超,崔思列. 鸡腹泻轮状病毒核酸电泳型及抗原性的研究[J]. 动物检疫,1990,1(1):1-4.
[13] 刁有祥,冯涛,崔治中,等. 鸡轮状病毒的分离鉴定及特性研究 [J]. 畜牧兽医学报,2004,35(4):434-438.
[14] QIU Y,CHEN J M,WANG T,et al. Detection of viromes of RNA viruses using the next generation sequencing libraries prepared by three methods [J]. Virus Research,2017,237:22-26.
[15] JOHNE R,OTTO P,ROTH B,et al. Sequence analysis of the VP6-encoding genome segment of avian group F and G rotaviruses [J]. Virology,2011,412(2):384-391.
[16] TAMURA K,STECHER G,PETERSON D,et al.MEGA6:Molecular evolutionary genetics analysis version 6.0 [J]. Molecular biology and evolution,2013,30(12):2725-2729.
[17] ARNOLDI F,CAMPAGNA M,EICHWALD C,et al.Interaction of rotavirus polymerase VP1 with nonstructural protein NSP5 is stronger than that with NSP2 [J]. Journal of Virology,2007,81(5):2128-2137.
Genome Analysis of the Pigeon Rotavirus Group G Isolated in China
Wang Kaicheng,Wang Suchun,Zhuang Qingye,Qiu Yuan,Hou Guangyu
(China Animal Health and Epidemiology Center,Qingdao,Shandong 266032,China)
To analyze the characterization of the pigeon rotavirus isolated in China,the genome of the pigeon rotavirus isolated from a meat pigeon in a living poultry market of Qingdao City,Shandong Province was sequenced by highthroughput sequencing. The genome of the isolate was analyzed and compared with other rotaviruses. The results showed that 11 genes were included in the genome with a total length of 17 803 bp,of which the genomic organization,structure and gene order were similar to other rotavirus. It was showed that the isolate was rotavirus group G through evolution analysis of VP6 amino acid sequence of 24 rotavirus representative strains and analysis of genetic evolution of other 5 structural protein amino acid sequences. The identity of the isolate and the group G rotavirus from Hongkong's pigeon in 2011 was the highest,while the identity of the capsid glycoprotein VP7 amino acid sequence between the two viruses was only 71.7%. The antigenicity of the two viruses was different.
pigeon;Rotavirus;genome;high-throughput sequencing
国家重点研发计划项目(2017YFC1200500);中国动物卫生与流行病学中心创新基金(2015IF-0004FF)
S851.3
A
1005-944X(2018)01-0085-06
10.3969/j.issn.1005-944X.2018.01.024
朱迪国)

