转基因水稻秸秆还田对土壤硝化反硝化微生物群落的影响*
王沛譞, 徐 焱, 宋亚娜
转基因水稻秸秆还田对土壤硝化反硝化微生物群落的影响*
王沛譞1,3, 徐 焱2, 宋亚娜1**
(1. 福建省农业科学院生物技术研究所 福州 350003; 2. 福州时代中学 福州 350007; 3. 福建师范大学附中福州 350007)
转基因作物可能通过根系分泌物和植株残体组成的改变及外源基因的转移释放令土壤微生物群落产生变化, 影响土壤微生物的生态功能。氨氧化细菌和反硝化细菌是驱动土壤硝化和反硝化过程的关键微生物, 其群落结构的变化直接关系土壤氮素的转化与利用。本研究利用荧光定量PCR和PCR-DGGE技术分析了转双价抗虫基因水稻‘Kf8’秸秆还田降解过程中, 土壤氨氧化细菌和反硝化细菌群落丰度与组成的变化, 探讨转基因水稻是否存在影响稻田土壤氮素转化与N2O排放的可能。结果显示: 无论是氨氧化细菌基因还是反硝化细菌基因, 其丰度在转基因水稻‘Kf8’与非转基因水稻‘Mh86’的秸秆还田土壤中都没有显著差异; 转基因水稻‘Kf8’和非转基因水稻‘Mh86’秸秆还田降解过程中0~10 cm土层中的基因丰度均显著高于10~20 cm及20~30 cm土层(<0.05); 各深度土层中的基因丰度均存在随秸秆还田时间延长而增加的趋势。水稻秸秆还田降解过程中, 转基因水稻‘Kf8’的土壤氨氧化细菌和反硝化细菌的群落多样性指数及组成, 均与非转基因水稻‘Mh86’没有显著差异。相关分析结果表明土壤氨氧化细菌和反硝化细菌群落组成均与水稻秸秆还田时间存在显著相关性(=0.002), 反硝化细菌群落组成还与土层深度显著相关(=0.024)。本研究表明转抗虫基因水稻秸秆还田对稻田土壤硝化和反硝化关键微生物群落不会产生明显影响。就土壤微生物群落而言, 转抗虫基因水稻秸秆还田不存在影响土壤氮素转化与N2O排放的可能。
转基因水稻; 秸秆还田; 氨氧化细菌;基因; 反硝化细菌;基因; 群落结构
秸秆还田能增加土壤有机质, 改良土壤结构, 促进微生物活力和作物根系的发育, 在杜绝了秸秆焚烧所造成的大气污染的同时具有增肥增产作用。转基因作物可能通过外源基因改变作物根系分泌物、根系及植株残体的组成及其降解过程, 从而在生长过程中及秸秆还田时影响土壤微生物群落组成及其功能[1]。
硝化和反硝化作用是由微生物驱动的土壤氮素转化过程, 硝化和反硝化关键微生物群落结构直接关系到土壤氮素转化与利用、氮肥有效性以及温室气体N2O排放等环境问题。由含有氨单加氧酶(amo)的氨氧化细菌驱动的氨氧化过程是硝化作用的速率限制性步骤[2], 氨单加氧酶基因()是检测环境中氨氧化细菌的标记基因[3-4]。反硝化作用是在硝酸盐还原酶、亚硝酸盐还原酶、氧化氮还原酶和氧化亚氮还原酶4种酶连续催化下完成的, 这4种酶的编码基因分别为、、和。目前亚硝酸盐还原酶基因()常用于反硝化细菌的分子标记[5-6]。
转抗虫基因水稻具有良好的抗虫和增产效果, 其外源基因及蛋白随水稻秸秆降解进入土壤后, 若改变了土壤氨氧化细菌或反硝化细菌的群落结构, 势必影响稻田土壤的硝化和反硝化过程, 从而关系到稻田氮肥利用及温室气体N2O的排放等问题。
目前, 已有研究表明转Bt基因水稻秸秆的降解对土壤细菌和真菌群落组成没有显著影响[7-8]。但有关转基因水稻秸秆降解对土壤中与氮素转化密切相关的硝化反硝化微生物群落的影响尚少见报道。本研究从硝化反硝化微生物功能基因着手, 开展了转基因水稻秸秆还田土壤中硝化反硝化微生物群落的研究。
我们以转双价抗虫基因水稻为对象, 利用分子生物学技术, 以和为标记基因, 对水稻秸秆还田降解过程中的土壤氨氧化细菌和反硝化细菌群落结构进行研究, 探讨转基因水稻是否存在影响稻田土壤氮素转化与N2O排放的可能。
1 材料与方法
1.1 供试水稻材料
供试水稻材料为转基因恢复系‘Kf8’及其对应的非转基因亲本恢复系‘Mh86’。‘Kf8’是以三系‘Mh86’为受体, 通过农杆菌介导法将双T-抗虫PCDMARUBAC载体中(苏云金芽胞杆菌杀虫晶体蛋白基因)和(修饰的豇豆胰蛋白酶抑制剂基因)两个抗虫基因导入水稻, 获得的能稳定遗传和表达的转基因恢复系。转基因恢复系‘Kf8’已于2006年获得农业部转基因安全委员会批准进行生产性试验。
1.2 田间试验设计
本研究在连续进行了6年的田间定位试验基础上进行。定位试验在具有防护措施的联动网室中进行, 试验地点位于福州市郊的吴凤村(26º01′N, 119º20′E)。该试验基地属暖湿亚热带季风气候, 年无霜期长达326 d, 年均气温19.6 ℃, 平均湿度77%, 年均降水量1 342.5 mm。供试土壤为砂壤, 土壤有机质含量为23.32 g×kg-1, pH 6.15, 全氮1.65 g×kg-1, 全磷(P2O5) 0.76 g×kg-1, 全钾(K2O) 18.28 g×kg-1, 速效氮154.53 mg×kg-1, 速效磷31.34 mg×kg-1, 速效钾70.52 mg×kg-1。6年定位试验均为中稻栽培, 5月播种, 6月插秧, 10月收获。2个水稻材料, 每个材料3次重复, 共6个小区随机排列, 每小区种植水稻350株, 株行距为20 cm×20 cm, 单本插。定位试验除不使用杀虫剂外其余水肥措施和田间管理等均按当地常规进行。本研究于定位试验第6年水稻收获后(2013年10月), 将水稻全部秸秆粉碎原位填埋于各田间小区内自然降解还田, 填埋深度约30 cm。
1.3 土壤样品采集
分别于秸秆还田后50 d、100 d、150 d和200 d采集0~10 cm、10~20 cm和20~30 cm深度土层的土壤。采用五点法取样, 每个小区选取5点, 用土钻分别采集不同深度土层土壤, 五点采集的土样混合为一个样品。田间采集的土样放入4 ℃冰盒中保存, 带回实验室后将混合后的土壤去除根系、杂草、土壤动物和石块等杂质后混匀, 装入50 mL塑料离心管中,-70 ℃冷冻保存用于土壤微生物群落分析。
1.4 土壤微生物总DNA提取
称取0.5 g解冻后的土壤样品, 采用FastDNAÒSPIN Kit For Soil (QBIOgene)试剂盒进行土壤微生物总DNA的提取, DNA样品-20 ℃冰箱保存待用。
1.5 荧光定量PCR
利用氨氧化细菌基因及反硝化细菌的基因的特异引物(表1), 在ABI PRISM7500 Real-Time PCR System扩增仪上进行了绝对荧光定量PCR分析。反应体系为25 µL, 其中2 µL稀释5倍的DNA模板加反应液23 µL, 反应液包括12.5 µL SYBR Premix Ex TaqTM(2 x), 0.5 µL ROX Reference Dye II(50 x), 前、后引物各1 µL(5 pmol×L-1, 英骏生物工程公司, 上海)和8 µL超纯水。标准曲线的建立方法为: 对土壤DNA用目的基因的特异引物进行PCR扩增, 将PCR扩增产物克隆到pMD-18(TaKaRa, 大连)载体中, 然后对插入正确片段的克隆进行质粒提取, 用Nanodrop ND-1000 UV-Vis测定质粒浓度, 计算质粒DNA拷贝数, 按10倍梯度稀释质粒DNA制作标准曲线[9]。本试验中和基因的标准曲线范围分别为9.09´108~9.09´104copies×µL-1和3.67´109~3.67´105copies×µL-1。和基因标准曲线的2均达0.99, 扩增效率分别为1.01和0.99。
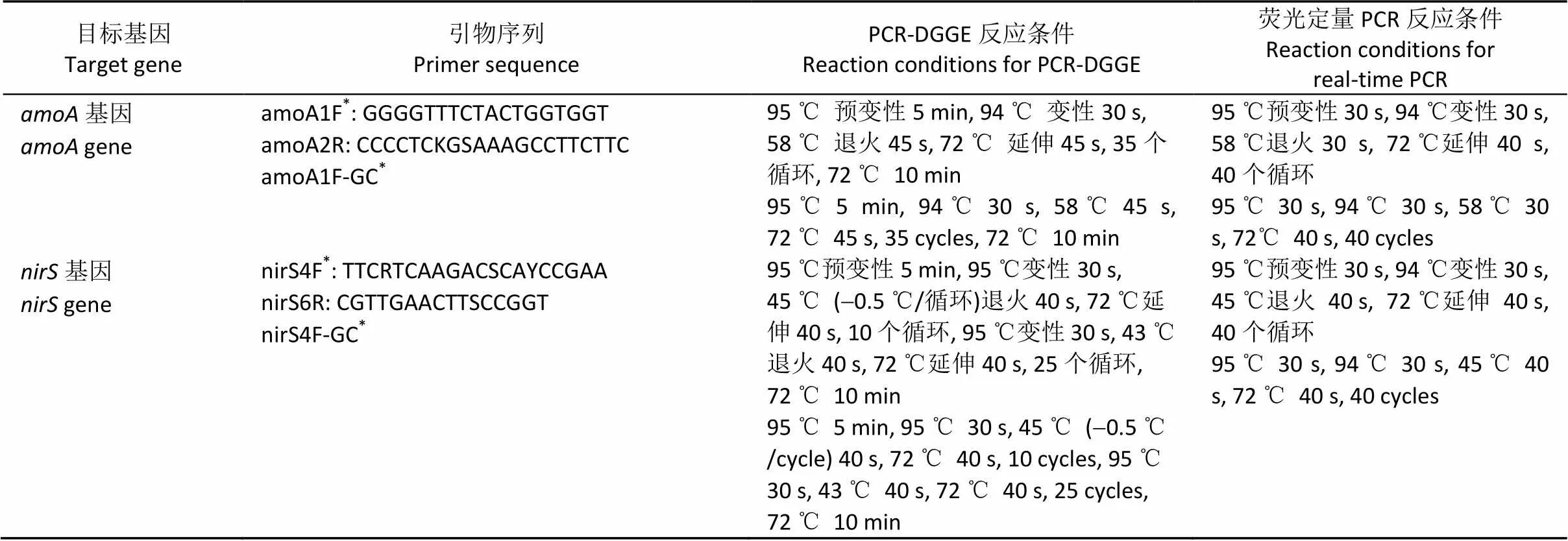
表1 PCR引物及反应条件
*: 分别在amoA1F和nirS4F引物序列前加一个GC夹序列(CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGG)用于DGGE的PCR扩增。*: A GC clamp (CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGG) was attached to the sequence of amoA1F or nirS4F for PCR of DGGE.
1.6 聚合酶链反应-变性梯度凝胶电泳(PCR- DGGE)
分别利用氨氧化细菌基因和反硝化细菌基因的带GC夹特异引物进行PCR扩增[6,10](表1)。反应体系50mL, 其中2mL稀释5倍的DNA模板加反应液48mL, 反应液包括1mL Taq DNA聚合酶(2.5 U×mL-1), 4mL dNTPs(2.5 mmol×L-1), 5mL 10×PCR- buffer(10倍的PCR缓冲液), 前、后引物各4mL(5 pmol×mL-1)和30mL超纯水。PCR产物经琼脂糖电泳EB(溴化乙腚)染色检查后, 用40mL的PCR产物进行变性梯度凝胶电泳, 采用梯度为35%~50%(基因)和40%~60%(基因)的8%聚丙烯酰胺凝胶[化学变性剂为100%尿素7 mol×L-1和40%(/)的去离子甲酰胺]在1倍TAE缓冲液中150 V 60 ℃下电泳5 h。电泳后用10 mL SyBR green I(1倍TAE稀释10 000倍)核酸染料染色45 min, 然后用FluoChem SP(Alpha Innotech)成像系统拍照。
1.7 数据分析
采用Quantity One (Bio-Rad)软件对DGGE图谱中DNA条带的位置和亮度进行数字化, 计算微生物群落多样性指数Shannon-Weiner index (′)=-å()× ln(/) (为每个带的亮度,为某一样品所有带亮度的和); 利用Canoco 4.5生态学软件进行微生物群落结构的主成分分析(PCA); 采用SPSS 13.0软件进行数据方差分析。
2 结果与分析
2.1 土壤硝化反硝化微生物丰度变化
利用荧光定量PCR技术分析了土壤硝化作用关键微生物氨氧化细菌基因与反硝化细菌基因丰度, 结果如图1所示。从图1A可见, 在水稻秸秆还田过程中稻田0~10 cm土壤中氨氧化细菌丰度较高, 4个取样时间内的基因数量都可以达106copies∙g-1(干土)以上; 而10~20 cm及20~30 cm土层中的基因数量只到达104~105copies∙g-1(干土); 前者显著(<0.05)高于后者。在水稻秸秆还田的各个取样时间内, 转基因水稻‘Kf8’的氨氧化细菌基因丰度与其对应的常规水稻‘Mh86’没有显著差异, 0~10 cm、10~20 cm和20~30 cm 3个深度土层中‘Kf8’秸秆还田土壤的基因数量与‘Mh86’均处于同一数量级上, 且差异不显著。说明‘Kf8’与‘Mh86’两个品种水稻秸秆降解过程中土壤氨氧化细菌基因丰度变化是一致的。
水稻秸秆还田过程中土壤反硝化细菌基因丰度变化如图1B所示, 结果表明秸秆还田的各个取样时间内, 转基因水稻‘Kf8’与非转基因水稻‘Mh86’在各个深度土层中的反硝化细菌基因丰度都没有显著差异。随秸秆还田时间的延长各深度土层的基因丰度存在逐渐增加的趋势, ‘Kf8’处理的0~10 cm、10~20 cm和20~30 cm土层中基因数量在秸秆还田200 d时分别是50 d时的1.58倍、1.91倍和1.41倍, ‘Mh86’处理分别是1.70倍、1.77倍和1.26倍。说明随着‘Kf8’与‘Mh86’两品种水稻秸秆的降解土壤反硝化细菌基因丰度都在增高。
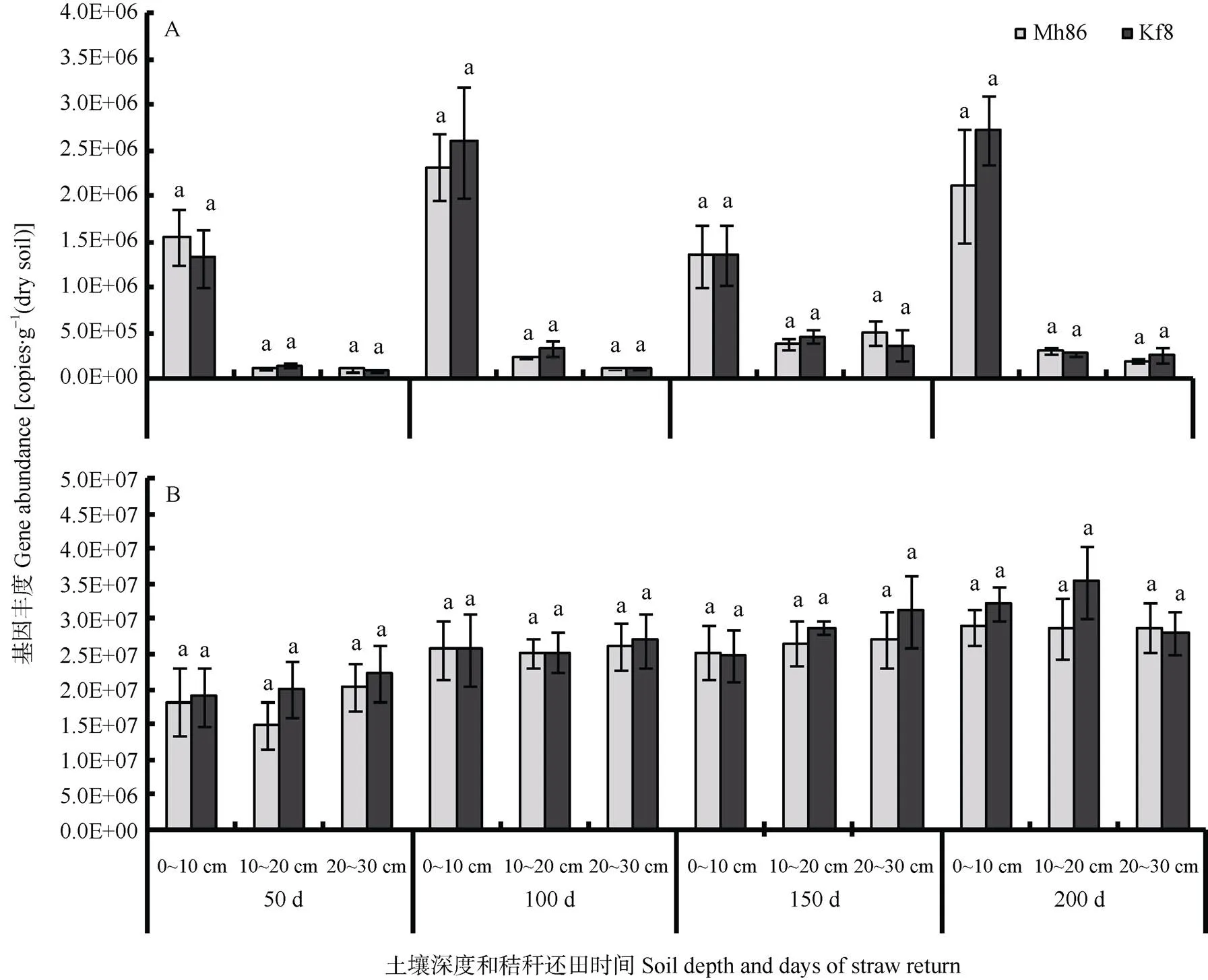
图1 转基因水稻‘Kf8’和常规水稻‘Mh86’秸秆还田后不同时间不同深度土壤氨氧化细菌amoA基因(A)和反硝化细菌nirS基因(B)丰度的变化
同一深度土层不同字母表示差异显著(0.05)。Values of the same depth of soil in the same time followed by different letters are significantly different (< 0.05).
2.2 稻田土壤硝化反硝化微生物群落多样性变化
通过对稻田土壤中基因和基因的PCR扩增及其产物的变性梯度凝胶电泳(DGGE), 分析了稻田土壤中硝化和反硝化细菌的群落多样性。图2A显示在水稻秸秆还田降解的200 d内土壤氨氧化细菌群落多样性指数的变化。在各取样时间内及各深度土层中氨氧化细菌群落多样性指数在‘Kf8’与‘Mh86’间均没有显著差异; 同一品种水稻的氨氧化细菌群落多样性指数在不同取样时间及不同深度土层中也都没有明显差异; 秸秆还田降解的200 d内2个品种水稻的土壤氨氧化细菌群落多样性指数基本维持在2.0左右。
在水稻秸秆还田降解的200 d内, 各深度土层的反硝化细菌群落多样性指数在‘Kf8’和‘Mh86’间也均没有差异(图2B); 同一秸秆还田时间内, 各个品种水稻的土壤反硝化细菌群落多样性指数在不同深度土层间同样没有明显差异(图2B)。秸秆还田降解的200 d内2个品种水稻的土壤反硝化细菌群落多样性指数基本维持在2.5左右。
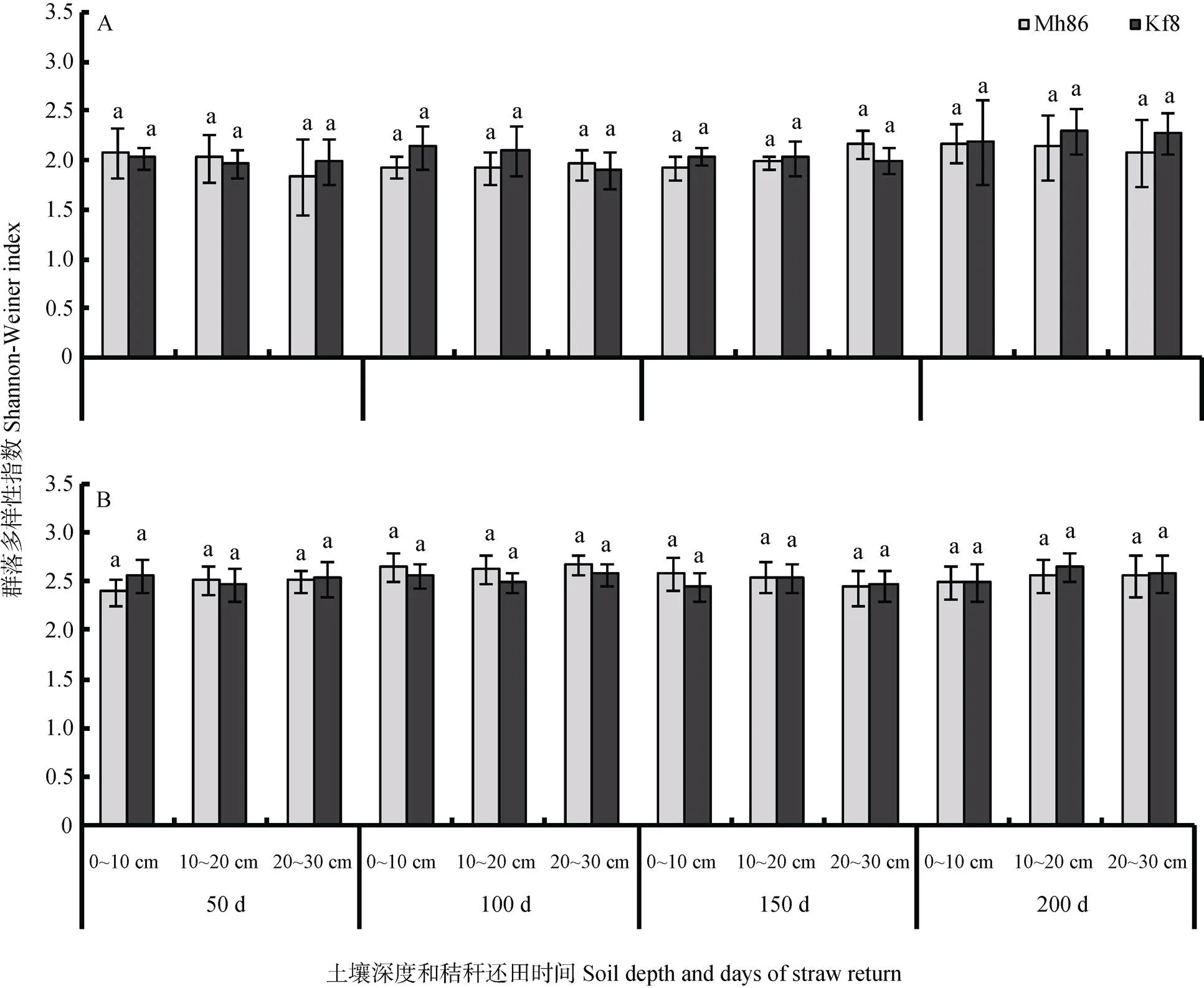
图2 转基因水稻‘Kf8’和常规水稻‘Mh86’秸秆还田后不同时间不同深度土壤氨氧化细菌(A)和反硝化细菌(B)多样性指数的变化
同一深度土层不同字母表示差异显著(0.05)。Values of the same depth of soil in the same time followed by different letters are significantly different (< 0.05).
2.3 稻田土壤硝化反硝化微生物群落组成的变化
基于DGGE图谱利用Canoco 4.5生态学软件进行了秸秆还田过程中土壤硝化和反硝化微生物群落组成的主成分分析(PCA), 并将微生物群落组成与水稻品种、取样时间和土层深度3个因素进行了相关性分析。
图3a显示, 2个品种水稻各深度土层4个取样时间的氨氧化细菌群落组成在变异较大的主成分1(PC1)方向上存在分布差异, 其中秸秆还田100 d和150 d的分布较为接近, 并分别与50 d、200 d的分布差异较大。同一取样时间内水稻‘Kf8’与水稻‘Mh86’间以及不同深度土层间的氨氧化细菌群落组成的分布差异均表现在变异较小的主成分2(PC2)方向上。同时, 相关性分析结果表明土壤氨氧化细菌的群落组成仅与取样时间即秸秆还田时间长短存在显著相关(=0.002), 与水稻品种和土层深度都没有显著相关性。
土壤反硝化细菌群落组成的变化趋势与土壤氨氧化细菌一致(图3b), 50 d、100 d、150 d和 200 d的不同水稻品种及不同深度土层的反硝化细菌群落组成均在PC1方向上分布差异明显, 同一取样时间内同一品种水稻的不同深度土层的反硝化细菌群落组成在PC2方向上也存在较明显的分布差异。相关性分析结果表明, 土壤反硝化细菌的群落组成与取样时间(=0.002)和土层深度(=0.024)均存在显著相关, 但与水稻品种没有明显相关性。
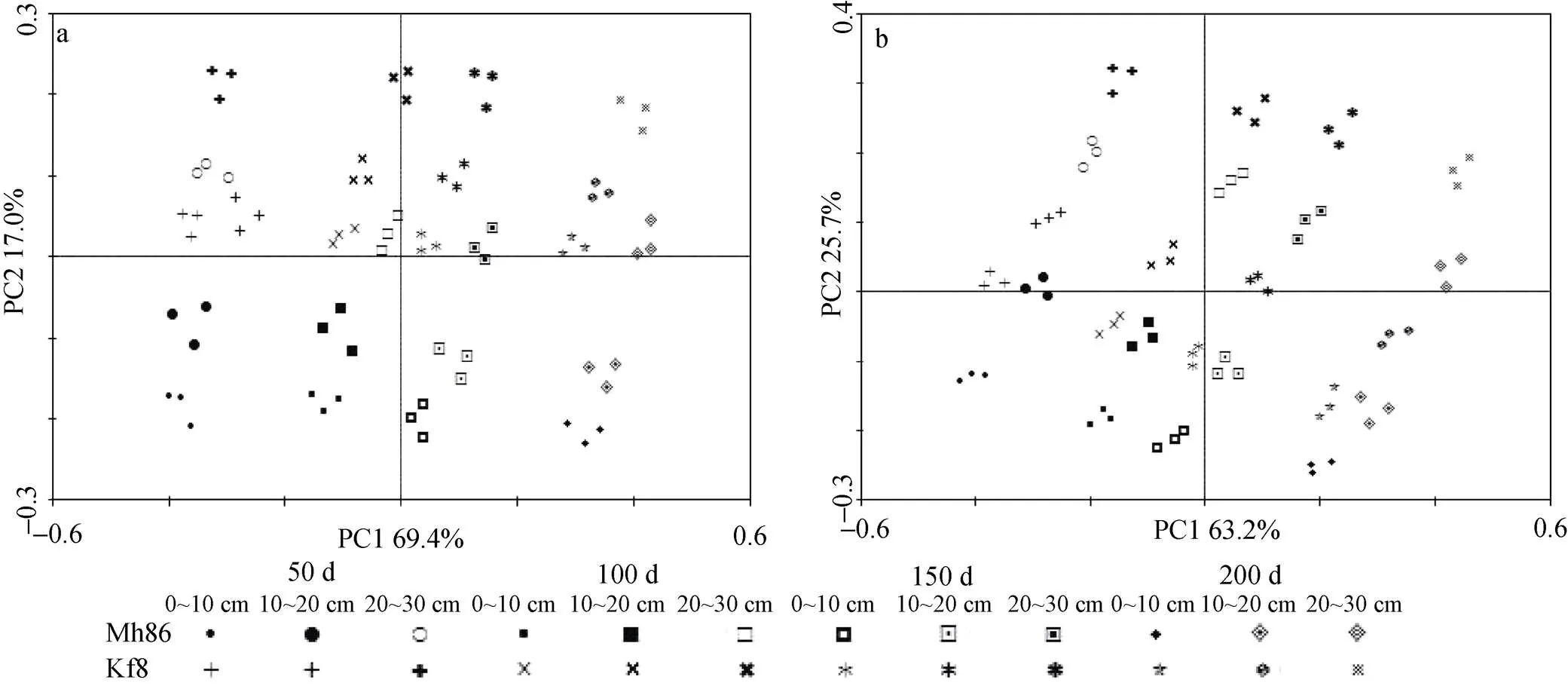
图3 转基因水稻‘Kf8’和常规水稻‘Mh86’秸秆还田后不同时间不同深度土壤氨氧化细菌(a)和反硝化细菌(b)群落组成的主成分分析
3 讨论与结论
目前, 关于转基因作物对环境微生物影响的研究结果依据作物种类、目标基因、微生物种类等因素而存在差异, 且研究多集中于对土壤总微生物如细菌或真菌的群落方面。如: 在转抗病基因木瓜的研究中发现, 转基因品种的种植能够改变土壤真菌或细菌的群落组成[11]; 而土壤微生物群落结构在转Bt基因抗虫玉米和非转基因玉米间没有差异[12]。本研究所在定位试验的前期研究证实了转双价抗虫基因水稻定位种植4年后对土壤细菌和真菌的群落组成与丰度都没有显著影响[13]。还有研究表明转Bt基因抗虫水稻的秸秆或根系还田降解过程中对土壤细菌或真菌的群落组成没有影响[7-8]。
近年来对转基因作物土壤中功能微生物群落变化的研究逐渐增多, 如对驱动土壤氮素转化关键微生物群落结构的研究。本研究定位试验前期工作已证实在一定时期内种植转双价抗虫基因水稻不会影响土壤氨氧化细菌的群落组成与丰度[14]。本研究进一步分析了转双价抗虫基因水稻秸秆还田降解过程中对土壤硝化和反硝化微生物群落组成与丰度的影响。结果表明转双价抗虫基因水稻秸秆还田降解对土壤氨氧化细菌和反硝化细菌群落丰度与结构都没有明显影响。土壤氨氧化细菌和反硝化细菌的群落结构仅与秸秆还田降解的时间长短或土层深度相关, 而与转基因与非转基因的水稻品种没有相关性。另外, 由于氨氧化是个好氧的微生物转化过程, 所以秸秆还田降解过程中通气性较好的表层土壤中含有更丰富的氨氧化细菌。
转基因技术研发的目的是将一些有利于作物生长发育的外源基因导入目标作物, 使其具有抗虫、抗病、抗逆、改善品质及增产的优势。但外源基因的转入有可能引起作物根系分泌物、根系及植株残体组成的改变, 外源基因及其表达蛋白也可以通过根系分泌物或植株残体降解直接进入到土壤中, 这些过程都有可能使种植转基因作物的土壤的微生物群落及其生态功能发生变化, 从而影响到土壤环境的生态可持续性。
转Bt基因作物中的杀虫晶体蛋白主要通过根系分泌物、花粉飘落和秸秆还田释放到环境中[15]。进入土壤的Bt蛋白能够迅速被土壤活性颗粒吸附, 1~3 h就能达到吸附平衡[16]。吸附态Bt蛋白的杀虫活性可保持数周或数月, 转Bt基因玉米的盆栽和大田试验证明 Bt毒素的杀虫活性能保持180 d以上[17]; 转Bt基因玉米、棉花和马铃薯秸秆分解释放的Bt毒素杀虫活性都可保持数周以上[18-20]。Bt毒蛋白在土壤环境中失活或消除的主要途径为进入昆虫体内后被降解、被阳光中紫外线照射分解及微生物的降解和最终矿化作用, 其中土壤微生物是影响Bt毒蛋白在土壤中存留累积的最关键因子[21]。
本研究结果初步揭示转双价抗虫基因水稻的秸秆还田降解对土壤氮素转化关键微生物群落丰度与组成没有明显影响, 不存在影响土壤氮素转化与N2O排放的可能。但由于土壤N2O的产生有多种途径, 由多个酶催化进行, 且不同途径的不同酶催化反应间存在相互制约或促进的复杂关系[22-24]。因此, 还需要通过对参与土壤N2O产生的关键微生物功能基因多样性及其作用进行系统的分析研究, 才能深入揭示转基因水稻对稻田温室气体N2O排放的影响及机制。
[1] MASOERO F, MOSCHINI M, ROSSI F, et al. Nutritive value, mycotoxin contamination and in vitro rumen fermentation of normal and genetically modified corn () grown in northern Italy[J]. Maydica, 1999, 44(3): 205–209
[2] CAPONE D G. Methane, nitrogen oxides and halomethanes[M]// Rodgers J E, Whitman W B. Microbial Consumption of Greenhouse Gases. Washington, DC: American Society for Microbiology, 1991: 225–275
[3] STEPHEN J R, McCAIG A E, SMITH Z, et al. Molecular diversity of soil and marine 16S rRNA gene sequences related to β-subgroup ammonia-oxidizing bacteria[J]. Applied and Environmental Microbiology, 1996, 62(11): 4147–4154
[4] WOESE C R, WEISBURG W G, HAHN C M, et al. The phylogeny of purple bacteria: The gamma subdivision[J]. Systematic and Applied Microbiology, 1985, 6(1): 25–33
[5] ZUMFT W G. Cell biology and molecular basis of denitrification[J]. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533–616
[6] 宋亚娜, 林智敏, 林艳. 氮肥对稻田土壤反硝化细菌群落结构和丰度的影响[J]. 中国生态农业学报, 2012, 20(1): 7–12 SONG Y N, LIN Z M, LIN Y. Response of denitrifying bacteria community structure and abundance to nitrogen in paddy fields[J]. Chinese Journal of Eco-Agriculture, 2012, 20(1): 7–12
[7] LU H H, WU W X, CHEN Y X, et al. Decomposition oftransgenic rice residues and response of soil microbial community in rapeseed-rice cropping system[J]. Plant and Soil, 2010, 336(1/2): 279–290
[8] WU W X, YE Q F, MIN H, et al. Bt-transgenic rice straw affects the culturable microbiota and dehydrogenase and phosphatase activities in a flooded paddy soil[J]. Soil Biology and Biochemistry, 2004, 36(2): 289–295
[9] 宋亚娜, 陈在杰, 林智敏. 水稻生育期内红壤稻田氨氧化微生物数量和硝化势的变化[J]. 中国生态农业学报, 2010, 18(5): 954–958 SONG Y N, CHEN Z J, LIN Z M. Abundance of ammonia-oxidizer and potential nitrification rate of quaternary red-clay paddy soil during rice growth[J]. Chinese Journal of Eco-Agriculture, 2010, 18(5): 954–958
[10] 袁飞, 冉炜, 胡江, 等. 变性梯度凝胶电泳法研究我国不同土壤氨氧化细菌群落组成及活性[J]. 生态学报, 2005, 25(6): 1318–1324 YUAN F, RAN W, HU J, et al. Ammonia-oxidizing bacteria communities and their influence on the nitrification potential of Chinese soils measured by denaturing gradient gel electrophoresis (DGGE)[J]. Acta Ecologica Sinica, 2005, 25(6): 1318–1324
[11] WEI X D, ZOU H L, CHU L M, et al. Field released transgenic papaya effect on soil microbial communities and enzyme activities[J]. Journal of Environmental Sciences, 2006, 18(4): 734–740
[12] GRIFFITHS B S, CAUL S, THOMPSON J, et al. A comparison of soil microbial community structure, protozoa and nematodes in field plots of conventional and genetically modified maize expressing theCryIAb toxin[J]. Plant and Soil, 2005, 275(1/2): 135–146
[13] SONG Y N, SU J, CHEN R, et al. Diversity of microbial community in a paddy soil withtransgenic rice[J]. Pedosphere, 2014, 24(3): 349–358
[14] 宋亚娜, 苏军, 林艳, 等. 转1Ac基因水稻对土壤氨氧化细菌群落组成和丰度的影响[J]. 生物安全学报, 2012, 21(1): 67–73 SONG Y N, SU J, LIN Y, et al. Effect of1Actransgenic rice on community composition and abundance of ammonia-oxidizing bacteria in paddy soil[J]. Journal of Biosafety, 2012, 21(1): 67–73
[15] 王建武, 骆世明, 冯远娇, 等. 转Bt基因作物Bt毒素在土壤中的环境去向及其生态效应[J]. 生态学报, 2003, 23(4): 797–804 WANG J W, LUO S M, FENG Y J, et al. Environmental fate and ecological effects of Bt toxin from transgenic Bt crops in soil[J]. Acta Ecologica Sinica, 2003, 23(4): 797–804
[16] ZHOU X Y, HUANG Q Y, CHEN S W, et al. Adsorption of the insecticidal protein ofon montmorillonite, kaolinite, silica, goethite and red soil[J]. Applied Clay Science, 2005, 30(2): 87–93
[17] SAXENA D, FLORES S, STOTZKY G. Bt toxin is released in root exudates from 12 transgenic corn hybrids representing three transformation events[J]. Soil Biology and Biochemistry, 2002, 34(1): 133–137
[18] SIMS S R, HOLDEN L R. Insect bioassay for determining soil degradation ofsubspCryIA (b) protein in corn tissue[J]. Environmental Entomology, 1996, 25(3): 659–664
[19] DONEGAN K, PALM C J, FIELAND V J, et al. Changes in levels, species and DNA fingerprints of soil microorganisms associated with cotton expressing thevarendotoxin[J]. Applied Soil Ecology, 1995, 2(2): 111–124
[20] DONEGAN K K, SCHALLER D L, STONE J K, et al. Microbial populations, fungal species diversity and plant pathogen levels in field plots of potato plants expressing thevarendotoxin[J]. Transgenic Research, 1996, 5(1): 25–35
[21] 白耀宇, 蒋明星, 程家安, 等. 转Bt基因作物Bt毒蛋白在土壤中的安全性研究[J]. 应用生态学报, 2003, 14(11): 2062–2066 BAI Y Y, JIANG M X, CHENG J A, et al. Advances in safetystudies of soil Bt toxin proteins released from transgenic Bt crops[J]. Chinese Journal of Applied Ecology, 2003, 14(11): 2062–2066
[22] FIRESTONE M K, DAVIDSON E A. Microbial basis of NO and N2O production and consumption in soils[M]//Andreae M O, Schimel D S. Exchange of Trace Gases Between Terrestrial Ecosystems and the Atmosphere. New York: John Wiley, 1989: 7–21
[23] ZHU X, BURGER M, DOANE T A, et al. Ammonia oxidation pathways and nitrifier denitrification are significant sources of N2O and NO under low oxygen availability[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(16): 6328–6333
[24] 朱永官, 王晓辉, 杨小茹, 等. 农田土壤N2O产生的关键微生物过程及减排措施[J]. 环境科学, 2014, 35(2): 792–800ZHU Y G, WANG X H, YANG X R, et al. Key microbial processes in nitrous oxide emissions of agricultural soil and mitigation strategies[J]. Environmental Science, 2014, 35(2): 792–800
Effect of transgenic rice straw return to soil on nitrification and denitrification microbial community*
WANG Peixuan1,3, XU Yan2, SONG Yana1**
(1. Institute of Biological Technology, Fujian Academy of Agricultural Sciences, Fuzhou 350003, China; 2. Fuzhou Shidai Middle School, Fuzhou 350007, China; 3. Affiliated High School of Fujian Normal University, Fuzhou 350007, China)
The cultivation of genetically modified plants may have the potential to alter microbial community structure and function in soils through root exudates and plant residues. Ammonia-oxidizing bacteria or denitrifying bacteria are the key microbes for nitrification or denitrification. A change in community structure of ammonia-oxidizing bacteria or denitrifying bacteria can affect the conversion and utilization of nitrogen in soil. The purpose of this study was to explore the possibility of transgenic rice to induce change in nitrogen transformation and N2O emission in paddy soils. In the study, the abundance and composition of ammonia-oxidizing bacteria or denitrifying bacteria in paddy soils under straw return to soil undertransgenic gene rice ‘Kf8’ or non-transgenic rice ‘Mh86’ were analyzed by real-time PCR and denaturing gradient gel electrophoresis (DGGE) based ongene orgene. The results showed that there were no differences in the abundance ofgene orgene in the soil with returned transgenic rice ‘Kf8’ straw and non-transgenic rice ‘Mh86’ straw. The abundance ofgene in the 0–10 cm soil layer was significantly (< 0.05) higher than that in the 10–20 cm and 20–30 cm soil layers under degraded transgenic rice ‘Kf8’ straw or non-transgenic rice ‘Mh86’ straw. The abundance ofgene in the soil increased with the time of returned straw to soil for either transgenic rice ‘Kf8’ or no-transgenic rice ‘Mh86’. At the same time, the Shannon-Weiner index and composition of ammonia-oxidizing bacteria or denitrifying bacteria in the soil under degraded transgenic rice ‘Kf8’ straw were similar to those under degraded non-transgenic rice ‘Mh86’ straw. The composition of ammonia-oxidizing bacteria in the soil was significantly correlated with the time of rice straw return (= 0.002). Also the abundance of denitrifying bacteria was significantly correlated with the time of rice straw return (= 0.002) and depth of soil (= 0.024). The findings demonstrated that there was no significant effect of returnedtransgenic rice straw to soil on key microbial communities for nitrification or denitrification in soil. In terms of soil microbial community, there was no signiifanct effect of returnedtransgenic rice straw to soil on nitrogen transformation and N2O emission in paddy soils.
Transgenic rice; Straw return; Ammonia-oxidizing bacteria;gene; Denitrifying bacteria;gene; Community structure
Corresponding author, E-mail: syana@sina.com
Feb. 13, 2017; accepted May 18, 2017
10.13930/j.cnki.cjea.170794
S154.3
A
1671-3990(2018)01-0008-08
通信作者:宋亚娜, 主要从事土壤微生物分子生态学研究。E-mail: syana@sina.com 王沛譞, 主要从事土壤微生物分子生态学研究。E-mail: 1092546064@qq.com
2017-02-13
2017-05-18
*This study was supported by the Natural Science Foundation of Fujian Province (2015J01105), the Public Welfare Foundation of Fujian Province (2015R1019-12) and the 30thYouth Innovation Competition Project in Fujian Province.
*福建省自然科学基金项目(2015J01105)、福建省公益类科研院所专项(2015R1019-12)和第30届福建省青少年创新大赛项目资助
王沛譞, 徐焱, 宋亚娜. 转基因水稻秸秆还田对土壤硝化反硝化微生物群落的影响[J]. 中国生态农业学报, 2018, 26(1): 8-15
Wang P X, Xu Y, Song Y N. Effect of transgenic rice straw return to soil on nitrification and denitrification microbial community[J]. Chinese Journal of Eco-Agriculture, 2018, 26(1): 8-15

