基于SNP标记桃抗蚜性状的基因定位
张南南,鲁振华,崔国朝,潘磊,曾文芳,牛良,王志强
基于SNP标记桃抗蚜性状的基因定位
张南南,鲁振华,崔国朝,潘磊,曾文芳,牛良,王志强
(中国农业科学院郑州果树研究所/国家桃葡萄改良中心/农业部果树育种技术重点实验室,郑州 450009)
挖掘与桃抗蚜性状紧密连锁的SNP位点及候选基因,为桃抗性分子标记辅助选择育种奠定基础,并为进一步揭示桃抗蚜性状的遗传基础和分子机制提供依据。以来源于‘粉寿星’的抗蚜桃‘01-77-3’为母本,栽培品种‘中油桃13号’为父本,杂交获得F1代分离群体进行基因定位。以‘96-5-1’(抗蚜)为母本,‘10-7’(感蚜)为父本杂交获得F1分离群体作为验证定位位点准确性的材料。参考桃基因组序列(Genome v2.0.a1)开发基于Sanger测序的SNP标记,在亲本(‘01-77-3’‘中油桃13号’)及各4个子代中PCR扩增后进行Sanger测序,获得候选SNP后,扩大群体验证,实现对抗性基因的初步定位。对亲本(‘01-77-3’‘中油桃13号’‘96-5-1’‘10-7’)进行覆盖度约为70×的全基因组深度测序,并基于重测序数据,在初定位区间内开发基于Sanger测序与HRM分析的SNP标记,筛选多态性标记,对‘01-77-3’ב中油桃13号’杂交后代141株实生苗进行基因分型,以获得与桃抗蚜型基因紧密连锁的分子标记。通过双亲(‘96-5-1’‘10-7’)表型与基因型一致,在精细定位区间内开发与桃抗蚜性状紧密连锁的InDel位点,以验证InDel标记与抗蚜表型是否连锁。最后,参考桃基因组分析定位区间的候选基因。通过人工接种观察蚜虫对桃F1后代单株新梢危害表明,抗蚜与感蚜比例接近1﹕1(值为0.556;χ2为0.348),符合孟德尔遗传规律。基于Sanger测序结果,初步将抗蚜基因定位在桃基因组Pp01_38011783与Pp01_47231340之间,物理距离约为9.22 Mb。亲本全基因组深度测序分别产生Clean data 17.109 Gb,平均覆盖度为75.19×,在Pp01初定位区间内开发基于HRM与Sanger测序的SNP标记共29对,筛选后得到11对紧密连锁的标记。基因分型结果表明,与桃抗蚜基因紧密连锁的标记位于桃基因组Pp01的45.66 Mb和46.12 Mb处,Pp01_45.66处等位基因为T和C,Pp01_46.12处等位基因为G和C,遗传距离分别为1.4 cM、2.1 cM;与抗蚜基因完全连锁的标记SNP_Pp01_45712702,位于桃基因组Pp01_45.71处,等位基因为G和T。基于亲本(‘96-5-1’‘10-7’)重测序数据,在精细定位区间内开发紧密连锁的InDel标记KYYZ_Pp01_45799758,位于Pp01_45.79处。对验证群体92个单株进行PCR扩增,经聚丙烯酰胺凝胶电泳检测表明,仅1个单株基因型与表型不符,分子鉴定准确率为98.91%。利用亲本深度测序与SNP标记技术相结合的方法,精细定位了控制桃抗蚜性状的基因,将抗蚜基因缩小到物理区间460 Kb内,共包含56个转录本,包括52个候选基因。
桃;SNP标记;抗蚜;基因定位
0 引言
【研究意义】桃[(L.)Batsch]是中国栽培面积较大的落叶果树之一。据2014年统计中国桃栽培面积799 500 hm2,占世界栽培总面积的53.3%(FAO)。蚜虫是危害桃产业发展的主要虫害之一,其危害新梢和幼果,严重的会导致叶片卷曲、果实畸形、品质下降、树体生长势减弱甚至死亡[1]。发掘和培育抗蚜品种是防治蚜虫的重要途径,利用现有遗传资源开展相关抗性基因的定位工作具有重要意义。【前人研究进展】蚜虫为半翅目昆虫,包含近5 000种,是地球上最大、分布最广、破坏力最强的植食性昆虫之一[2]。其繁殖力强,生活周期短,常常在短期内就能形成较高的种群密度[3]。据报道,桃绿蚜(GPA)具有很强的抗药性,因为杀虫剂的广泛使用,使得GPA能抵抗众多的杀虫剂[4-6]。法国农业科学院(INRA)最先对‘Weeping Flower Peach’(S2678)、‘Rubira®’(S2605)、‘’(P1908)、‘Summergrand’(S39712)和‘Malo konare’(S5392)5种桃抗蚜资源开展了一系列研究工作,发现桃‘Weeping Flower Peach’和‘Rubira®’具有很强的抗蚜能力,进一步研究表明其中有5种抗蚜基因型[7-8]。初步明确了与抗蚜相关的显性遗传机制,同时阐明3种不同来源的抗蚜资源抗性机制不同[9-11]。目前,中国已经发现了多种抗性资源。其中,牛良等[1]对‘粉寿星’抗蚜性的遗传及其稳定性进行分析,明确了其抗蚜性状由一对显性基因控制。【本研究切入点】尽管中国有丰富的抗蚜资源,但相关遗传资源的研究和利用起步较晚,且对抗蚜机制没有深入研究。基于来源中国重要的抗蚜资源‘粉寿星’,开展抗蚜基因的精细定位是明确桃抗蚜机制与遗传改良的前提。【拟解决的关键问题】本研究在明确‘粉寿星’抗蚜遗传特征的基础上,继续对来源中国‘粉寿星’的桃抗蚜基因进行定位。通过获得与抗蚜基因紧密连锁的分子标记实现早期对目标性状的分子鉴定,提高育种效率,为桃抗蚜资源的挖掘和抗性品种的培育奠定基础。
1 材料与方法
田间试验于2014—2016年在中国农业科学院郑州果树研究所新乡试验基地进行,其他试验于中国农业科学院郑州果树研究所农业部果树育种技术重点实验室进行。
1.1 试验材料
以‘01-77-3’为母本,来源于‘粉寿星’,具有抗蚜性状,基因型为Rr;以‘中油桃13号’为父本,感蚜,基因型为rr。2014年4月手工去雄后并人工授粉,获得杂交后代141株实生苗作为基因定位群体。另外,以‘96-5-1’(抗蚜,Rr)为母本,‘10-7’(感蚜,rr)为父本,人工去雄授粉,获得F1分离后代116株实生苗,用于验证标记与抗蚜表型的连锁性。
1.2 表型鉴定与DNA提取
参考PASCAL等[12]的抗蚜表型鉴定方法和标准确定各单株的抗蚜型和感蚜型。2015年4月上旬将两群体F1代实生苗分别放入网室中进行接种处理,人工接种混合桃绿蚜10头后套上小纱网,蚜虫来自试验园感蚜株,2周后观察蚜虫对新梢的危害,确定抗蚜与感蚜表型(图1)。2016年4月上旬将全部F1代实生苗分别放入网室中进行接种处理,重复鉴定。
采用改良CTAB法[13]提取亲本及F1群体各单株基因组DNA,略作修改。加入100 μL体积的0.1×TE溶解沉淀DNA,同时加入0.5 μL的RNase,37℃放置1 h,去除RNA污染(长期保存在-20℃冰箱,常用则存于4℃冰箱);采用NanoDrop 1000 spectrophotometer(Thermo)和1%的琼脂糖胶对提取的DNA纯度、浓度和完整度进行检测,并稀释成工作液浓度(25 ng∙μL-1),用于后续研究。

左边为感蚜,右边为抗蚜 Left:Sensitive type to green aphid, Right:Resistant type to green aphid
1.3 抗蚜基因的初步定位
参考Genome Database for Rosaceae数据库的桃基因组(Genome v2.0.a1)序列,采用primer3 Web Version 4.0(http://primer3.ut.ee/)设计引物,引物的退火温度在60—63℃,长度20—23 bp,开发基于Sanger测序的SNP标记,选择约每1—2 Mb设计1对引物,扩增片段长度约为1 600 bp或750 bp。引物由生工生物工程(上海)股份有限公司合成。对亲本及各4个子代(抗蚜型和感蚜型)进行PCR扩增(步骤参数详见TaKaRa Ex Taq说明书),PCR产物经琼脂糖凝胶电泳检测,合格产物送生工生物工程(上海)股份有限公司进行测序。将测序结果在CExpress软件中打开,序列对齐后寻找与抗蚜性状紧密连锁的SNP标记,表现为抗蚜型母本基因型为Rr,感蚜型父本基因型为rr,且子代表型与基因型一致。
1.4 亲本深度测序
将检验合格的亲本(‘01-77-3’‘中油桃13号’‘96-5-1’‘10-7’)基因组DNA样品(无污染,样品量>2 μg,样品浓度>50 ng∙μL-1)送至北京诺禾致源科技(天津)股份有限公司进行文库构建,采用Hiseq4000(Illumina,CA)进行基因组重测序,测序深度为70×,下机数据经质量控制及过滤后,获得Clean Data。参考桃基因组,采用IGV软件对生成的数据进行分析,寻找与亲本表型一致的SNP位点。
1.5 抗蚜性状紧密连锁标记的开发
基于两亲本(‘01-77-3’ב中油桃13号’)深度测序数据,在初步定位区间内依据双亲的基因型和表型开发更多的SNP标记,平均每200 Kb处设计1对引物,引物的退火温度在60—63℃,引物长度20—25 bp,扩增片段长度约为150 bp或700 bp。采用HRM(High Resolution Melting)和Sanger测序技术对杂交组合‘01-77-3’ב中油桃13号’后代群体单株进行SNP基因分型,确立紧密连锁的SNP标记。HRM master mix试剂购自Roche,在LightCycler 480II定量PCR仪(Roche)上进行SNP基因分型,HRM基因分型参考鲁振华等[14]的方法。
1.6 基于分子标记的植株表型鉴定
根据已经获得的紧密连锁的SNP标记的物理位置,参考桃基因组数据在亲本(‘96-5-1’‘10-7’)中开发表型和基因型一致的InDel标记KYYZ_Pp01_ 45799758。即在同一位点,抗蚜亲本基因型为Rr,感蚜基因型为rr。随机选取92个杂交F1代单株进行PCR扩增,采用8%非变性聚丙烯酰胺凝胶电泳对扩增片段进行分离、检测。目的是利用定位区间内的分子标记对杂交后代单株的表型进行鉴定。
2 结果
2.1 抗蚜表型鉴定
观察蚜虫对桃新梢的危害,确定抗蚜和感蚜的表型,其中呈红点状无其他危害症状的为抗蚜类型,具有明显危害症状的为感蚜类型。对‘01-77-3’ב中油桃13号’杂交后代141株实生苗的表型评价表明,67株实生苗抗蚜特性明显,无蚜虫侵害,无卷叶,新梢蚜虫刺探部位有明显的红色斑点,为抗蚜型;74株实生苗新稍叶背均有蚜虫,出现不同程度的卷叶(2—4级),为感蚜型;二者比例接近1﹕1(值为0.556,χ2为0.348),符合孟德尔遗传规律。‘96-5-1’ב10-7’杂交获得F1后代116株实生苗,经表型鉴定后随机选取92个单株(抗蚜型33株,感蚜型59株),用于验证定位区间的准确性。
2.2 基因的初步定位
基于Genome Database for Rosaceae数据库的桃基因组(Peach v2.0)序列,依据双亲重测序数据,开发与抗蚜表型一致的SNP标记,共设计引物108对,在亲本及感蚜型、抗蚜型各4个子代中PCR扩增后进行Sanger测序。将测序结果在CExpress软件中打开,序列对齐后根据亲本与子代的基因型和表型寻找候选SNP标记,筛选出基因型与表型一致的引物68对,初步获得可能连锁的标记;通过扩大杂交群体,在抗蚜型和感蚜型各20个后代单株中进一步验证连锁关系,进而在全部样品中验证。最终获得了与抗蚜性状连锁的SNP标记SNP_Pp01_38011783与SNP_Pp01_47231340,分别位于桃参考基因组(v2.0.a1)Pp01的38.02 Mb与47.23 Mb处,将目的基因初步定位至Pp01_38011783与Pp01_47231340之间,遗传距离约为9.22 Mb。
2.3 亲本深度测序数据分析
本次测序共产生Raw data均为17.956 Gb,过滤后的Clean data 17.109 Gb,测序质量较高,其中Q20≥94.85%,Q30≥88.57%,全基因组GC含量为39.04%。参考基因组大小为227 411 381 bp,所有样本的比对率为96.75%,对参考基因组(排除N区)的平均覆盖深度为75.19×,1×覆盖度(至少有一个碱基的覆盖)在98.87%以上。
分析结果显示,样本测序质量合格,GC分布正常,建库和测序成功,可用于后续的变异检测及相关分析。
2.4 基因的精细定位
根据深度测序数据,开发杂交群体亲本‘01-77-3’ב中油桃13号’的SNP。在Pp01初定位区间9.22 Mb内,依据桃参考基因组设计引物,避开Poly区域、重复序列和微卫星区域,以便能够顺利完成测序并获得更多的SNP信息,在141个后代单株中扩增,抗蚜生长型为Rr,感蚜生长型为rr。29对引物(表1)中,26对引物存在多态的SNP位点,约占总引物数的89.66%,其中11对引物的基因型与表型一致(图2)。
结合Sanger测序与HRM对141个单株进行基因分型,获得的重组单株如图3。在抗蚜基因位点两翼各筛选出1对引物SNP_Pp01_45665389与SNP_Pp01_ 46120950,与抗蚜基因紧密连锁。分别位于桃参考基因组Pp01的45.66 Mb和46.12 Mb位置,在母本‘01-77-3’中引物SNP_Pp01_45665389的SNP位点为C/T,在父本‘中油桃13号’中SNP位点为T/T(图4);而在母本‘01-77-3’中引物SNP_Pp01_ 46120950的SNP位点为G/C,在父本‘中油桃13号’中SNP位点为C/C。

表1 基因定位引物序列
另外,在以上两个紧密连锁的标记之间获得一对引物SNP_Pp01_45712702,与抗蚜基因完全连锁,位于桃基因组Pp01的45.71 Mb处,在母本‘01-77-3’中SNP位点为G/T,在父本‘中油桃13号’中SNP位点为G/G(图5)。
2.5 基于分子标记的植株表型鉴定
根据已经获得的紧密连锁的SNP标记的物理位置,在亲本(‘96-5-1’‘10-7’)中开发表型和基因型一致的InDel标记KYYZ_Pp01_45799758(表1),并在92个后代单株中进行PCR扩增,产物经聚丙烯酰胺凝胶电泳检测后,抗蚜单株呈现明显的两条带,分别为150 bp与200 bp,感蚜单株呈单条带,为150 bp;且150 bp处不同表型对应条带具有明显的多态性(图6),证明该InDel可用于验证标记与表型是否连锁。通过标记基因型和表型比较发现,在92个样品中,仅1个样品基因型与表型不一致,验证符合率为98.91%。

图2 与目标性状的连锁SNP标记在LG01的位置
基于已将目的基因定位在Pp01_45665389—Pp01_46120950,物理距离约460 kb,介于Prupe. 1G559300与Prupe.1G564800两基因之间,共包含56个转录本,52个候选基因,其中包括虫漆酶14、β-D-木糖苷酶、γ-微管蛋白、Loricrin相关蛋白、DNA结合蛋白、P环核苷三磷酸水解酶蛋白超家族、磷酸吡哆醛(PLP)端依赖转移酶蛋白超家族等(详见基因注释v2.0,https://www.rosaceae.org/search/genes)。
3 讨论
单核苷酸多态性(single nucleotide polymorphism,SNP)是指单个核苷酸的变异导致的DNA序列多态性,且等位基因频率不低于1%[15]。目前,SNP标记已被广泛用于遗传图谱构建、分子标记辅助选择育种[16]和全基因组关联分析[17-20]。VERDE等[21]通过对56个桃进行测序,获得了大量SNP位点,分布于桃的8对染色体,平均约每26.7 Kb一个SNP。本研究参考桃基因组数据,利用亲本全基因组深度测序数据开发SNP标记,多态率为40%。本研究基于初步定位的区间,利用双亲重测序数据有助于开发更多SNP标记,解决了亲本亲缘关系较近时,难以获得多态的SNP标记的问题;且获得的SNP标记具有针对性,更准确、有效,有利于高效、精确地定位抗蚜基因。
桃蚜是春季危害桃树最常见的害虫之一,4—5月为蚜虫繁殖高峰期,其群集于新梢嫩叶背面危害,抑制新梢生长,危害幼果,导致果实畸形,品质下降[22]。关于桃蚜的防治已有多种报道,化学农药的残留会对环境产生威胁、蚜虫的抗药性等问题的出现,使得化学防治在桃园管理方面的作用逐渐减小,生物防治对桃蚜虫的控制也具有很大的局限性[23-25]。因此,研究桃抗蚜遗传机制,培育抗蚜新品种是防治蚜虫的一种有效途径。已有报道表明甜瓜、番茄、苹果等多种植物中存在抗蚜基因[26-29]。Monet等[30]最早发现桃‘Weeping Flower Peach’具有抗蚜性状,受显性单基因控制,并将该抗蚜基因命名为。随后法国农业科学院(INRA)对抗蚜资源‘Rubira®’进行抗性遗传特征及基因定位研究,发现其受显性单基因控制,并将抗性基因定位在pchgms29与UDAp-467两标记之间,位于LG1末端Pp01_ 43499521—Pp01_46502361区间内,物理距离约3 Mb[12,31]。PASCAL等[32]研究发现来源于‘Weeping Flower Peach’的抗蚜基因也位于LG1末端,定位区间为物理距离2 880 352 bp(遗传距离7.2 cM),并认为与位于染色体的同一位点。本研究以来源于中国的重要抗性资源‘粉寿星’为材料,将抗蚜基因定位至Pp01,物理距离仅460 kb的区间内(遗传距离约3.5 cM),但来源于‘粉寿星’的抗蚜基因与与是否位于Pp01的同一位点二者是否存在不同的变异形式以及来源是否相同,还需进一步研究。另外,虽然将精细定位区间缩小至460 Kb(包含52个候选基因),但控制抗蚜性状的候选基因还不能确定,仍需进一步研究。

GY表示感蚜,KY表示抗蚜;黄色表示基因型为Rr;绿色表示基因型为rr;红色表示重组单株
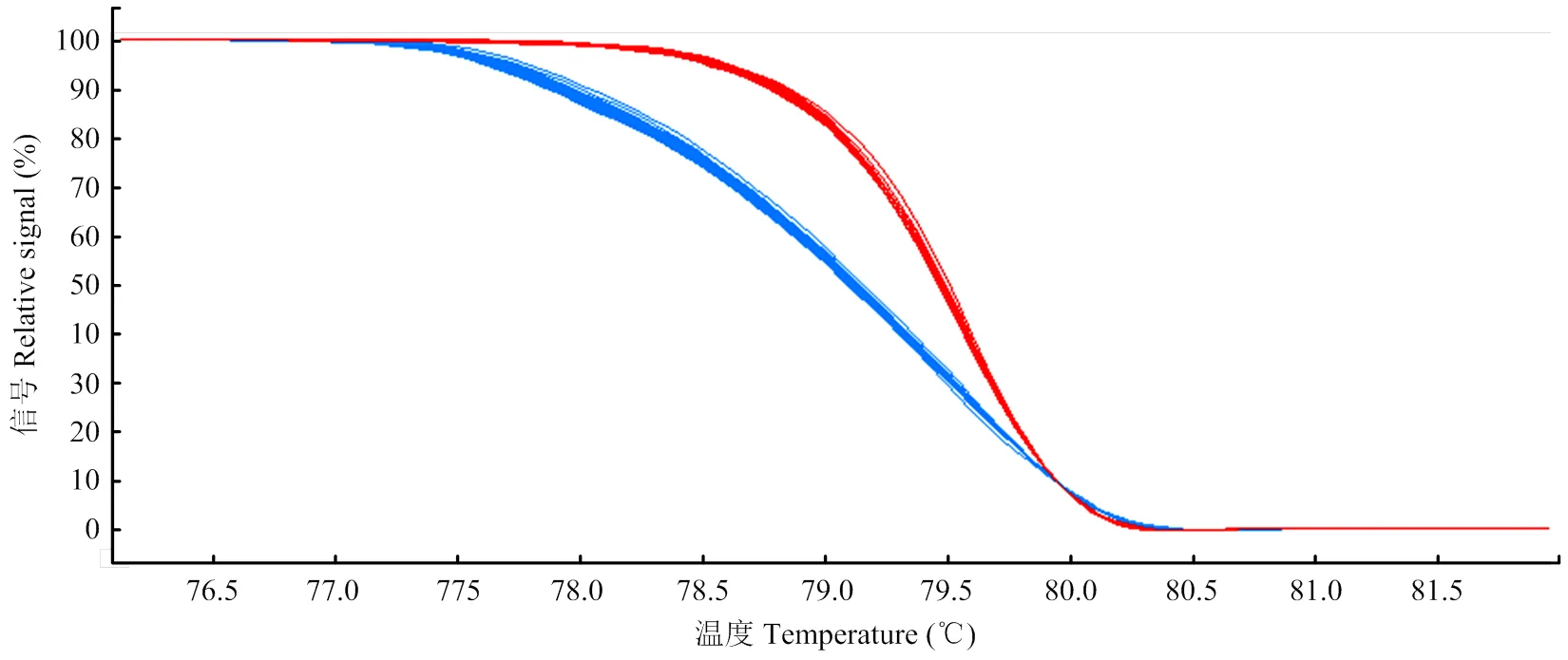
图4 基于HRM抗蚜C/T(蓝色曲线)和感蚜T/T(红色曲线)位点的SNP鉴定

A:抗蚜基因型G/T;B:感蚜基因型G/G A: Aphid resistant genotype-G/T; B: Aphid susceptibility genotype-G/G

M表示DNA标记500;R表示抗蚜;S表示感蚜;♀:母本;♂:父本
抗蚜性状鉴定具有多种途径。其中,传统的田间观察是最直观、简便的方法,但由于抗蚜表型易受外界环境影响,且蚜虫的发生具有季节性[22],表型鉴定耗时长,且需对群体单株逐一调查,需投入大量人力物力[33]。分子标记是一种有效的性状鉴定方法,本研究基于精细定位区间,开发了与性状连锁的InDel标记,缺失大小为2 bp,准确、有效地完成了表型的分子鉴定。
4 结论
本研究基于亲本全基因组深度测序与SNP分型相结合的方法,对目标性状进行基因定位。通过基因定位初步将目标性状定位在桃基因组Pp01,物理区间约9.22 Mb。在初步定位区域内利用重测序数据开发与抗蚜表型一致的SNP标记,利用基于高分辨率熔解曲线(HRM)结合Sanger测序进一步进行精细定位,将目标基因缩小至Pp01_45665389—Pp01_46120950区域内,物理距离460 kb,包含56个转录本,52个候选基因。
[1] 牛良, 鲁振华, 曾文芳, 崔国朝, 潘磊, 徐强, 李国怀, 王志强. ‘粉寿星’对桃绿蚜抗性的遗传分析. 果树学报, 2016, 33(5): 578-584.
NIU L, LU Z H, ZENG W F, CUI G C, PAN L, XU Q, LI G H, WANG Z Q. Inheritance analysis of resistance to green peach aphid ((Sulzer)) for peach cultivar ‘’ (var.)., 2016, 33(5): 578-584. (in Chinese)
[2] SMITH C M, CHUANG W B. Plant resistance to aphid feeding: Behavioral, physiological, genetic and molecular cues regulate aphid host selection and feeding., 2014, 70(40): 528-540.
[3] BLACKMAN R L, EASTOP V F.:. John Wiley & Sons Ltd, 2000.
[4] DEVONSHIRE A L, FIELD L M, FOSTER S P. The evolution of insecticide resistance in the peach-potato aphid,., 1998, 353(1376): 1677-1684.
[5] FUENTES-CONTRERAS E, FIGUEROA C C, REYES M.Genetic diversity and insecticide resistance of() populations from tobacco in Chile: evidence for the existence of a single predominant clone., 2004, 94(1): 11-18.
[6] FOSTER J C, RIDEOUT W. Storm enhanced density: Magnetic conjugacy effects., 2007, 25(8): 1791-1799.
[7] MASSONIÉ G, MAISON P, MONET R. Résistance au puceron vert du pêcherSulzer () chezL. Batsch. et d’autres espèces de prurrus., 1982, 2(1): 63-70.
[8] MONET R, MASSONIÉ G. Déterminisme génétique de la résistance au puceron vert () chez le pêcher Résultats complémentaires., 1994, 14(3): 177-182.
[9] SAUGE M H, KERVELLA J, RAHBÉ Y. Probing behaviour of the green peach aphidon resistantgenotypes., 1998, 89(3): 223-232.
[10] SAUGE M H, LACROZE J P, POËSSEL J L. Induced resistance byin the peach cultivar ‘Rubira’., 2002, 102(1): 29-37.
[11] SAUGE M H, MUS F, LACROZE J P. Genotypic variation in induced resistance and induced susceptibility in the peach-aphid system., 2006, 113(2): 305-313.
[12] PASCAL T, PFEIFFER F, KERVELLA J, LACROZE J P, Sauge M H, Weber W E. Inheritance of green peach aphid resistance in the peach cultivar ‘Rubira’., 2002, 121(5): 459-461.
[13] LU Z, NIU L, CHAGNÉ D. Fine mapping of the temperature- sensitive semi-dwarf () locus regulating the inter-node length in peach ()., 2016, 36(2): 1-11.
[14] 鲁振华, 牛良, 张南南, 崔国朝, 潘磊, 曾文芳, 王志强. 基于HRM获得与桃紧密连锁的SNP标记. 中国农业科学, 2017, 50(8): 1505-1513.
LU Z H, NIU L, ZHANG N N, CUI G C, PAN L, ZENG W F, WANG Z Q. SNP marker tightly linked tofor peach using high resolution melting analysis., 2017, 50(8): 1505-1513. (in Chinese)
[15] BROOKES A J. The essence of SNPs., 1999, 234(2): 177-186.
[16] MURANTY H, JORGE V, BASTIEN C.Potential for marker-assisted selection for forest tree breeding: lessons from 20 years of MAS in crops., 2014, 10(6): 1491-1510.
[17] DIRLEWANGER E, COSSON P, BOUDEHRI K. Development of a second-generation genetic linkage map for peach [(L.) Batsch] and characterization of morphological traits affecting flower and fruit., 2006, 3(1): 1-13.
[18] PICAÑOL R, EDUARDO I, ARANZANA M J. Combining linkage and association mapping to search for markers linked to the flat fruit character in peach., 2013, 190(2): 279-288.
[19] MARTÍNEZ GARCÍA J S. Fracaso escolar, PISA y la difícil ESO., 2013, 2(1): 56-85.
[20] HAN Y, CHAGNÉ D, GASIC K. BAC-end sequence-based SNPs and Bin mapping for rapid integration of physical and genetic maps in apple., 2009, 93(3): 282-288.
[21] VERDE I, BASSIL N, SCALABRIN S. Development and evaluation of a 9K SNP array for peach by internationally coordinated SNP detection and validation in breeding germplasm.2012, 7(4): e35668.
[22] 许建兰, 张斌斌, 马瑞娟, 俞明亮, 沈志军, 周懋. 春季异常气候对桃树蚜虫发生及坐果率的影响. 江西农业学报, 2014, 26(11): 21-23, 28.
XU J L, ZHANG B B, MA R J, YU M L, SHEN Z J, ZHOU M. Effects of Spring abnormal climate on aphid occurrence and fruit setting rate of peach., 2014, 26(11): 21-23, 28. (in Chinese)
[23] 乔岩, 董杰, 王品舒, 岳瑾, 张保常, 张金良, 袁志强, 杨建国. 三种生物源农药对桃树蚜虫的防治效果研究. 生物技术进展, 2015, 5(6): 468-470.
QIAO Y, DONG J, WANG P S, YUE J, ZHANG B C, ZHANG J , YUAN Z Q, YANG J G. Control effects of three biogenic pesticides on controlling peach aphids., 2015, 5(6): 468-470. (in Chinese)
[24] MENG J Y, ZHANG C Y, CHEN X J. Differential protein expression in the susceptible and resistant(Sulzer) to imidacloprid., 2014, 115: 1-8.
[25] CRAVEDI P, CERVATO P. Resistance to insecticides of the green peach aphid and integrated fruit production guidelines., 1997, 20: 75-78.
[26] PAUQUET J, BURGET E, HAGEN L. Map-based cloning of thegene from melon conferring resistance to both aphid colonization and aphid transmission of several viruses//:, 2004: 325-329.
[27] KALOSHIAN I, YAGHOOBI J, LIHARSKA T. Genetic and physical localization of the root-knot nematode resistance locusin tomato., 1998, 257(3): 376-385.
[28] MILLIGAN S B, BODEAU J, YAGHOOBI J. The root knot nematode resistance genefrom tomato is a member of the leucine zipper, nucleotide binding, leucine-rich repeat family of plant genes., 1998, 10(8): 1307-1319.
[29] ALSTON F H, BRIGGS J B. Resistance to(in apple., 1968, 17(3): 468-472.
[30] MONET R, GUYE A, MASSONIE G. Breeding for resistance to green aphidin the peach., 1997: 171-176.
[31] LAMBERT P, PASCAL T. Mappinggene conferring resistance to the green peach aphid (Sulzer) in the peach cultivar ‘Rubira®’., 2011, 7(5): 1057-1068.
[32] PASCAL T, ABERLENC R, CONFOLENT C. Mapping of new resistance () and ornamental () Mendelian trait loci in peach., 2017, 213(6): 132.
[33] FURBANK R T. Plant phenomics: from gene to form and function., 2009, 36(10): 5-6.
(责任编辑 赵伶俐)
Gene Mapping of Aphid-resistant for Peach Using SNP Markers
Zhang NanNan, Lu ZhenHua, Cui GuoChao, Pan Lei, Zeng WenFang, Niu Liang, Wang ZhiQiang
(Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences/National Peach and Grape Improvement Center/ Key Laboratory of Fruit Breeding Technology of Ministry of Agriculture, Zhengzhou 450009)
The objective of this study is to identify the SNP loci tightly linked to peach((L.) Batsch ) aphid((Sulzer)) resistance traits, revealing its genetic basis and laying a foundation for the marker assistant selection in resistance breeding of peach.In this study, the population used for the mapping study consisted of141 individuals which were obtained from a cross between female parent (‘01-77-3’ ) and male parent (‘CN13’). Referencing the peach genome and using Sanger sequencing, single nucleotide polymorphism (SNP) markers were developed in female and male parents and 8 progenies to obtain markers linked to the target loci which were tested on the whole population. Subsequently, using whole genome re-sequencing data of two parents, SNPs for fine mapping were selected based on the genotype of two parents, andemployed to conduct genotyping to obtain the SNP marker linked to resistance traits. Ultimately, the fine mapping region was validated by using an InDel marker to verify the genotype of F1population generated from ‘96-5-1’ × ‘10-7’.As a result of phenotypeidentification of 141 progenies, the segregation ratio of resistance to aphid to susceptible ones showed 1﹕1 (: 0.556; χ2: 0.348). Using Sanger sequencing we mapped the resistant gene to an approximate 9.92 Mb physical distance between two SNP markers, Pp01_38011783 and Pp01_47231340 on Pp01. For fine mapping, a total of 17.109 Gb clean data was generated from genome re-sequencing and the average coverage depth is 75.19×. 11 of 29 pairs of primers which were designed based on genome re-sequencing data were effective and linked to target trait. As a result of genotyping, we obtained two SNP makers tightly linked to desired trait, SNP_Pp01_45665389 and SNP_Pp01_46120950, with genetic distance of 1.4 cM and 2.1 cM, respectively. The target locus was between these two markers, an approximate physical distance of 460 Kb, and the gene was co-segregating with another marker SNP_Pp01_45712702. With fine gene mapping region, an InDel marker, KYYZ_Pp01_45799758, was designed and used to verify the phenotype of 92 individuals generated from an F1segregation population of ‘96-5-1’ ב10-7’ with 98.91% accuracy.The SNP loci and candidate genes related closely with aphid-resistant gene of peach were identified in this study. The resistant gene had been mapped to an approximate 460 kb physical distance between two SNP markers, SNP_Pp01_45665389 and SNP_Pp01_46120950 at the bottom of Pp01 which contains 56 transcripts (52 candidate genes).
; SNP markers; aphid-resistant; gene mapping
2017-06-02;
2017-09-07
国家自然科学基金(31470679)、中国农业科学院科技创新工程(CAAS-ASTIP-2016-ZFRI)、河南省科技计划项目(152102110110)
联系方式:张南南,Tel:18638212579;E-mail:18763895031@163.com。通信作者牛良,Tel:13786834196,E-mail:niuliang@caas.cn;通信作者王志强,Tel:13703841063,E-mail:wangzhiqiang@caas.cn

