水稻OsHKT基因表达模式分析
崔立新 和亚男 李亚萍 谢先芝,*
(1山东师范大学 生命科学学院,济南 250014;2山东省水稻研究所,济南 250100;*通讯联系人,E-mail: xzhxie2010@163.com)
水稻OsHKT基因表达模式分析
崔立新1,2和亚男2李亚萍1,2谢先芝1,2,*
(1山东师范大学 生命科学学院,济南 250014;2山东省水稻研究所,济南 250100;*通讯联系人,E-mail: xzhxie2010@163.com)
【目的】OsHKT(High-affinity K+transporter)是与水稻耐盐胁迫有关的一类Na+或K+转运体或Na+-K+共转运体,对水稻体内Na+再循环,维持植株地上部特别是叶片中的低Na+浓度和低Na+/K+比具有重要作用。本研究的目的是分析OsHKT基因家族表达的组织特异性、昼夜节律性以及盐、ABA对其表达的影响。【方法】利用荧光定量PCR技术分析OsHKT家族基因的表达模式。【结果】OsHKT基因家族中不同成员的表达具有明显的组织特异性,OsHKT1;1、OsHKT1;3、OsHKT2;3以及OsHKT2;4主要在水稻的叶片中表达,其余基因在根中表达水平较高。分析盐处理后OsHKT家族基因在水稻根和叶中的表达模式,结果表明OsHKT家族基因的表达均受盐胁迫的影响,但是不同OsHKT家族成员对盐胁迫反应不同。盐处理后OsHKT1;5、OsHKT2;1和OsHKT2;2基因在根和叶中的表达模式是一致的,而其他家族成员在根和叶中存在差异。所有 OsHKT基因的表达在根或叶片中均受ABA调控,其中只有 OsHKT1;3和 OsHKT1;5在根和叶片中表达模式相似。光周期表达模式结果表明,OsHKT基因家族成员的表达具有相似的昼夜节律性。【结论】本研究结果表明水稻 OsHKT家族不同成员的表达具有组织特异性和相似的昼夜节律性,并且不同成员对盐胁迫和ABA应答反应不同。这种表达模式可能与水稻在不同环境下的盐胁迫反应相适应。本研究结果为进一步揭示OsHKT基因的作用机制奠定了基础。
OsHKT;表达模式;盐胁迫;ABA;组织特异性;光周期
HKT转运蛋白(high-affinity K+transporter)是质膜上的一种离子运输体,是生物界普遍存在的负责Na+或K+转运或Na+-K+共转运的一种跨膜蛋白。在结构上,HKT有8个跨膜结构域,中间是4个孔环(pore-loop,P-loop)组成的核心区,这4个孔环与1个中心孔状区域相连。其中第1个孔环最重要,可分为两种类型。类型Ⅰ:第1个孔环中含有丝氨酸,丝氨酸是 Na+特异性载体,该家族成员主要来自双子叶植物[1],大多是对于 Na+有特异性的低亲和转运蛋白;类型Ⅱ:第 1个孔环中含有甘氨酸,是K+选择性载体[2-4],该家族成员都来自单子叶植物,是 Na+-K+的协同运输体或 Na+/K+的单一运载体。HKT蛋白含有4个MPM(membrane-pore-membrane)基序,即每个基序有2个跨膜区域(M1和M2)和1个孔状区域组成。K+通道是由4个亚基组成的聚合体,而Na+通道则由4个MPM基序和多个跨膜区域组成。
在拟南芥(Arabidopsis thaliana)中仅发现一个HKT类蛋白AtHKT1;1,主要参与Na+在维管组织中的转运[5]。在一定的盐胁迫下,AtHKT1;1对于钠离子的摄取具有较低的亲和性,且受钾离子浓度的影响,在调控植物体内的钠钾分配起着很重要的作用[6]。Wang等[7]提出假设,在正常钾离子浓度范围内,AtHKT1;1的主要功能是卸载钠离子;而在低浓度的钾离子浓度下,它对于钠离子的摄取起着重要的作用。Sunarpi等[8]采用GUS染色和免疫定位技术证明,AtHKT1;1主要在木质部薄壁细胞中表达,在韧皮部组织中仅有少量表达,因而提出AtHKT1;1从根木质部汁液中卸载 Na+到周围薄壁细胞中,防止过多Na+上运至地上部。
1994年,Schachtman[9]从单子叶植物小麦中分离出TaHKT1基因。Horie等[2]发现了水稻中 2个HKT基因(OsHKT1和 OsHKT2)。目前已经在日本晴中发现了9个HKT类基因(OsHKT1~OsHKT9)。早期对水稻中 HKT的分类和命名是根据其发现顺序进行的。如水稻中发现的第 2个 HKT,写成OsHKT2。随着HKT基因家族研究的不断深入,逐渐发现了这种命名法的一些问题,特别是它不能反映HKT的相关功能。基于HKT的分子结构研究,Platten等[10]将OsHKT蛋白分为两类:第1类为Na+转运蛋白[11,12],包括 OsHKT1;1、OsHKT1;2、OsHKT1;3、OsHKT1;4和OsHKT1;5等;第2类为Na+-K+共转运蛋白,包括水稻 OsHKT2;1、OsHKT2;2和OsHKT2;3等。有研究发现OsHKT2;4主要是钾转运蛋白,尤其是在低钠浓度下[13]。但OsHKT2;1有多种渗透模式,既属于Na+-K+共转运蛋白,也属于 Na+转运蛋白;它的转运状态,取决于外部的Na+和K+的浓度[14,15]。书写时在HKT后面第一个数字分别用1或2表示HKT属第一亚类或第二亚类,然后隔以分号(;),再写第 2个数字,该数字根据 HKT发现的次序先后编号排序。HKT基因通常含有两个内含子,且第一类上的内含子明显比第二类大。这种命名方法的优点在于可以分析不同物种相同HKT基因或相同物种不同HKT基因在进化和功能上的区别和联系[16]。
目前,对水稻 OsHKT基因的功能研究已有报道。盐胁迫下,OsHKT1;4在水稻的生殖生长阶段可以外排茎和叶片中的钠离子;而在营养生长阶段,外排钠离子的贡献非常低[17]。盐胁迫下,OsHKT2;1和OsHKT2;2在耐盐性水稻品种Nona Bokra中可以调节钠离子的进入,从而避免钠离子毒害[18]。盐胁迫下 OsHKT2;1的转录水平会下降[15]。OsHKT1;1主要在叶的韧皮部表达,盐胁迫下会提高表达量,减少水稻地上部Na+的积累;而且可被MYB类转录因子调控[13]。OsHKT1;5可以减少钠离子向地上部的转运,从而提高盐胁迫耐性[19]。也有研究表明,OsHKT2;4是HKT中的一个新成员,具有高的钾离子渗透性和低钠离子渗透性[20]。尽管 OsHKT基因功能有一些报道,但是研究均不系统。本研究对OsHKT基因表达的组织特异性、盐胁迫处理、ABA处理和昼夜节律性的表达模式进行了系统研究,为深入揭示 OsHKT基因在水稻生长发育中的作用提供参考。
1 材料与方法
1.1 植物材料
供试材料为水稻品种日本晴(Oryza sativa L.,cv. Nipponbare)。
材料的培养和处理:野生型(WT)水稻种子经过浸种、催芽后先种于光照培养箱(28℃,光照强度为7200 lx,6:00光照开始,23:00光照结束)下的去离子水中培养3 d,然后换成Yoshida培养液(pH=5.7)培养,分别挑选4叶期健壮一致的幼苗用于脱落酸处理、NaCl处理和光周期处理实验。
脱落酸(ABA)处理:将100 μmol/L的ABA溶液(含 0.2%的吐温 20)喷洒叶片表面并浇灌植株根部,分别在处理0 h、2 h和6 h后取叶片和根部样品。NaCl处理在营养液中进行,幼苗首先在营养液中生长到4叶期,随后加入含有200 mmol/L NaCl的Yoshida营养液,在处理0 h、1 h和24 h分别取叶片和根的样品。
光周期处理:在日本晴幼苗4叶期,分别于当日 6:00、10:00、14:00、18:00、22:00 及次日 2:00、6:00取叶片样品进行基因表达的光周期特性分析。以上每处理均设置3次重复。
1.2 RNA的提取与反转录
采用 RNAiso Plus试剂(TaKaRa,中国大连)提取总RNA,利用DNaseⅠ(TaKaRa)处理以除去样品中的DNA污染。样品质量经琼脂糖凝胶电泳和紫外分光光度计检测后,取 1 μg的总 RNA,参照cDNA合成试剂盒(TaKaRa)说明书反转录cDNA。
1.3实时RT-PCR分析
根据水稻ABC1以及OsHKT家族中的各个基因的cDNA序列设计引物(表1),实时RT-PCR分析使用生物染料法荧光定量试剂盒(SYBR Premix Ex TaqTM,TaKaRa)在ABI PRISM 7500实时PCR系统上进行。cDNA稀释约20倍后进行PCR扩增。每个反应包括 10 μL 2×SYBR Premix Ex Taq、2 μL cDNA模板以及 0.2 µmol/L基因特异性引物对(表1),总体积为20 μL。反应条件如下: 95℃下预变性3 min; 95℃下变性15 s,55℃下复性15 s,72℃下延伸45 s,40个循环。每个样品进行3次重复,采用 2-ΔΔCT方法计算目标基因相对表达量,其中ΔΔCT=处理(CT目标基因-CTActin)-对照(CT目标基因-CTActin)。
1.4数据统计分析
采用Microsoft Excel的单因素方差分析进行数据统计分析。

表1 本研究中所用的荧光定量PCR引物Table 1. Real time RT-PCR primers used in the study.
2 结果与分析
2.1OsHKT基因在水稻中的组织特异性表达
我们首先利用荧光定量PCR比较了OsHKT家族中的各个基因在日本晴水稻叶片和根中的相对表达水平。如图1所示,OsHKT1;1、OsHKT1;3、OsHKT2;3以及OsHKT2;4主要在水稻的叶片中表达;OsHKT1;5主要在根中表达;OsHKT1;2、OsHKT1;4、OsHKT2;1和OsHKT2;2在根和叶中均有表达,但在根中表达水平较高。由此可以看出,OsHKT家族不同成员基因的表达具有明显的组织特异性。
2.2盐处理对OsHKT家族基因表达水平的影响
OsHKT作为一种 Na+/K+转运蛋白,在调控水稻Na+、K+离子转运中发挥着功能。因此,我们利用荧光定量PCR比较NaCl处理前后OsHKT家族基因在水稻根和叶中的相对表达量差异。如图2-A所示,在根部,NaCl处理后 OsHKT1;2、OsHKT1;3、OsHKT1;4、OsHKT1;5和OsHKT2;3的表达受到抑制;OsHKT1;1、OsHKT2;1、OsHKT2;2 和 OsHKT2;4的表达在NaCl处理1 h后上调,而在NaCl处理24 h后表达受到抑制。如图 2-B所示,在地上部,OsHKT1;1、OsHKT1;2、OsHKT1;4、OsHKT2;1 和OsHKT2;2分别在NaCl处理1 h后上调表达,而在NaCl处理24 h后表达受到抑制;OsHKT1;5在NaCl处理后表达受到抑制;而OsHKT2;3和OsHKT2;4在盐处理1 h后表达受到抑制,在NaCl处理24 h表达量恢复到正常水平;OsHKT1;3基因的表达不受影响。
通过比较,我们发现NaCl处理前后OsHKT1;5、OsHKT2;1和OsHKT2;2在根和叶中的表达模式是一致的;而NaCl处理前后OsHKT1;1、OsHKT1;2、OsHKT1;3、OsHKT1;4、OsHKT2;3和 OsHKT2;4的表达在根和叶中存在差异。以上结果表明,OsHKT家族中基因的表达均受NaCl胁迫的影响,但是OsHKT家族不同转运蛋白基因的表达对NaCl胁迫反应不同。
2.3 ABA对OsHKT家族基因表达水平的影响
比较了ABA处理前后,OsHKT家族中的各个基因在日本晴水稻根中的相对表达水平(图 3-A)。
OsHKT2;3和OsHKT2;4的表达受ABA处理后上调;而OsHKT2;1 和OsHKT2;2的表达在ABA处理2 h后上调,而在ABA处理6 h后表达受到抑制。随后我们又比较了ABA处理前后,OsHKT家族中的各个基因在日本晴水稻地上部的相对表达水平。如图3-B所示,OsHKT家族中的OsHKT1;3和OsHKT1;5在 ABA处理之后表达量上调;OsHKT1;1、OsHKT1;2、OsHKT1;4、OsHKT2;1和 OsHKT2;2的表达量均下调;仅有OsHKT2;3和OsHKT2;4的表达量几乎不受 ABA处理的影响。通过比较,发现ABA处理前后OsHKT1;3和OsHKT1;5在根和地上部的表达模式是一致的;而OsHKT家族中的其他基因在根和叶中的表达存在差异。说明所有OsHKT基因的表达均受ABA调控。

图1 OsHKT基因家族在水稻根和叶片中的表达水平Fig. 1. Relative expression level of OsHKTfamily genes in rice root and leaf.
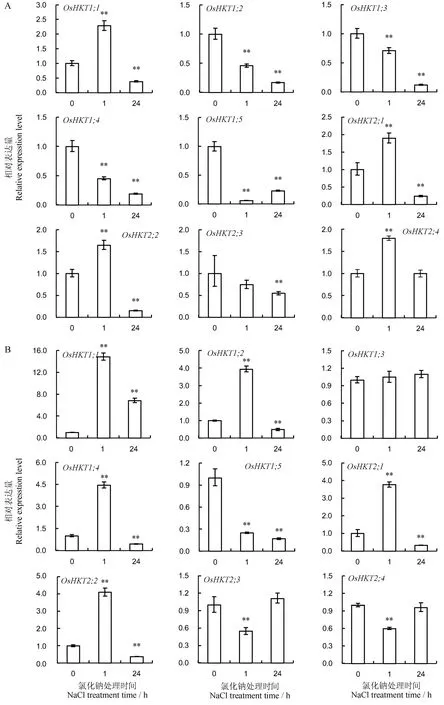
图2 NaCl处理对OsHKT基因家族表达水平的影响Fig. 2. Relative expression level of OsHKT family genes with the NaCl treatment.

图3 ABA处理对水稻苗期OsHKT家族基因表达水平的影响Fig. 3. Relative expression level of OsHKTfamily genes in rice seedling treated with ABA.
2.4光周期表达模式分析
比较了 OsHKT家族基因在长日照下的昼夜节律表达模式。结果表明,OsHKT家族中的所有成员的表达均存在昼夜节律现象,并且表达模式基本一致,即光期表达水平逐渐下降,在光期结束时达到最低(18:00-22:00),在暗期表达水平逐渐升高,在暗期结束时(6:00)达到最高(图4)。
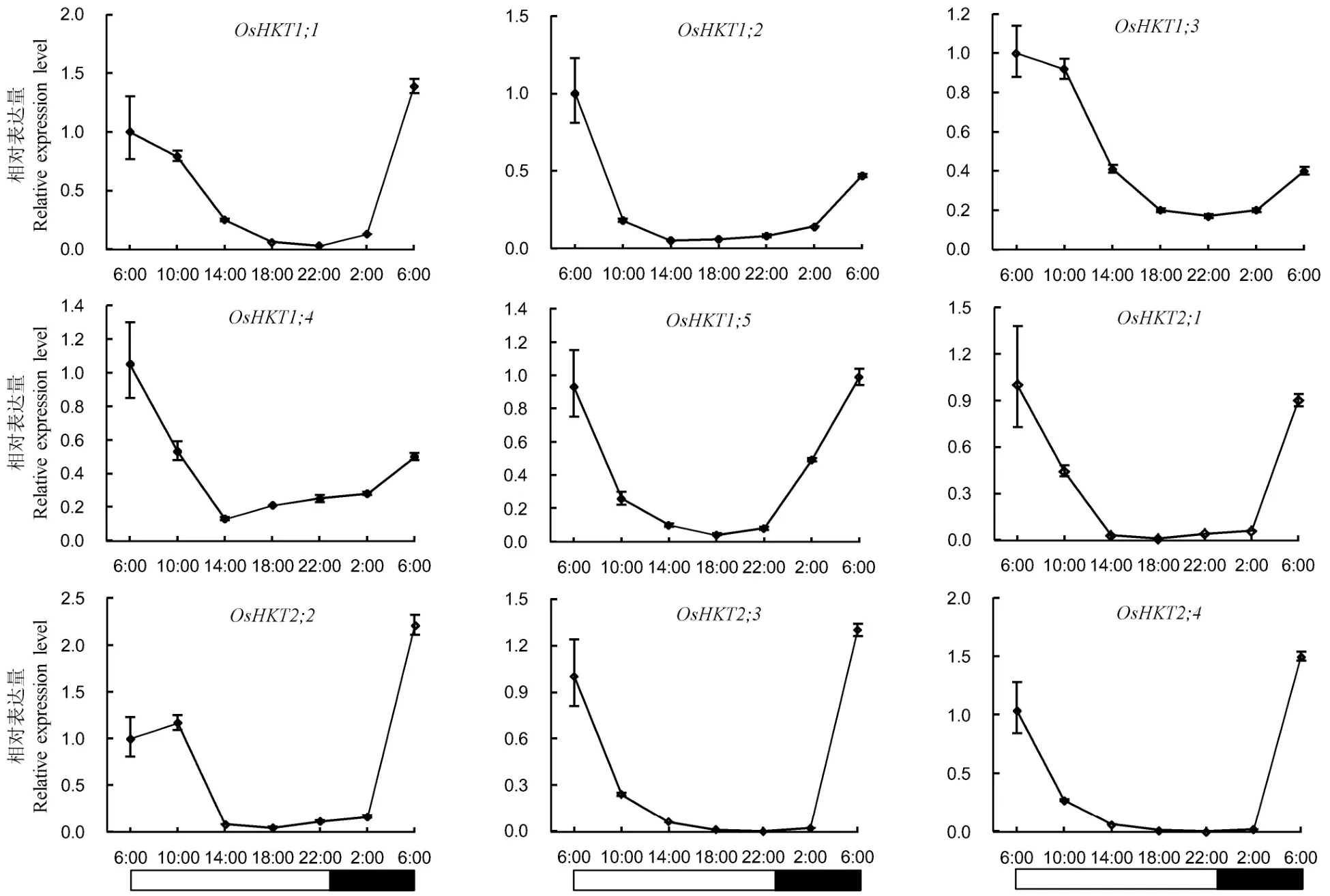
图4 长日照条件下OsHKT家族基因昼夜节律性表达模式Fig. 4. Diurnal rhythm expression patterns of OsHKT family genes under long-day conditions.
3 讨论
研究表明OsHKT1;1基因主要在地上部表达[18],而OsHKT1;5主要在根中表达[19],这与我们的结果一致(图1)。已有研究表明,OsHKT2;1主要在根的皮层和内皮层表达,在叶片的维管束区域表达[14],与此一致的是,我们的研究结果也表明 OsHKT2;1在根和叶中均有表达(图1);OsHKT2;3和OsHKT2;4基因主要在叶中表达(图 1),且在根中的表达量受ABA诱导(图3-A),这与胡一兵等[21]的报道一致。
拟南芥中的研究表明,盐胁迫反应可以分为脱落酸(ABA)依赖性的和 ABA 非依赖性的[22]。我们比较了ABA处理和盐胁迫下的OsHKT家族中的不同基因在根和叶中表达模式的差异,发现根中OsHKT1;1、OsHKT2;1和OsHKT2;2基因在盐胁迫下与ABA处理下的表达模式是一致的(图2-A和图3-A),推测这些基因可能参与 ABA依赖性的盐胁迫反应。而 OsHKT家族中的其他基因在盐胁迫下和 ABA处理下的表达模式均不一致,推测这些基因参与的盐胁迫反应可能与ABA途径无关。
有研究表明,OsHKT1;5可以将过多的 Na+从木质部中卸载到周围薄壁细胞中,降低木质部汁液中Na+含量,防止向地上部转运,间接使得K+向地上部运输,从而提高地上部的K+/Na+比,在Na+长距离运输过程中发挥重要作用[23-24]。OsHKT1;5的功能与其组织特异性相关,主要在根中表达。而我们的研究结果表明OsHKT1;5在日本晴水稻根和叶中的表达均受盐胁迫抑制(图2),这可能与日本晴是盐敏感品种相关。研究表明OsHKT2;1和OsHKT2;2在盐胁迫下发挥着重要功能,在盐胁迫下,OsHKT2;1在耐盐型水稻品种中表达量下调;OsHKT2;2在盐敏感型和耐盐型水稻品种中表达量均下调[25]。即盐胁迫下,OsHKT2;1和 OsHKT2;2的下调表达有利于植物生长。我们的结果表明OsHKT2;1和OsHKT2;2在盐处理24 h表达受到抑制,但在盐处理1 h表达上调(图2)。
植物中许多与盐胁迫相关基因的表达均受光调控,如小麦中的 TaGBF1基因[26]、拟南芥中的AtSTO基因[27]以及水稻中的OsDABH1基因[28]。有报道表明冰叶日中花的叶片中V-H+-ATPase的A、B、C亚基特别是C亚基编码基因的mRNA呈昼夜节律性变化,且在盐胁迫下变化幅度增大[29]。本研究结果显示了OsHKT家族成员基因的转录水平均具有昼夜节律性,据此推测水稻在光期和暗期对Na+和K+的转运能力不同,这可能与水稻光期和暗期的不同生理状态相适应。
OsHKT家族不同基因表达具有不同组织特异性,受盐胁迫处理后表达模式也不同。推测这些基因协同作用维持着体内离子平衡,特别是 Na+/K+平衡。但是除了OsHKT1;1和OsHKT1;5外,其余OsHKT家族基因的作用机制仍不清楚。本研究为深入探讨OsHKT家族基因的功能奠定了基础。
[1] Garciadeblas B,Senn M E,Banuelos M A,Rodríguez Navarro A. Sodium transport and HKT transporters: The rice model. Plant J,2003,34(6): 788-801.
[2] Horie T,Yoshida K,Nakayama H,Yamada K,Oiki S,Shinmyo A. Two types of HKT transporters with different properties of Na+and K+transport in Oryza sativa. Plant J,2001,27(2): 129-138.
[3] Maser P,Hosoo Y,Goshima S,Horie T,Eckelman B,Yamada K,Yoshida K,Bakker EP,Shinmyo A,Oiki S,Schroeder J I,Uozumi N. Glycine residues in potassium channel-like selectivity filters determine potassium selectivity in four-loop-per-subunit HKT transporters from plants. Proc Natl Acad Sci USA,2002,99(9):6428-6433.
[4] Garciadeblas B,Senn M E,Banuelos M A,Rodríguez N A. Sodium transport and HKT transporters: The rice model. Plant J,2003,34(6): 788-801.
[5] Uozumi N,Kim E J,Rubio F,Yamaguchi T,Muto S,Tsuboi A,Bakker E P,Nakamura T,Schroeder J I. The Arabidopsis HKT1 gene homolog mediates inward Na+currents in xenopuslaevis oocytes and Na+uptake in Saccharomyces cerevisiae. Plant Physiol,2000,122(4):1249-1259.
[6] Wang Q,Guan C,Wang P,Lü M L,Ma Q,Wu G Q,Bao A K,Zhang J L,Wang S M. AtHKT1;1 and AtHAK5 mediate low-affinity Na+,uptake in Arabidopsis thaliana,under mild salt stress. Plant Growth Regul,2015,75(3):615-623.
[7] Wang Q,Guan C,Wang S M. Coordination of AtHKT1;1 and AtSOS1 facilitates Na+,and K+,homeostasis in Arabidopsis thaliana,under salt stress. J Plant Biol,2014,57(5): 282-290.
[8] Sunarpi,Horie T,Motoda J,Kubo M,Yang H,Yoda K,Horie R,Chan W Y,Leung H Y,Hattori K,Konomi M,Osumi M,Yamagami M,Schroeder J I,Uozumi N.Enhanced salt tolerance mediated by AtHKT1 transporter-induced Na unloading from xylem vessels to xylem parenchyma cells. Plant J,2006,44(6): 928-938.
[9] Uozumi N,Kim E J,Rubio F,Yamaguchi T,Muto S,Tsuboi A,Bakker E P,Nakamura T,Schroeder J I. The Arabidopsis HKT1 gene homolog mediates inward Na+currents inxenopuslaevis oocytes and Na+uptake in Saccharomyces cerevisiae. Plant Physiol,2000,122(4):1249-1259.
[10] Platten J D,Cotsaftis O,Berthomieu P,Bohnert H,Davenport R J,Fairbairn D J,Horie T,Leigh R A,Lin H X,Luan S,Mäser P,Pantoja O,Rodríguez-Navarro A,Schachtman D P,Schroeder J I,Sentenac H,Uozumi N,Véry A A,Zhu J K,Dennis E S,Tester M. Nomenclature for HKT transporters,key determinants of plant salinity tolerance. Trends Plant Sci,2006,11(8): 372-374.
[11] Fairbairn D J,Liu W,Schachtman D P,Gomez-Gallego S,Day S R,Teasdale R D. Characterisation of two distinct HKT1-like potassium transporters from Eucalyptus camaldulensis. Plant Mol Biol,2000,43(4):515-525.
[12] Su H,Balderas E,Veraestrella R,Golldack D,Golldack D,Quigley F,Zhao C,Pantoja O,Bohnert H J.Expression of the cation transporter McHKT1 in a halophyte. Plant Mol Biol,2003,52(5): 967-980.
[13] Wang R,Jing W,Xiao L,Jin Y,Shen L,Zhang W. The OsHKT1;1 transporter is involved in salt tolerance and regulated by an MYB-Type transcription factor. Plant Physiol,2015,168(3): 1076-1090.
[14] Horie T,Yoshida K,Nakayama H,Yamada K,Oiki S,Shinmyo A. Two types of HKT transporters with different properties of Na+and K+transport in Oryza sativa. Plant J,2001,27(2): 129-138.
[15] Horie T,Costa A,Kim T H,Han M J,Horie R,Leung H Y,Miyao A,Hirochika H,An G,Schroeder J I. Rice OsHKT2;1 transporter mediates large Na+influx component into K+-starved roots for growth. EMBO J,2007,26(12): 3003-3014.
[16] Platten J D,Cotsaftis O,Berthomieu P,Bohnert H,Davenport R J,Fairbairn D J,Horie T,Leigh R A,Lin H X,Luan S,Mäser P,Pantoja O,Rodríguez-Navarro A,Schachtman D P,Schroeder J I,Sentenac H,Uozumi N,Véry A A,Zhu J K,Dennis E S,Tester M. Nomenclature for HKT transporters,key determinants of plant salinity tolerance. Trends Plant Sci,2006,11(8): 372-374.
[17] Suzuki K,Yamaji N,Costa A,Okuma E,Kobayashi N I,Kashiwagi T,Katsuhara M,Wang C,Tanoi K,Murata Y,Schroeder J I,Ma J F,Horie T. OsHKT1;4-mediated Na+,transport in stems contributes to Na+,exclusion from leaf blades of rice at the reproductive growth stage upon salt stress. BMC Plant Biol,2016,16(1): 22.
[18] Suzuki K,Costa A,Nakayama H,Katsuhara M,Shinmyo A,Horie T. OsHKT2;2/1-mediated Na+influx over K+uptake in roots potentially increases toxic Na+accumulation in a salt-tolerant landrace of rice Nona Bokra upon salinity stress. J Plant Res,2016,129(1):67-77.
[19] Ren Z H,Gao J P,Li L G,Cai X L,Huang W,Chao D Y,Zhu M Z,Wang Z Y,Luan S,Lin H X. A rice quantitative trait locus for salt tolerance encodes a sodium transporter. Nat Genet,2005,37(10): 1141.
[20] Sassi A,Mieulet D,Khan I,Moreau B,Gaillard I,Sentenac H,Véry A A. The rice monovalent cation transporter OsHKT2;4: Revisited ionic selectivity. Plant Physiol,2012,160(1): 498-510.
[21] 胡一兵,罗伟,吴延寿,徐国华. 水稻钠钾离子转运蛋白基因OsHKT2;3和OsHKT2;4的功能研究//2011年中国遗传学会大会论文摘要汇编. 乌鲁木齐: 中国遗传学会,2011: 46.Hu Y B,Luo W,Wu Y S,Xu G H. The functional study of rice Na+/K+transporter OsHKT2;3 and OsHKT2;4 genes//Proceedings of China Genetics Conference in 2011. Urumqi,China: Chinese Genetics Society,2011:46. (in Chinese)
[22] 顾建伟. 光敏色素B介导的光信号调控水稻ABA反应的研究. 郑州: 郑州大学,2012.Gu J W. Study on the regulation of ABA reaction of rice by photosensitive B mediated optical signal.Zhengzhou: Zhengzhou University,2012. (in Chinese with English abstract)
[23] Lin H X,Zhu M Z,Yano M,Gao J P,Liang Z W,Su W A,Hu X H,Ren Z H,Chao D Y. QTLs for Na+and K+uptake of the shoots and roots controlling rice salt tolerance. Theor Appl Genet,2004,108(2): 253-260.
[24] Cotsaftis O,Plett D,Johnson A A,Walia H,Wilson C,Ismail A M,Close T J,Tester M,Baumann U.Root-specific transcript profiling of contrasting rice genotypes in response to salinity stress.. Mol Plant,2011,4(1): 25-41.
[25] Kader M A,Seidel T,Golldack D,Lindberg S.Expressions of OsHKT1,OsHKT2,and OsVHA are differentially regulated under NaCl stress in salt-sensitive and salt-tolerant rice (Oryza sativa L.) cultivars. J Exp Bot,2006,57(15): 4257.
[26] Sun Y,Xu W,Jia Y,Wang M,Xia G. The wheat TaGBF1 gene is involved in the blue-light response and salt tolerance. Plant J Cell amp; Mol Biol,2016,84(6):1219-1230.
[27] Indorf M,Cordero J,Neuhaus G,Rodríguez-Franco M.Salt tolerance (STO),a stress-related protein,has a major role in light signaling. Plant J,2007,51(4): 563-574.
[28] Hasthanasombut S,Paisarnwipatpong N,Triwitayakorn K,Kirdmanee C,Supaibulwatana K. Expression of OsBADH1 gene in indica rice (Oryza sativa L.) in correlation with salt,plasmolysis,temperature and light stresses. Plant Omics,2011,4(7): 400-407.
[29] Luttge U,Ratajczak R. The physiology,biochemistry and molecular biology of the plant vacuolar ATPase. Adv Bot Res,1997,25(8): 253-296.
Expression Patterns of OsHKT Genes in Rice
CUI Lixin1,2,HE Yanan2,LI Yaping1,2,XIE Xianzhi1,2,*
(1College of Life Sciences,Shandong Normal University,Jinan 250014,China; 2Shandong Rice Research Institute,Jinan 250100,China; *Corresponding author,E-mail: xzhxie2010@163.com)
【Objective】OsHKTs (High Affinity K+Transporter) encode Na+or K+transporter or Na+-K+co-transporters that play an important role in Na+recirculation and maintaining the low concentration of Na+and low Na+/ K+ratio in above-ground tissues,especially leaves. Therefore,OsHKT family is related to salt stress tolerance in rice. In this study,we analyzed the tissue specificity and diurnal rhythm of OsHKTs gene expression and expression patterns in rice seedlings treated with either salt or ABA. 【Method】qRT-PCR technique was used to analyze OsHKTs expression patterns. 【Result】Individual member of OsHKT family genes had different expression levels in root and leaf. OsHKT1;1,OsHKT1;3,OsHKT2;3,and OsHKT2;4 mainly expressed in leaves,whereas other members had higher transcript levels in roots relative to those in leaves. Salt treatment affected transcript levels of OsHKT family genes both in leaves and roots. OsHKT1;5,OsHKT2;1,and OsHKT2;2 had similar expression patterns in both leaves and roots of seedlings treated with NaCl,whereas other members did not. Transcript levels of OsHKT family genes were regulated by ABA treatment either in leaves or in roots. OsHKT1;3 and OsHKT1;5 had similar expression patterns in both leaves and roots. In addition,expression patterns of OsHKT genes exhibited similar diurnal rhythm. 【Conclusion】Our results suggest that expression of OsHKT family genes has tissue-specificity and similar diurnal rhythm. Exogenous NaCl and ABA treatment have different influences on OsHKT transcripts,depending on individual member of the family. Our findings lay a foundation for further revealing the functional mechanism of OsHKT genes in rice.
OsHKT; expression patterns; salt stress; ABA; tissue specificity; photoperiod
Q945.78; S511.03
A
1001-7216(2017)06-0559-09
10.16819/j.1001-7216.2017.7070
2017-06-08; 修改稿收到日期:2017-07-07。
山东省科技重大专项(2015ZDJS03001-4); 山东省中青年科学家科研奖励基金资助项目(ZR2016CB17); 山东省农业重大应用技术创新项目[黄河三角洲中高度盐碱地水稻品种(系)筛选与淡水控量增效技术研究与示范]。

