橡胶树PEPC基因的克隆、生物信息学和表达分析
王岳坤,阳江华,丁 丽,黄红海,桂红星
(中国热带农业科学院橡胶研究所∕农业部橡胶树生物学与遗传资源利用重点实验室,海南 儋州571737)
橡胶树PEPC基因的克隆、生物信息学和表达分析
王岳坤,阳江华,丁 丽,黄红海,桂红星*
(中国热带农业科学院橡胶研究所∕农业部橡胶树生物学与遗传资源利用重点实验室,海南 儋州571737)
【目的】克隆橡胶树磷酸烯醇式丙酮酸羧化酶(PEPC)基因的全长cDNA,通过生物信息学和基因表达分析,为基因功能研究奠定基础。【方法】根据橡胶树PEPC基因片段序列设计引物,通过RACE获得cDNA全长,用DNAMAN软件预测编码多肽序列,用ProtParam工具分析多肽基本理化性质,用NCBI-Blast工具搜索同源的核酸及多肽序列,用ClustalX2.0软件比对不同植物来源的多肽序列,并用MEGA 6.06软件构建系统进化树,用Real-time RT-PCR分析基因表达模式。【结果】从橡胶树胶乳中克隆到一个新的PEPC基因的全长cDNA,其长度为3206 bp,包含一个2898 bp的开放读码框(ORF),112 bp的 5’UTR和196 bp 的3’UTR。预测该基因编码965个氨基酸的多肽(HbPPC2),其分子量为110.35 kDa,等电点为5.76;该基因的编码区核苷酸序列和其编码多肽氨基酸序列分别与早期克隆的橡胶树HbPPC1基因(KJ721228)的编码区和编码多肽序列有81.3 %和92.1 %的同源性。预测HbPPC2定位于细胞质,为不稳定蛋白,其活性受多种因素调控。预测HbPPC2酶活性中心是其第172位残基His,磷酸化位点是其第11位残基Ser,单泛素化位点是其第628位残基Lys。其第178、179、226和366位残基Arg在三维结构中靠近,构成葡萄糖-6-磷酸(G6P)的结合位点;其第450、641、753、767位残基Arg在三维结构中靠近,构成PEP的结合位点。HbPPC2基因在叶片、树皮、胶乳、雄花、雌花均有表达,在胶乳表达量最高;乙烯利处理显著下调该基因在胶乳的表达。【结论】本研究可为橡胶树PEPC功能研究提供理论基础。
橡胶树;磷酸烯醇式丙酮酸羧化酶基因;克隆;生物信息学;基因表达

1 材料与方法
1.1 供试材料
1.1.1 植物材料 本研究所用的植物材料为橡胶树热研7-33-97,种植在中国热带农业科学院试验农场。不同组织样品采自1997年定植的橡胶树,采样年份橡胶树采用“S/2 d/3 ET 1.5 % Ga2(1) 16/y(2w)”割制(半螺旋割线,3日割,施1.5 %乙烯利,拔胶线施药,每次2 g,带宽1 cm,每年施 16 次,每2周施1次),采样前2 w不涂乙烯利;乙烯利处理的胶乳样品采自2007年定植的未开割树,采样时橡胶树达开割标准。
1.1.2 质粒、菌株、酶和试剂盒 质粒pMD18-T、大肠杆菌JM109、逆转录酶、Taq酶、DNase I、RNase抑制剂、SYBR Green qRT-PCR试剂盒购自Takara公司,SMARTTMRACE 试剂盒购自Clontech公司,凝胶回收试剂盒购自Axygen公司,引物合成和核酸测序委托上海生工公司完成。
1.2 试验方法
1.2.1 材料处理 不同组织样品采自9株橡胶树,每样品采自3株树,3个重复,同一天采样并冻存。乙烯利处理的胶乳样品采自54株橡胶树,设6种处理,每处理3株树,3个重复。6种处理分别在采样前0、4、8、24、48、72 h涂1.5 %乙烯利(每株树 2 mL),同一时间采样,每株采集2 mL胶乳,同处理3株树胶乳混合后置于冰上备用。
1.2.2 总RNA提取及cDNA合成 参照唐朝荣等[11]的方法提取胶乳和其它组织的总RNA,取2 μl 胶乳总RNA根据逆转录试剂盒说明书合成cDNA。取2 μl 胶乳总RNA根据SMARTTMRACE 试剂盒说明书合成5′-和3′-RACE-Ready cDNA。
1.2.3 基因克隆 用蓖麻PEPC的cDNA序列(EF634317.1)搜索橡胶树胶乳EST数据库(本实验室建立),发现contig06486序列1~753区段与蓖麻PEPC mRNA序列的2251~3003区段有91.1 %的同源性。根据contig06486序列设计引物86-322A(5′-CCAAATCAAT GGTGACCCTA AAGAAAGG-3′)和86-241A(5′-TGACCTGTTT AAATGCTGCT CCAAAGC-3′),按照SMARTTMRACE试剂盒说明书进行5′-RACE。第1轮PCR以5′-RACE-Ready-cDNA为模板,以86-322A和通用引物UPM(试剂盒提供)组合扩增。第2 轮PCR以第1 轮PCR产物的50倍稀释液为模板,以86-241A和通用引物NUP(试剂盒提供)组合扩增。回收第2轮PCR产物后连接pMD18-T载体,转化大肠杆菌,挑取阳性克隆测序,获得5′-RACE片段A。
根据5′-RACE片段A设计3′-RACE引物86-2242F(5′-GCAATCATTT GGAGAGGAGC ACTTGTG-3′)和86-2353F(5′-GCTGATGGAT GAAATGGCTG TTGTTGC-3′),按照SMART? RACE试剂盒说明书进行3′-RACE。第1轮PCR以3′-RACE-Ready-cDNA为模板,以86-2242F和通用引物UPM组合扩增,第2轮PCR以第1轮PCR产物的50倍稀释液为模板,以86-2353F和通用引物NUP组合扩增。回收第二轮扩增产物后克隆、测序,获得3′-RACE片段B。拼接5′-RACE片段A和3′-RACE片段B得到拼接序列C。根据拼接序列C设计引物86-nF1-115(5′-TCCTCTTTCC CATCCCAACC-3′)和86-nR1-3325(5′-CAGCAAACAC TGGCAGCCCT AAG-3′),以胶乳cDNA为模板进行PCR扩增,回收PCR产物后克隆、测序,获得验证序列D。
1.2.4 生物信息学分析 用Primer Premier 5软件进行引物设计,用DNAman 5.22 软件进行序列拼接和序列分析。用BLAST在线工具(http://www.ncbi.nlm.nih.gov/Blast.cgi/)搜索GenBank的同源序列,用ClustalX 2.0进行蛋白序列比对,用MEGA 6.06软件构建蛋白序列进化树(用Clustalw进行序列比对,Neighbor-Jioning算法构建进化树,用自展值1000检验各分支的置信度)。用ProtParam在线工具(http://www.expasy.org/tools/ protparam.html)预测蛋白的基本物理化学性质,用PredictProtein在线工具(http://www.predictprotein.org/)预测蛋白是否存在信号肽和亚细胞定位。
1.2.5 表达分析 根据测序结果,设计引物对HbPPC2-qF1427(5′-CTGAAGAACG CCGACAAGAG T-3′)和HbPPC2-qR1602 (5′-TGGTGCAGTT GCCATAGAAA T-3′),以18S rRNA为内参基因(引物对5′-GCTCGAAGAC GATCAGATAC C-3′和5′-TTCAGCCTTG CGACCATAC-3′),用Real-time PCR分析HbPPC2的表达模式。为比较HbPPC2与HbPPC1的表达差异,两个基因同时进行表达分析。HbPPC1的引物为HbPPC1-qF101 (5′-ATGCTCTGTT ATTGGACCGA TTT-3′)和HbPPC1-qR218 (5′-GGGTCACGCT TCCCTTCATA C-3′)。扩增程序为94 ℃预变性30 s;94 ℃变性5 s,60 ℃退火20 s,72 ℃延伸20 s,扩增40个循环。反应结束后,根据Ct值,用Pfaffl法[12]计算相对表达量。
2 结果与分析
2.1 基因克隆
根据蓖麻PEPC基因的核苷酸序列,在橡胶树胶乳EST数据库中搜索,获得一段753 bp的同源序列。根据该序列设计引物,以胶乳RNA制备的5′-RACE-Ready cDNA为模板进行5′-RACE,获得2612 bp的5′-RACE片段A。根据片段A序列设计引物,以胶乳RNA制备的3′-RACE-Ready cDNA为模板进行3′-RACE,获得1023 bp的3′-RACE片段B。拼接片段A和片段B,获得3375 bp的拼接序列C。根据拼接序列C设计引物,以胶乳cDNA为模板进行PCR,获得3206 bp的序列D。序列D含有2898 bp的阅读框,编码965个氨基酸,5′UTR为112 bp,3′UTR为196 bp (图1)。将序列D命名为HbPPC2,提交GenBank,登录号为KP742954。
2.2 HbPPC2基因生物信息学分析
同源性分析显示,HbPPC2的编码区和多肽序列分别与橡胶树HbPPC1 (KJ721228)的编码区和多肽序列有81.3 %和92.1 %的同源性;分别与蓖麻RcPPC3(EF634317.1) 的编码区和多肽序列有90.6 %和95.3 %的同源性;分别与胡杨PePPC(XM011031267.1)的编码区和多肽序列有89.0 %和94.6 %的同源性。
NCBI-CDD搜索显示,HbPPC2编码多肽的第162~965个残基是PEPC超家族pfam00311的保守域,多肽的N端第9~17位残基是植物型PEPC可逆磷酸化的特征序列XXSIDAQLR,C端是保守四肽序列QNTG,第774位残基为C3-PEPC的特有残基Ala,表明HbPPC2为C3-PEPC基因。

图1 HbPPC2的核苷酸及编码的多肽序列Fig.1 Nucleotide and deduced polypeptide sequences of HbPPC2
ProtParam工具分析表明,HbPPC2的编码多肽相对分子量为110.35 kDa,等电点为5.76,吸光度为1.083,不稳定系数为45.57,脂肪系数为90.56,总平均亲水性为-0.396。Prosite和PredictProtein分析表明,编码多肽没有二硫键结构,不含信号肽,定位在细胞质。
2.3 氨基酸序列比对和功能位点预测
鉴于蓖麻[6-7]、玉米[4-5]和拟南芥[13]PEPC功能的深入研究,本研究将HbPPC2的氨基酸序列与蓖麻RcPPC3 (ABR29876.1, C3)、RcPPC4 (ABR29877.1, BTPC)、玉米ZmPPC1 (CAA43709.1, C3)、ZmPPC2 (CAA33316.1, C4)、拟南芥AtPPC1 (CAD58725, C3)和橡胶树HbPPC1 (KJ721228, C3)进行多序列比对,预测HbPPC2的功能位点。图2显示,蓖麻RcPPC4的氨基酸序列与其他6条序列的差别最大,插入和缺失片段较长较多,原因是蓖麻RcPPC4属于BTPC,其他蛋白属于PTPC;玉米ZmPPC2序列与5个C3-PEPC很相似,原因是C4-和C3-PEPC同属于PTPC,它们由共同的祖先基因进化而来,氨基酸序列差别较小。
根据PTPC序列特征、蓖麻RcPPC3功能位点和玉米ZmPPC2晶体结构,预测HbPPC2的第172位残基His是其酶活性中心;第11位残基Ser是磷酸化修饰位点;第628位残基Lys是单泛素化位点;第178、179、226和366位残基Arg在三维结构中靠近,构成1个G6P结合位点;第450、641、753、767位残基Arg在三维结构中靠近,构成PEP结合位点;第641、888位残基Arg和第963位残基Asn构成Asp结合位点;第560位残基Glu和第597位残基Asp构成Mg2+结合位点。这些位点的残基替代或三维结构变化均可引起酶活性明显变化。
2.4 分子进化分析
根据HbPPC2氨基酸序列,搜索NCBI数据库中同源序列,选取基因功能比较明确的15条序列(兼顾PEPC分型)和橡胶树HbPPC1序列,用ClustalX 2.0对蛋白序列进行比对,然后用MEGA 6.06软件中邻接法构建进化树(图3)。结果显示,17条序列的进化树呈明显的阶梯形排列,最基部是大肠杆菌的细菌型PEPC,其次是蓖麻、拟南芥、水稻的细菌型PEPC,往上是玉米、高粱的C4-PEPC,高粱、水稻的C3-PEPC,拟南芥、蓖麻、橡胶树、玉米的C3-PEPC和冰叶日中花的CAM-PEPC聚在一起,位于阶梯形进化树的最上部,橡胶树和蓖麻的4条C3-PEPC序列进化距离最近。

图2 不同植物PEPC序列比对Fig.2 Sequence alignment of PEPCs from different plants

图3 HbPPC2的进化树分析Fig.3 Phylogenetic analysis of HbPPC2
2.5 表达分析
表达分析表明,2个基因在胶乳的表达量均明显高于其它组织;HbPPC2在树皮、雄花、雌花也有较高的表达量,在叶片表达量最低;HbPPC1在叶片具有较高表达量,在树皮几乎不表达,与HbPPC2表达模式存在差异(图4)。乙烯利处理明显下调胶乳HbPPC2的表达,在处理后4 h,表达量降至处理前的5 %~10 %,此后一直维持这种低水平的表达量至乙烯利处理后72 h,HbPPC1基因也呈这种应答模式(图5)。
3 讨 论

图4 HbPPC基因在橡胶树不同组织的相对表达量Fig.4 Expression profiles of HbPPC in different tissues of Hevea
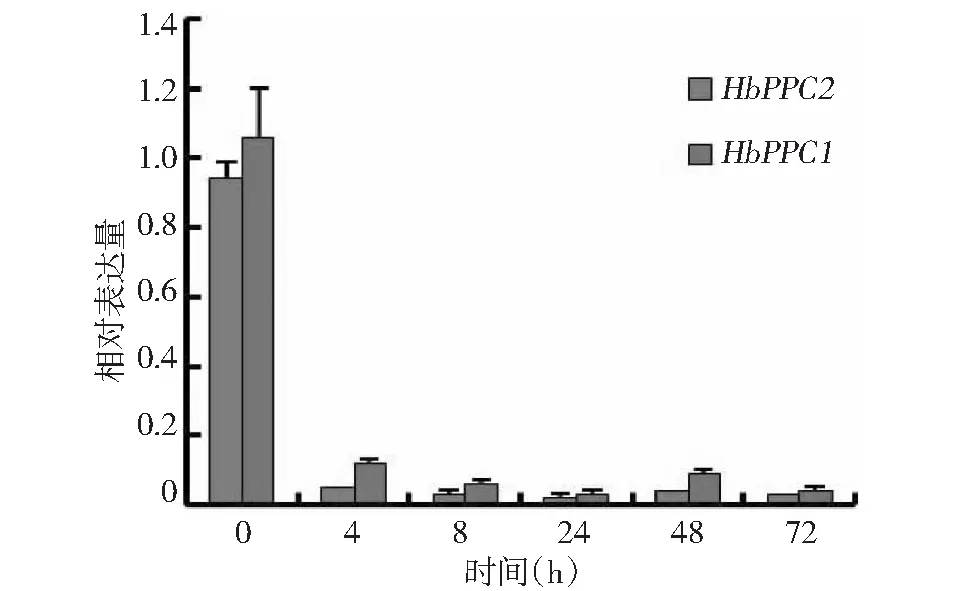
图5 胶乳HbPPC基因对乙烯利处理的表达应答Fig.5 Expression responds of latex HbPPC to ethephon treatment
PEPC具有复杂的转录后活性调控途径,能够敏感地根据环境因素的变化调节活性。现已发现,PEPC至少有以下5种转录后活性调控途径:①异构调节。双子叶植物PEPC被葡萄糖-6-磷酸(G6P)激活,被苹果酸、Asp抑制;单子叶植物PEPC还能被Gly和Ala激活。②磷酸化调节。在PTPC多肽N端的Ser残基磷酸化上调酶活性,同时降低苹果酸和L-Asp的抑制效果,提高G6P的激活效果。③单泛素化调节。正在萌发的蓖麻[16]、高梁[17]种子PTPC多肽分别在Lys-628、Lys-624残基单泛素化后活性下降,该位点单泛素化和其N端Ser残基的磷酸化是相互排斥的反应。④PTPC和BTPC亚基的互作调节。C3-PEPC呈活性状态时一般由完全相同亚基组成四聚体(Class-1 PEPC),但当PTPC与BTPC亚基结合形成异源八聚体(Class-2 PEPC)之后,酶的异构调节不敏感[7,18]。⑤细胞溶质pH值的调节。细胞溶质pH值越偏碱性,PEPC活性越高[1-3]。
橡胶树茎干树皮的乳管组织(乳管系统)糖代谢很旺盛,割胶后排出的胶乳,不仅包含20 %~50 %的橡胶烃,还包含橡胶烃生物合成所需的糖、酶类物质和细胞器(橡胶粒子和黄色体)。因此,乳管的胶乳再生能力是维系橡胶树生长发育和胶乳产量的一个重要构成因素。糖酵解是胶乳再生原料(蔗糖)降解为异戊二烯前体物(乙酰-CoA)的必经代谢途径,PEP是糖酵解的重要中间产物,其代谢方向及强度极大影响糖酵解效率和胶乳再生能力。在健康的常规割胶树茎干乳管系统中,PEP旁路代谢流和主路代谢流的比率及其变化幅度是多少,这是一个有待深入研究的科学问题。上世纪法国学者Jacob等认为,在一般生理条件下,PEP旁路代谢流不超过主路代谢流的10 %,因为强劲的PEP旁路代谢流不利于橡胶烃的生物合成[19]。乙烯利(ET)处理可有效地提高胶乳产量,其原因主要是:⑴延长割胶后的排胶时间,使胶树排出更多胶乳;⑵促进乳管的糖代谢,使乳管胶乳再生能力增强。胶乳pH值一般保持在6.6~6.9范围,在ET处理后的1~3 d,pH值可提升0.3~0.4个单位。由于催化蔗糖转化为葡萄糖和果糖的转化酶活性对pH值很敏感,pH值稍微提升便导致转化酶活性显著上升,导致乳管糖代谢和胶乳再生能力显著增强[19]。值得注意的是,胶乳pH值提高,也显著激活PEPC,把较多的糖带入PEP旁路代谢,这在物质和能量方面均不利于橡胶烃的生物合成,况且相对强劲的PEP旁路代谢产生较多的H+,使胶乳pH值较快回复至ET处理前的水平。本研究发现,ET处理显著下调胶乳PEPC基因的表达,使胶乳PEPC数量下降,这可能部分抵消了胶乳pH值上升PEPC活性提高的效果,使胶乳pH值较长时间保持在较高水平,并使乳管糖代谢保持有利于胶乳再生的方向。
4 结 论
本研究从橡胶树胶乳中克隆到1个PEPC基因的全长cDNA,其长度为3206 bp,包含2898 bp的阅读框,编码965个氨基酸。该cDNA的编码多肽的第162~965个残基是PEPC超家族pfam00311的保守域,N端第9~17位残基是植物型PEPC可逆磷酸化的特征序列XXSIDAQLR,C端是保守的四肽序列QNTG,第774位残基为C3-PEPC的特有残基Ala。该cDNA的编码区和编码多肽序列分别与早期克隆的橡胶树HbPPC1 (KJ721228)基因的编码区和编码多肽序列有81.3 %和92.1 %的同源性;分别与蓖麻RcPPC3(EF634317.1)的编码区和编码多肽序列有90.6 %和95.3 %的同源性。因此,确认该cDNA为橡胶树一个新的PEPC基因的全长cDNA,命名为HbPPC2,登录号为KP742954。预测其编码多肽的分子量为110.35 kDa,等电点为5.76,定位于细胞质,为不稳定蛋白,其活性受多种因素调控。预测编码多肽的第172位残基His是酶的活性中心;第11位残基Ser是酶的磷酸化位点;第628位残基Lys是酶的单泛素化位点;第178、179、226、366位残基Arg在三维结构中靠近,构成G6P的结合位点;第450、641、753、767位残基Arg在三维结构中靠近,构成PEP的结合位点。HbPPC2在叶片、树皮、胶乳、雄花、雌花均有表达,在胶乳表达量最高;乙烯利处理显著下调其在胶乳的表达。本研究可为橡胶树PEPC功能研究提供一些理论基础。
[1]Chollet R, Vidal J, O′Leary M H. Phosphoenolpyruvate carboxylase:a ubiquitous, highly regulated enzyme in plants[J].Annu Rev Plant Physiol Plant Mol Biol, 1996, 47: 273-298.
[2]Izui K, Matsumura H, Furumoto T, et al. Phosphoenolpyruvate carboxylase: a new era of structural biology[J]. Annu Rev Plant Biol, 2004, 55: 69-84.
[3]O′Leary B, Park J, Plaxton W C. The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase): recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs[J].Biochemical Journal, 2011, 436(1): 15-34.
[4]Matsumura H, Xie Y, Shirakata S, et al. Crystal structures of C4 form maize and quaternary complex ofE.coliphosphoenolpyruvate carboxylases[J]. Structure, 2002, 10(12): 1721-1730.
[5]Kai Y, Matsumura H, Izui K. Phosphoenolpyruvate carboxylase: three-dimensional structure and molecular mechanisms[J]. Arch Biochem Biophys, 2003, 414(2): 170-179.
[6]O′Leary B, Fedosejevs E T, Hill A T, et al. Tissue-specific expression and post-translational modifications of plant- and bacterial-type phosphoenolpyruvate carboxylase isozymes of the castor oil plant,RicinuscommunisL[J]. J Exp Bot., 2011, 62(15): 5485-5495.
[7]O′Leary B, Rao S K, Plaxton W C. Phosphorylation of bacterial-type phosphoenolpyruvate carboxylase at Ser-425 provides a further tier of enzyme control in developing castor oil seeds[J]. Biochemical Journal, 2011, 433(1): 65-74.
[8]Muramatsu M, Suzuki R, Yamazaki T, et al. Comparison of Plant-Type Phosphoenolpyruvate Carboxylases from Rice: Identification of Two Plant-Specific Regulatory Regions of the Allosteric Enzyme[J]. Plant and Cell Physiology, 2015, 56(3): 468-480.
[9]Masumoto C, Miyazawa S, Ohkawa H, et al. Phosphoenolpyruvate carboxylase intrinsically located in the chloroplast of rice plays a crucial role in ammonium assimilation[J]. Proc Natl Acad Sci U S A, 2010, 107(11): 5226-5231.
[10]王岳坤,阳江华. 橡胶树磷酸烯醇式丙酮酸羧化酶基因HbPPC1的克隆和表达分析[J]. 热带作物学报,2015,36(5):895-900.
[11]Tang C, Huang D, Yang J, et al. The sucrose transporter HbSUT3 plays an active role in sucrose loading to laticifer and rubber productivity in exploited trees ofHeveabrasiliensis(para rubber tree)[J]. Plant Cell Environ, 2010, 33(10): 1708-1720.
[12]Pfaffl M. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research, 2001, 29(9): 2002-2007.
[13]Gregory A L,Hurley B A, Tran H T, et al. In vivo regulatory phosphorylation of the phosphoenolpyruvate carboxylase AtPPC1 in phosphate-starvedArabidopsisthaliana[J]. Biochem J, 2009, 420(1): 57-65.
[14]Plaxton W C. The organization and regulation of plant glycolysis[J]. Annu. Rev. Plant Physiol.Plant Mol.Biol., 1996, 47: 185-214.
[15]Sakano K. Revision of Biochemical pH-Stat-Involvement of Alternative Pathway metabolisms[J]. Plant Cell Physiol, 1998, 39(5): 467-473.
[16]Uhrig R G, O′Leary B, Spang H E, et al. Coimmunopurification of Phosphorylated Bacterial- and Plant-Type Phosphoenolpyruvate Carboxylases with the Plastidial Pyruvate Dehydrogenase Complex from Developing Castor Oil Seeds[J].Plant Physiology, 2008, 146(3): 1346-1357.
[17]Ruiz-Ballesta I, Feria A B, Ni H, et al. In vivo monoubiquitination of anaplerotic phosphoenolpyruvate carboxylase occurs at Lys624 in germinating sorghum seeds[J]. Journal of Experimental Botany, 2014, 65(2): 443-451.
[18]Gennidakis S, Rao S, Greenham K, et al. Bacterial-and plant-type phosphoenolpyruvate carboxylase polypeptides interact in the hetero-oligomeric Class-2 PEPC complex of developing castor oil seeds[J]. The Plant Journal, 2007, 52(5): 839-849.
[19]d′Auzac J, Jacob J, Chrestin H. Physiology of Rubber Tree Latex. The Laticiferous Cell and Latex-A Model of Cytoplasm[M]. Boca Raton, Florida: CRC Press, Inc., 1989, 101-144: 295-320.
(责任编辑 陈 虹)
Cloning,Bio-informaticsandExpressionAnalysisofPEPCGenefromHeveabrasiliensis
WANG Yue-kun, YANG Jiang-hua, DING Li, HUANG Hong-hai, GUI Hong-xing*
(Key Laboratory of Biology and Genetic Resources of Rubber Tree, Ministry of Agriculture / Rubber Research Institute, Chinese Academy of Tropical Agricultural Sciences, Hainan Danzhou 571737, China)
【Objective】This study aimed to clone the full-length cDNAs of phosphoenolpyruvate carboxylase (PEPC) genes ofHeveabrasiliensis, elucidate the sequence characters of encoded proteins and their expression profiles, with a view to lay the foundations of the research on biologic function ofHeveaPEPC genes.【Method】 Primers were designed according to the partial sequences ofHeveaPEPC gene and the full-length cDNA of PEPC gene was cloned fromHevealatex by RACE technology. The amino acid sequence of encoded protein was predicted by DNAMAN software and its basic physical and chemical characteristics were predicted by ProtParam tool. Similar nucleotide and protein sequences were obtained by NCBI-Blast. Protein sequences from different plants were multiple-aligned by ClustalX 2.0 software and the phylogenetic tree was constructed by MEGA 6.06. The expression profiles of the PEPC genes were analyzed by real-time RT-PCR. 【Result】 A full-length cDNA of a new PEPC gene was cloned fromHevealatex which contained a 2898 bp open reading frame (ORF), a 112 bp 5′UTR and a 196 bp 3′UTR. It was predicted to encode a polypeptide of 965 amino acids (HbPPC2) with a theoretical molecular weight of 110.35 kDa and a PI of 5.76. The sequences of its coding region and coded polypeptide shared 81.3 % and 92.1 % homology respectively toHeveaHbPPC1 gene (KJ721228) which had been cloned previously. HbPPC2 was predicted to locate in cytoplasm, belonging to non-stable protein whose enzymatic activity might be regulated by diverse factors. The His172residue of HbPPC2 was predicted to be the enzymactic activity site; the Ser11residue to be the phosphorylation site; the Lys628residue to be the mono-ubiquination site; the group of Arg178, Arg179, Arg226, and Arg336residue-close in three-dimension structure-to form the glucose-6-phosphate (G6P) biding site; and the group of Arg450, Arg641, Arg753, and Arg767residue-close in three-dimension structure-to form the phosphoenolpyruvate (PEP) biding site.HbPPC2 gene was ubiquitously expressed in bark, latex, leaf, male and female flower, with the highest expression in latex, however its expression in latex reduced significantly after ethephon treatment onHeveatrunk.【Conclusion】This result may provide theoretical basis for the research of HbPPC.
Heveabrasiliensis; Phosphoenolpyruvate carboxylase (PEPC) gene; Cloning; Bio-informatics; Gene expression
S794.1
A
1001-4829(2017)11-2436-08
10.16213/j.cnki.scjas.2017.11.008
2017-03-20
中国热带农业科学院基本科研业务费专项资金(163 0022012011,1630022017023)
王岳坤(1969-),男,广东普宁人,副研究员,从事橡胶树胶乳生理和分子生物学研究;*为通讯作者,E-mail:xwangyk@126.com,guihongxing@263.net,电话:0898-23306599。

