基于ITS2序列的博落回属植物鉴定研究△
黄鹏,刘金凤,徐敏,夏丽琼,柳亦松,3,唐其,唐昭山,曾建国*
(1.湖南农业大学 兽用中药资源与中兽药创制国家地方联合工程研究中心,湖南 长沙 410128;2.湖南中医药大学 药学院,湖南 长沙 410208;3.湖南农业大学 动物医学院,湖南 长沙 410128;4.湖南美可达生物资源股份有限公司,湖南 长沙 410005)
·基础研究·
基于ITS2序列的博落回属植物鉴定研究△
黄鹏1,刘金凤1,徐敏1,夏丽琼2,柳亦松1,3,唐其1,唐昭山4,曾建国1*
(1.湖南农业大学 兽用中药资源与中兽药创制国家地方联合工程研究中心,湖南 长沙 410128;2.湖南中医药大学 药学院,湖南 长沙 410208;3.湖南农业大学 动物医学院,湖南 长沙 410128;4.湖南美可达生物资源股份有限公司,湖南 长沙 410005)
目的采用ITS2序列对博落回属药用植物进行鉴定。方法通过对来自湖南、江西、浙江、湖北、河南、陕西、重庆的30个博落回属植物ITS2序列进行扩增和测序,并对其进行扩增效率及测序成功率、碱基构成、种内和种间变异分析及NJ系统进化树分析来评估ITS2序列鉴定效果。结果博落回属药用植物ITS2序列长度在500 bp以内,扩增效率和测序成功率为100%,并获得30条博落回ITS2序列。结论ITS2序列可以有效准确鉴别博落回属植物。
博落回属;ITS2序列;DNA条形码;物种鉴定;遗传分化
博落回Macleayacordata(Willd.)R.Br.与小果博落回M.microcarpa(Maxim.)Fedde.属于罂粟科博落回属植物。现代药理研究表明博落回属植物中的4种主要生物碱:原阿片碱、别隐品碱、白屈菜红碱、血根碱对治疗多种炎症有效,并对畜禽类有较好的肠道菌群调节作用,目前已作为饲用抗菌药的替代品在欧洲等地区销售[1]。博落回与小果博落回主要分布于中国、东亚、北美洲和欧洲,两者的形态区别主要是果荚部分,博落回的果荚呈倒卵形,而小果博落回的果荚呈近圆形。前期研究显示博落回与小果博落回中的4种主要生物碱具有组织分布特异性,其中血根碱和白屈菜红碱主要分布在博落回的果荚和小果博落回的叶片中,而原阿片碱和别隐品碱主要分布在博落回叶片、根和小果博落回果荚中[2]。由于在博落回属植物中一般只有博落回的果荚才是用来提取血根碱的原料,因此开发一种简单、准确的方法区分博落回与小果博落回具有重要的意义。
DNA条形码技术是一种基于DNA分子进化原理,通过一段通用的标准短序列对物种进行鉴定的新生物学技术[3]。近年来,DNA条形码作为物种鉴定的工具已广泛应用于动植物的物种鉴定及种间亲缘关系的分析,在众多的DNA条形码中,ITS2序列具有序列片段较短、进化速率较快等特点,已经成功运用于许多传统中药材的鉴定[4-9]。这些研究均证明ITS2特征序列在中药材的鉴定方面具有广泛的应用价值[10]。虽然应用ITS2条形码特征序列鉴别物种的研究越来越多,但是系统地用于博落回属植物鉴别的报道很少。因此,利用ITS2序列片段建立一种准确鉴定博落回属植物的分子鉴定体系,不仅可以对其进行准确分类,而且对用药安全具有十分重要的意义。本研究对博落回属植物ITS2序列进行比较分析和快速准确鉴定,旨在为博落回属植物分子鉴定提供理论依据。
1 材料
共收集30份博落回属植物根部材料,采自湖南、江西、浙江、湖北、河南、陕西、重庆等地,均用网袋及泥土包裹根部材料运送至湖南省长沙市湖南农业大学国家中药材生产(湖南)技术中心。在种植1年后待长出地上部分再采集叶片进行DNA提取,具体样本信息见表1。
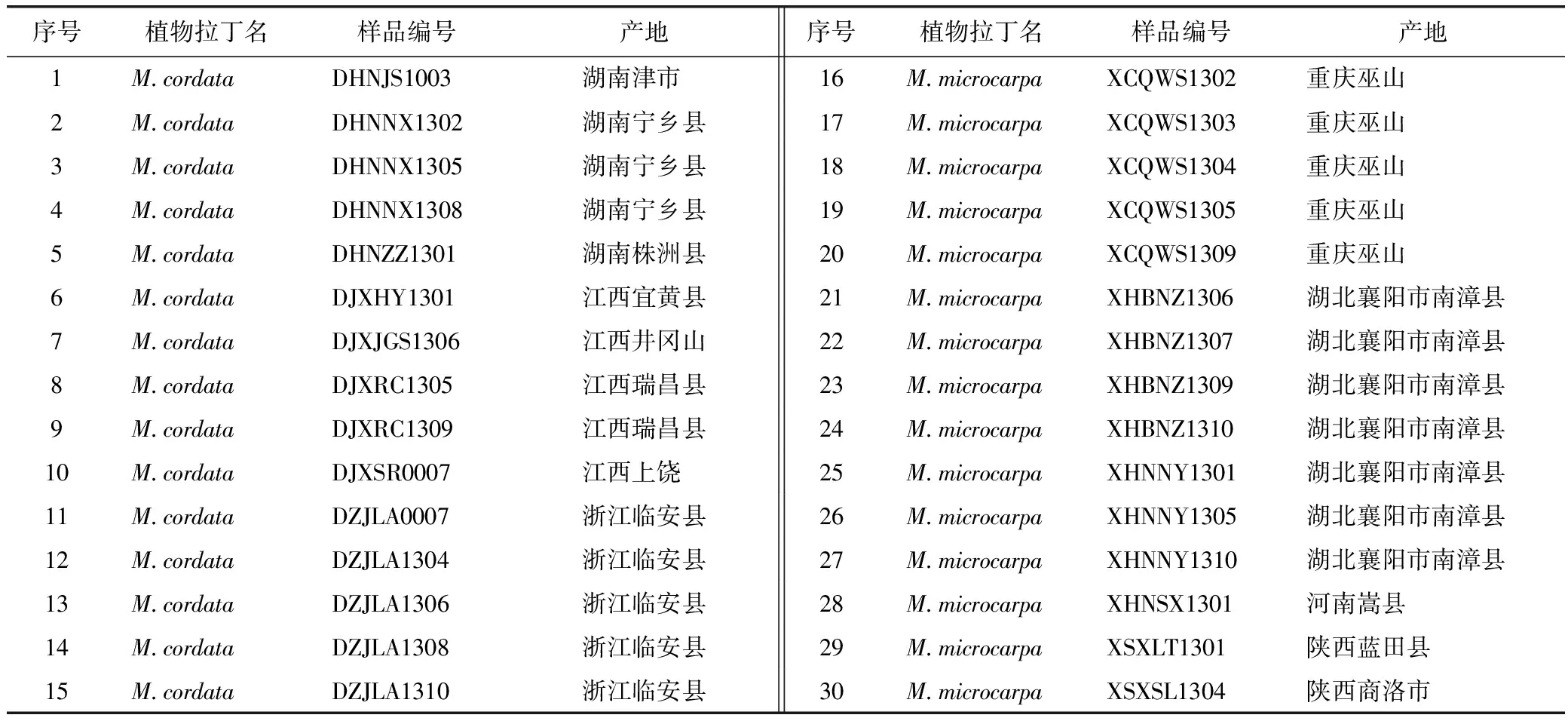
表1 博落回属植物样本信息
2 方法
2.1 DNA提取和序列扩增
博落回与小果博落回新鲜叶片用超纯水洗净之后,用灭菌滤纸吸净表面水分。加入适量液氮充分研磨成粉末;再用取样小勺称取200~300 mg粉末样本置1.5 mL离心管中,使用磁珠法植物基因组DNA提取试剂盒提取总DNA(TIANGEN#DP342-01)。在凝胶成像仪下观察拍照并筛选出提取效果较好的样品用于下一步的PCR扩增。PCR反应体积为25 μL,体系内包含:ddH2O 12 μL、2 Taq mastermix 10 μL、DNA模板1 μL、正向引物1 μL、反向引物1 μL。本实验参照远志属研究结果[11],引物正向为:ATGCGATACTTGGTGTGAAT,反向为:GACGC-TTCTCCAGACTACAAT。扩增程序:94 ℃变性5 min,再进行35个循环(94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸45 s),最后72 ℃延伸7 min。
2.2 电泳检测
称取0.5 g琼脂糖凝胶于锥形瓶中,加入50 mL 50×TAE缓冲液,中高火微波2 min,将锥形瓶置于水中5 min,加入0.5 μL GelRed,混匀后倒于凝胶模具中20 min后使用。取PCR产物点样电泳,100 V,23 min后,在凝胶成像系统中对凝胶拍照,得到结果。
2.3 数据处理
选择PCR扩增成功的DNA样品交由擎科生物有限公司进行测序,将测序结果输入计算机后应用Clustal W软件对所有ITS2序列对位排列,并用Geneious 10.0.2对校对后的序列进行分子进化遗传分析。
3 结果与分析
3.1 DNA提取、PCR扩增
对30份博落回样品的ITS2序列分析发现,所有实验样本的PCR扩增及测序成功率均为100%,序列获得率(有效序列比例)亦为100%,经琼脂糖凝胶电泳得到PCR扩增电泳图(见图1),扩增效果较好,条带较亮,没有拖尾现象,产物大小在500 bp以内。
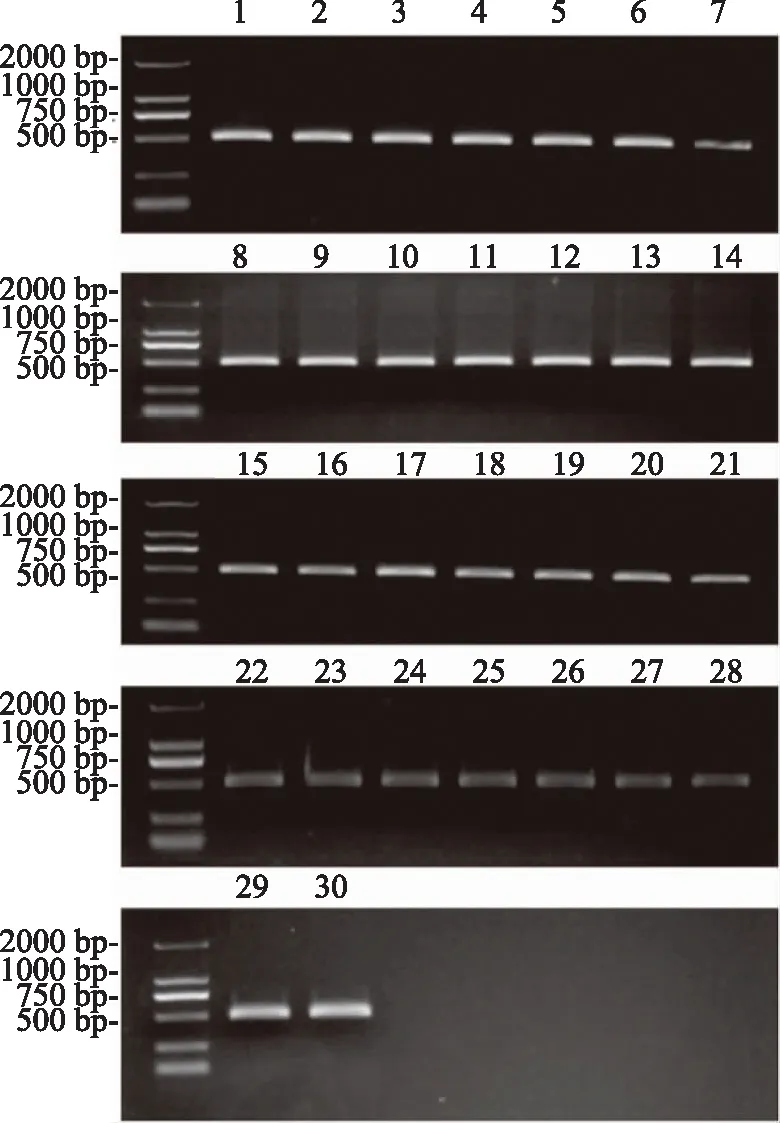
注:泳道上序号对应样本序号。图1 30份博落回样本的ITS2序列凝胶电泳图
3.2 测序及序列分析
博落回属的30份样本ITS2序列的PCR及测序成功率均为100%。ITS2序列博落回种内遗传距离为0.001 4~0.009 4,平均值为0.003 2。种间的遗传距离为0.077 2~0.084 8,平均值为0.079 3。表明其种间遗传距离要明显大于种内遗传距离。
3.3 碱基构成分析
由表2可知,博落回属植物ITS2序列中A、C、G、T碱基的变化范围分别为1.27%、2.17%、0.9%、2.99%。其中,湖南株洲县产地的博落回A碱基含量最高,为21.3%;江西瑞昌县产地的博落回A碱基含量最低,为16.7%。河南嵩县产地的小果博落回C碱基含量最高,为33.9%。浙江临安县产地的博落回C碱基含量最低,为28.9%。浙江临安县产地的博落回以及重庆巫山、湖北襄阳市南漳县、陕西商洛市产地的小果博落回G碱基含量最高,为32.6%;湖南株洲县产地的博落回G碱基含量最低,为29.4%。江西瑞昌县产地的博落回T碱基含量最高,为21.9%。湖北襄阳市南漳县小果博落回T碱基含量最低,为14.7%。
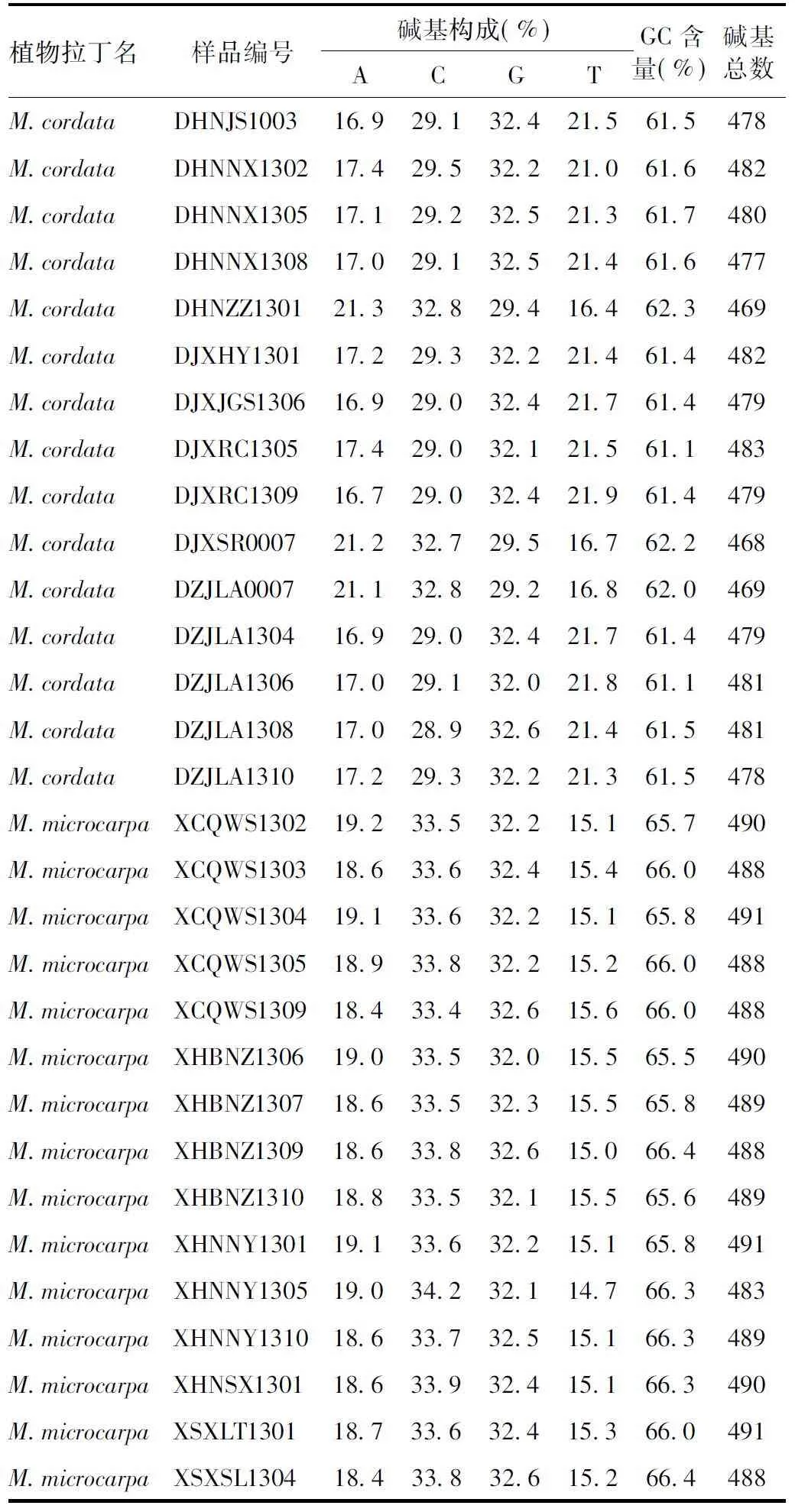
表2 博落回属植物ITS2序列碱基构成
3.4 构建系统进化发育树
利用Geneious10.0.2软件,采用Neighbor-Joining方法构建系统发育树(见图2)。拓扑结构的可靠性用1000次重复的自展检验(Bootstrap analysis)来评估,分支的支持值低于50%的略去。结果显示博落回分为一支,小果博落回被分为另一支。

图2 基于ITS2序列构建的NJ树
4 讨论
近年来,随着DNA条形码技术的不断发展及应用,越来越多的药用植物都使用该技术进行物种鉴定,如野豌豆属[11]、远志属[12]、千斤拔属[13]和黄芩属[14]等大量的药用植物均已成功使用DNA条形码技术进行物种鉴定。陈士林等[10]认为应当建立以ITS2为核心、psbA-trnH为辅的植物类药材DNA条形码鉴定体系和以COI为主、ITS2为辅的动物类药材DNA条形码鉴定体系,这为中药材准确、可靠鉴定及临床用药安全提供新的技术手段。
本课题组已于2012年完成了博落回属植物的转录组高通量测序[1],对不同时间的根叶果材料的转录组以及生物碱生源合成可能的机理进行了探索,之后又对博落回进行了全基因组测序,为下一步的功能基因挖掘和分子育种提供基础数据。由于博落回是目前最主要的血根碱来源植物,在博落回属植物中只有博落回的果荚才是用来提取血根碱的原料。而分辨博落回与小果博落回的主要区别也是果荚部位,因此在博落回属植物生长早期区分两者困难较大。本实验运用ITS2序列鉴定博落回属植物,结果显示所有实验样本的PCR扩增和测序成功率均达到100%,表明ITS2序列适合于博落回属植物的鉴定且稳定性较好。除了运用ITS2序列以外,本实验样本涵盖了湖南、江西、浙江、湖北、河南、陕西、重庆等多个产地,鉴定结果表明不同产地的博落回属植物种内差异较小,说明博落回属植物遗传多样性较低并且符合本实验室之前的SSR实验结果[15]。从最终数据分析,ITS2条形码序列构建的NJ进化树显示ITS2序列能够准确鉴别博落回与小果博落回,保证了原料收集的准确性。
[1] Zeng J,Liu Y,Liu W,et al.Integration of transcriptome,proteome and metabolism data reveals the alkaloids biosynthesis in Macleaya cordata and Macleaya microcarpa[J].Plos One,2013,8(1):2159-2163.
[2] 刘秀斌.博落回属植物中生物碱代谢累积研究[D].长沙:湖南农业大学,2012.
[3] Hebert P D,Ratnasingham S,DeWaard J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proc Biol Sci,2003,270(Suppl 1):S96-S99.
[4] Chen J,Zhao J,Erickson D L,et al.Testing DNA barcodes in closely related species of Curcuma(Zingiberaceae)from Myanmar and China[J].Mol Ecol Resour,2015,15(2):337-348.
[5] Ashfaq M,Asif M,Anjum Z I,et al.Evaluating the capacity of plant DNA barcodes to discriminate species of cotton(Gossypium:Malvaceae)[J].Mol Ecol Resour,2013,13(4):573-582.
[6] Yan L J,Liu J,Moller M,et al.DNA barcoding of Rhododendron(Ericaceae),the largest Chinese plant genus in biodiversity hotspots of the Himalaya-Hengduan Mountains[J].Mol Ecol Resour,2015,15(4):932-944.
[7] Yang J B,Wang Y P,Moller M,et al.Applying plant DNA barcodes to identify species of Parnassia(Parnassiaceae)[J].Mol Ecol Resour,2012,12(2):267-275.
[8] Yuan Q J,Zhang B,Jiang D,et al.Identification of species and materia medica within Angelica L.(Umbelliferae)based on phylogeny inferred from DNA barcodes[J].Mol Ecol Resour,2015,15(2):358-371.
[9] 辛天怡,雷美艳,宋经元.中药材DNA条形码鉴定研究进展[J].中国现代中药,2015,17(2):170-176.
[10] Chen S,Pang X,Song J,et al.A renaissance in herbal medicine identification:from morphology to DNA[J].Biotechnol Adv,2014,32(7):1237-1244.
[11] 李骁,赵龙,王佩,等.野豌豆属蒙药药用植物的rDNA-ITS序列分析[J].中草药,2015,46(12):1814-1818.
[12] 樊杰,白妍,束明月.远志属7种药用植物ITS1和ITS2序列分析[J].中草药,2015,46(4):562-565.
[13] 张忠廉,宋美芳,李海涛,等.千斤拔属药用植物DNA条形码鉴定研究[J].中草药,2015,45(1):118-122.
[14] 夏至,高致明,张红瑞,等.黄芩及其同属近缘种的DNA条形码鉴定研究[J].中草药,2014,45(1):107-112.
[15] 朱鹏程,柳亦松,黄鹏,等.博落回SSR引物的开发以及遗传多样性分析[J].生命科学研究,2013,17(2):120-124.
IdentificationofPlantsinMacleayaspp.BasedonITS2Sequence
HUANG Peng1,LIU Jinfeng1,XU min1,XIA liqiong2,LIU Yisong1,3,TANG Qi1,TANG Zhaoshan4,ZENG Jianguo1*
(1.NationalandLocalUnionEngineeringResearchCenterfortheVeterinaryHerbalMedicineResourcesandInitiative,
HunanAgriculturalUniversity,Changsha410128,China;2.Schoolofpharmacy,HunanUniversityofChineseMedicine,Changsha410208,China;3.VeterinaryFacultyofHunanAgriculturalUniversity,Changsha410128,China;4.MicolltaBioresourceInc,Changsha410005,China)
Objective:To identify the medicinal plantMacleayaspp.by using ITS2 sequence.MethodsThe ITS2 sequence and sequence of 30 samples collected from Hunan,Jiangxi,Zhejiang,Hubei,Henan,Shanxi,Chongqing was amplified and the amplification efficiency,the sequencing success rate,the constitution of base pair,intra- and inter-specific variation and NJ(Neighbor-Joining) tree were analyzed,in order to evaluate the ITS2 sequencing results.ResultsThe ITS2 sequence length ofMacleayaspp.was within 500 bp;the amplification efficiency and the success sequencing rate was 100% and 30 ITS2 sequences were obtained.ConclusionThe ITS2 sequence can be effective and accurate for identification ofMacleayaspp..
Macleayaspp.;ITS2 sequence;DNA barcode;Species identification;Genetic differentiation
国家重点实验室培育基地开放项目(15KFXM09);湖南省科技重点计划(2016SK3002)
*
曾建国,博士生导师,教授,研究方向:中药资源与综合利用;Tel:(0731)84673824,E-mail:zengjianguo@hunau.edu.cn
10.13313/j.issn.1673-4890.2017.10.003
2017-02-20)

