基于GEO数据库的EGFR或KRAS突变型非小细胞肺癌耐药基因表达谱分析
吴 峰 朱圣明 胡承浩 邱 力 王 科
基于GEO数据库的EGFR或KRAS突变型非小细胞肺癌耐药基因表达谱分析
吴 峰 朱圣明 胡承浩 邱 力 王 科
目的通过对相关数据库数据分析,探究NSCLC中EGFR和KRAS突变与耐药基因表达的关系,以及对患者生存和预后的影响。方法对NCBI中GEO数据库中的数据集数据进行表达量分析、生存分析和相关性分析。结果①研究分析表明,ABCC1、GSTP1、MGMT1和RRM1基因在NSCLC组织中存在明显的高表达(P<0.0001;P<0.0001;P=0.0014;P<0.0001);而ERCC1和TUBB3基因表达差异没有统计学意义(P=0.3922;P=0.4583)。②在KM组中,TUBB3、ABCC1和XPA基因存在明显的高表达(P=0.0169;P=0.033;P=0.0165),而在EM组中只有ABCC1存在明显的高表达(P=0.0002)。③在KM组中ABCC1和XPA的表达情况呈正相关(P=0.025,γ=0.4992);而它们与TUBB3的表达情况呈负相关(P=0.0075,γ=-0.5789;P=0.0012,γ=-0.6696)。④ABCC1和TUBB3的高表达会导致肺癌患者的总生存率和无复发生存率明显降低。结论在NSCLC中EGFR或KRAS基因的突变会导致部分相关耐药基因的差异表达,这些差异表达的基因之间存在相关性,而且对患者的生存及预后又具有重要影响。
非小细胞肺癌;EGFR;KRAS;耐药基因
(ThePracticalJournalofCancer,2017,32:1411~1415)
非小细胞肺癌(NSCLC)40%~80%存在表皮生长因子受体(EGFR)的过度表达和突变。针对该靶点的药物主要为EGFR酪氨酸激酶抑制剂(EGFR-TKI)[1]。尽管EGFR突变的NSCLC患者大多会对其首次治疗有响应,但在此后肿瘤又会有不同程度的复发,甚至出现耐药[2-3]。KRAS也是NSCLC的驱动突变,其结果是信号通路的持续激活,KRAS的突变与抗EGFR抗体药物的治疗效果也有密切关系[4]。
本研究旨在通过相关NSCLC的耐药基因的表达研究及相关样本的预后情况,了解EGFR和KRAS突变与NSCLC耐药性的相关性,以期为临床EGFR和KRAS突变的NSCLC患者的用药和治疗提供一个可靠的理论基础。
1 材料与方法
1.1 研究材料
本研究所用到的材料均来自于NCBI的GEO数据库,具体信息见表1。
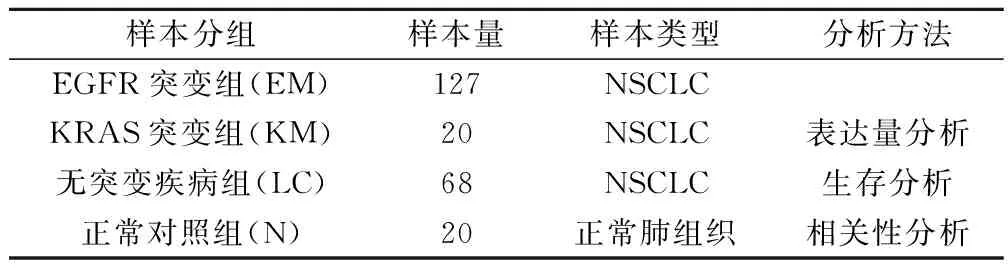
表1 GES31210数据集样本分组统计表
1.2 分析方法
1.2.1 表达量分析 基于数据库特定研究样本数据集中特定目标基因的表达量的相对值进行表达量的分析,所有数据都经过对数转换和中位数中心化处理。根据表达量处理后数据进行聚类和差异分析。文中热图由HemI软件制作,比较差异分析图由GraphPad Prism 5软件制作(P<0.05表示差异有统计学意义)。
1.2.2 生存分析 基于数据库特定研究样本数据集中特定目标基因的表达量的相对值及对应样本的总生存(OS)和无复发生存(RFS)信息进行生存分析,所有数据都经过对数转换和中位数中心化处理,并采用Kaplan-Meier分析方法进行分析。
1.2.3 相关性分析 基于数据库特定研究样本数据集中特定目标基因的表达量的相对值进行相关性分析,所有数据都经过对数转换和中位数中心化处理(P<0.05表示差异有统计学意义,γ<0表示负相关,γ>0表示正相关)。
2 结果
2.1 相关耐药基因在NSCLC患者中的表达分析
为了评估NSCLC耐药性与EGFR和KRAS基因突变的关系,我们选取了7个已有报道过的NSCLC相关的耐药基因,分别为切除修复交叉互补基因1(excision repair cross complement-1,ERCC1)、核糖核苷还原酶亚单位1 (ribonucleotide reductase M1,RRM1)、细胞骨架微管蛋白Ⅲ(classⅢ-tubulin,TUBB3)、多药耐药相关蛋白基因1(multidrug resistence-associated protein,MRP1或ABCC1)、谷胱甘肽硫转移酶1(glutathione-S-transferase P1,GSTP1),O6-甲基鸟嘌呤-DNA甲基转移酶(O6-methy lguanine-DNA methyltransferase,MGMT)和DNA修复基因(DNA damage recognition and repair factor,XPA)。据以往的相关研究表明,这些基因在NSCLC的耐药性调控方面都起着非常重要的作用。据此,我们对GSE31210数据集中的不同分组的NSCLC样本和正常样本的这些基因的表达进行了表达谱的聚类分析,结果表明,不同的耐药相关基因在不同分组间的表达存在差异,如RRM1和GSTP1基因在NSCLC组织中存在明显的高表达,这也与以往的类似研究结果相一致。这些表明,耐药基因的表达在不同突变类型NSCLC分组间存在差异,可能与NSCLC的耐药性及EGFR和KRAS基因的突变有关。
2.2 正常对照组与NSCLC组间耐药基因的表达差异比较
通过上述表达谱显示,耐药基因在不同分组间的表达量确实存在差异。所以,为了进一步具体探究这些表达差异,我们在不同分组间进行了具体的比较和统计学分析。结果表明,与正常肺组织相比,ABCC1、GSTP1、MGMT1和RRM1基因在NSCLC组织中存在明显的高表达(ABCC1,P<0.0001;GSTP1,P<0.0001;MGMT1,P=0.0014;RRM1,P<0.0001);而ERCC1和TUBB3基因在正常组织和NSCLC组织间的表达差异没有统计学意义(ERCC1,P=0.3922;TUBB3,P=0.4583),甚至XPA基因在正常组织中存在一定程度的高表达(XPA,P=0.0004)(图1)。
2.3 不同基因突变组间耐药基因的表达差异比较
为了进一步明确耐药相关基因的表达与EGFR和KRAS基因突变间的联系,我们又对EGFR基因突变组(EM)、KRAS基因突变组(KM)和无突变NSCLC组(LC)间的耐药基因的表达差异进行了比较。结果显示,不同分组间耐药基因的表达存在明显的差异,尤其是在KM组中,与LC组相比,TUBB3、ABCC1和XPA基因存在明显的高表达(TUBB3,P=0.0169;ABCC1,P=0.033; XPA,P=0.0165),而在EM组中只有ABCC1存在明显的高表达(ABCC1,P=0.0002),TUBB3反而呈现出明显的低表达现象(TUBB3,P=0.0003)(表2和图2)。这些结果表明,相关耐药基因的表达与NSCLC中EGFR或KRAS基因的突变存在着一定的联系,而且通过这一比较分析可以看到不同的耐药基因在不同的突变组中表达不同,各耐药相关基因也不会同时处于同样的表达趋势,也许它们之间还存在功能互补的关系。
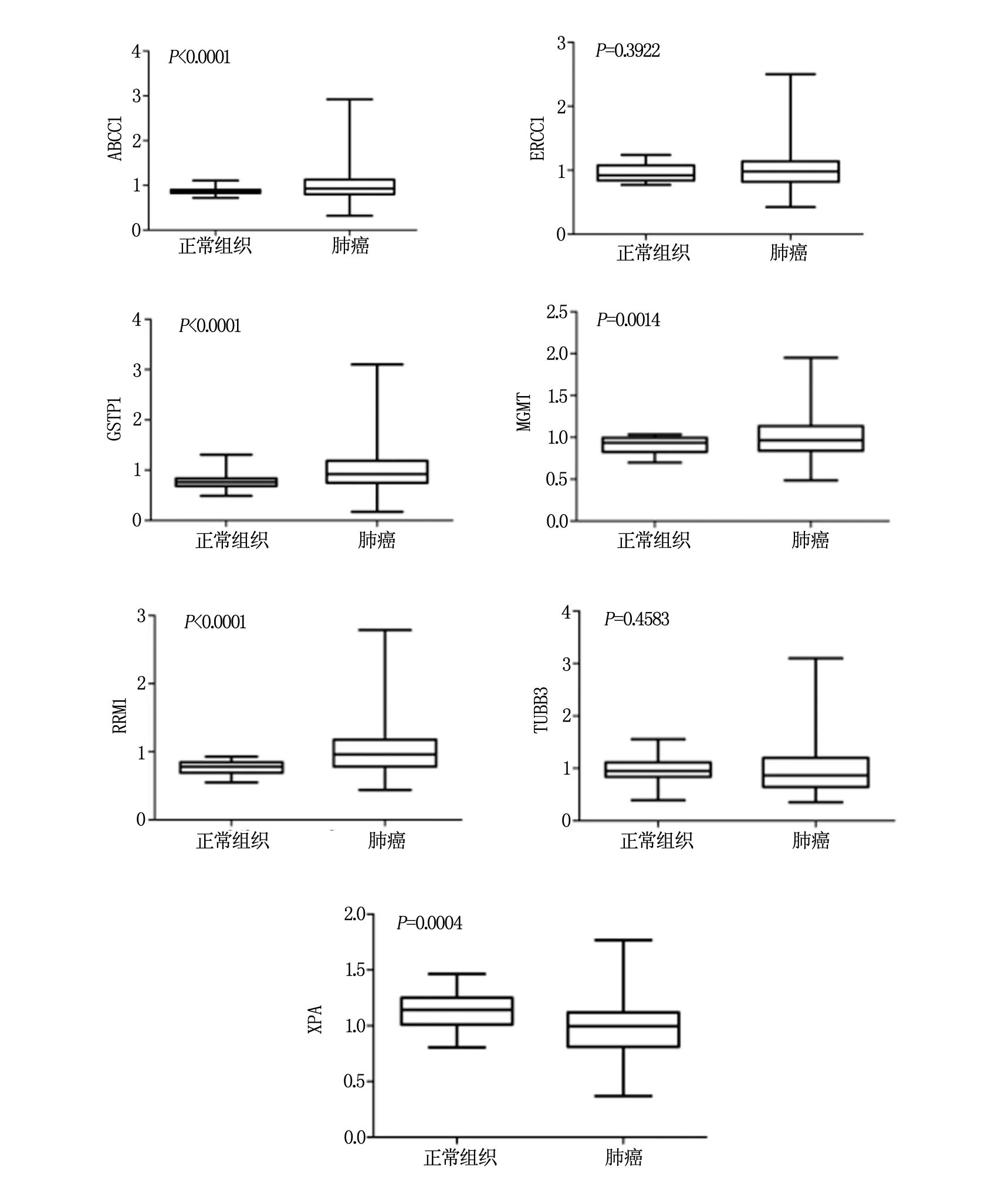
图1 正常组与NSCLC组间耐药相关基因的表达差异比较分析
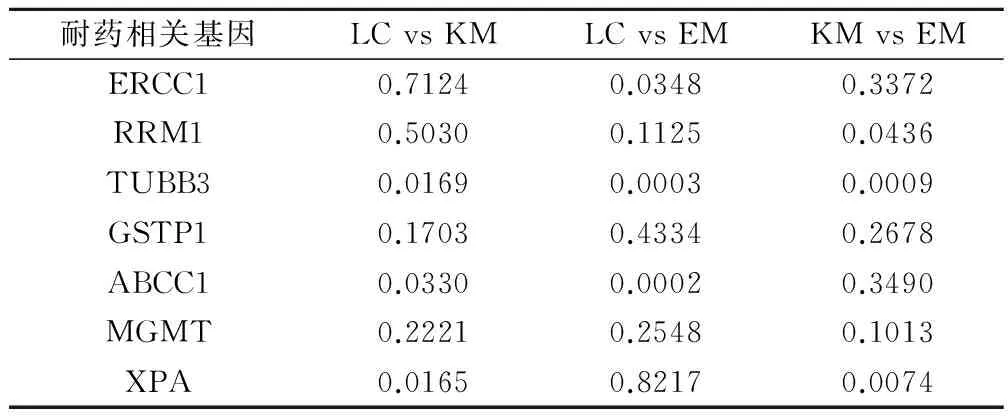
耐药相关基因LCvsKMLCvsEMKMvsEMERCC10.71240.03480.3372RRM10.50300.11250.0436TUBB30.01690.00030.0009GSTP10.17030.43340.2678ABCC10.03300.00020.3490MGMT0.22210.25480.1013XPA0.01650.82170.0074
2.4 耐药基因表达间的相关性分析
鉴于上述的研究结果,为了进一步探明不同差异表达的耐药相关基因之间的关系,我们选取KM组中明显高表达的3个基因TUBB3、ABCC1和XPA,并对其进行了相关性分析。结果显示,在KM组中ABCC1和XPA的表达情况呈正相关(P=0.025,γ=0.4992);而它们与TUBB3的表达情况呈负相关(P=0.0075,γ=-0.5789;P=0.0012,γ=-0.6696)(图3)。这就说明,在KM组中,ABCC1和XPA可能存在协同高表达的情况,而它们与TUBB3之间则可能是一种耐药功能上的互补关系。因此即便是在同样高表达的KM组中,各耐药基因的高表达情况并不完全同步,且可能存在互补的现象。
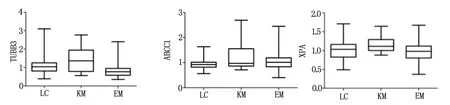
图2 不同基因突变组间耐药相关基因的表达差异比较分析
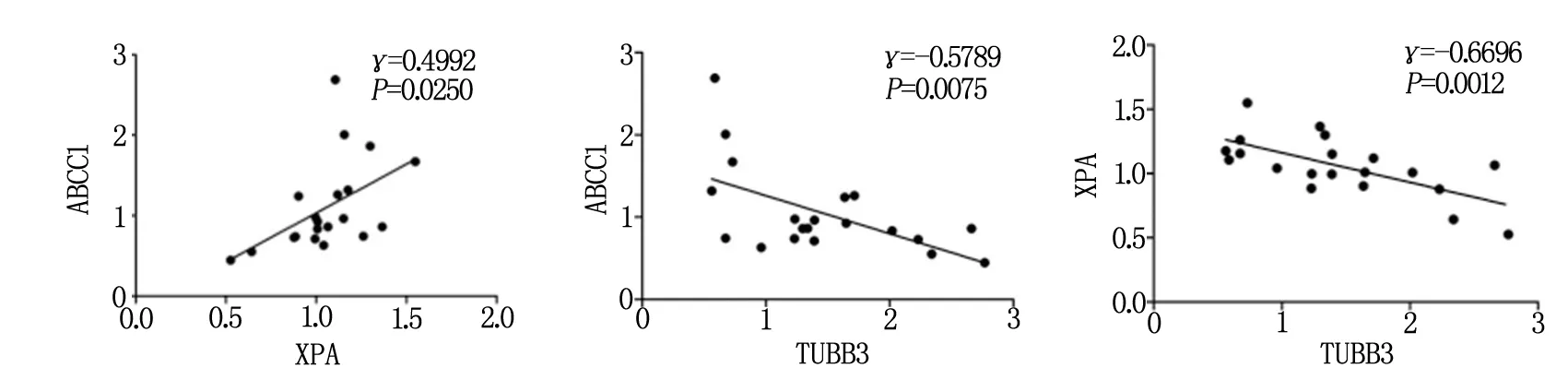
图3 KRAS突变组中相关耐药基因表达相关性分析
2.5 耐药基因异常表达对NSCLC患者生存及预后的影响
通过我们对部分高表达耐药相关基因的相关性分析,我们在一定程度上揭示了其在表达上的协同或互补的规律。基于此,我们进一步对ABCC1和TUBB3基因表达对患者生存和预后的影响进行了分析。结果表明,在EGFR突变的NSCLC患者中,ABCC1的高表达会导致肺癌患者的总生存率明显降低(P=0.0186),并且它的高表达也同样会导致肺癌患者的无复发生存率明显降低(P=0.0331);与此类似,TUBB3的高表达也会导致肺癌患者的总生存率明显降低(P=0.0018),并且它的高表达也同样会导致肺癌患者的无复发生存率明显降低(P=0.0071)。见图4。
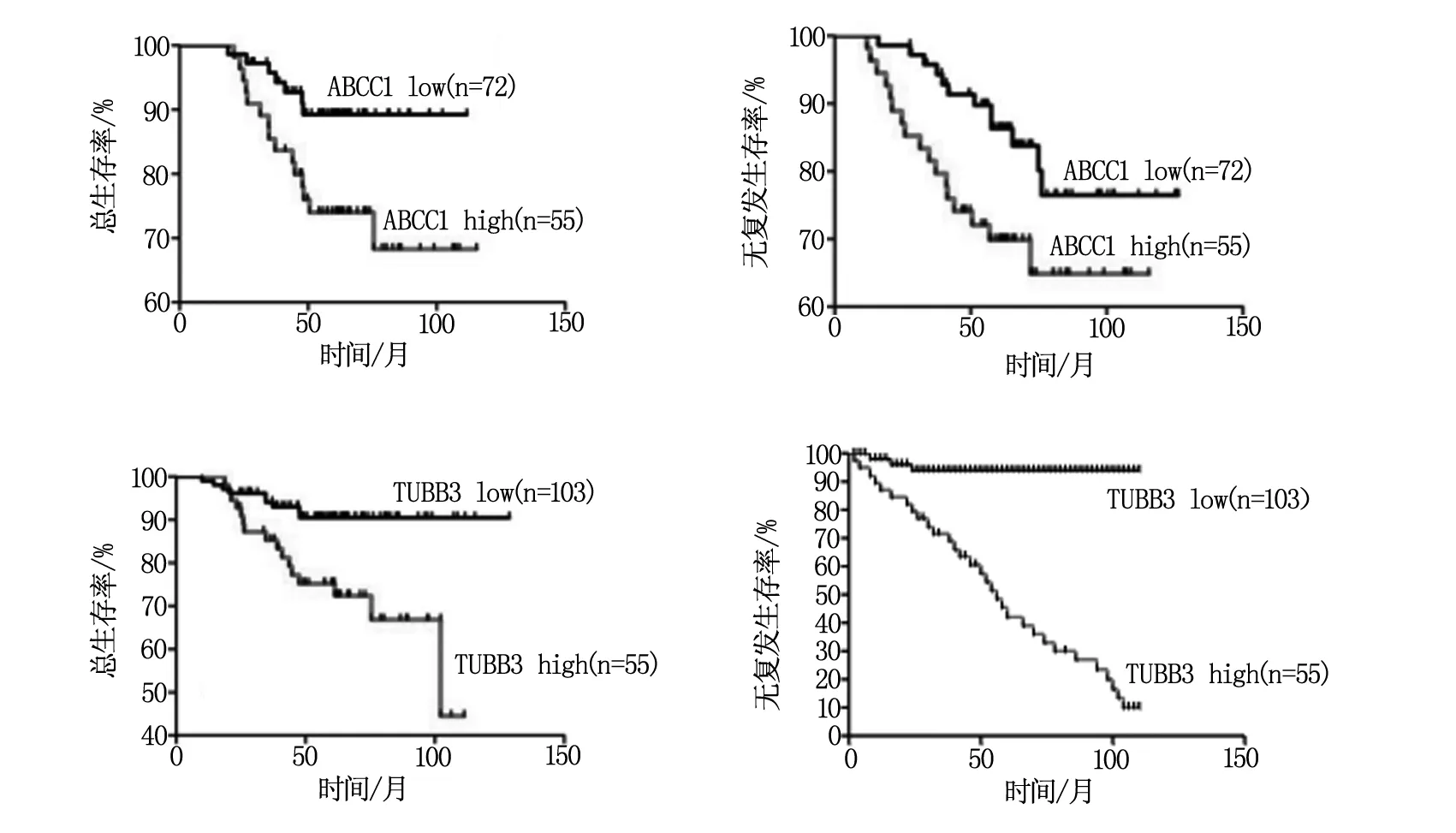
图4 ABCC1和TUBB3高表达对NSCLC患者的生存分析
3 讨论
基于NSCLC患者多存在EGFR的过表达或突变,目前所采用EGFR-TKIs靶向治疗NSCLC患者仍然属于较为有效的治疗手段,可以使患者免受传统化疗手段所带来的痛苦[5]。但在初期对EGFR-TKIs敏感性阶段的治疗后,耐药性问题的出现困扰了NSCLC患者的继续治疗[6]。有研究者发现,KRAS基因的突变可能与NSCLC耐药性相关,而且随着分子生物学的发展,与耐药性有关的一些基因也不断被发现,正如本研究中所分析那些耐药相关基因,它们在DNA修复、细胞增殖和细胞运动等方面发挥着不同的功能[7-8]。由于研究发现的众多基因、蛋白和信号通路等均参与同一种或者多种肿瘤的发生和发展,所以尽可能地阐明这些耐药基因与EGFR或KRAS基因突变的相关性及其对患者生存和预后的影响将有利于寻找解决EGFR-TKIs耐药性问题以及靶向治疗NSCLC患者的分子靶点和治疗策略,通过这样阻断治疗过程中出现的耐药现象,提高靶向药物治疗NSCLC患者的疗效。鉴于此,本研究基于数据库,对EGFR和KRAS基因突变的NSCLC样本中相关耐药基因进行了分析。
我们的研究分析表明,ABCC1、GSTP1、MGMT1和RRM1基因在NSCLC组织中存在明显的高表达;而ERCC1和TUBB3基因在正常组织和NSCLC组织间的表达差异没有统计学意义,且XPA基因反而在正常组织中存在一定程度的高表达。这一表达情况的分析证明了耐药基因异常表达的情况存在于NSCLC病例中,且有可能与疾病的某些进程相关。与此同时,部分耐药相关基因在此之间存在差异表达,尤其是在KM组中,TUBB3、ABCC1和XPA基因存在明显的高表达,而在EM组中只有ABCC1存在明显的高表达,TUBB3反而呈现出明显的低表达现象。由此,相关耐药基因的表达与NSCLC中EGFR或KRAS基因的突变存在着一定的联系,而且通过这一比较分析可以看到不同的耐药基因在不同的突变组中表达不同。针对于此,进一步的研究结果显示,在KM组中ABCC1和XPA的表达情况呈正相关;而它们与TUBB3的表达情况呈负相关。这一结果就说明了在KM组中,ABCC1和XPA可能存在协同高表达的情况,而它们与TUBB3之间则可能是一种耐药功能上的互补关系。最终,我们对本研究分析筛选出的耐药基因ABCC1和TUBB3进行了生存分析,在EGFR突变的NSCLC患者中,ABCC1、TUBB3的高表达会导致肺癌患者的总生存率明显降低,并且它们的高表达也同样会导致肺癌患者的无复发生存率明显降低。
通过本研究对相关数据库中NSCLC样本数据的分析和归纳,发现在NSCLC中EGFR或KRAS基因的突变会导致部分相关耐药基因的差异表达,尤其是高表达,这就与某些NSCLC病例的耐药性相关联了。此外,这些耐药基因的表达还存在协同或互补的关系,通过这样的表达关系可以更大化地维持肿瘤的增殖和存活等功能,以进一步增强肿瘤的耐药性。鉴于此,在今后的研究和临床治疗中,可以以这些差异表达的耐药基因作为针对的依据及靶向治疗的靶点。
[1] Gainor JF,Shaw AT.Emerging paradigms in the development of resistance to tyrosine kinase inhibitors in lung cancer〔J〕.J Clin Oncol,2013,31(31):3987-3996.
[2] Rosell R,Carcereny E,Gervais R,et al.Erlotinib versus sta-ndard chemotherapy as first-line treatment for European patients with advanced EGFR mutation-positive non-small-cell lung cancer(EURTAC):a multicentre,open-label,randomised phase 3 trial〔J〕.Lancet Oncol,2012,13(3):239-246.
[3] Sequist LV,Yang JC,Yamamoto N,et al.Phase Ⅲ study of afatinib or cisplatin plus pemetrexed in patients with metastatic lung adenocarcinoma with EGFR mutations〔J〕.J Clin Oncol,2013,31(27):3327-3334.
[4] Ohashi K,Sequist LV,Arcila ME,et al.Lung cancers with acquired resistance to EGFR inhibitors occasionally harbor BRAF gene mutations but lack mutations in KRAS,NRAS,or MEK1〔J〕.Proc Natl Acad Sci USA,2012,109(31):E2127-2133.
[5] Takezawa K,Pirazzoli V,Arcila ME,et al.HER2amplificati-on:a potential mechanism of acquired resistance to EGFR inhibition in EGFR-mutant lung cancers that lack the second-site EGFRT790M mutation〔J〕.Cancer Discov,2012,2(10):922-933.
[6] Yu HA,Arcila ME,Rekhtman N,et al.Analysis of tumor s-pecimens at the time of acquired resistance to EGFR-TKI therapy in 155 patients with EGFR-mutant lung cancers〔J〕.Clin Cancer Res,2013,19(8):2240-2247.
[7] Bosc C,Ferretti GR,Cadranel J,et al.Rebiopsy during disease progression in patients treated by TKI for oncogene-addicted NSCLC〔J〕.Target Oncol,2015,10(2):247-253.
[8] Del Re M,Vasile E,Falcone A,et al.Molecular analysis of cell-free circulating DNA for the diagnosis of somatic mutations associated with resistance to tyrosine kinase inhibitors in non-small-cell lung cancer〔J〕.Expert Rev Mol Diagn,2014,14(4):453-468.
(编辑:甘艳)
AnalysisofResistantGenesExpressionProfilinginEGFRorKRASMutantNon-smallCellLungCancer(NSCLC)BasedonGEODatabase
WUFeng,ZHUShengming,HUChenghao,etal.
TaiheHospital,Taihe,442000
ObjectiveTo explore the relationship between EGFR or KRAS mutant NSCLC and resistance genes and the impact on the survival and prognosis of NSCLC patients,based on relevant database data analysis.MethodsBased on data sets of GEO in the NCBI database,to perform express analysis,survival analysis and correlation analysis.Results①Analysis showed thatERCC1,GSTP1,MGMT1 and RRM1 genes significantly higher expression in NSCLC(P<0.0001;P<0.0001;P=0.0014;P<0.0001);and the expression of ERCC1 and TUBB3 had no statistically significance(P=0.3922;P=0.4583).②In the KM group,the expression of TUBB3,ABCC1 and XPA genes were significantly higher(P=0.0169;P=0.033;P=0.0165),but in the EM group only ABCC1 significant higher expression(P=0.0002).③In the KM group,the expression of ABCC1 and XPA were positively correlated(P=0.025,γ=0.4992);and they were negatively correlated with TUBB3(P=0.0075,γ=-0.5789;P=0.0012,γ=-0.6696).④High expression of ABCC1 and TUBB3 will lead to overall survival and relapse-free survival of patients with lung cancer were significantly reduced.ConclusionThe mutations of EGFR or KRAS gene in NSCLC could lead to differences in part related resistance gene expression,in which the genes expression are correlated,but also has a significant impact on the survival and prognosis of patients.
NSCLC;EGFR;KRAS;Resistant genes
442000 湖北省十堰市太和医院(湖北医药学院附属医院)
王 科
10.3969/j.issn.1001-5930.2017.09.005
R734.2
A
1001-5930(2017)09-1411-05
2016-10-24
2017-04-10)

