基于花椒转录组序列SSR分子标记开发及花椒种质鉴定
李立新,司守霞,魏安智,刘玉林,冯世静 ,杨途熙
(1.西北农林科技大学,陕西 杨凌 712100;2.河南林业职业学院,河南 洛阳 471002)
基于花椒转录组序列SSR分子标记开发及花椒种质鉴定
李立新1,司守霞2,魏安智1,刘玉林1,冯世静1,杨途熙1
(1.西北农林科技大学,陕西 杨凌 712100;2.河南林业职业学院,河南 洛阳 471002)
为拓展分子标记在花椒种质资源分析中的应用、开发花椒EST-SSR功能性分子标记、分析花椒DNA指纹图谱,利用凤县大红袍花椒的茎尖节点转录组cDNA数据库中45 057条长度大于200 bp的非冗余 Unigene序列,使用MIcroSAtellite(MISA)软件搜索SSR位点,分析花椒cDNA序列中SSR位点的频率和密度、SSR重复基元种类及比例、SSR重复次数与数量等分布特征;用Primer 3.0软件在线设计SSR引物并经PCR扩增筛选适合的多态性引物;用Quantity One软件统计多态性条带和分子量大小;NTsys 2.0软件分析12份花椒种质的遗传距离、构建树状聚类图及DNA指纹图谱库。结果表明,45 057条序列中有3 315 条Unigene序列包含SSR位点,共3 814个,3 315条序列中SSR位点出现频率为7.07%;在检索出的 SSR位点中,二核苷酸、三核苷酸是主要重复类型,分别占29.42%和58.58%,二核苷酸重复中以AG/TC、CT/GA 出现频率最高,三核苷酸重复中GAA/CTT、AGA/TCT出现频率最高;二、三、四、五、六核苷酸重复基元总数随着重复次数的增加呈明显下降趋势。利用Primer 3.0设计的64对EST-SSR引物中,55对引物能产生预期片段大小的 PCR 产物,其中18对引物具有多态性;利用18对引物对12份花椒种质进行PCR扩增,共产生81条扩增条带,其中多态性条带73条,多态率为90.12%;各花椒种质的遗传相似系数为0.552 6~0.894 7,平均为0.725 0;经UPGMA聚类分析在相似系数0.70处12份花椒种质共分为3类,即顶坛花椒为Ⅰ类、竹叶椒为Ⅱ类、其他花椒为Ⅲ类;指纹图谱分析中,8对引物在5份种质中能扩增出特征带型,最少用3对引物进行组合即可将12份花椒种质区分开。成功在凤县大红袍花椒的茎尖节点转录组cDNA 序列中开发SSR标记,设计并筛选出8对能扩增出特征带型的引物,最少用3对引物进行组合即可将12份花椒种质区分开,这为今后花椒遗传多样性分析、遗传图谱构建等方面提供新的引物序列并奠定了基础。
花椒;EST-SSR;引物开发;指纹图谱
花椒(ZanthoxylumbungeanumMaxim)为芸香科花椒属植物,是我国的特色辛香料和中药材,也是目前退耕还林中重要的生态型山地栽培经济树种[1]。花椒为单性结实,无融合生殖率较高[2],因此遗传稳定性较高,这对花椒的良种选育提供了有力的理论依据。目前关于花椒的研究主要集中在花椒精油提取与分析、成分功能及开发利用等方面[3-5],在DNA分子水平上对花椒的研究虽然已有RAPD[6]、ISSR[7]、SRAP[8]及转录组测序[9]等方面的报道,但尚未见有关EST-SSR的报道。
EST-SSR(Expressed sequence tag based simple sequence repeat)是基于EST序列或cDNA数据开发的一种标记[10];它来源于功能基因,可直接反映功能基因的多样性,具备基因组SSR标记的优点和引物开发成本低、通用性好等特点,而且与生理生化特征和某些形态特征相关联[11];现已广泛应用在植物遗传多样性分析、遗传图谱构建及分子标记辅助育种等方面的研究[12]。该标记已在小麦[10-13]、柑橘[14-15]、枳壳[16]、苹果[17-19]等植物中得到开发和应用。近年来,随着生物技术的进展,基于EST序列的SSR标记的开发也在日趋增加[20],收录的EST数目也由1991年的不足2 000条发展到2010年的65 255 769条(共涉及1 978个物种),目前,NCBI数据库中收录的植物EST的数量已多达76 165 384条。
但是,迄今为止NCBI数据库中收录的花椒EST 序列数量还很少,可供选择性范围较小;鉴于此,本试验对获得的花椒cDNA数据库中非冗余序列进行了SSR信息发掘和EST-SSR标记引物开发,分析了花椒指纹图谱,希望能为花椒EST-SSR分子标记的研究、遗传图谱构建及品种鉴定、良种选育提供理论依据。
1 材料和方法
1.1试验材料
转录组序列样本材料为凤县大红袍花椒的茎尖节点,共包含45 057条 Unigenes序列。SSR扩增所用花椒样品(种质)来源于国家林业局花椒工程技术研究中心凤县花椒试验示范站 (表1)。
选择生长良好、无病虫害的植株进行采样。每个样品取5株重复。采样时,采集不同花椒种质的新鲜嫩叶,用冰袋保鲜,带回实验室置于-70 ℃冷冻保存备用。
1.2试验方法
1.2.1 花椒基因组DNA提取 试验方法参照李晓等[21]的CTAB法对花椒嫩叶进行基因组DNA提取,取1.5 μL 所提取的DNA 于超微量核酸分析仪上测定其在紫外光波长260,280 nm处的吸光比值以检测所提取DNA的纯度和DNA浓度。将基因组DNA于-20 ℃保存,用于后续SSR-PCR分析。
1.2.2 序列搜索及SSR位点查找 cDNA序列来源于蒋弘刚等[9]使用二代高通量测序技术获得的凤县大红袍花椒茎尖节点转录组测序数据,以组装出来的 Unigene 作为参考序列,使用 MIcroSAtellite(MISA)软件找出所有的 SSR位点[22]。本试验SSR位点的查找参照刘博等[23]SSR位点搜索的标准:二、三、四、五、六核苷酸重复基元的重复次数分别大于或等于6,5,4,4,3次;一般查找到的SSR重复基元的重复次数越多,相关的多态性也就越高[24],并分析 SSR 在cDNA序列中的分布特征。

表1 花椒试验材料Tab.1 Zanthoxylum bungeanum accessions used in this study
1.2.3 EST-SSR引物设计 引物设计参照崔海荣等[25]引物开发的方法和原则:cDNA序列的长度大于100 bp;选择二、三、四、五、六核苷酸重复基元的重复次数分别大于或等于10,6,5,5,4 次的序列且SSR位点的开始和结束位置距5′和3′端均不少于50 bp;引物GC 含量为40%~60%,最佳为50%;退火温度为50~ 60 ℃,最佳温度为55 ℃,上下游引物相差不大于5 ℃;引物长度为 18~25 bp,最佳为20 bp;PCR扩增产物长度100~500 bp;应尽量避免引物二聚体(Dimer)、发夹结构(Hairpin)以及连续6个碱基配对的出现;用Primer 3.0[26]软件在线设计SSR 侧翼区域引物,其他参数采用默认值。引物设计时先用中括号将 SSR 位点括起来,以确保产物序列中包括 SSR 位点。引物由生工生物工程(上海)公司合成。
1.2.4 PCR扩增及多态性引物筛选及检测 本试验PCR反应体系为:总体积为20 μL:包含10 μL 2×TaqMasterMix(CWBIO北京生物技术有限公司(中国)有限公司),正反向引物(10 μmol/L)各1 μL,双蒸水7 μL和基因组DNA(50 ng/μL)1 μL。PCR循环参照Feng 等[8]的程序。扩增产物用10%的非变性聚丙烯酰胺凝胶电泳[25]、0.5×TBE的缓冲液、稳压250 V、室温电泳4 h、银染显色、胶片观察灯照相观察的方法进行检测。
1.2.5 数据处理 选取可重复且清晰可辨的扩增条带,有谱带计为1,无谱带计为0,构建初始数据矩阵,计算扩增条带和多态性条带及比率;用NTSYS-pc2.10e软件[27]SimQual 程序求 Jaccard 相似系数,用SHAN 程序中的 UPGMA(非加权平均法)进行聚类分析。并通过 Treeplot模块生成聚类图,并对其进行Cophenetic相关性检验;使用POPGENE[28]软件计算花椒种质的遗传多样性指标,观测等位基因数(Na)、有效等位基因数(Ne)、Nei′s遗传多样性(H)以及Shannon′s信息指数(I);根据条带特征分析花椒DNA指纹图谱。
2 结果与分析
2.1花椒cDNA序列SSR位点分布特征
2.1.1 花椒EST-SSR频率和密度 在凤县大红袍花椒茎尖节点转录组测序及数据分析得到的cDNA 序列中,长度大于200 bp的非冗余Unigene序列共45 057条,其总长度约为32.5 Mbp,平均长度为610 bp;在全部的 Unigene 序列中有3 315条包含简单重复序列,占全部序列的 7.36%,其中422 条包含2个及2个以上简单重复基元的序列,占全部序列的0.94%;在全部非冗余序列中共检测到3 814个SSR位点,SSR的出现频率为 7.07%,平均分布距离约7.2 kb,花椒的SSR位点分布距离比杨树(Populus)14.0 kb[29]、杜仲11.6 kb[30]、银杏(Ginkgobiloba) 12.02 kb[31]的要大,比柑橘(Citrus) 5.7 kb[32]、茶树(Camelliasinensis) 3.68 kb[33]、橡胶树(Heveabrasiliensis)3.93 kb[34]等木本植物的分布距离要小。
2.1.2 花椒EST-SSR重复基元及比例 在获得的SSR位点中,二-六核苷酸重复基元都能被检出,但出现的频率有明显的差异。其中,二、三、四、五、六核苷酸重复基元分别有 5,31,29,42,175共5种类型(表2)。SSR重复类型最多的为三核苷酸,共有1 866个,占全部SSR的58.58%,出现频率为4.14%;其次是二核苷酸重复类型,共有937个,占全部SSR的29.42%,出现频率为2.08%;五核苷酸重复类型出现频率较低,共64个,占全部SSR总数的2.01%,出现频率0.14%。可见,在花椒SSR位点中,二、三核苷酸重复类型占主导地位(图1-A)。
在含有二核苷酸重复基元的花椒 EST 序列中,AG/TC和CT/GA在二核苷酸重复基元,分别占二核苷酸总数的42.53%和37.81%,所占比例最高;在花椒三核苷酸重复基元中,GAA/CTT基序出现频率最高,其次为AGA/TCT和ACT/TGA,这3类重复基元占三核苷酸重复类型总量的33.58%,27.03%和13.69%(图1-B、C)。

表2 花椒EST-SSR类型及分布频率Tab.2 Repeat types and frequency of the EST-SSR in Zanthoxylum bungeanum

A.核苷酸重复类型及比例;B.二核苷酸重复中不同重复基元所占比例;C.三核苷酸重复中不同重复基元所占比例;D.不同 SSR 重复基元的数量与其重复次数。A.The nucleotide repeat type and proportion;B.The proportion of different repeating primers in the dinucleotide repeats;C.The proportion of different repeat motifs in trinucleotide repeats;D.The number of repetitive primitives for different SSRs and their number of repetitions.
在四核苷酸重复基元中AAGA/TTCT和ATAC/TATG基序出现频率最高,分别占四核苷酸总数的14.29%和10.00%。五核苷酸基元中TTCTT/AAGAA出现频率最高,出现5次,出现频率为7.81%;六核苷酸重复基元中GCAACA/CGTTGT出现频率最高,出现7次,出现频率为2.82%。
2.1.3 花椒EST-SSR重复次数与数量 由图1-D可知,不同SSR重复基元的数量与其重复次数的统计结果表明:重复基元的重复次数在4~12次,二核苷酸重复次数种类最多,重复次数在6~12次;六核苷酸重复次数种类最少,只有重复次数4次这一种;在所有重复基元中以5次重复的核苷酸最多,占SSR总数的29.98%;另外,在重复基元相同时,随着重复基元重复次数(或 SSR 长度)的增加,EST-SSR 的数量减少,随着重复次数的增加,二、三、四、五核苷酸重复基元的数量和重复基元总数均呈明显下降趋势。
2.2花椒DNA提取及SSR产物有效性检测
本试验所提取样本 DNA 吸光度比值OD260/280均为1.8~2.0,试验结果表明,CTAB法能提取到纯度较高的DNA。本试验随机挑选了符合筛选条件的序列,用Primer 3.0在线设计了包括二、三、四、五、六核苷酸重复基元的SSR位点在内的共64 对引物来检测所发掘出的 EST-SSR 标记的可利用性。以12份花椒材料DNA 为模板对64对SSR引物进行多态性引物及其最适 PCR 退火温度的筛选。其中,55对引物能扩增出理想产物,扩增产物大小符合预期;其中18对引物能扩增出多态性条带(表3),占可扩增出的34.55%,占设计引物总数的29.68%。18对引物共扩增出81条清晰可见的条带,其中多态性条带73条,平均多态率为90.12%,DNA片段大小为120~300 bp,平均每对引物的扩增条带数为4.5条。多态性引物所对应的重复基元结果表明:二核苷酸重复类型有1个,三核苷酸重复类型8个,四核苷酸重复类型有6个,五核苷酸重复类型有1个,六核苷酸重复类型有2个。说明利用花椒EST序列开发EST-SSR标记是高效可行的。

表 3 花椒 18 对 EST-SSR 引物信息Tab.3 Information of 18 pairs of EST-SSR primers from Zanthoxylum bungeanum Maxim
2.3花椒种质间遗传多样性分析
花椒的遗传相似系数结果表明:各花椒种质间遗传相似系数为0.552 6~0.894 7,平均为0.725 0。其中顶坛花椒与油椒、顶坛花椒与竹叶椒、无刺花椒与竹叶椒之间的遗传相似系数最小均为0.552 6,凤县大红袍与武都大红袍、山西盂县大红袍与黄金椒之间的遗传相似系数最大均为0.894 7。遗传相似系数能很好地体现了花椒种质之间的亲缘关系(表4),且12份花椒种质总的观测等位基因数(Na)、有效等位基因数(Ne)、Nei′s遗传多样性H以及Shannon′s信息指数(I)分别为1.763 2,1.399 3,0.240 1,0.367 9,结果表明花椒种质具有较强的遗传多样性[35]。

表4 12份花椒种质的遗传相似系数Tab.4 Genetic similarity coefficient of 12 Zanthoxylum varieties
用NTSYS-pc2.10软件UPGMA法得聚类分析树状图(图2)。由图2可知,在相似系数为0.70处,所有的花椒样品可以聚为3类:顶坛花椒为第Ⅰ类,竹叶椒为第Ⅱ类,其他花椒为第Ⅲ类。该结果与Feng等[8]报道的结果一致,说明所开发的花椒EST-SSR是高效可行的。

图2 基于SSR标记的12份花椒种质聚类图Fig.2 Cluster result of 12 Zanthoxylum varieties based on SSR markers
聚类分析的相关性检验结果见图3:相关性分析r=0.89,t=6.56,P=1.00,说明遗传是正相关,即遗传相似系数越大,亲缘关系越近,说明试验结果是可靠的。
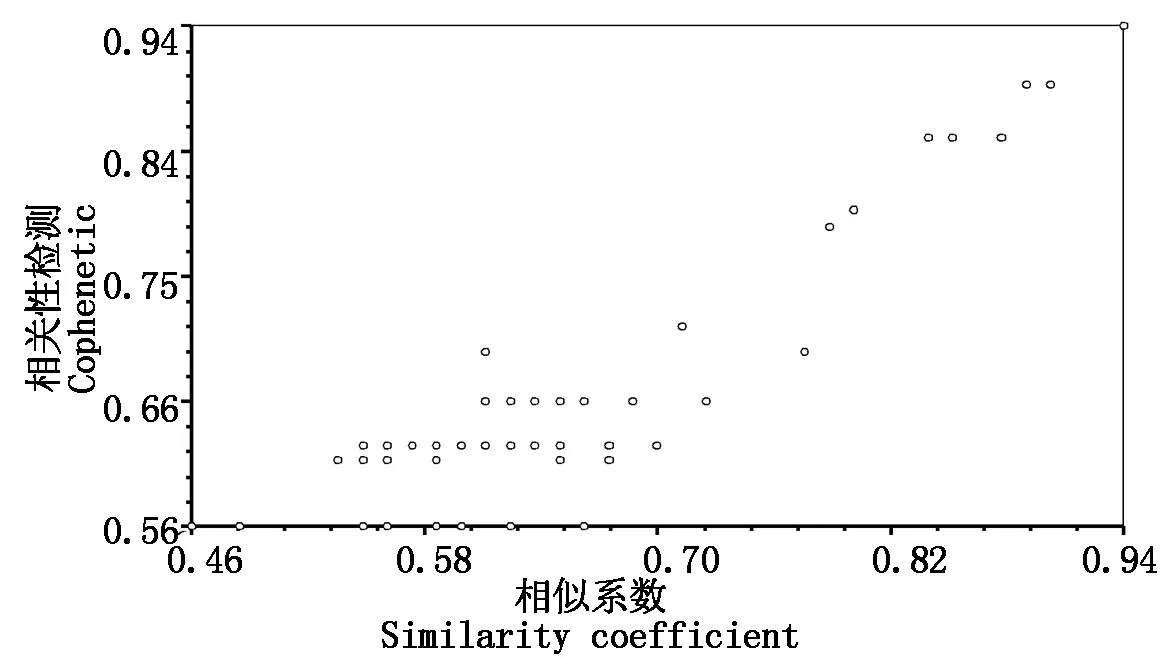
图3 聚类分析的相关性Fig.3 Correlation analysis of cluster analysis
2.4花椒指纹图谱分析
采用以上18对引物对12份花椒种质进行指纹分析,5份种质具有特征谱带(表5),仅用1个特征引物即可与其他种质区分开。其中,无刺花椒、凤县大红袍、汉源花椒具有 1 个特征引物,顶坛花椒具有2个特征引物,竹叶椒具有 4个特征引物。引物FX-27在顶坛花椒、凤县大红袍上均表现出特征谱带,表明该引物不仅多态性丰富,且特征谱带也多,在进行品种指纹鉴定时可优先采用(图4)。从18对引物中挑选多态性相对丰富的引物进行组合鉴别,选择FX-15、FX-27和FX-40以及FX-60、FX-40和FX-15 2组引物进行组合,根据特征引物对应特定种质所扩增出的特征条带的相对位置可以将12份花椒种质完全区分开。

表5 具有特征谱带的花椒种质及对应引物Tab.5 Corresponding primers with specific amplifications for Zanthoxylum varieties
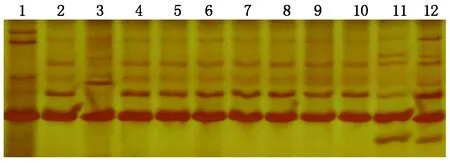
图4 引物 FX-27对 12 份花椒种质的扩增结果Fig. 4 DNA fragments amplified by SSR primer FX-27 in 12 Zanthoxylum varieties
3 讨论与结论
3.1花椒cDNA序列SSR位点特征分析
本试验所得花椒的SSR位点出现频率为7.07%,低于同科植物柑橘EST-SSRs出现频率(21.73%),高于枳壳EST-SSRs出现频率(2.96%),造成这种位点差异的原因:一可能由于基因组中转录部分的比例及低拷贝序列出现的频率不同、基因组大小不同、重复DNA序列所占的比例不同,二可能是由于搜索SSR位点的标准(SSR重复类型、长度等)不同等原因[36],如李小白[37]研究发现把油菜(Brassicanapus)EST-SSR 最小SSR长度标准由12 bp增加到20 bp,EST-SSR 的频率从15. 58%降低到11. 61%。
本研究发现,三核苷酸是SSR重复基元中的主导类型,且出现频率最多的是(GAA/CTT)n,这与对同科植物柑橘[38]、甜橙[39]中的研究结果一致。从目前报道的各类研究论文结果来看,大多数植物EST-SSRs的主导重复基元都是以二、三核苷酸为主,但主导重复基元的类型不同。以二核苷酸为主导重复基元的植物主要有猕猴桃[40]、茶树[41]、白菜等[42];以三核苷酸为主导重复基元的植物有水稻[43]、葡萄[44]、百合[45]等。这是因为EST序列大部分由外显子组成,是表达序列,且功能单位密码子是由3个核苷酸组成,在转录翻译成蛋白质的过程中会产生移码突变使三核苷酸发生位移,但是在基因表达的过程中不会影响表达基因的阅读框的位移[46]。在谷类作物的相关研究中已经验证了该理论[47],本研究与前人研究结果一致。
3.2花椒SSR引物多态性分析
18对多态性引物所对应的重复基元中,二核苷酸重复基元有1个,三核苷酸重复基元有8个,四核苷酸重复基元有6个,五核苷酸重复基元有1个,六核苷酸重复基元有2个。有研究发现,多态性与SSR重复基元的长度有关。由短重复基元组成的SSR获得(或失去)重复基元的速率要比长重复基元组成的SSR快,将具有更高的多态性[48],本试验结果与前人研究结果是一致的。早在2002年,Eujayl[49]通过对来自基因组和 EST 数据库的 SSR 多态性的比较,就发现了来自 EST 数据库的 SSR 多态性(25%)低于来自基因组的 SSR 多态性(53%),本试验获得了与Eujayl同样的结果。
从本试验的研究结果来看,花椒EST-SSRs出现频率较高,且类型丰富;所建立的cDNA数据库中的序列也具有较高的可用性,所设计引物的多态性较高而且可信。EST-SSR具有引物开发成本低、信息含量丰富、应用范围广泛等优点。本试验研究结果为花椒EST-SSR标记鉴定了基础,丰富了花椒分子标记类型,筛选出EST-SSR引物可用于花椒遗传多样性分析、遗传图谱构建等,为花椒的开发利用奠定基础。
3.3花椒遗传多样性分析
3.3.1 花椒相似性系数分析 相似系数也称为遗传一致度,是衡量品种之间遗传关系远近的一个重要参数,通过遗传相似系数能对个体或品种间亲缘关系远近有一个大概的了解。相似系数分析结果表明:不同地理来源的花椒遗传相似系数差异较大,为0.552 6~0.894 7,说明花椒种质之间的遗传多样性还是很丰富的,Nei′s遗传多样性H也支撑了这个结论,本试验材料遗传背景比较复杂,这可能是因为花椒无融合生殖、单性结实的特点造成的;其中顶坛花椒、竹叶椒与其他花椒之间的遗传相似系数小,即青花椒与红花椒之间遗传相似系数小。本试验结果与前人研究结果一致[50]。
3.3.2 花椒聚类结果分析 由基于SSR标记建立的不同来源的花椒种质树状聚类图可知:所有供试材料的遗传距离都大于零,又能够聚类在一起,说明不同花椒种质之间既有相同遗传背景但又相互之间存在着差异。树状图显示不同花椒种质的遗传距离为0.64~0.89,在遗传距离0.70处除顶坛花椒和竹叶椒之外,其他红花椒聚为一类,说明红花椒之间的亲缘关系较近。在聚类图上不同花椒种质相互交错排布,即来自同一省份的花椒与来自其他省份的花椒相互交织,花椒间的遗传距离与其生长的实际地理位置之间的关系不明显,是因为不同省份之间由于长期的引种过程造成品种的变异,所以出现这种结果。
3.4花椒指纹图谱分析
DNA 指纹图谱分析主要有 3 种方法,即引物组合法、特征谱带法与核心引物组合法[51]。本研究中,8对引物在5份花椒种质上具有特征谱带,仅用1对特征引物即可鉴别相应的种质,在进行DNA指纹检测时,使用起来简单方便。本研究中,只有少部分种质具有特征引物,需进行大量的引物筛选工作获取其他种质的特征引物,随着种质范围的扩大,原来在某种质上表现为特征带的引物,有可能在其他种质上出现相同带型,即所谓特征谱带是相对的,只有在固定的材料范围内有效,鉴别能力相对有限。引物组合法通过不同引物的有限组合,可以在很大程度上提高引物的鉴别能力,本研究采用3对引物组合进行鉴别可将12份花椒完全区分开,随着种质资源数量的进一步增加,这3对引物组合的鉴别能力可能会逐渐减低,可根据实际检测情况适当增加引物组合的数量。
3.5结论
在花椒cDNA全部非冗余序列中共检测到3 814个SSR位点。在花椒SSR位点中,二、三核苷酸重复类型占主导地位,其中:TC/AG和CT/GA基序在二核苷酸重复基元中所占比例最高,GAA/CTT基序在三核苷酸重复基元中出现频率最高;不同SSR重复基元的重复次数在4~12次,随着重复次数的增加,二、三、四、五核苷酸重复基元的数量和重复基元总数均呈明显下降趋势;利用花椒 EST 序列开发 EST-SSR 标记高效与可行;指纹图谱分析中,8对引物在5份种质中能扩增出特征带型,最少用3对引物进行组合即可将12份花椒种质区分开。本研究成功开发花椒EST-SSR标记,并应用于花椒遗传多样性研究和指纹图谱构建中,表明利用花椒转录组数据开发SSR标记是可行的,本研究开发的引物将为花椒遗传多样性分析、育种群体的建立、分子标记辅助育种等奠定基础。
[1] 毕 君,王春荣,赵京献,等. 北方花椒主产区种质资源考察报告[J]. 河北林果研究,2003,18(2):165-168.
[2] 王双贵,赵京献,毕 君,等. 国内外花椒的研究现状及其发展趋势[J]. 内蒙古林业科技,2003(2):32-34.
[3] 白小鸣,王 华,曾小峰,等. 气相色谱-质谱法结合保留指数对比初步分析梁平柚麻味物质的组成成分[J]. 食品科学,2015,18(36):103-107.
[4] 杨 静,赵 镭,史波林,等. 青花椒香气快速气相电子鼻响应特征及GC-MS物质基础分析[J]. 食品科学,2015,36(22):69-74.
[5] 游玉明,任文瑾,刘庆庆,等. 花椒精有效成分对高脂膳食大鼠脂质代谢的影响[J]. 营养学报,2015(3):288-293.
[6] 郑海星,李周岐,薛惠丹,等. 花椒种质资源的RAPD分析[J]. 西北林学院学报,2011,26(2):96-100.
[7] 汉素珍,王有科,李 捷,等. 甘肃省主产花椒品种ISSR遗传多样性分析[J]. 甘肃农业大学学报,2011,46(6):46-51.
[8] Feng S J,Yang T X,Liu Z S,et al. Genetic diversity and relationships of wild and cultivatedZanthoxylumgermplasms based on sequence-related amplified polymorphism (SRAP) markers[J]. Genetic Resources and Crop Evolution,2015,62(8):1193-1204.
[9] 蒋弘刚,魏安智,杨途熙,等. 花椒茎尖节点转录组测序及基因注释[J]. 西北林学院学报,2014,29(6):94-99.
[10] 潘海涛,汪俊君,王盈盈,等. 小麦EST-SSR标记的开发和遗传作图[J]. 中国农业科学,2010,43(3):452-461.
[11] 孟清照,李仕金,董转年,等. SSR引物开发方法概述[J]. 大众科技,2007(12):116-117,107.
[12] 杨东娟,马瑞君. SSR分子标记在作物遗传多样性研究中的应用现状[J]. 甘肃科技,2007,23(8):99-102.
[13] Zhuang L F,Song L X,Feng Y G,et al. Development and chromosome mapping of new wheat EST-SSR markers and application for characterizing rye chromosomes added in wheat[J]. Acta Agronomica Sinica,2008,34(6):926-933.
[14] Chai L J,Biswas M K,Yi H L,et al. Transferability,polymorphism and effectiveness for genetic mapping of the Pummelo (CitrusgrandisOsbeck) EST-SSR markers[J]. Scientia Horticulturae,2013,155(155):85-91.
[15] 曾柏全,邓子牛,熊兴耀,等. 湖南宽皮柑橘EST-SSR反应体系研究[J]. 中国农学通报,2009,25(21):244-247.
[16] 杨春霞,温 强,叶金山,等. 枳壳EST-SSR标记的开发[J]. 分子植物育种,2011(1):123-127.
[17] Yao L H,Zheng X Y,Cai D Y,et al. Exploitation ofMalusEST-SSRs and the utility in evalution of genetic diversity inMalusandPyrus[J].Genetic Resources and Crop Evolution,2010,57(6):841-851.
[18] 张俊娥. 苹果EST中微卫星分析[J]. 广西农业生物科学,2008,27(4):378-380.
[19] Celton J M,Tustin D S,Chagné D,et al. Construction of a dense genetic linkage map for apple rootstocks using SSRs developed fromMalusESTs andPyrusgenomic sequences[J]. Tree Genetics & Genomes,2009,5(1):93-107.
[20] 许玉兰,蔡年辉,康向阳,等. EST-SSR标记的开发及其在木本植物中的分布特点[J]. 中国农学通报,2012,28(4):1-7.
[21] 李 晓,杨途熙,魏安智,等. 花椒DNA提取方法的研究[J]. 北方园艺,2011(7):130-132.
[22] Mortazavi A,Williams B A,Mccue K,et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods,2008,5(7):621-628.
[23] 刘 博,邵艳卿,滕爽爽,等. 缢蛏(Sinonovaculaconstricta)EST-SSR分布特征及引物开发利用[J]. 海洋与湖沼,2012,43(1):132-137.
[24] 曾庆国,陈艺燕. 微卫星位点筛选方法综述[J]. 生态科学,2005,24(4):368-372.
[25] 崔海荣,刘金义,佟兆国,等. 砂梨EST-SSR引物开发及其应用[J]. 西北植物学报,2010,30(8):1551-1556.
[26] Rozen S,Skaletsky H. Primer3 on the WWW for general users and for biologist programmers[J]. Methods in Molecular Biology,2000,132(3):365-386.
[27] Rohlf F J. NTSYSpc:numerical taxonomy and multivariate analysis system,version2.02.[M].New York:Exeter software,1998:2010-2015.
[28] Yeh F C,Yang R C,Boyle T B,et al. POPGENE,the user-friendly shareware for population genetic analysis[M]. Edmonton:University of Alberta,1997.
[29] Cardle L,Ramsay L,Milbourne D,et al. Computational and experimental characterization of physically clustered simple sequence repeats in plants[J]. Genetics,2000,156(2):847-854.
[30] 黄海燕,杜红岩,乌云塔娜,等. 基于杜仲转录组序列的SSR分子标记的开发[J]. 林业科学,2013,49(5):176-181.
[31] 樊洪泓,李廷春,李正鹏,等. 银杏EST序列中微卫星的分布特征[J]. 基因组学与应用生物学,2009,28(5):869-873.
[32] Chen C,Zhou P,Choi Y A,et al. Mining and characterizing microsatellites fromCitrusESTs[J]. Theoretical and Applied Genetics,2006,112(7):1248-1257.
[33] 杨 华,陈 琪,韦朝领,等. 茶树转录组中SSR位点的信息分析[J]. 安徽农业大学学报,2011,38(6):882-886.
[34] 安泽伟,赵彦宏,程 汉,等. 橡胶树EST-SSR标记的开发与应用[J]. 遗传,2009,31(3):311-319.
[35] 吴岐奎,邢世岩,王 萱,等. 核用银杏品种遗传关系的AFLP分析[J]. 园艺学报,2015,42(5):961-968.
[36] Morgante M,Hanafey M,Powell W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes[J]. Nature Genetics,2002,30(2):194-200.
[37] 李小白. 油菜(BrassicanapusL.)EST-SSR标记的开发及应用研究[D]. 杭州:浙江大学,2007.
[38] Zhuang L. Development and chromosome mapping of 81 new wheat EST-SSR markers and application for characterizing rye chromosomes added in wheat[J]. Acta Agronomica Sinica,2008,34(6):926-933.
[39] Chen C A. Mining and characterizing microsatellites fromcitrusESTs[J].Theoretica and Applied Genetics,2006,7(112):1248-1257.
[40] Fraser L G,Harvey G H,Crowhurst R N. et al. EST-derived microsatellites fromActinidiaspecies and their potential for mapping [J].Theoretical and Applied Genetics,2004,108(6):1010-1016.
[41] 王丽鸳,姜燕华,段云裳,等. 茶树EST-SSRs分布特征及引物开发[J]. 植物遗传资源学报,2009,10(4):511-516.
[42] 忻 雅,崔海瑞,张明龙,等. 白菜EST-SSR标记的通用性[J]. 细胞生物学杂志,2006,28(2):248-252.
[43] Kantetyr V,Larotam L,Matthewsd E,et al. Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat[J]. Plant Molecular Biology,2002,48(5-6):501-510.
[44] Cordeiro G M,Casu R,Mcintyre C L,et al. Microsatellite markers from sugarcane (Saccharumspp.) ESTs cross transferable to erianthus and sorghum[J]. Plant Science,2001,160(6):1115-1123.
[45] 杨素丽,明 军,刘 春,等. 基于EST信息的百合SSR标记的建立[J]. 园艺学报,2008,35(7):1069-1074.
[46] Metzgar D,Bytof J,Wills C. Selection against frameshift mutations limits microsatellite expansion in coding DNA[J]. Genome Research,2000,10(1):72-80.
[47] Gao L F,Jing R L,Huo N X,et al. One hundred and one new microsatellite loci derived from ESTs (EST-SSRs) in bread wheat[J]. Theoretical and Applied Genetics,2004,108(7):1392-1400.
[48] 李淑娴,张新叶,王英亚,等. 桉树EST序列中微卫星含量及相关特征[J]. 植物学报,2010,45(3):363-371.
[49] Eujayl I,Sorrells M E,Baum M,et al. Isolation of EST-derived microsatellite marker for genotying the A and B genomes of wheat[J].Theor Appl Genet,2002,104(2-3):399-407.
[50] Thiel T,Michalek W,Varshney R K,et al. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (HordeumvulgareL.[J]. Theoretical and Applied Genetics,2003,106(3):411-422.
[51] 王凤格,赵久然,郭景伦,等. 比较三种DNA指纹分析方法在玉米品种纯度及真伪鉴定中的应用[J]. 分子植物育种,2003,1(5):655-661.
StudyonDevelopmentofSSRMolecularMarkersBasedonTranscriptomeSequencingandGermplasmIdentificationinZanthoxylumGermplasm
LI Lixin1,SI Shouxia2,WEI Anzhi1,LIU Yulin1,FENG Shijing1,YANG Tuxi1
(1.Northwest Agriculture and Forestry University,Yangling 712100,China;2.Henan Forestry Vocational College,Luoyang 471002,China)
To improve the application of molecular markers in theZanthoxylumgenus,the stuty developed functional EST-SSR markers and analyzed DNA fingerprint ofZanthoxylumgermplasm.Microsatellite software was used to scan the SSR loci from 45 057 non-redundant Unigenes among length above 200 bp derived from the node tissue of stem tip transcriptome sequences ofZanthoxylumbungeanumMaxim Fengxiandahongpao. Then the frequency and density of SSR loci,the type and proportion of SSR motifs and the number of SSR repetitions were also analysed. The SSR primers were then designed by Primer 3.0 online and polymorphic primer were screened by PCR;the polymorphic bands and molecular size were evaluated by using Quantity One software;the genetic distance and clustering map were analyzed by using software NTsys 2.0 and then constructed Fingerprints by Quantity One. The results showed that 3 315 Unigene sequences contained a total of 3 814 SSR loci(7.07%) and the dinucleotide repeat and trinucleotide repeat were the main types and accounted for 29.42% and 58.58% of the total SSRs,respectively. Among dinucleotides,AG/TC and CT/GA were the most frequent repeats;among trinucleotides,GAA/CTT and AGA/TCT appeared high frequency;with the increased of the number of repetition,the total number of repeat motifs showed a clear downward trend. 64 pairs of SSR primers were designed and 55 primer pairs were successfully amplifying DNA fragments. Out of 55 primer pairs,18 pairs of polymorphic primer were used for PCR amplification in 12Zanthoxylumgermplasm. A total of 81 clear bands were amplified and the percentage of polymorphic bands was 90.12%.Genetic similarity coefficients of eachZanthoxylumgermplasm among 0.552 6-0.894 7,with an average of 0.725 0. The UPGMA clustering showed that all theZanthoxylumgermplasm was divided into three main groups at the similarity coefficient 0.70:(Ⅰ)ZanthoxylumarmatumDC,(Ⅱ)ZanthoxylumbungeanumMaxim,and (Ⅲ)Zanthoxylumplanispinumvar. dingtanensis;In the fingerprint analysis,8 pairs of primers could amplify characteristic bands on 5Zanthoxylumgermplasms and 3 pairs of primers could be used to separate the 12Zanthoxylumgermplasm at least. We successfully developed SSR markers in the node tissue of stem tip transcriptome sequences ofZanthoxylumbungeanumMaxim Fengxiandahongpao. 8 pairs of primers could amplify characteristic bands were designed and screened,a minimum of 3 pairs of primers could be used to separate 12 pepper cultivars,these new EST-SSR markers fromZanthoxylumgermplasm provided a new primer sequence,basis for genetic analysis and fingerprint construction ofZanthoxylumgermplasm.
ZanthoxylumbungeanumMaxim;EST-SSR;Primer development;Fingerprint
2017-08-16
国家林业局林业公益性行业科研专项(201304706);西北农林科技大学产业技术集成与示范推广(TGZX 2016-08);西北农林科技大学试验示范站(基地)科技成果推广专项(TGZX2015-46)
李立新(1990-),女,河北沧州人,在读硕士,主要从事林木遗传育种研究。
杨途熙(1963-),男,陕西杨凌人,教授,硕士,主要从事林木遗传育种研究。
Q78;S573.03
A
1000-7091(2017)05-0069-09
10.7668/hbnxb.2017.05.011

