马铃薯扩展蛋白基因家族的鉴定与表达分析
理向阳,代丹丹,杨铁钢,郝 西
(河南省农业科学院 经济作物研究所,河南 郑州 450002)
马铃薯扩展蛋白基因家族的鉴定与表达分析
理向阳,代丹丹,杨铁钢,郝 西
(河南省农业科学院 经济作物研究所,河南 郑州 450002)
为了分析马铃薯扩展蛋白基因家族的进化与功能,鉴定了马铃薯的扩展蛋白基因家族,分析了其基因结构特征、系统发育及表达模式等。结果表明,马铃薯基因组包含33个扩展蛋白基因,包括A (21)、B (5)、LA (1)和LB (6) 4个亚家族,分布在马铃薯11条染色体上。扩展蛋白氨基酸长度为194~488 aa,编码蛋白质具有保守的结构域,蛋白质亚细胞定位预测结果表明所有蛋白均定位于细胞外。基因结构分析表明,马铃薯扩展蛋白家族有23个基因(69.7%)含有2~3个内含子。基因表达分析发现,该家族基因在马铃薯根、茎、匍匐茎、果实及块茎发育过程中表达丰度较高,并且其表达受到高温、盐、激素及病害等逆境的调控。
马铃薯;扩展蛋白;生物信息学;基因家族
扩展蛋白(Expansins)是植物细胞壁的重要组成部分,通过引起细胞壁组分间的松驰和细胞壁柔韧性增加,参与植物的许多生长发育过程[1]。1992年,Mcqueen-Mason等[2]首次从黄瓜胚轴中分离出扩展蛋白。随着植物基因组测序工作的开展,燕麦[3]、番茄[4]、玉米[5]、大豆[6]和黄瓜[7]等物种的扩展蛋白基因已经完成了全基因组鉴定。按照进化关系,扩展蛋白分为4个亚家族:EXPA、EXPB、EXLA和EXLB (也称α、β、γ和δ亚家族)[8]。扩展蛋白的长度一般为205~275个氨基酸,大多数在氨基末端含有一个信号肽,长度为20~30个氨基酸[9]。研究表明,扩展蛋白在植物的多个生理过程中发挥重要功能,比如水果成熟软化[10]、木质部形成[11]、叶片脱落[12]、种子发芽[13]和花粉管伸长[14-15]、根毛的起始和伸长[16-17]、棉花纤维发育[18-19]、抗逆[20-21]等,扩展蛋白基因的进化与功能研究已成为国内外的研究热点之一。
马铃薯(SolanumtuberosumL.)是世界上最重要的非谷物类粮食作物,据统计2009年全球马铃薯产量约为3.3亿t[22]。其中我国马铃薯种植面积2009年达到508.30万hm2,总产量为7 328.19万t,种植面积和总产量均居世界第1位,种植范围遍及全国[23]。马铃薯基因组测序计划的完成[24],为深入研究该物种扩展蛋白家族的进化和其他特征提供了充足的数据资源。本研究依据已发表的马铃薯全基因组测序数据,鉴定马铃薯扩展蛋白基因家族成员33个,并分析了其染色体分布、基因结构、组织表达模式等生物学信息,以期为进一步开展该基因家族的功能和进化研究提供参考。
1 材料和方法
1.1马铃薯expansin基因家族的鉴定
拟南芥和水稻expansin家族的基因和蛋白序列分别从TAIR网站(http://arabidopsis.org/)和RGP网站(http://rapdb.dna.affrc.go.jp)下载。利用下载的拟南芥和水稻expansin蛋白序列,分别比对PHYTOZOME v11 database (www.phytozome.net/)中的马铃薯预测(Solanumtuberosumv3.4),满足E-value≤-10的序列被作为候选序列;最后利用Smart(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和Pfam(http://pfam.xfam.org/search)网站预测这些候选序列是否同时存在PF03330和PF01357结构域。番茄expansin家族基因序列从PHYTOZOME v11 database (www.phytozome.net)下载。
1.2马铃薯expansin基因家族蛋白特征分析
利用ProtParam tool在线工具(http://web.expasy.org/protparam)分析每个马铃薯扩展蛋白的分子量、等电点等参数;分别利用CELLO v2.5 server (http://cello.life.nctu.edu.tw)和Euk-mPLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/euk/multi-2/#)进行蛋白的亚细胞定位预测。
1.3马铃薯expansin基因家族的染色体定位与基因结构分析
从PHYTOZOME数据库中获得33个马铃薯扩展蛋白基因的注释信息,进行染色体定位;利用Gene Structure Display Server在线工具(http://gsds.cbi.pku.edu.cn/)分析每个基因的结构。
1.4马铃薯expansin基因的保守motif分析
利用MEME(multiple expectation maximization for motif elicitation)在线分析工具(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)预测马铃薯expansin的保守motif。利用此软件分析时,最大模体数量(Maximum number of motifs)设为10,其他参数默认。
1.5马铃薯expansin家族基因的进化分析
首先利用Clustal X2程序进行多序列比对分析,再使用MEGA 5.1软件对序列比对结果生成系统发育树(Bootstrap值设置为1 000,其他参数为默认值)。
1.6马铃薯expansin基因家族的表达模式分析
利用已发表的马铃薯转录组测序数据分析基因的表达特征[24]。该转录数据包含不同组织及生物和非生物胁迫下的基因表达数据。转录本数据通过取对数(log2)转换,利用Genesis软件作热图。
2 结果与分析
2.1马铃薯扩展蛋白基因家族的筛选
通过Blastp比对,从马铃薯基因组数据库中共获得77个扩展蛋白候选基因,进一步通过Pfam和Smart在线软件剔除不含扩展蛋白保守结构域的序列,最终鉴定出33个马铃薯扩展蛋白基因(表1)。33个马铃薯扩展蛋白基因分为EXPA、EXPB、EXLA和EXLB 4个亚家族,其中EXPA亚家族数目最多为21个,EXPB、EXLA和EXLB 3个亚家族基因数目分别为5,1,6个。马铃薯扩展蛋白基因数目相比拟南芥少2个,分别是拟南芥(35)、水稻(56)、大豆(49)、苹果(34)、菜豆(25)的0.94,0.59,0.67,0.97,1.32倍(表2)。马铃薯与其近缘物种番茄相比总扩展蛋白基因数目少5个,二者EXLA亚家族基因数目均为1个。
马铃薯33个扩展蛋白基因的蛋白分子质量从20.75 kDa到54.49 kDa不等,等电点最小值为4.68,最大为9.87;其蛋白氨基酸残基的长度为194~488,平均值为272,跨度较大。其中19个(占57.6%)蛋白序列中含有长度为17~26个氨基酸的信号肽。
分别利用CELLO v2.5 server和Euk-mPLoc localization在线工具进行扩展蛋白的亚细胞定位预测。根据Euk-mPLoc软件预测,33个马铃薯扩展蛋白均定位于细胞外;从CELLO v2.5 server软件分析结果看,30个蛋白(占90.9%)定位于细胞外,2个蛋白(StEXPA4和StEXPB4)同时定位于细胞外和质膜,1个蛋白(StEXLB1)同时定位于细胞质、线粒体和细胞核。

表1 马铃薯扩展蛋白基因家族基本信息Tab.1 Identification of expansin gene family members from potato

表2 不同植物扩展蛋白亚家族数目分析Tab.2 Sizes of the four expansin subfamilies in different plants
2.2马铃薯扩展蛋白基因家族的染色体定位
利用扩展蛋白基因在染色体上的物理位置信息,分别把33个马铃薯扩展蛋白基因定位于11条染色体上(第11号染色体除外),并且33个基因在11条染色体上的分布是不均衡的(图1);其中8号染色体含有最多的基因(7个),3,6号染色体含4个基因,1,7,10号染色体包含3个基因,2,5,9,12号染色体含2个基因,4号染色体含1个基因,11号染色体上不含基因。与近缘物种番茄相比,马铃薯和番茄均是11号染色体上不含扩展蛋白基因;所不同的是,番茄3号染色体含有最多的基因(9个),而马铃薯则是8号染色体含有最多的扩展蛋白基因(7个)。

图1 马铃薯扩展蛋白基因的染色体定位Fig.1 Chromosome distribution of potato expansin genes
2.3马铃薯扩展蛋白基因结构分析
通过马铃薯扩展蛋白基因的内含子外显子结构分析,发现其含有的内含子较少(表1、图2),平均为2.4个;其中6个基因(18.2%)含有1个内含子,23个基因(69.7%)含有2~3个内含子,4个基因(12.1%)含有3个以上的内含子。马铃薯扩展蛋白4个亚家族的平均内含子数目分别为A类1.8个,B类3.6个,LA类4.0个和LB类3.3个。

图2 马铃薯扩展蛋白基因的进化与结构分析Fig.2 Phylogenetic and structural analysis of expansin genes of potato
通过MEM软件在线分析,发现每个马铃薯扩展蛋白基因含有4~9个motif。4个亚家族均含有motif 3和motif 6;除了StEXPB4外,EXPA、EXPB 2个亚家族均含有motif 1;除了StEXPA1外,EXPA亚家族均含有motif 2、motif 4和motif 7。除了StEXPB5,EXPB、EXLA和EXLB亚家族均含有motif 8和motif 9。EXLA和EXLB亚家族均含有motif 10。
从图2可以看出,同一亚家族内的扩展蛋白基因含有保守的内含子外显子结构和motif特征,而不同亚家族之间则差异较大。
2.4马铃薯扩展蛋白家族的系统发育树分析
为了分析33个马铃薯扩展蛋白家族成员之间的进化关系,利用Mega 5程序构建了该家族蛋白的系统发育树。结果表明,33个扩展蛋白被划分为EXPA、EXPB、EXLA、EXLB 4个亚家族(图2)。从系统发育树共发现12个同源基因对,其中9个同源基因对的步长值90%,8个同源基因对的步长值为100%;在12个同源基因对中,5个姐妹基因对间有80%~86%的蛋白序列相似性,4个姐妹基因对具有75%~79%的蛋白序列相似性,2个姐妹基因对具有71%~73%的蛋白序列相似性,1个姐妹基因对具有53%的蛋白序列相似性。
马铃薯与番茄同属茄科,亲缘关系较近,为研究二者扩展蛋白家族之间的进化关系,利用33个马铃薯扩展蛋白和38个番茄扩展蛋白的氨基酸序列构建了系统发育树(图3)。从系统发育树可知,71个扩展蛋白基因被明显分为4个亚家族,并且具有较高的相似性。共鉴定物种间的直系同源蛋白29对,其中有27对直系同源蛋白间的步长值为100%;而物种内的旁系同源蛋白数目为0。
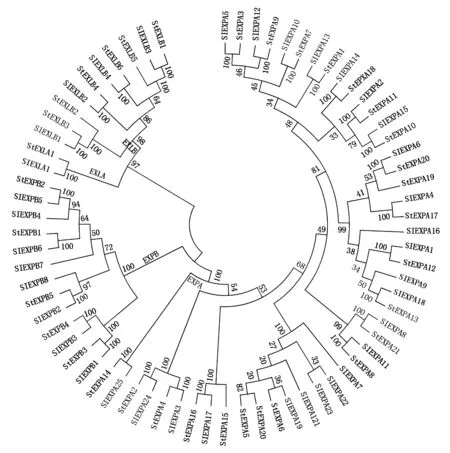
图3 马铃薯与番茄的系统发育树分析Fig.3 The phylogenetic tree of potato and tomato expansin family
2.5马铃薯扩展蛋白家族成员表达模式
下载已发表的马铃薯15个组织的转录组数据[24],找到33个扩展蛋白基因对应转录本的RPKM值,先进行对数值转换(log2),然后利用Genesis软件作热图(图4-A)。图中深灰色代表基因表达丰度较高,浅灰色代表基因表达水平较低。由图4-A可知,有11个扩展蛋白基因在15个组织中都表达(占33.3%);19个基因在10种以上组织表达(占57.6%);有14个基因在10种以下组织中表达(占42.4%),未发现具有组织特异性表达特点的扩展蛋白基因。此外,StEXPA14、StEXPA17和StEXLA1这3个基因在所有的15个组织均有较高的表达量,StEXPA19和StEXPA21 2个基因在所有的15个组织表达量均很低;共有23个基因在块茎中表达,其中5个基因(StEXPA9、StEXPA12、StEXPA14、StEXLA1和StEXLB6)在块茎中的表达量非常高。

A.在不同组织或器官中的表达;B.不同激素或胁迫条件下的诱导表达情况。A.Expression within different tissues or organs; B.Induced expression by different plant hormones or stress.
33个马铃薯扩展蛋白基因在35 ℃高温、甘露醇(260 μmol/L)、盐(150 μmol/L NaCl)、激素诱导(50 μmol/L 赤霉素、10 μmol/L生长素、50 μmol/L脱落酸和10 μmol/L细胞分裂素)及生物胁迫(初生组织损伤、次生组织损伤、晚疫病菌侵染、2 mg/mL β-氨基丁酸、100 mg/mL二叔丁对甲酚)条件下的表达如图4-B所示。在盐和甘露醇胁迫诱导下,StEXPA10均显著上调表达;高温胁迫下,StEXPA15和StEXLB5显著上调表达;在高温和脱落酸胁迫下,StEXLB5均显著上调表达,而StEXPA8和StEXPA11均显著下调表达;在脱落酸、生长素和赤霉素诱导下,StEXLB1均表现为显著上调表达。在BAP诱导下,除StEXPA10、StEXPA16和StEXPB5这3个基因上调外,其余基因的表达多表现为下调。StEXPA1和StEXPB2在所有胁迫条件下RPKM值均为0。在脱落酸诱导下,除StEXLB3基因下调外,其余5个EXLB亚家族基因的表达一致表现为显著上调;StEXLB3和StEXLB4的表达同时受到细胞分裂素、初生组织损伤、次生组织损伤3种胁迫诱导的显著抑制;在初生组织损伤和次生组织损伤2种条件下,多数基因均上调表达,其中StEXPA3、StEXPA7、StEXPA8、StEXPA9和StEXPA11均显著受到初生组织损伤和次生组织损伤2种胁迫诱导条件的显著诱导上调表达;在初生组织损伤诱导下,除StEXLB4基因外,其余5个EXLB亚家族基因的表达一致,表现为显著下调;在晚疫病菌侵染侵染胁迫下,StEXLB2和StEXLB5均表现为显著下调表达。
3 结论与讨论
扩展蛋白广泛存在于植物基因组中,主要参与细胞扩张和一系列发生细胞壁修饰的发育过程。本研究共鉴定出马铃薯扩展蛋白家族基因33个,包含4个亚家族;不同亚家族之间在基因内含子外显子结构、motif模式等方面具有一定的保守性,代表了亚家族的重要特征。23个扩展蛋白基因(69.7%)含有2~3个内含子;EXPA、EXPB、EXLA和EXLB 4个亚家族均含有motif 3和motif 6;除了StEXPB5,EXPB、EXLA和EXLB亚家族均含有motif 8和motif 9。相比EXPA和EXPB,EXLA和EXLB 2个亚家族特异含有motif 10。同一亚家族内的扩展蛋白基因含有保守的内含子与外显子结构和motif特征,而不同亚家族之间motif的多样化也表明了马铃薯扩展蛋白基因广泛参与细胞内的多种代谢途径和反应。
扩展蛋白在马铃薯许多生长发育过程中发挥重要功能。通过分析已发表的马铃薯公共转录组数据,发现19个StExp基因在10种以上组织表达(占57.6%),未发现组织特异性表达的StExp基因;共有23个基因在块茎中表达(占69.7%),其中5个基因(StEXPA9、StEXPA12、StEXPA14、StEXLA1和StEXLB6)在块茎中的表达量非常高。由于扩展蛋白基因与马铃薯块茎膨大过程关系密切[25-26],今后可以通过构建特异的突变体或转基因等手段来研究扩展蛋白基因的功能,并探讨通过转基因技术调控马铃薯块茎膨大的可行性。
激素和胁迫处理的转录数据分析表明,94%的马铃薯扩展蛋白基因(31/33)都受到一种或多种胁迫条件的显著诱导或抑制。StEXPA8和StEXPA11的表达受到初生组织损伤、次生组织损伤和二叔丁对甲酚的显著诱导,但在高温、脱落酸、细胞分裂素和β-氨基丁酸处理下均明显下调;StEXLB1的表达受到脱落酸、生长素和赤霉素3种激素的显著诱导,但在初生组织和次生组织损伤、晚期病菌侵染和β-氨基丁酸处理条件下均表现为下调;StEXLB6的表达受到脱落酸和β-氨基丁酸的显著诱导,但在高温、初生组织损伤和二叔丁对甲酚处理条件下均表现为下调。这些结果表明马铃薯扩展蛋白基因在激素和逆境胁迫响应过程中具有重要功能。
植物扩展蛋白基因功能已成为近年来的研究热点之一,但是有关马铃薯扩展蛋白基因的全基因组鉴定尚未见报道。本研究基于马铃薯全基因组测序数据,鉴定了33个扩展蛋白基因,并对其基因结构、进化与表达模式等进行了初步分析,将为开展该基因家族的深入研究提供参考。
[1] Cosgrove D J,Li L C,Cho H T,et al. The growing world of expansins[J]. Plant and Cell Physiology,2002,43(12):1436-1444.
[2] Mcqueen-Mason S,Durachko D M,Cosgrove D J. Two endogenous proteins that induce cell wall extension in plants[J]. The Plant Cell,1992,4(11):1425-1433.
[3] Li Z C,Durachko D M,Cosgrove D J. An oat coleoptile wall protein that induces wall extension in vitro and that is antigenically related to a similar protein from cucumber hypocotyls[J]. Planta,1993,191(3):349-356.
[4] Keller E,Cosgrove D J. Expansins in growing tomato leaves[J]. Plant Journal,1995,8(6):795-802.
[5] Wu Y,Sharp R E,Durachko D M,et al. Growth maintenance of the maize primary root at low water potentials involves increases in cell-wall extension properties,expansin activity,and wall susceptibility to expansins[J]. Plant Physiology,1996,111(3):765-772.
[6] Zhu Y,Wu N N,Song W L,et al. Soybean (glycine max) expansin gene superfamily origins:segmental and tandem duplication events followed by divergent selection among subfamilies[J]. BMC Plant Biology,2014,14:93.
[7] 郝 西,理向阳,腊贵晓,等. 黄瓜扩展蛋白基因家族的鉴定与生物信息学分析[J]. 分子植物育种,2015,13(10):2280-2289.
[8] Kende H,Bradford K,Brummell D,et al. Nomenclature for members of the expansin superfamily of genes and proteins[J]. Plant Molecular Biology,2004,55(3):311-314.
[9] Sampedro J,Cosgrove D J. The expansin superfamily[J]. Genome Biology,2005,6(12):12.
[10] Cosgrove D J. Loosening of plant cell walls by expansins[J]. Nature,2000,407(6802):321-326.
[11] Gray-Mitsumune M,Mellerowicz E J,Abe H,et al. Expansins abundantin secondary xylem belong to subgroup A of the α-expansin gene family[J]. Plant Physiology,2004,135(3):1552-1564.
[12] Belfield E J,Ruperti B,Roberts J A,et al. Changes in expansin activity and gene expression during ethylene-promoted leaflet abscission inSambucusnigra[J]. Journal of Experimental Botany,2005,56(413):817-823.
[13] Chen F,Bradford K J. Expression of an expansin is associated with endosperm weakening during tomato seed germination[J].Plant Physiol,2000,124(3):1265-1274.
[14] Cosgrove D J,Bedinger P,Durachko D M. Group Ⅰ allergens of grass pollen as cell wall-loosening agents[J]. Proc Natl Acad Sci,1997,94(12):6559-6564.
[15] Pezzotti M,Feron R,Mariani C. Pollination modulates expression of the PPAL gene,a pistil-specific β-expansin[J]. Plant Molecular Biology,2002,49(2):187-197.
[16] Yu Z M,Kang B,He X W,et al. Root hair-specific expansins modulate root hair elongation in rice[J]. Plant Journal,2011,66(5):725-734.
[17] Guo W B,Zhao J,Li X X,et al. A soybean beta-expansin geneGmEXPB2 intrinsically involved in root system architecture responses to abiotic stresses[J]. Plant Journal,2011,66(3):541-552.
[18] 李晓东,李 波,范 玲. 基因工程改良棉花纤维品质研究进展[J]. 生物技术通报,2012(10):8-14.
[19] An C F,Saha S,Jenkins J N,et al. Transcriptome profiling,sequence characterization,and SNP-based chromosomal assignment of the EXPANSIN genes in cotton[J]. Molecular Genetics and Genomics,2007,278(5):539-553.
[20] Han Y Y,Li A X,Li F,et al. Characterization of a wheat (TriticumaestivumL.) expansin gene,TaEXPB23,involved in the abiotic stress response and phytohormone regulation[J]. Plant Physiology and Biochemistry,2012,54:49-58.
[21] Zhao M R,Han Y Y,Feng Y N,et al. Expansins are involeved in cell growth mediated by abscisic acid and indole-3-acetic acid under drought stress in wheat[J]. Plant Cell Reports,2012,31(4):671-685.
[22] Florian J,Leighton P,Graham J E,et al. Identification and localisation of the NB-LRR gene family within the potato genome [J]. BMC Genomics,2012,13:75.
[23] 贾晶霞,杨德秋,李建东,等. 中国与世界马铃薯生产概况对比分析与研究[J]. 农业工程,2011,1(2):84-86.
[24] Xu X,Pan S,Cheng S,et al. Genome sequence and analysis of the tuber crop potato[J]. Nature,2011,475(7355):189-195.
[25] 毛彦芝. 番茄和马铃薯扩展蛋白研究进展[J]. 黑龙江八一农垦大学学报,2013,25(5):5-8,47.
[26] Jieun J,Erin M O D,Paul P D,et al. Expression of multiple expansin genes is associated with cell expansion in potato organs [J]. Plant Science,2010,179:77-85.
Genome-wideIdentificationandExpressionAnalysisoftheExpansinGeneFamilyinPotato
LI Xiangyang,DAI Dandan,YANG Tiegang,HAO Xi
(Industrial Crop Institute,Henan Academy of Agricultural Sciences,Zhengzhou 450002,China)
As an important component of plant cell wall,expansins have effects on loosening and increasing the flexibility of cell wall,and are thought to have function in many process of plant growth. To study potential function of expansin gene family in potato,we investigated expansin gene family with the whole genome database to analyse its structure,phylogenetic relationship and expression profiles. Potato genome contained 33 expansin genes which belonged to 4 subfamilies (EXPA (21),EXPB (5),EXLA (1) and EXLB (6)) located on 11 different chromosomes. Expansin proteins ranged from 194 aa to 488 aa in size and had conserved domains. All 33 Expansin proteins were predicted to localize in extracellular. Gene structure analysis revealed that seventy percent(23/33) expansin genes had 2-3 introns. Transcriptome analysis showed that the potato expansin genes expressed highly in root,stem,stolon,mature fruit and tuber.
Potato;Expansin;Bioinformatics;Gene family
2017-07-02
河南省农业科学院自主创新项目(2016ZZ20)
理向阳(1975-),男,河南西华人,助理研究员,主要从事作物生理与分子生物学研究。
郝 西(1979-),男,河南平顶山人,副研究员,博士,主要从事作物生理研究。
Q78
A
1000-7091(2017)05-0037-08
10.7668/hbnxb.2017.05.007

