基于NRPSs基因筛选与鉴定海芦笋内生细菌
李代婧,殷跃,刘梦婕,卫婷,郭佳,辛志宏
(南京农业大学 食品科技学院,江苏 南京,210095)
基于NRPSs基因筛选与鉴定海芦笋内生细菌
李代婧,殷跃,刘梦婕,卫婷,郭佳,辛志宏*
(南京农业大学 食品科技学院,江苏 南京,210095)
利用形态学观察和分子生物学相结合的方法分离鉴定海芦笋内生细菌,并筛选含有NRPS基因的菌株(nonribosomal peptide synthetase,NRPS),以期获得能够产生环肽化合物的菌株。研究结果表明,共从海芦笋中分离到6株内生细菌Srtli-F1~Srtli-F6,分别鉴定为绿针假单胞菌橙黄亚种(Pseudomonaschlororaphissubsp.aurantiaca)、组氨酸节杆菌(Arthrobacterhistidinolovorans)、庆盛芽孢杆菌(Bacillusqingshengii)、类芽孢杆菌羽扇豆根瘤菌(Paenibacilluslupini)、假蕈状芽孢杆菌(Bacilluspseudomycoides)、高山麝病原性阿尔伯蒂埃希氏杆菌(Escherichiaalbertii),其中菌株Srtli-F5检测到含有NRPS基因,经系统发育树分析,预测该菌株可能产生环肽类化合物,该株菌经过发酵,有机溶剂萃取发酵产物,并用电喷雾质谱ESI-MS (electrospray mass,ESI-MS)分析Srtli-F5能产生环肽化合物——杆菌肽,证实了该预测结果。该研究为定向筛选能够产生环肽类化合物的菌种开拓了新思路。
内生细菌;分离鉴定;系统发育分析;非核糖体多肽合成酶;质谱;环肽
盐生海芦笋(SalicorniabigeloviiTorr),是通常生长在含盐率高的湖泊及土壤附近[1]的绿色蔬菜,味道鲜美,质脆多汁。海芦笋有茎无叶,不生虫子,提示其组织内部或表面可能含有能产生抗菌活性物质的内生菌或附生菌[2]。目前,有关海芦笋的研究大多集中在对其化学成分的分离鉴定及其生物活性研究方面,有关海芦笋内生细菌的研究鲜有报道。
传统的植物内生细菌的筛选方法多是采用活性筛选、化学筛选或二者的组合[3],这些方法过去在筛选可以产生活性次级代谢产物的菌株研究过程中非常有效,但在研究不断深入的情况下,传统方法的弊端也日益明显:重复地发现已知的菌株和化合物,使得新菌株、新化合物的发现受阻,造成了时间和资源的极大浪费[4-5]。
近年来,随着测序技术和生物信息学的快速发展,许多微生物的生物合成机制逐步解密,新的植物内生菌筛选方法应运而生。非核糖体肽类化合物合成酶(nonribosomal peptide synthetase,NRPS)是生物合成次级代谢产物过程中的多功能关键酶,由催化氨基酸之间肽键形成的缩合(condensation,C)结构域、负责底物识别和活化的腺苷酰化(adenylation,A)结构域、硫醇(thiolation,T)结构域以及 C 末端终止延伸反应的硫酯酶(thioesterase,TE)结构域组成[6]。其中 A 结构域有很强的保守性,可以通过PCR扩增该区域基因,并通过系统发育分析预测化合物的结构类型。因此,以NRPS功能基因为靶点,定向筛选含有环肽化合物的菌株,成为从“基因-合成酶系-环肽”的筛选新方法。
本实验用采自新疆盐湖的野生海芦笋,采用平板划线法分离纯化培养得到内生细菌,提取其DNA,经PCR扩增16S rRNA目标序列后,建立系统发育进化树。通过观察菌落形态,并对菌株进行系统发育学分析,明确其种属地位。在此基础上,以NRPS(nonribosomal peptide synthetase)功能基因为指标,定向筛选含有NRPS基因的内生细菌,筛选到的菌株经发酵,ESI-MS(electrospray mass)分析发酵产物证实预测结果,以期为定向筛选能够产生环肽化合物的菌株提供新的思路。
1 材料与方法
1.1实验器材
盐生海芦笋,取自新疆盐湖。
Microfuge 22R台式微量冷冻离心机,美国Beckman公司;TP600型梯度PCR仪,日本TaKaRa公司;DYCP-31DN电泳仪,北京市六一仪器厂;JS-380C全自动数码凝胶成像分析仪,上海培清科技有限公司。
D3350-01 E.Z.N.A细菌DNA微量提取试剂盒,美国Omega公司;Cat#DP1502多功能DNA纯化回收试剂盒(进口离心柱型),南京华普生物科技有限公司。
本实验主要采用如下培养基对细菌进行初步的分离培养:
细菌用培养基:牛肉膏蛋白胨培养基(1 L):牛肉膏5.0 g、蛋白胨10.0 g、NaCl 5.0 g、去离子水1 000 mL、琼脂18 g,pH 7.2~7.4。
放线菌用培养基:高氏一号培养基(1 L)。可溶性淀粉20 g、KNO31.0 g、K2HPO40.5 g、MgSO4·7H2O 0.5 g、NaCl 0.5 g、FeSO4·7H2O 0.01 g、去离子水 1 000 mL、琼脂18 g。
真菌用培养基:孟加拉红培养基(1 L):葡萄糖10.0 g、1/3 000孟加拉红水溶液 100 mL、蛋白胨5.0 g、KH2PO41.0 g、琼脂20 g、MgSO4·7H2O 0.5 g、去离子水900 mL,氯霉素0.1 g。
1.2实验步骤
1.2.1 内生细菌的分离纯化
无菌条件下,先后将海芦笋浸在75%酒精、质量分数为1%的NaClO溶液、75% 酒精中浸泡1 min,然后用无菌水清洗3次,用灭菌后的吸水纸除去表面的水分。用灭菌后的剪刀将其剪成7~9 mm小段,分别放在上述3种培养基上,每板3~5段,其中牛肉膏蛋白胨培养基、高氏一号培养基于37 ℃恒温培养2 d至茎末端长出菌落,孟加拉红培养基于30 ℃恒温培养5 d直到茎末端长出菌落。用划线分离的方法纯化培养得到单菌落。
1.2.2 形态学观察
将长有细菌菌落的平板,置于37 ℃培养2 d,观察菌落的形态特征。
1.2.3 细菌DNA的提取
按照生产厂家提供的试剂盒说明书进行。
1.2.4 16SrRNA基因片段的PCR扩增
16S区域的扩增选择细菌16S rRNA的通用扩增引物16S(F)(5’-AGAGTTTGATCCTGGCTCAG-3’)/16S(R)(5’-GGTTACCTTGTTACGACTT-3’),PCR反应条件为:94 ℃预变性3 min,94 ℃变性30 s,45 ℃退火1 min,72 ℃延伸1 min 40 s,共35个循环,最后72 ℃延伸7 min[7]。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×TaqMaster Mix 12.5 μL、10 μmol/L Primer 16S(F) 1 μL、10 μmol/L Primer 16S(R) 1 μL、模板DNA 1 μL。使用1%琼脂糖凝胶电泳检测PCR扩增产物,4 ℃保存备用。
1.2.5 NRPS基因片段的PCR扩增
NRPS基因片段的扩增选择的引物为NP1(5’-CCTAATTCAATACGAAAACCACGAADYTTNAYYTG-3’)/NP2(5’-TGTATGTTATTTATACTTCTGGTTCTACTGGTMRNCCANARGG-3’),PCR反应条件为:94 ℃预变性5 min,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸10 min。PCR扩增反应采用25 μL的反应体系,包括ddH2O 9.5 μL、2×TaqMaster Mix 12.5 μL、10 μmol/L Primer NP1 1 μL、10 μmol/L Primer NP2 1 μL、模板DNA 1 μL。使用1%琼脂糖凝胶电泳检测PCR扩增产物,4 ℃保存备用。
1.2.6 PCR产物的回收和克隆
按照生产厂家提供的试剂盒说明书进行。
1.2.7 序列测定与系统发育学分析
将含有目的DNA序列的菌液交由上海美吉生物有限公司进行测序。获得序列后,通过Ezbiocloud(http://eztaxon-e.ezbiocloud.net/)网站与所有已发表的典型菌株进行序列相似性分析,下载与供试菌株序列同源性相近的菌株序列,利用MEGA 6.0软件NJ(neighbor joining)方法构建系统发育树,自展数:1 000。
1.2.8 质谱测定方法
将筛选得到的含有NRPS基因的菌株发酵7 d,丙酮提取发酵产物,经旋转蒸发器浓缩,过滤,进样分析。质谱参数:电喷雾离子源(ESI),离子源温度500 ℃,气帘气25.0 psi,喷雾电压5 500.0 V,离子源气1∶55.0 psi,离子源气2∶50.0 psi。
2 结果与分析
2.1菌落形态特征
挑取平板上长出的菌落,在对应的培养基平板上划线分离纯化,得到单个菌落,进行形态学观察。结果如图1所示。
高氏一号培养基平板划线纯化后得到3个纯的菌株Srtli-F1、Srtli-F2和Srtli-F4。Srtli-F1菌落较小而且扁平,呈浅黄色,在光照下显现出荧光,边缘规则,半透明,菌落湿润易挑起。菌株Srtli-F2的菌落呈黄色,大而隆起,黏稠,不透明,边缘不规则。菌株Srtli-F4为白色大菌落,微凸,边缘规则,半透明,湿润易被挑起。
经过孟加拉红培养基平板划线纯化后仅得到1个纯的菌株Srtli-F3。Srtli-F3菌落大且呈圆红色,微微凸起,黏稠,不透明。
经牛肉膏蛋白胨培养基平板划线纯化后得到2个纯的菌株Srtli-F5和Srtli-F6。Srtli-F5菌落微小,呈白色圆点状,边缘光滑,微微凸起、干燥、不透明,菌株Srtli-F6的菌落呈白色圆形,小而扁平,表面半透明,边缘光滑。
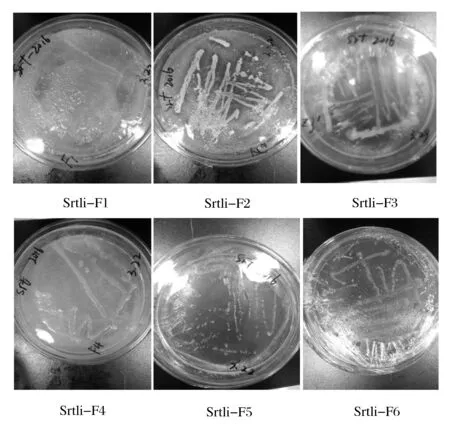
图1 内生细菌Srtli-F1~Srtli-F6的菌落形态特征Fig.1 The morphological characters of Srtli-F1-Srtli-F6
2.2系统发育学分析
经过PCR扩增、切胶回收测序,获得了菌株Srtli-F1~Srtli-F6的16S rDNA,序列长度均在1 500 bp左右。将菌株Srtli-F1的16S rDNA序列在EzTaxon数据库中进行同源性搜索,选取并下载同源性较高的模式菌种16S rDNA序列,利用MEGA 6.0软件构建系统发育树,如图2所示。从图2可见,与外组Aztobacterarmeniacus(AB175655)相比,同一属的菌株聚为一个大类,不同种的菌株聚在不同的分枝。菌株Srtli-F1与Pseudomonaschlororaphissubsp.aurantiacaT(DQ682655)聚为一枝,自展值为94,亲缘关系极其相近,可鉴定为该种。以相同的方法构建系统发育树对其他菌种进行鉴定。发现菌株Srtli-F2与ArthrobacterhistidinolovoransT(X83406)聚为一支,自展值为98(图3),菌株Srtli-F3与BacillusqingshengiiT(JX293295)聚为一支,自展值99(图4),菌株Srtli-F4与PaenibacilluslupiniT(KF769449)聚为一支,自展值100(图5),菌株Srtli-F5与BacilluspseudomycoidesT(ACMX01000133)聚为一支,自展值87(图6),菌株Srtli-F6与EscherichiaalbertiiT(ABKX01000030)聚为一支,自展值95(图7)。综上所述,将菌株Srtli-F1~Srtli-F6分别鉴定为绿针假单胞菌橙黄亚种P.chlororaphissubsp.aurantiaca、组氨酸节杆菌A.histidinolovorans、庆盛芽孢杆菌B.qingshengii、类芽孢杆菌羽扇豆根瘤菌P.lupini、假蕈状芽孢杆菌B.pseudomycoides、高山麝病原性阿尔伯蒂埃希氏杆菌E.albertii。
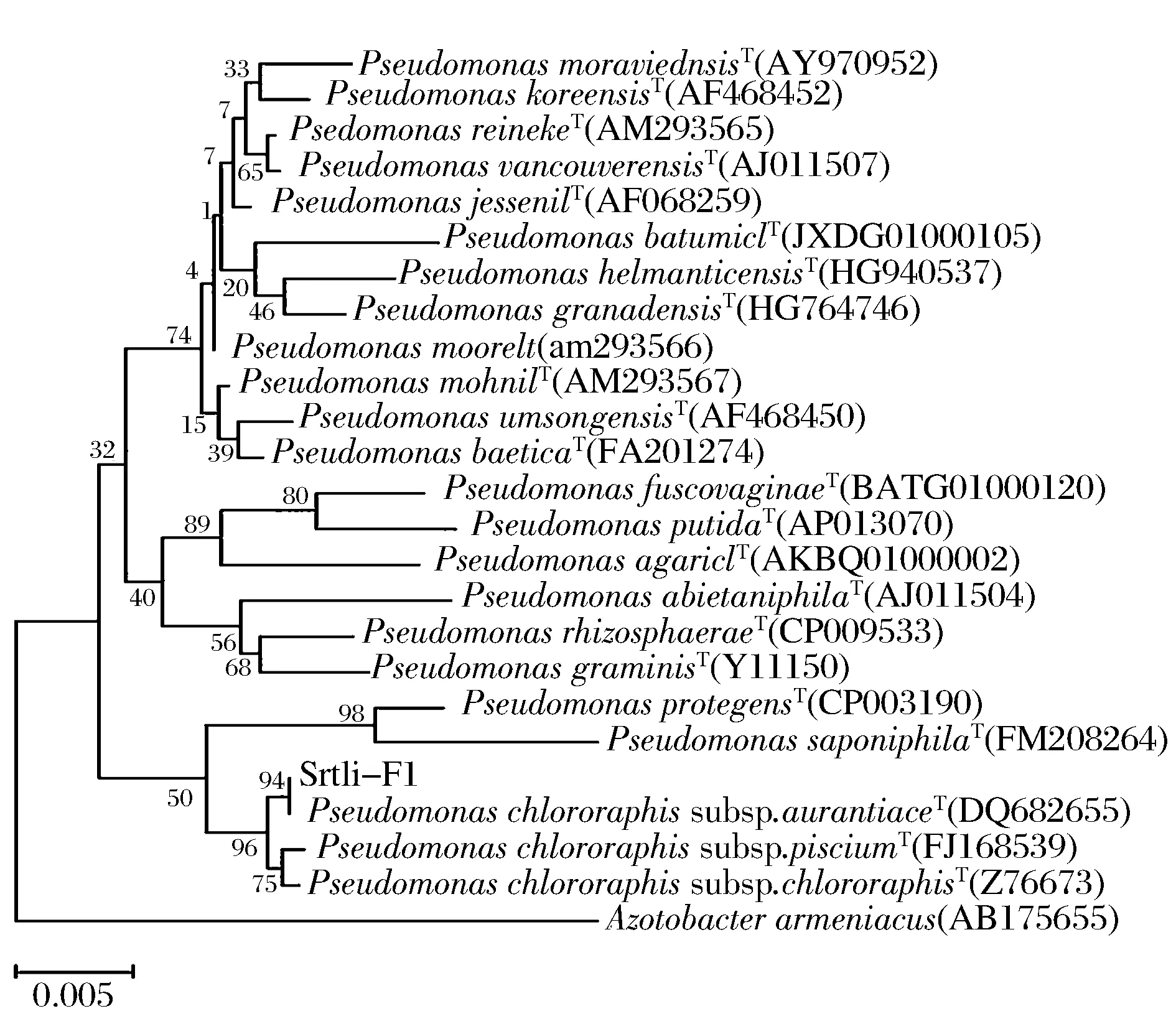
图2 菌株Srtli-F1的系统发育进化树(基于16S rDNA 基因序列)Fig.2 Phylogenetic tree of strain Srtli-F1 based on 16S rDNA gene sequence
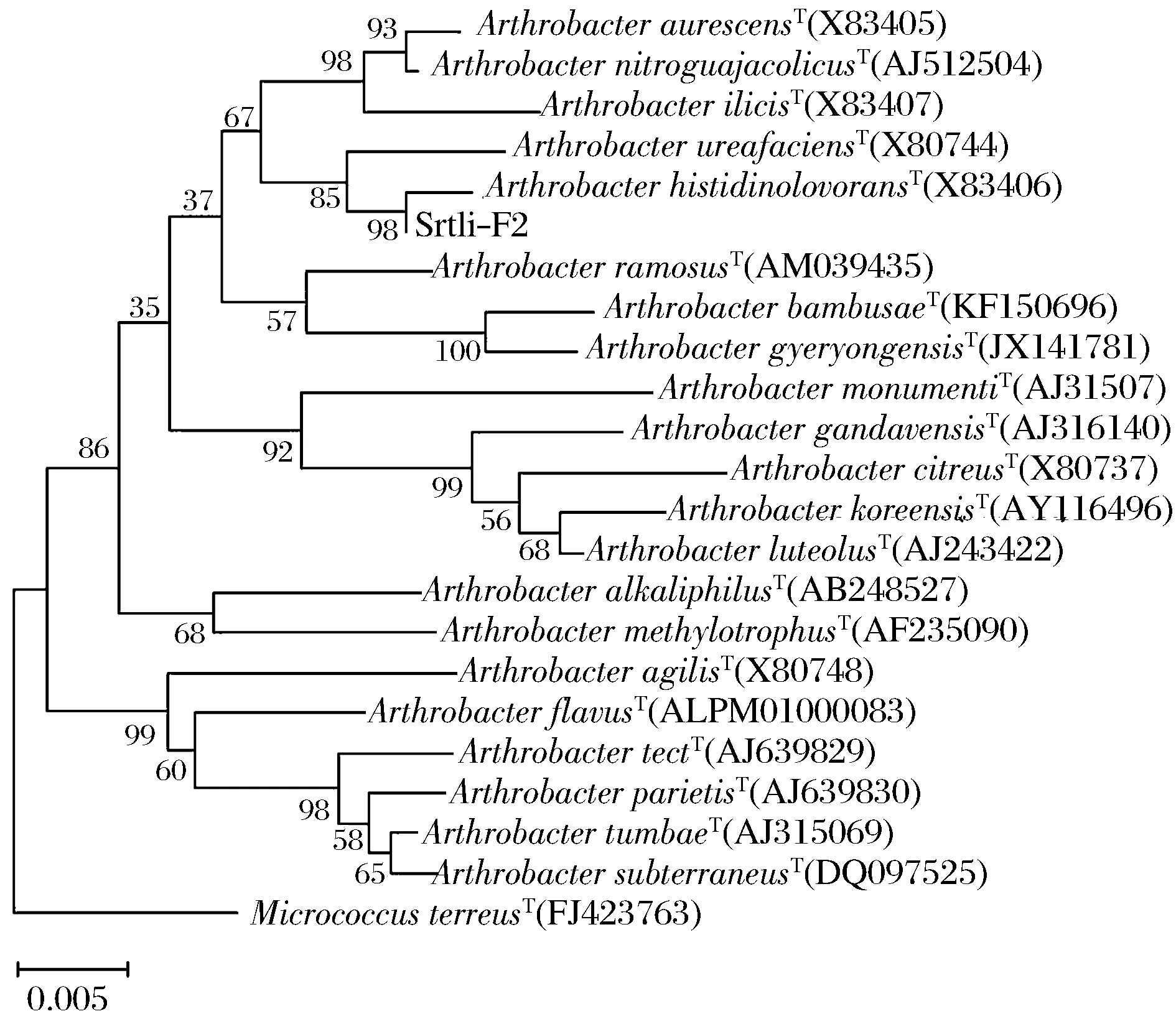
图3 菌株Srtli-F2的系统发育进化树(基于16S rDNA 基因序列)Fig.3 Phylogenetic tree of strain Srtli-F2 based on 16S rDNA gene sequence

图4 菌株Srtli-F3的系统发育进化树(基于16S rDNA 基因序列)Fig.4 Phylogenetic tree of strain Srtli-F3 based on 16S rDNA gene sequence

图5 菌株Srtli-F4的系统发育进化树(基于16S rDNA 基因序列)Fig.5 Phylogenetic tree of strain Srtli-F4 based on 16S rDNA gene sequence
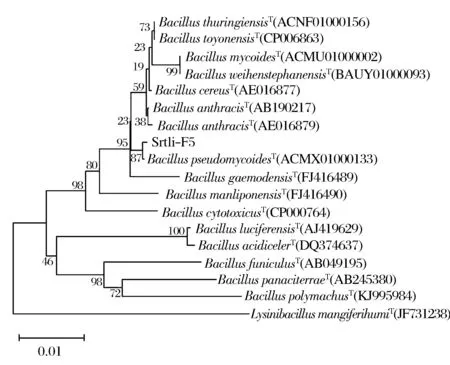
图6 菌株Srtli-F5的系统发育进化树(基于16S rDNA 基因序列)Fig.6 Phylogenetic tree of strain Srtli-F5 based on 16S rDNA gene sequence
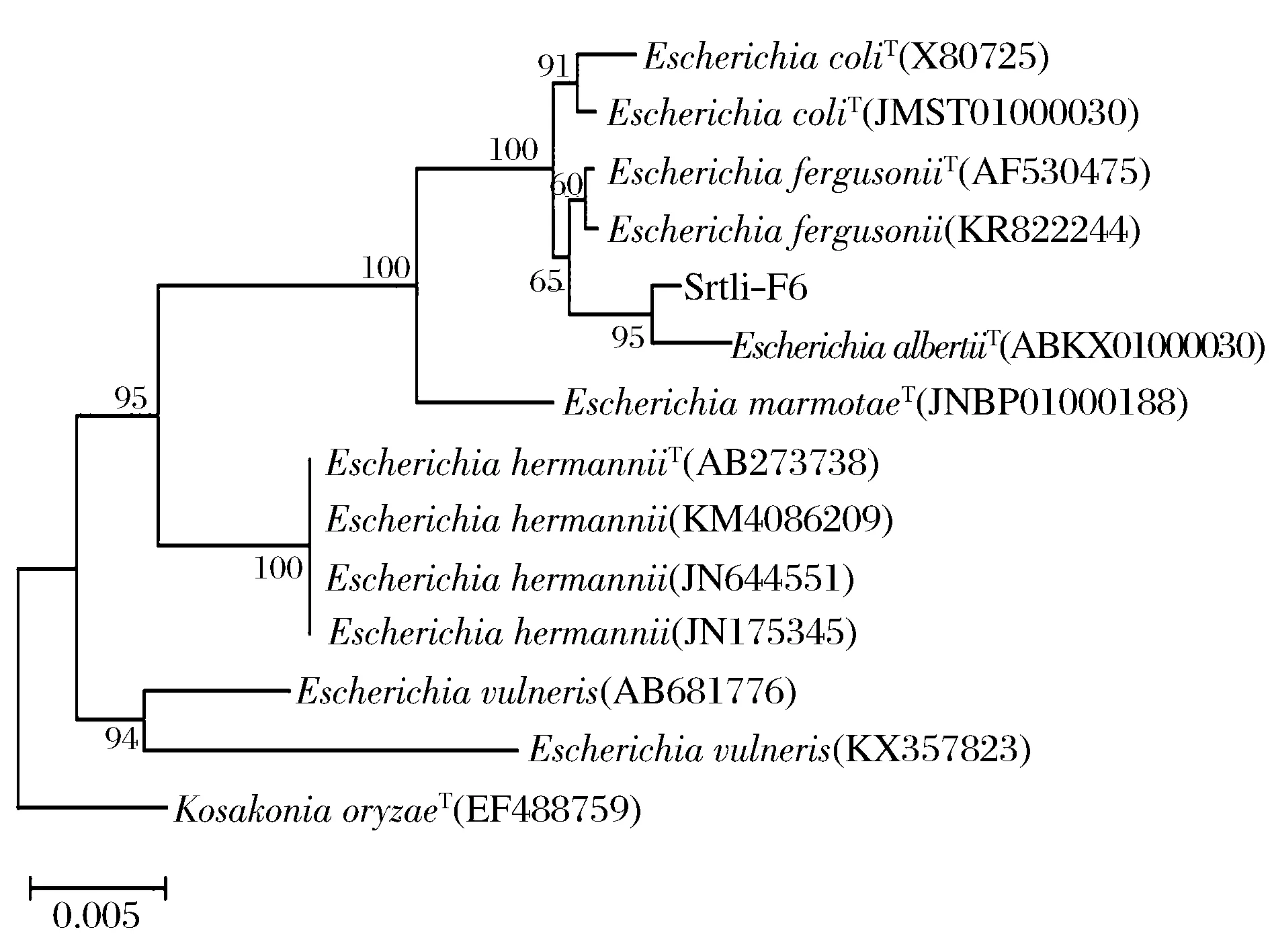
图7 菌株Srtli-F6的系统发育进化树(基于16S rDNA 基因序列)Fig.7 Phylogenetic tree of strain Srtli-F6 based on 16S rDNA gene sequence
2.3NRPS功能基因筛选与系统发育学分析
在确定6株内生细菌分类地位的基础上,分别PCR扩增菌株Srtli-F1~Srtli-F6的NRPS功能基因,并对NRPS基因进行克隆测序。结果表明菌株Srtli-F3和Srtli-F5含有目的基因片段,片段大小均为780bp,利用NCBI Orf finder(Open Reading Frame Finder)将核酸序列转化为蛋白序列,下载同源性较高的蛋白序列,用MEGA6.0软件构建系统发育树,分别如图8和图9所示。由图可见,种属相同的不同菌株聚为一支,提示可能产生相同类型的化合物,如Brevibacillusparabrevis(Q70LM6)和Brevibacillusparabrevis(Q70LM7)都能产生杆菌肽(Bacitracin),而不同的菌种也可聚为一支,提示也能产生相同的化合物,如Aneurinibacillusmigulanus(P0C063)和Brevibacillusbrevis(P0C064) 都能产生化合物Gramicicin。菌株Srtli-F3和Srtli-F5在2个进化树中均与B.licheniformis(O68008)聚为一枝,自展值分别为40和44,表明菌株Srtli-F5和Srtli-F3可能产生环肽类抗生素——杆菌肽(Bacitracin)。

图8 菌株Srtli-F3的系统发育进化树(基于NRPS基因序列)Fig.8 Phylogenetic tree of strain Srtli-F3 based on NRPS gene sequence

图9 菌株Srtli-F5的系统发育进化树(基于NRPS基因序列)Fig.9 Phylogenetic tree of strain Srtli-F5 based on NRPS gene sequence
2.4菌株发酵产物的质谱分析
将分离得到的菌株发酵7 d,丙酮提取发酵产物,经旋转蒸发器浓缩后,过滤,进行ESI-MS分析。结果分别如图10所示。由图10(A)可知,菌株Srtli-F5的粗提物在阳离子ESI-MS模式下在m/z1 102.7、m/z1 103.7、m/z1 104.7处产生碎片离子峰,由图10(B)可知,菌株Srtli-F5的粗提物在阴离子ESI-MS 模式下在m/z1 034.6、m/z1 035.6、m/z1 036.6处产生碎片离子峰,为杆菌肽化合物的特征分子离子质量,因此推测Srtli-F5可产生杆菌肽类物质。然而,菌株Srtli-F3的发酵产物经ESI-MS分析并没有出现环肽化合物特征离子峰,这可能是该菌株中含有的NRPS基因是沉默的,在实验室条件下无法表达环肽[8]。
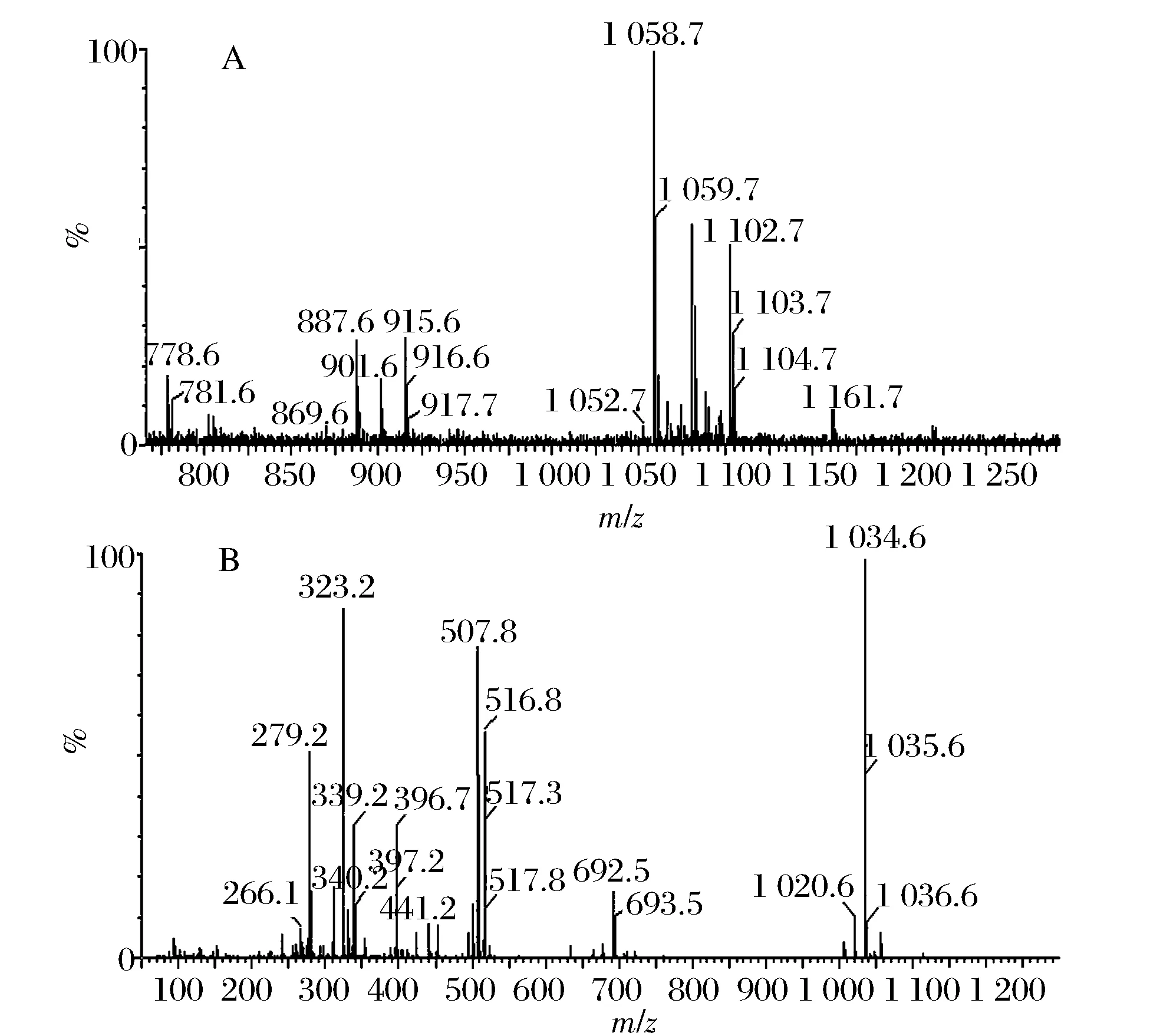
图10 菌株Srtli-F5发酵产物质谱图Fig.10 Mass spectrum diagram about the fermentation products of Srtli-F5
3 讨论
研究表明,微生物能否产生有价值化合物是由其功能基因决定的[9]。因此,通过靶向扩增生物合成次级代谢产物过程中的多功能关键酶——NRPS,实现产环肽菌株的定向筛选,不仅减少了资源的浪费,也大大提高了筛选效率。本研究项目从海芦笋中分离出6株内生细菌Srtli-F1~Srtli-F6,利用PCR扩增这6个菌的16S rDNA目标序列,接着通过系统发育树分析与形态学观察,将其分别鉴定为绿针假单胞菌橙黄亚种P.chlororaphissubsp.aurantiaca、组氨酸节杆菌A.histidinolovorans、庆盛芽孢杆菌B.qingshengii、类芽孢杆菌羽扇豆根瘤菌P.lupini、假蕈状芽孢杆菌B.pseudomycoides、高山麝病原性阿尔伯蒂埃希氏杆菌E.albertii。基于NRPS型功能基因对这6个菌株进行筛选,发现其中编号为Srtli-F3和Srtli-F5的菌株含有NRPS基因,提示可能产生环肽类化合物,对这2个菌种分别进行发酵,利用ESI-MS分析发酵产物,发现Srtli-F5能够产生杆菌肽,而Srtli-F3发酵产物没有出现环肽类化合物分子离子峰,提示在实验室条件下,产生环肽类化合物的NRPS基因可能是沉默的而无法表达。
杆菌肽,是一种环肽类化合物。因为这种多肽来源于地衣芽孢杆菌的发酵产物,所以被称为“杆菌肽”[10]。杆菌肽是一种广谱抗菌素[11],能够抑制动物肠道中的许多有害菌,起到防治动物肠道疾病的效果,保证了饲料的充分利用,有利于动物生长。与传统抗生素相比,这种抗生素安全性高、残留少,不产生抗药性、毒副作用弱[12],是饲料添加剂中传统抗生素的有效替代品,已被广泛应用在国内外动物养殖和饲料生产中[13-14],也被作为生物学的研究工具应用于生物表面活性剂、饲料添加剂、食品防腐剂研究中[15]。因此,发现能够产生杆菌肽的微生物,对于拓宽获得杆菌肽的途径,具有重要的实际应用价值。本研究首次从海芦笋中分离鉴定到假蕈状芽孢杆菌,今后将对其发酵产物进行深入研究,研究其除产生环肽杆菌肽外,是否能够产生其他环肽,验证同一微生物产生多种环肽的能力。
[1] 冉火苗,孔望君,蒋会芳,等.盐生海芦笋抗菌内生细菌的筛选与鉴定[J].食品与发酵工业,2016,42(3):79-86.
[2] 陆东和,张慜,蔡金龙,等.海芦笋营养价值及其采后品质变化[J].食品工业科技,2010,31(6):303-305.
[3] 朱鹏.珍珠膜海绵共附生微生物PKS与NRPS的筛选与模块结构研究[D].杭州:浙江大学,2008.
[4] MOORE B S,Biosynthesis of marine natural products:microorganisms (Part A)[J].NATURAL PRODUCT REPORTS,2005,22(5):580-593.
[5] 文艳苹.基于PKS、NRPS基因的抗生素paenimacrolidin和嗜铁素paenibactin研究[D].杭州:浙江大学,2011.
[6] LANGE A,SUN H,PILGERJ,et al.Predicting the structure of cyclic lipopeptides by Bioinformatics:structure revision of Arthrofactin[J].CHEMBIOCHEM,2012,13(18):2 671-2 675.
[7] 刘怡君,潘沐,王明洋,等.海芦笋内生真菌基于PKSⅠ型功能基因的分离与鉴定[J].食品科学,2014(11):115-119.
[8] K YAMANAKA,KA REYNOLDS,RD KERSTEN,et al.Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J].Proceedings of the National Academy of Sciences,2014,111(5):1 957-1 962.
[9] 周培瑾,徐毅,马允卿,等.极端嗜盐菌 16S rDNA的PCR扩增[J].微生物学报,1994,24(1):6-8.
[10] 胡尚勤.杆菌肽高产的代谢调控[J].国外医药(抗生素分册),2004(4):160-162.
[11] 周岩民,王龙昌.杆菌肽产品在动物饲料中的应用研究进展[J].饲料工业,2012(18):1-5.
[12] 陈吉红,庞玉红,郭荣富.杆菌肽锌在畜禽中的应用及其安全性评价[J].中国饲料,2004(9):28-29.
[13] RAVINDRAN V,THOMAS D V,THOMAS D G,et al.Performance and welfare of broilers as affected by stocking density and zinc bacitracin supplementation[J].Animal Science Journal,2006,77(1):110-116.
[14] 周臣飞,李俊辉,季朝金.杆菌肽类饲料添加剂在畜禽养殖中的应用[J].中国动物保健,2011(12):8-11.
[15] 周欣,李宝库.杆菌肽的研究进展及应用[J].科技信息,2010(14):92-93.
ScreeningandidentificationofendophyticbacteriafromSalicorniabigelovii.TorrbasedonaNRPSfunctionalgeneassay
LI Dai-jing,YIN Yue,LIU Meng-jie,WEI Ting,GUO Jia,XIN Zhi-hong1*
(College of Food Science and Technology,Nanjing Agricultural University,Nanjing 210095,China)
In the current research, endophytic bacteria were isolated fromSalicorniabigelovii.Torr and characterized by morphological observation and molecular methods, and the NRPS gene was screened to obtain the strains that could produce cyclic peptide compounds. The results showed that six endophytic bacteria were isolated and identified asPseudomonaschlororaphissubsp.Aurantiaca,Arthrobacterhistidinolovorans,Bacillusqingshengii,Paenibacilluslupini,Bacilluspseudomycoides,Escherichiaalbertii, respectively. Among them, the strain Srtli-F5 contained NRPS genes and was predicted to have capability of producing cyclopeptide compounds by phylogenetic tree analysis. It was further confirmed by the detection of fermentation production using ESI-MS (electrospray mass, ESI-MS) that the strain really produced bacitracin. The study provides a new method for directional screening strains with the potential to produce cyclic peptide compounds.
endophytic bacillus; isolation and identification; phylogenetic analysis; nonribosomal peptide synthetase; mass spectrum;cyclopeptide
10.13995/j.cnki.11-1802/ts.014643
本科生(辛志宏教授为通讯作者,E-mail:xzhfood@njau.edu.cn)。
2017年农产品质量安全风险评估项目(GJFP201701102);2016年南京农业大学大学生创新训练计划(SRT)项目(1618C01)
2017-04-27,改回日期:2017-05-17

