长链非编码RNA SUMO1P3在非小细胞肺癌中的表达及其预后价值*
刘 晓,胡 岗,陈一凡,刘 波,徐 敏,廖俊喆
(四川省成都市第五人民医院呼吸科 611130)
长链非编码RNA SUMO1P3在非小细胞肺癌中的表达及其预后价值*
刘 晓,胡 岗△,陈一凡,刘 波,徐 敏,廖俊喆
(四川省成都市第五人民医院呼吸科 611130)
目的探讨非小细胞肺癌lncRNA SUMO1P3的表达与临床意义。方法应用qRT-PCR法检测126例非小细胞肺癌患者肿瘤组织及配对癌旁组织中SUMO1P3的表达水平及其与临床病理特征和预后的关系。结果与癌旁组织、健康组织相比,非小细胞肺癌组织中SUMO1P3表达水平明显升高(P<0.01)。其表达水平与TNM分期(χ2=12.49,P<0.01)及肿瘤体积(χ2=10.20,P<0.01)显著相关。ROC曲线下面积为0.728 4[95%CI(0.664,0.761);P=0.001 2]。Kaplan-Meier生存分析显示SUMO1P3高表达组患者的总生存时间(OS)和疾病无进展生存期(PFS)显著缩短(P=0.000 1,P=0.003 1)。Cox回归分析显示SUMO1P3表达水平、组织学分级和TNM分期是OS、PFS的独立预测指标。结论SUMO1P3的高表达与非小细胞肺癌患者的不良预后密切相关,可能是非小细胞肺癌患者诊断及治疗的潜在靶点。
肺肿瘤;长链非编码RNA;SUMO1P3;预后
肺癌是世界范围内癌症死亡的首要原因,而非小细胞肺癌(NSCLC)约占全部肺癌的80%~85%[1]。NSCLC 5年总体生存率仅约11%~15%[2],这与NSCLC高侵袭和高转移性密切相关,并且缺少有效的分子靶点及分子标志物[3]。长非编码RNA(lncRNAs)是近几年由高通量转录组分析发现的一类非编码RNA,其参与多种生物学过程,包括肿瘤的发生和发展[4-5]。小泛素样修饰物(SUMO)1假基因3(SUMO1P3)是lncRNAs家族一员,近年来已证实其与膀胱癌、胃癌的不良预后、肿瘤生长和转移密切相关[6-7]。但是,lncRNA SUMO1P3与NSCLC之间的相关性尚不明确。本研究应用qRT-PCR检测126例NSCLC患者肿瘤组织及对应癌旁组织中SUMO1P3的表达水平,通过分析SUMO1P3表达与NSCLC的临床病理指标以及预后之间的相关性,探讨SUMO1P3在NSCLC发生和发展中的作用,为寻找NSCLC潜在的预后判断指标和治疗靶点提供新思路。
1 资料与方法
1.1一般资料 126例新鲜癌组织及配对的癌旁正常组织取自2010年6月至2012年5月在四川省成都市第五人民医院确诊的NSCLC患者。标本通过经支气管镜黏膜活检、经皮肺穿刺活检、经支气管肺活检以及手术切除获得,所有患者在获取标本前均未接受肿瘤的相关治疗。标本采集后立即存放在无RNA酶的冻存管中,-80 ℃保存备用。选取57例未患有NSCLC的志愿者作为健康对照。采用2010年WHO病理分级标准对肿瘤进行组织学分级;根据2009年IASLC肺癌分期系统对患者进行分期;对所有患者进行严密随访,前两年3个月随访1次,之后6个月随访1次。计算总生存期(OS)和疾病无进展生存期(PFS),OS为确诊到死亡的间隔时间,PFS为确诊到首次发现局部、区域性或远处肿瘤进展的间隔时间。本研究通过本院医学研究伦理委员会批准,所有研究对象均签署知情同意书。
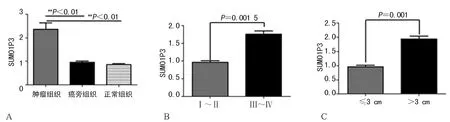
图1 SUMO1P3基因在NSCLC组织中的表达和相关的临床参数
1.2RNA抽提与qRT-PCR 应用Trizol试剂(Invitrogen,中国上海)从组织样本中提取总RNA。用NanoDrop ND-2000分光光度计(Thermo Scientific)在260 nm处测定总RNA的浓度和纯度,电泳检测显示,提纯的RNA质量良好。应用反转录试剂盒(TaKaRa,中国大连)将总RNA转化为cDNA。采用SYBR Green法进行荧光定量PCR(TaKaRa,中国大连),并应用ABI PRISM 7000 荧光定量PCR系统(Agilent Technologies)进行数据收集。引物由Biosune(中国上海)合成,SUMO1P3引物序列:上游5′- ACT GGG AAT GGA GGA AGA-3′,下游5′-TGA GAA AGG ATT GAG GGA AAA G -3′;GAPDH引物序列:上游5′-CGC TCT CTG CTC CTC CTG TTC-3′,下游5′-ATC CGT TGA CTC CGA CCT TCA C-3′。取3次测量的平均值用2-ΔΔCt法计算SUMO1P3的相对量。
2 结 果
2.1NSCLC患者肿瘤组织中SUMO1P3的表达 SUMO1P3在NSCLC肿瘤组织中的表达与癌旁组织(P<0.01)和正常组织(P<0.01)相比明显上调。这表明SUMO1P3的表达差异可能同NSCLC的发病机制相关(图1A)。患者TNM分期越高,肿瘤体积越大,SUMO1P3表达水平越高(图1B、C)。
2.2SUMO1P3的表达水平与患者临床病理特征之间的关系 SUMO1P3水平同患者TNM分期(0.960 3±0.153 3vs. 1.757 0±0.270 5,P=0.001 5)和肿瘤体积存在正相关性(0.971 2±0.143 3vs. 1.937 1±0.302 1,P=0.001 0)。肿瘤大于3 cm,或TNM分期为Ⅲ、Ⅳ期的患者,其SUMO1P3水平较高,而肿瘤小于3 cm,或TNM分期为Ⅰ、Ⅱ期的患者,其SUMO1P3水平较低。同时,根据癌组织中SUMO1P3表达水平的中位数,笔者将样本分成SUMO1P3高表达组(高于中位数,n=63)和SUMO1P3低表达组(低于中位数,n=63)。利用χ2检验评估两组间临床病理因素。SUMO1P3表达水平同肿瘤体积 (χ2=10.20,P<0.01)和TNM分期(χ2=12.49,P<0.01),存在相关性。而SUMO1P3表达水平同其他因素之间不存在相关性(P>0.05),例如性别、年龄、组织学分级(低、中、高)、组织学类型、淋巴结转移情况、吸烟史,见表1。
2.3SUMO1P3的诊断价值 应用ROC曲线和ROC曲线下面积对比NSCLC肿瘤组织和癌旁组织SUMO1P3的表达水平。两组ROC曲线明显分离,SUMO1P3的AUC为0.728 4[95%CI(0.664,0.761);P=0.001 2],说明SUMO1P3可以作为NSCLC的一种潜在的分子标志物,见图2。
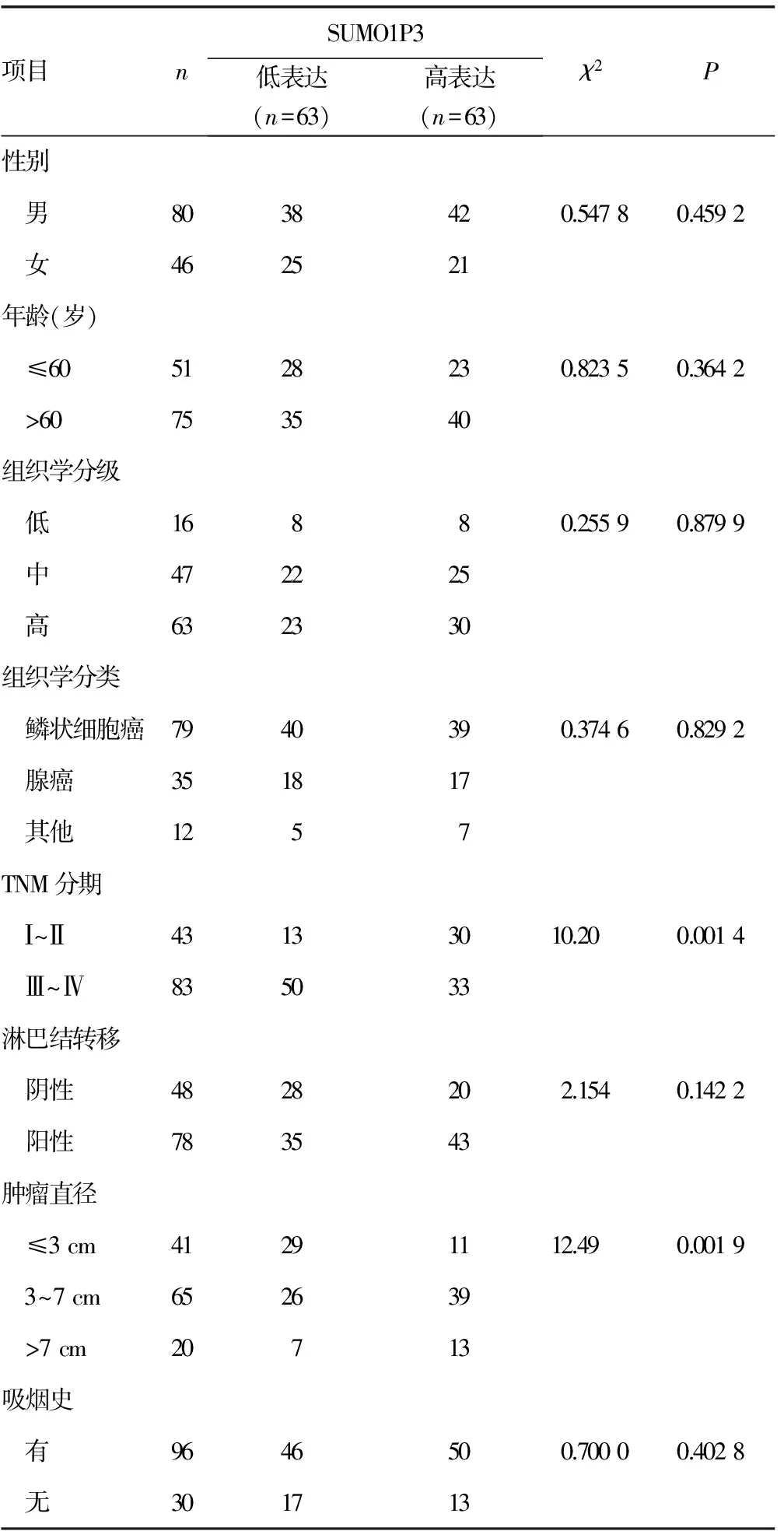
表1 SUMO1P3的表达与患者临床病理特征之间的关系
2.4SUMO1P3高表达与NSCLC患者不良预后的相关性 为明确SUMO1P3表达水平同患者预后之间的关系,笔者首先应用Kaplan-Meier生存分析和log-rank秩检验评估了SUMO1P3表达水平同OS和PFS之间的相关性。如图3A和3B所示,SUMO1P3低表达组5年OS为20.6%,而高表达组只有11.5%;高表达组的中位生存时间为23个月,而低表达组为48个月;高表达组中位PFS为19个月,低表达组为36个月。值得注意的是,SUMO1P3高表达组的OS (P=0.000 1)和PFS(P=0.003 1)都较差。这些都提示NSCLC中SUMO1P3表达上调与患者的生存时间密切相关。
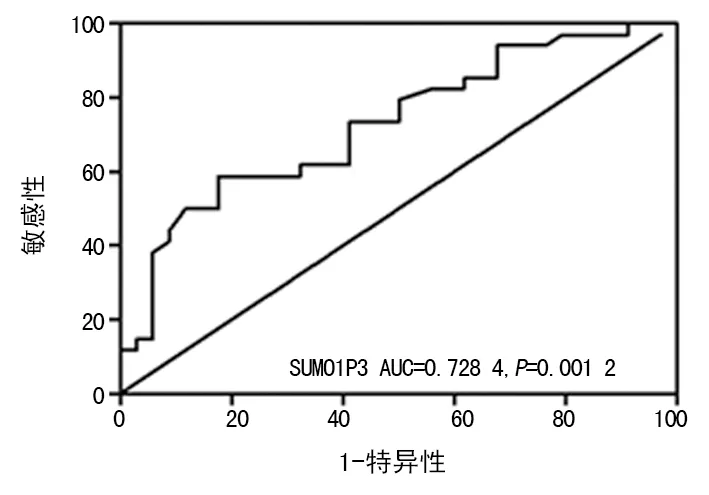
图2 SUMO1P3表达的ROC曲线
2.5SUMO1P3的高表达是NSCLC患者的独立预后指标 为了进一步明确SUMO1P3在NSCLC中的预后价值,笔者对126例NSCLC患者的PFS和OS分别进行了单变量和多变量生存分析(COX风险回归模型)。单变量分析明确了3个预后指标(组织学分级、TNM分期、SUMO1P3表达)是影响患者OS和PFS具有统计学意义的危险因素。其他临床病理因素,例如,性别、肿瘤大小、组织学类型、吸烟史、淋巴结转移情况等因素都不具有统计学意义(P>0.05)。多因素分析进一步揭示影响NSCLC患者OS、PFS的独立预测指标包括SUMO1P3表达情况、组织学分级和TNM分期,见表2。
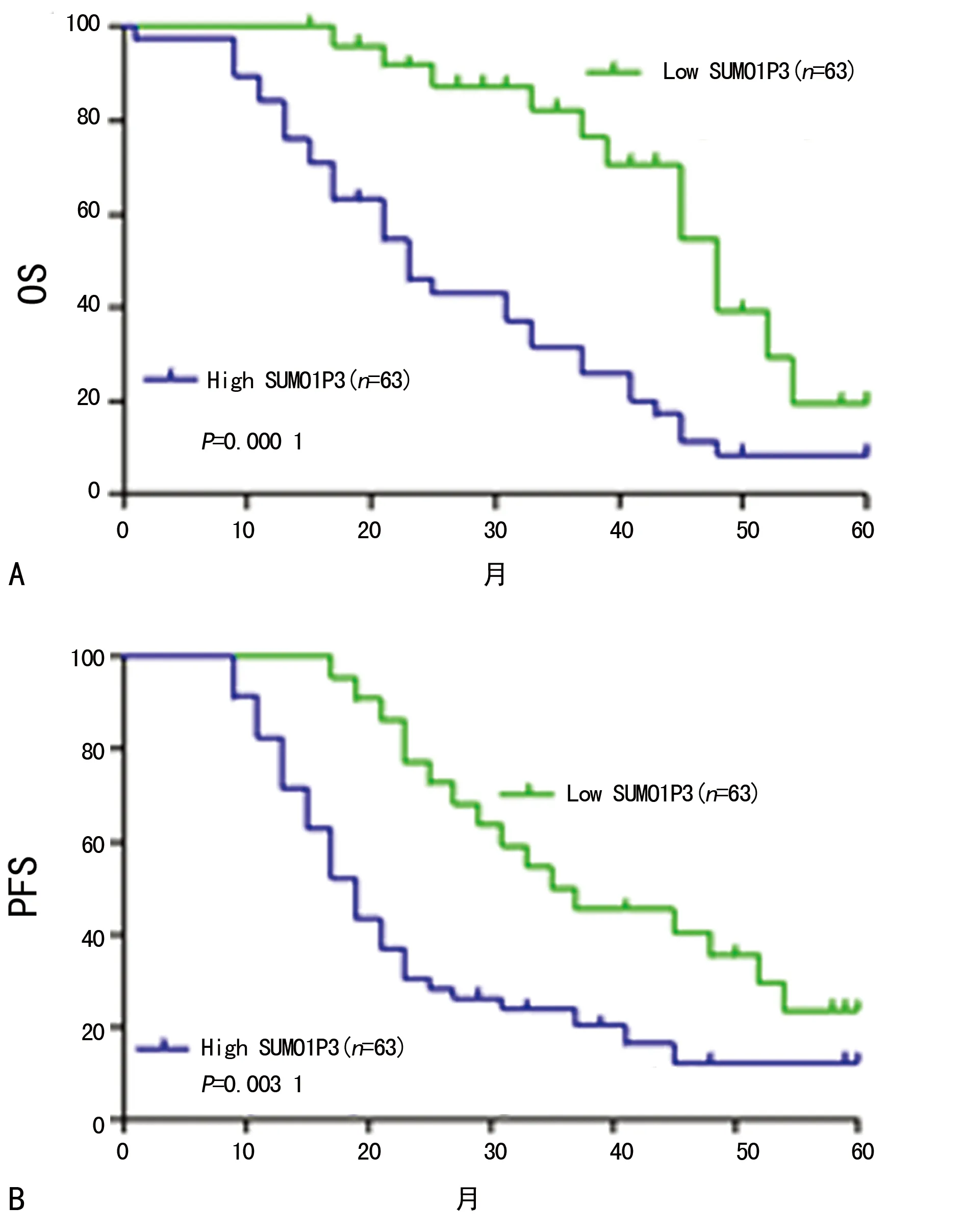
图3 SUMO1P3的表达与NSCLC患者OS与PFS的关系
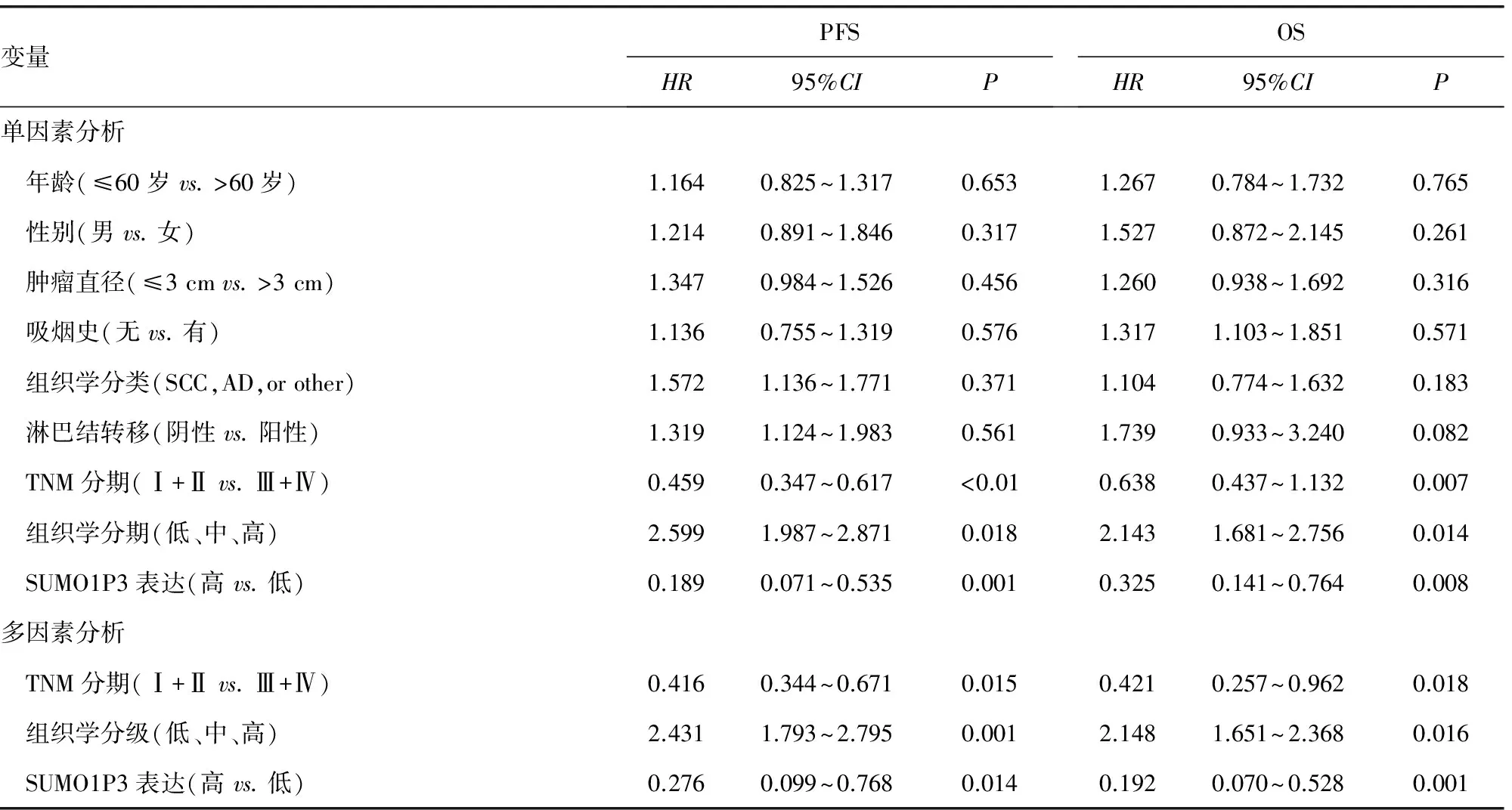
表2 单因素及多因素分析NSCLC患者临床病理特征与PFS和OS的关系
3 讨 论
近几年发现了许多lncRNAs,大量研究探讨其在广泛的生物学过程中的作用,从转录到mRNA剪接、RNA衰减及翻译,基因生命周期的每一步几乎都可受到这些分子的影响[8-9]。此外,近期研究开始鉴定癌症相关lncRNAs,并探讨它们在癌症中的临床意义和生物学功能。例如,HOTAIR是一种lncRNA,在多种肿瘤中发生过表达,并与癌症转移显著相关[10-12]。lncRNA CCAT2过表达与结直肠癌(CRC)和NSCLC患者的不良预后具有相关性,可用作患者生存率的独立预后指标[13-14]。MALAT1表达上调是NSCLC生存的独立预后指标[15]。与之相反,其他一些lncRNAs如MEG3、GAS5和lncRNA H19则在多种人类癌症中具有肿瘤抑制作用[16-18]。这些发现表明,像蛋白质编码基因和miRNAs一样,lncRNAs可作为诊断和预后判断生物标志物。但是,lncRNAs在NSCLC中的总体病理生理作用目前尚不明确。SUMO1P3是SUMO假基因家族的成员之一,是一种新发现的长链非编码RNA[19]。SUMO1P3在胃癌和膀胱癌中表达上调,是胃癌和膀胱癌潜在的预后判断和治疗靶点[6-7]。在胃癌中,SUMO1P3与肿瘤大小、分化、淋巴结转移以及浸润具有相关性[7]。此外,SUMO1P3 siRNA转染的膀胱细胞出现增殖抑制、凋亡增多以及迁移抑制[6]。但是,SUMO1P3在NSCLC中的表达和临床意义目前尚不明确。
本研究结果表明:与癌旁组织相比,NSCLC肿瘤组织SUMO1P3的表达水平明显增高(P<0.01),另外通过进一步分析NSCLC患者肿瘤组织中SUMO1P3表达与临床病理特征之间的关系,发现SUMO1P3高表达与TNM分期及肿瘤体积有关(P<0.01),而与其他临床病理特征如患者性别、年龄、组织学分级(低、中、高)、组织学类型、淋巴结转移情况、吸烟史无关。ROC曲线和ROC曲线下面积显示ROC曲线显示明显分离,SUMO1P3的AUC为0.728 4,说明SUMO1P3可以作为NSCLC的一种潜在的生物标志物。Kaplan-Meier生存分析和log-rank秩检验结果显示SUMO1P3高表达与患者的不良预后相关,SUMO1P3高表达组患者的OS和PFS显著缩短。单变量和多变量生存分析结果显示组织学分级、TNM分期、SUMO1P3表达是影响患者PFS和OS具有统计学意义的危险因素和独立预测指标。本研究仅为单中心研究,纳入样本量较少,有待今后进一步扩大样本量对实验结果进一步验证。本研究将样本中位数(n=63)的SUMO1P3表达水平作为SUMO1P3高表达和低表达的临界值,这需要进一步扩大样本,确定一个更为敏感度和特异性的SUMO1P3值,进而进入临床推广应用。在随后的研究中,笔者将扩大样本量作进一步探讨,并尝试鉴定lncRNA SUMO1P3的生物学功能,阐明其在NSCLC肿瘤增殖、侵袭及转移中的分子机制。
综上所述,与配对癌旁组织和健康组织相比,NSCLC常发生SUMO1P3表达上调,提示SUMO1P3可能具有关键的致癌作用,有望用作NSCLC患者低生存率的预测指标以及负性预后因子。这些新发现表明,SUMO1P3可能成为NSCLC潜在的预后和治疗靶点。
[1]Siegel RL,Miller KD,Jemal A.Cancer statistics,2016[J].CA Cancer J Clin,2016,66(1):7-30.
[2]Chen Z,Fillmore CM,Hammerman PS,et al.Non-small-cell lung cancers:a heterogeneous set of diseases[J].Nat Rev Cancer,2014,14(8):535-546.
[3]Rosell R,Bivona TG,Karachaliou N.Genetics and biomarkers in personalisation of lung cancer treatment[J].Lancet,2013,382(9893):720-731.
[4]Huarte M.The emerging role of incRNAs in cancer[J].Nat Med,2015,21(11):1253-1261.
[5]Rinn JL,Chang HY.Genome regulation by long noncoding RNAs[J].Annu Rev Biochem,2012,81(2):145-166.
[6]Zhan YH,Liu YC,Wang C,et al.Increased expression of SUMO1P3 predicts poor prognosis and promotes tumor growth and metastasis in bladder cancer[J].Oncotarget,2016,7(13):16038-16048.
[7]Mei D,Song H,Wang K,et al.Up-regulation of SUMO1 pseudogene 3 (SUMO1P3) in gastric cancer and its clinical association[J].Med Oncol,2013,30(4):709.
[8]Flynn A,Chang Y.Long noncoding RNAs in cell-fate programming and reprogramming[J].Cell Stem Cell,2014,14(6):752-761.
[9]Turner M,Galloway A,Vigorito E.Noncoding RNA and its associated proteins as regulatory elements of the immune system[J].Nat Immunol,2014,15(6):484-491.
[10]Gupta RA,Shah N,Wang KC,et al.Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J].Nature,2010,464(7291):1071-1076.
[11]Hajjari M,Salavaty A.HOTAIR:an oncogenic long non-coding RNA in different cancers[J].Cancer Biol Med,2015,12(1):1-9.
[12]Du M,Wang W,Jin H,et al.The association analysis of lncRNA HOTAIR genetic variants and gastric cancer risk in a Chinese population[J].Oncotarget,2015,6(31):31255-31262.
[13]Ling H,Spizzo R,Atlasi Y,et al.CCAT2,a novel noncoding RNA mapping to 8q24,underlies metastatic progression and chromosomal instability in colon cancer[J].Genome Res,2013,23(9):1446-1461.
[14]He X,Tan X,Wang X,et al.C-Myc-activated long noncoding RNA CCAT1 promotes colon cancer cell proliferation and invasion[J].Tumour Biol,2014,35(12):12181-12188.
[15]Gutschner T,Hämmerle M,Eissmann M,et al.The noncoding RNA MALAT1 is a critical regulator of the metastasis phenotype of lung cancer cells[J].Cancer Res,2013,73(3):1180-1189.
[16]Sun M,Jin FY,Xia R,et al.Decreased expression of long noncoding RNA GAS5 indicates a poor prognosis and promotes cell proliferation in gastric cancer[J].BMC Cancer,2014,14(3):319.
[17]Liang C,Fu M,Wong W,et al.The lncRNA H19 promotes epithelial to mesenchymal transition by functioning as miRNA sponges in colorectal cancer[J].Oncotarget,2015,6(26):22513-22525.
[18]Yin D,Liu J,Zhang E,et al.Decreased expression of long noncoding RNA MEG3 affects cell proliferation and predicts a poor prognosis in patients with colorectal cancer[J].Tumour Biol,2015,36(6):4851-4859.
[19]Wright MW,Bruford EA.Naming junk:human non-protein coding RNA (ncRNA) gene nomenclature[J].Hum Genomics,2011,5(2):90-98.
10.3969/j.issn.1671-8348.2017.27.030
R734.2
B
1671-8348(2017)27-3839-04
2016-12-14
2017-04-29)
四川省医学会青年创新项目(Q16008)。
刘晓(1982-),硕士,主治医师,主要从事肺癌的早期诊断与治疗。△
,E-mail:hu13684038833@163.com。

