一碳化合物利用新途径的设计和体外构建
陈 阳,杨 雪,袁倩倩,罗 浩,李培顺,马红武
(中国科学院 天津工业生物技术研究所,天津 300308)
一碳化合物利用新途径的设计和体外构建
陈 阳,杨 雪,袁倩倩,罗 浩,李培顺,马红武
(中国科学院 天津工业生物技术研究所,天津 300308)
一碳化合物作为糖类的廉价替代底物在工业生物技术中具有广泛的应用前景。传统途径中由一碳化合物或糖类生产乙酰辅酶A衍生产品时会有1/3的碳损失,导致产品得率较低,因此找到没有碳损失的新途径非常重要。基于MetaCyc数据库的代谢反应集和通量平衡分析的途径分析方法,计算得到一条由一碳化合物甲醛生成乙酰辅酶A衍生产品的新途径,该途径不仅无碳损失,并且无能量(ATP)和还原力消耗。为了验证途径的可行性,筛选途径相关酶,纯化后进行体外实验,通过乙酸的产量来间接表征途径的运行情况。结果发现:乙酸的生成浓度会随着体系初始甲醛加入量的增加而增加,在一定条件下,乙酸的总碳摩尔得率可以达到90%,超过了传统路径的最大理论得率67%。
代谢网络模型;一碳化合物;乙酰辅酶A;新途径;体外验证
近年来,一碳化合物作为很有应用前景的绿色 能源物质得到广泛研究[1-3],基本思路是借鉴与利用生物界现已存在的天然一碳利用途径,以一碳化合物作为底物生产化学品和生物能源。自然界中最常见的一碳物质利用途径是存在于甲基营养代谢细菌中的核酮糖一磷酸(RuMP)途径[4]。第一步是利用5-磷酸核酮糖(Ru5P)固定甲醛(FALD)得到6-磷酸己糖(H6P),H6P经过异构酶作用生成6-磷酸果糖(F6P),然后需要ATP将F6P磷酸化成果糖二磷酸,随后裂解成3-磷酸甘油醛(G3P)和磷酸二羟丙酮(DHAP),其中 G3P可以脱去 ATP和NADH得到丙酮酸,丙酮酸则会脱掉CO2而形成乙酰辅酶A(AcCoA),此过程中生成的DHAP和F6P则可以循环回补再形成初始底物Ru5P。因此,整个途径的净反应可以归纳为3分子甲醛缩合成1分子丙酮酸,再脱羧形成乙酰辅酶A,中间过程会损失1分子的CO2,最终碳效率为67%,并且涉及的磷酸化反应会需要ATP的额外供应。某些酵母体内的木酮糖一磷酸(XuMP)途径[5],与 RuMP途径的差异在于用5-磷酸木酮糖(Xu5P)固定甲醛,因此同样存在类似CO2造成的碳损失以及依赖ATP的问题。丝氨酸途径尽管没有碳损失的问题,却需要额外供应还原力和ATP。总之,这些天然途径都不可避免地存在碳损失以及额外消耗ATP或还原力的问题[6-7]。
Bogorad等[8]构建了一条从甲醇出发到乙酰辅酶A的体外甲醇缩合循环途径(MCC途径),整个过程完全没有碳损失,同时还不依赖ATP,在优化途径中关键酶加入量的基础上证明了体系的可循环性,应用这条途径,他们成功在体外低温条件下将甲醇直接转化成乙酰辅酶A下游衍生物乙醇和丁醇。如果再对这条途径的稳定性进行优化和调控,可进一步用于工业放大,因此应用前景十分广阔。
近几年,基于模型指导的理性途径设计越来越成为生物工程领域的研究热点[9-11],对实验具有重要的指导意义。Bar-Even等[12]利用KEGG数据库提取的数据构建了复合网络模型,并针对固碳途径进行计算设计,得到20余条固碳新途径。笔者所在课题组的Lin等[13]利用大肠杆菌iJO1366模型对乙酰辅酶A衍生产品聚3-羟基丁酸酯(PHB)最优合成途径进行预测,发现了一条通过碳再利用途径来避免碳损失从而提高PHB得率的新途径。
因此,本研究中,笔者希望结合代谢网络模型构建的方法,预测出一条从一碳化合物甲醛出发,过程不依赖ATP和还原力,且没有碳损失的体外合成催化新途径,再通过体外验证的方法证明其可行性。
1 材料与方法
1.1 代谢网络模型分析算法及工具
通量平衡分析算法(FBA)利用线性规划算法求解拟稳态条件下的通量分布[14]。本工作中的途径预测即基于FBA算法。计算平台基于Matlab的COBRA工具包[15],该工具包可以直接对Excel或者SBML格式的模型进行计算,并且包含多种模型分析算法,如FBA、通量可变性分析、基因敲除算法和基因必需性分析等。
1.2 菌株和质粒
E.coli MG1655基因组作为基因扩增的模板,E.coli BL21(DE3)用于蛋白表达的宿主,质粒载体pET28a用来连接基因序列。
1.3 材料与试剂
甲醛(质量分数 37%)、5-磷酸核糖(R5P)、Xu5P、G3P、F6P、NADH、NADP、咪唑、二羟基丙酮(DHA)、甘油磷酸脱氢酶(兔肌肉来源)和己糖激酶(源自Saccharomyces cerevisiae),Sigma-Aldrich公司;乙酸(Ace)检测试剂盒(K-ACET),Megazyme公司;
蛋白纯化过程洗脱液为Buffer A(25 mmol/L Tris,150 mmol/L NaCl,20 mmol/L 咪唑,pH 7.5)与Buffer B(25 mmol/L Tris,150 mmol/L NaCl,500 mmol/L 咪唑,pH 7.5)。
1.4 表达载体的构建与蛋白纯化
所有酶蛋白基因的引物序列列于表1中,基因各自连接在带有His标签的质粒pET28a上,然后转化到E.coli BL21(DE3)感受态细胞中。挑取阳性重组菌株,在37℃、220 r/min条件下培养2.5 h,当OD600达到 0.6~0.8 时,加入 IPTG 至浓度为 0.5 mmol/L,17℃诱导 20 h后,8 000 r/min、4 ℃条件下,离心20 min。将下层菌体沉淀用Buffer A重悬,经JN-3000 plus型高压均质机(广州聚能生物科技有限公司)破碎后,10 000 r/m、4℃离心30 min,取上清液过镍柱进行咪唑溶液的梯度洗脱。洗脱液经超滤浓缩后,用磷酸缓冲液(PBS,pH7.0)洗去残留的咪唑,在-80℃储存待用。
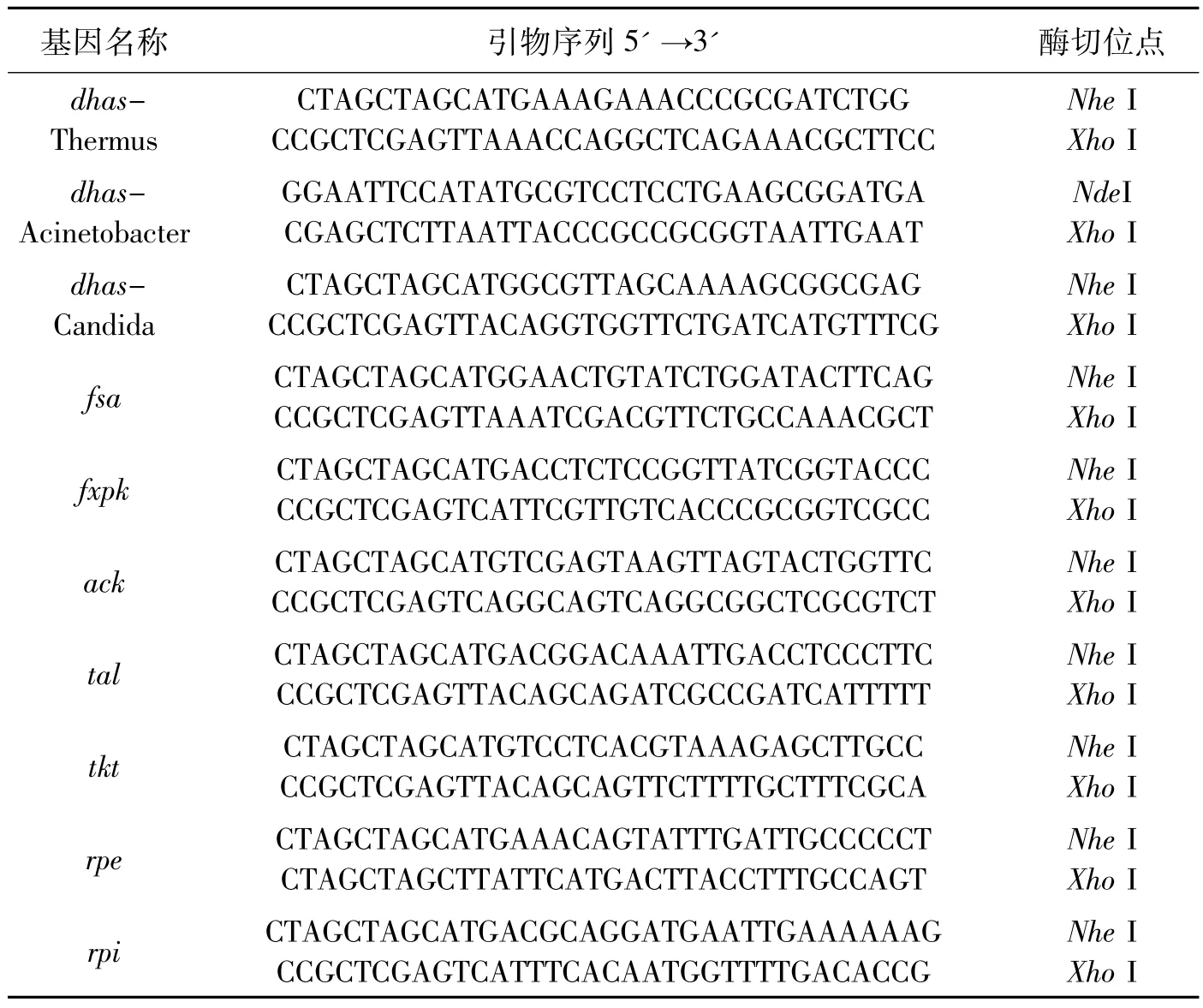
表1 引物序列Table 1 Primer sequences
1.5 甲醛利用途径中所有酶的活性测定
二羟基丙酮(Dha)合成酶(Dhas)酶活检测方法[16]。200 μL反应体系包含 50 mmol/L 磷酸缓冲液(pH 7.0)、0.5 mmol/L X5P、2 mmol/L FALD、0.16 mmol/L NADH、5 mmol/L MgCl2、0.5 mmol/L硫胺素焦磷酸(TPP)、13 U磷酸三糖异构酶和0.1 U甘油磷酸脱氢酶。在30℃条件下,通过加入10 μL适当稀释的Dhas酶液起始反应,检测OD340。
6-磷酸果糖酶(Fsa)酶活检测方法。200 μL反应体系包含50 mmol/L磷酸盐缓冲液(pH 7.0)、50 mmol/L DHA、3 mmol/L G3P、0.5 mmol/L NADP+、偶联磷酸葡萄糖异构酶(Pgi)和6-磷酸葡萄糖脱氢酶(Zwf),在30℃条件下,通过加入10 μL适当稀释的Fsa酶液起始反应,检测OD340。
磷酸转酮酶(Fxpk)酶活检测方法。200 μL反应体系包含 50 mmol/L磷酸缓冲液(pH 7.0)、5 mmol/L MgCl2、0.2 mmol/L NADP+、1 mmol/L TPP、0.2 mmol/L ADP、20 mmol/L Glucose、30 μg Ack、2 U葡萄糖激酶(Glk)、2 U Zwf和10 mmol/L F6P(或5-磷酸核糖(R5P)10 mmol/L,5-磷酸核糖异构酶(Rpi)、5-磷酸核糖差向异构酶(Rpe)各 10 μg)。在30℃条件下,通过加入10 μL适当稀释的Fxpk酶液起始反应,检测OD340。
乙酸激酶(Ack)酶活检测方法。200 μL反应体系包含 50 mmol/L磷酸盐缓冲液(pH 7.0)、5 mmol/L ADP、5 mmol/L乙酰磷酸(AcP)、1 mmol/L NADP+、5 mmol/L 葡萄糖、0.5 U Glk、0.5 U Zwf。在30℃条件下,通过加入10 μL适当稀释的Ack酶液起始反应,检测OD340。
Rpi、Rpe、转醛醇酶(Tal)和转酮醇酶(Tkt) 酶活检测方法。200 μL反应体系包含50 mmol/L磷酸盐 buffer(pH 7.0)、5 mmol/L R5P、5 mmol/L MgCl2、1 mmol/L TPP、0.26 mmol/L NADP+、0.4 U Zwf、0.4 U Pgi、260 μg Tal、8 μg Tkt、14 μg Rpe 和 2 μg Rpi。在30℃条件下,通过加入10 μL适当稀释的相应酶液起始反应,检测OD340(在测Rpi、Rpe、Tal和Tkt酶活时调整各自适当酶量)。
1.6 途径中相关代谢物的检测方法
1)NADH和NADPH浓度的检测。利用Tecan公司Infinite M200 PRO多功能酶标仪进行在线测定,紫外检测波长为340 nm;
2)乙酸浓度的检测。乙酸浓度利用乙酸试剂盒(K-ACET,Megazyme公司)检测;
3)甲醛浓度的检测。甲醛浓度通过Nash[17]显色法检测;
4)F6P浓度的检测。样品经甲氧铵盐酸盐及硅烷化试剂衍生后,利用GC-MS进行检测。气质联用色谱仪为Agilent 7890A GC/7200 Q-TOF MS,色谱柱为 DB-5MS(30 m × 0.25 mmol/L,0.25 μm),利用Agilent Mass Hunter B.07.00软件进行样品分析。
2 结果与讨论
2.1 基于模型的甲醛利用新途径设计
在数据库中下载所有反应,其中包含反应名称、反应方程及可逆性信息,在上述信息基础上,去除了末端反应(即任何条件下都不参与代谢的反应),构建了包含6 636个反应的代谢网络模型。以甲醛为底物,以乙酸为优化目标,利用该模型进行通量平衡分析,模型模拟约束条件设定甲醛为唯一底物,吸收速率设为2 mmol/(g·h)(以细胞干质量计),氮、硫、磷源不受限制。预测出了两条无碳损失,并且无NADH和ATP生成和消耗的新途径,如图1所示。图中箭头的上方(或右方)为催化反应的酶,图中箭头的下方(或左方)为计算得到的途径通量分布数值。从图1中通量分布结果可以看出,这两条途径的净反应都是消耗2分子甲醛生成1分子乙酸,理论碳得率达到了100%。图1(a)中预测出的途径正是Liao课题组研究的MCC途径,该途径结合了RuMP途径和非氧化糖酵解(NOG)途径[18],而图1(b)中是本文预测出的一条新途径,与MCC类似,该途径结合了XuMP途径与非氧化糖酵解(NOG)途径。两条途径的不同之处为最初固定甲醛的反应不同,MCC途径中甲醛与Ru5P结合生成H6P,进一步生成6-磷酸果糖(F6P),而在新途径中甲醛首先与5-磷酸木酮糖(Xu5P)结合,经过二羟基丙酮合成酶(Dhas)和磷酸果糖酶(Fsa)两步生成F6P,生成 F6P后两条途径都是经磷酸转酮酶(Fxpk)不可逆裂解得到 AcP和 4-磷酸赤藓糖(E4P),E4P可与F6P经过碳重排反应再生得到两分子底物Xu5P,如此往复循环继续。AcP可以经过Ack催化得到乙酸,可以通过检测乙酸验证这条体外新途径的可行性及可循环性。
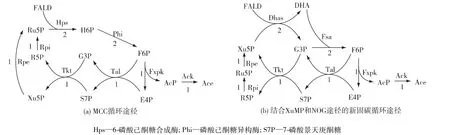
图1 基于模型预测的甲醛固碳循环途径Fig.1 Formaldehyde assimilation pathway calculated from metabolic network model
2.2 新途径中酶的选择及反应体系的确定
在设计的新途径中,从甲醛出发得到乙酸的过程一共需要8种酶。其中有6种可直接来源于大肠杆菌,其余两种分别是途径第一步醛缩酶(二羟基丙酮合成酶)和裂解F6P的磷酸转酮酶,均有多种来源可供选择。
2.2.1 磷酸二羟基丙酮合成酶(Dhas)
新途径中对甲醛固定步骤具有关键作用的是磷酸二羟基丙酮合成酶(Dhas),是甲基营养型生物体内XuMP途径中用于固定甲醛的关键一步,用来催化甲醛和Xu5P生成G3P和DHA。该途径最初发现于甲基营养型酵母,后被证实,在一些可以利用甲醇的细菌内也存在。文献报道的主要有假丝酵母和汉逊酵母来源的酵母属以及细菌来源的不动杆菌属[19-21],其中已测定的Dhas比酶活最高的微生物是假丝酵母[16]。因此本研究初步选定假丝酵母和不动杆菌这两种基因来源作为目标。此外,基于酶的热稳定性考虑,以假丝酵母来源的Dhas为参照序列通过NCBI序列比对,找到了一种与之氨基酸序列相似度仅为47%的嗜热细菌来源Dhas。分别克隆了假丝酵母(Candida boidinii),不动杆菌(Acinetobacter)和嗜热细菌(Thermus thermophilus)3种Dhas。
在30℃、50 mmol/L磷酸钾缓冲液、pH 7.0的条件下,测不同来源的Dhas的酶活,结果见表2。由表2可知:假丝酵母来源的Dhas比酶活为0.10 U/mg;不动杆菌来源的 Dhas比酶活为 0.076 U/mg;嗜热细菌来源的Dhas比酶活为0.14 U/mg(表2)。同时,可以发现3种不同来源的Dhas的比酶活相差不大,但基于比酶活最高且相对较为稳定的特点,最终选取了嗜热细菌来源的Dhas进行体系中甲醛的固定。
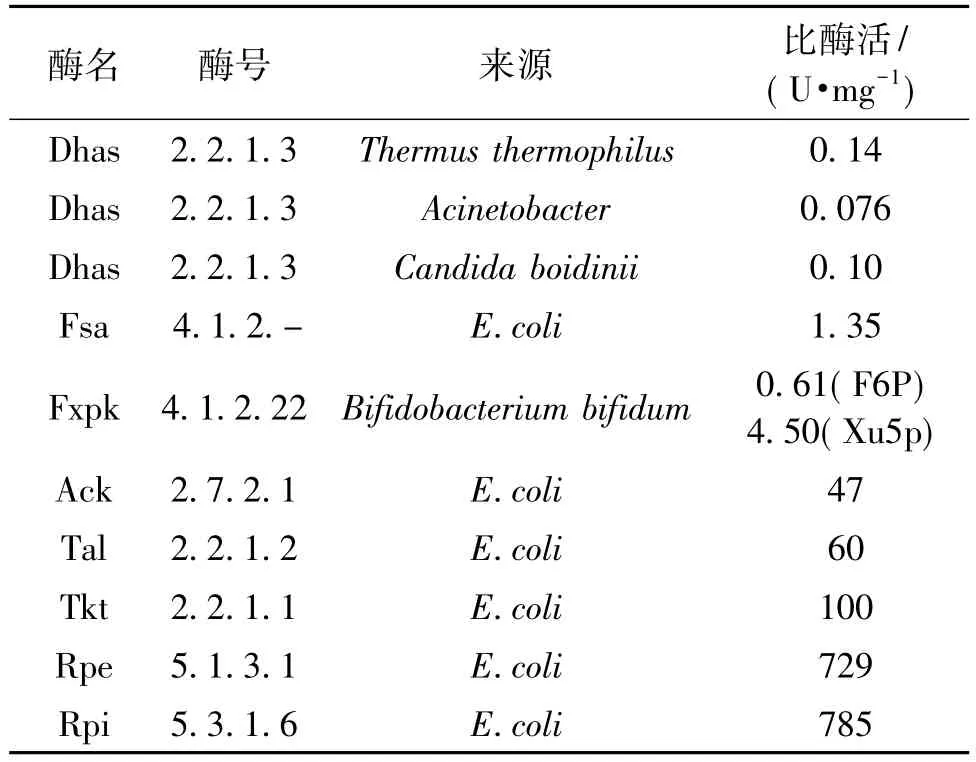
表2 反应体系所有酶的活性测定Table 2 The measured specific activities of enzymes in the pathway
2.2.2 多酶反应体系的条件确定
首先,分别测定体系中所有酶的比酶活,具体见表2。由于整条体外合成途径中较为关键的是催化第一步反应的Dhas(最适反应条件为30℃,磷酸钾缓冲液,pH7.0),比酶活相对较低,会是整个体系的限速步骤。因此我们将其最适反应条件设定为整个体系的反应条件。体系中的两种主要底物是甲醛和R5P,其中R5P用于启动反应并且在体系里可循环再生,所以加入量不需太高,为1 mmol/L。Dhas对于甲醛 Km值为 0.43 ~ 1.86 mmol/L[21],因此,甲醛加入浓度为5 mmol/L以保证Dhas催化反应速率不受甲醛浓度限制。考虑到Dhas和Fxpk两种酶会同时竞争底物Xu5P,应使体系里Fxpk的酶活略低一些。基于上述考虑,反应体系中的代谢物浓度和酶量确定为1 mmol/L R5P和5 mmol/L FALD,5 mmol/L MgCl2,1 mmol/L TPP,5 mmol/L ADP,0.2 U Dhas,0.34 U Fsa,0.1 U Fxpk(F6P 底物),44 U Rpe,22 U Rpi,1.34 U Tkt,0.78U Tal,119 U Ack,结果见图2。由图2可知,在30℃反应3 h后,乙酸生成量低于1 mmol/L,原因可能是新途径中存在的双功能酶Fxpk直接分解掉Xu5P形成AcP和G3P,使体系中Xu5P浓度很低,因此Dhas催化的反应速率也很低,无法有效利用甲醛。
2.3 两步法操作方式优化
为了验证是否是由于Fxpk酶的竞争造成乙酸产量无法提升,我们尝试在体系中采取分步操作。第一步,先加入Dhas和Fsa,单独反应1 h。这个过程积累了F6P,同时避免了底物Xu5P被Fxpk直接裂解。第二步,加入Fxpk裂解F6P以及Ack、Tal和Tkt等酶用于底物的再生,反应2 h后对反应体系中乙酸浓度进行检测,结果如图2所示。
由图2可知:与一步法相比,甲醛消耗明显增多,乙酸浓度显著上升,由不足1 mmol/L提高至2.5 mmol/L。对体系中的碳元素进行计量学分析,当Xu5P直接被Fxpk消耗时会造成底物无法循环再生,体系产生的乙酸基本来自于Xu5P直接裂解,因此最大为1 mmol/L。如果体系中的甲醛先经过Xu5P固定并依照途径循环下去,当甲醛被完全转化时,最多会得到2.5 mmol/L乙酸。而实验结果检测到两步反应实际产生了2.5 mmol/L乙酸且有少量甲醛残留,说明系统确实进行了循环,仅有少部分的乙酸是通过Xu5P直接裂解而来。因此,通过两步法的改进策略确实促进了系统的高效循环。
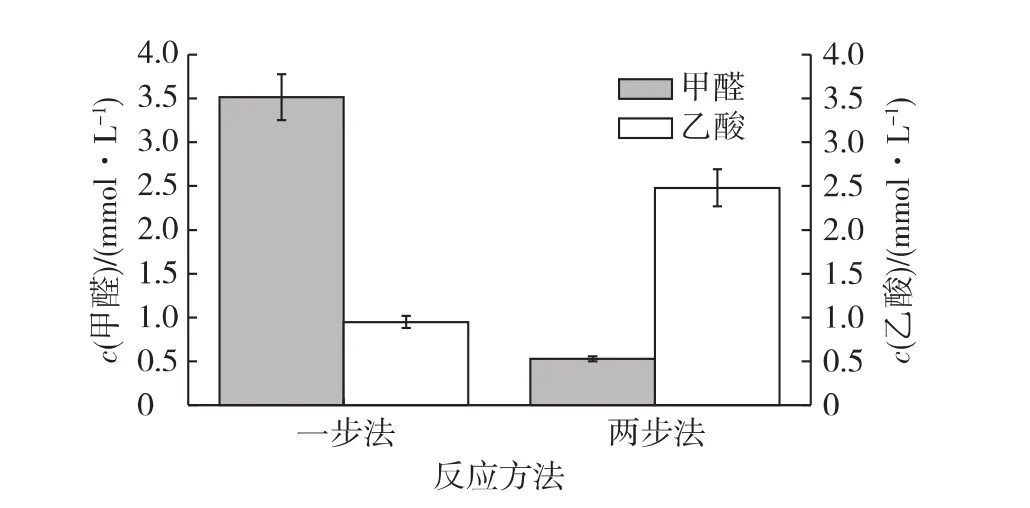
图2 一步和两步反应体系生成的乙酸和体系剩余的甲醛浓度Fig.2 Acetate concentration and the residual formaldehyde concentration from one or two step reaction systems
为了确定两步法各自的最佳反应时间,对于1 mmol/L R5P和5 mmol/L甲醛的底物反应体系进行最佳时间的探索。对于第一步反应体系的产物F6P利用GC-MS在0.5、2及4 h 3个时间点行测定,对反应第二阶段终产物乙酸浓度在 0、0.5、1.0、1.5、2.0、2.5 和3.0 h 多点取样检测。实验结果发现,第一阶段最佳反应时间为2 h;第二阶段为1.5 h。优化后,乙酸浓度可达到3.2 mmol/L,整个反应周期为 3.5 h。
2.4 体系中初始底物甲醛的浓度变化对系统循环效率的影响
在两步法操作的基础上,笔者通过考察乙酸的产量来评估体系中的甲醛浓度对系统循环效率的影响,结果如图3所示。由图3可知:在体系中残留的甲醛随初始甲醛浓度的升高而略有增加,乙酸的产量与甲醛的添加量呈正相关,证明甲醛的确在系统里得到了有效利用。
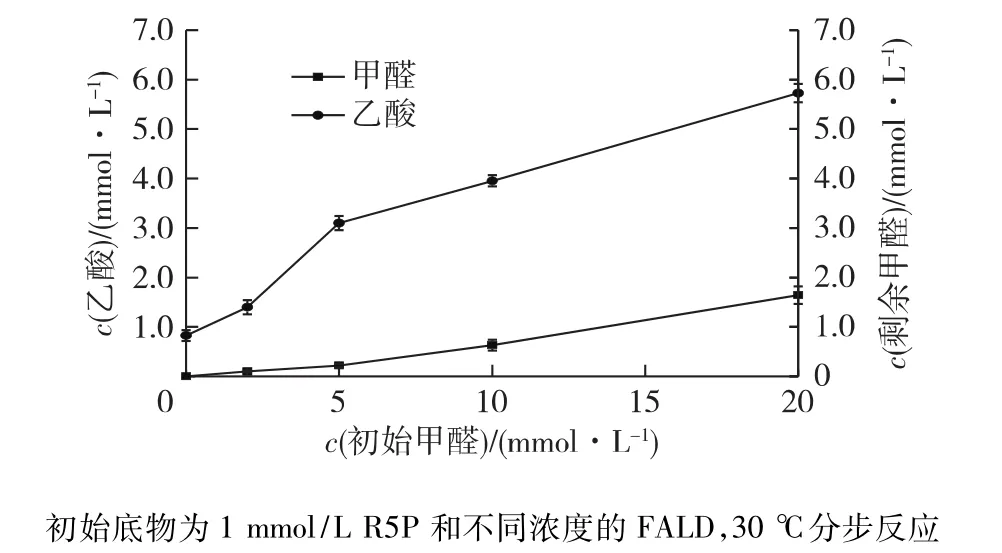
图3 随甲醛浓度提高乙酸浓度的变化情况Fig.3 Changes of produced acetate concentration at different initial FALD concentrations
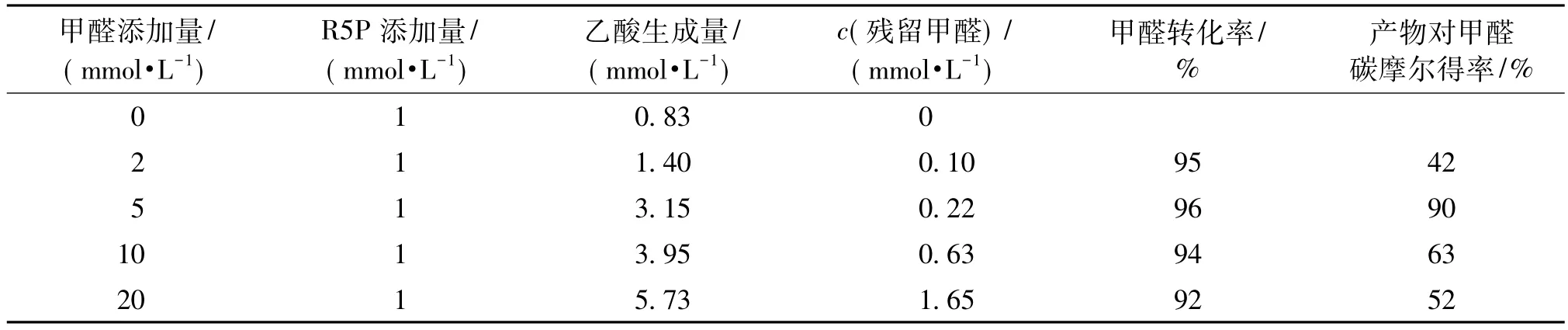
表3 不同甲醛浓度时的产物碳摩尔得率Table 3 Product carbon yield from different initial FALD concentrations
表3列出了不同甲醛浓度体系中乙酸的生成量并算出相应碳摩尔得率以及甲醛转化率。对于新途径而言,体系生成的乙酸应该全部来自底物甲醛的转化,而另一种底物R5P在体系中应该是不断循环再生且不被消耗的,然而结合图2的结果分析,体系中的乙酸还有部分来自于Xu5P的直接裂解,即来源于R5P。因此,在计算实际碳摩尔得率时,将R5P底物来源的乙酸产量按照1 mmol/L进行了扣减,最终的碳摩尔得率是针对体系中甲醛发挥作用的部分。由表3可知:底物甲醛浓度为5 mmol/L时,得率最高且可达到90%,并超过了天然存在途径的最大理论得率67%。
此外,由表3不难发现,尽管不同体系中甲醛的转化率都比较高,但随着甲醛初始添加浓度的提高,实际的碳得率却在明显下降,可能是由于高浓度甲醛会影响途径中部分酶的活性及稳定性,且随着甲醛不断循环固定的过程,途径中间代谢物会积累增多导致体系偏离稳态,导致途径中的可逆反应的平衡状态被改变,最终制约了系统的高效循环运转,体现为乙酸的转化率大大下降。因此如何进一步提高系统在高浓度甲醛条件下的稳定性及高效性,还有待进一步实验优化。
3 结论
本研究基于代谢网络模型设计的方法,构建了一条全新的从甲醛出发到乙酰辅酶A的体外催化合成的可循环途径。该途径没有碳损失,并且不依赖能量ATP和还原力。通过对乙酸的快速检测证明了途径的可行性及可循环性,因此可广泛应用于乙酸或者其他乙酰辅酶A下游衍生化合物的体外生产。利用两步法策略解决了两种酶对同一底物的竞争问题,最终,在 1 mmol/L 5-磷酸核糖与 5 mmol/L甲醛的底物浓度下,得到3.15 mmol/L乙酸,总碳摩尔得率达90%,证明甲醛在新途径中确实得到了较为高效转化利用。
[1] WHITAKER W B,JONES J A,BENNETT R K,et al.Engineering the biological conversion of methanol to specialty chemicals in Escherichia coli[J].Metab Eng,2016,39:49-59.
[2] MULLER J E,MEYER F,LITSANOV B,et al.Engineering Escherichia coli for methanol conversion[J].Metab Eng,2015,28:190-201.
[3] ANTONOVSKY N,GLEIZER S,NOOR E,et al.Sugar synthesis from CO2in Escherichia coli[J].Cell,2016,166:115-125.
[4] DIJKHUIZEN L,LEVERING P R,VRIES G E D.The physiology and biochemistry of aerobic methanol-utilizing gram-negative and gram-positive bacteria[M]∥ MURRELL J C,DALTON H.Methane and methanolutilizers.Biotechnology Handbooks.Boston:Springer,1992:149-181.
[5] KONING W D,HARDER W.Methanol-utilizing yeasts[M]∥MURRELL J C,DALTON H.Methane and methanol utilizers.Biotechnology Handbooks.Boston:Springer,1992:207-244.
[6] YURIMOTO H,KATO N,SAKAI Y.Assimilation,dissimilation,and detoxification of formaldehyde,a central metabolic intermediate of methylotrophic metabolism[J].Chem Record,2005,5(6):367-375.
[7] WHITAKER W B,SANDOVAL N R,BENNETT R K,et al.Synthetic methylotrophy:engineering the production of biofuels and chemicals based on the biology of aerobic methanol utilization[J].Curr Opin Biotechnol,2015,33:165-175.
[8] BOGORAD I W,CHEN C T,THEISEN M K,et al.Building carbon-carbon bonds using a biocatalytic methanol condensation cycle[J].Proc Natl Acad Sci USA,2014,111:15928-15933.
[9] EDWARDS J S,PALSSON B O.Systems properties of the Haemophilus influenzae Rd metabolic genotype[J].J Biol Chem,1999,274:17410-17416.
[10] BOYLE N R,MORGAN J A.Computation of metabolic fluxes and efficiencies for biological carbon dioxide fixation[J].Metab Eng,2011,13:150-158.
[11] ORTH J D,CONRAD T M,NA J,et al.A comprehensive genomescale reconstruction of Escherichia coli metabolism:2011[J].Mol Syst Biol,2011,7(1):535.
[12] BAR-EVEN A,NOOR E,LEWIS N E,et al.Design and analysis of synthetic carbon fixation pathways[J].Proc Natl Acad Sci USA,2010,107:8889-8894.
[13] LIN Z,ZHANG Y,YUAN Q,et al.Metabolic engineering of Escherichia coli for poly(3-hydroxybutyrate) production via threonine bypass[J].Microb Cell Fact,2015,14:185.
[14] ORTH J D,THIELE I,PALSSON B Ø.What is flux balance analysis?[J].Nat Biotechnol,2010,28(3):245-248.
[15] SCHELLENBERGER J,QUER,FLEMING R M,etal.Quantitative prediction of cellular metabolism with constraintbased models:the COBRA Toolbox v2.0[J].Nat Protoc,2011,6:1290-1307.
[16] BYSTRYKH L V,KONING W D,HARDER W.Dihydroxyacetone synthase from Candida boidinii KD1[J].Methods Enzymol,1990,188:435-445.
[17] NASH T.The colorimetric estimation of formaldehyde bymeans of the Hantzsch reaction[J].Biochem J,1953,55:416-421.
[18] BOGORAD I W,LIN T S,LIAO J C.Synthetic non-oxidative glycolysis enables complete carbon conservation[J].Nature,2013,502:693-697.
[19] SAKAI Y,NAKAGAWA T,SHIMASE M,et al.Regulation and physiological role of the DAS1 gene,encoding dihydroxyacetone synthase,in the methylotrophic yeast Candida boidinii[J].J Bacteriol,1998,180:5885-5890.
[20] SEO J G,PARK S W,PARK H,et al.Cloning,characterization and expression of a gene encoding dihydroxyacetone synthase in Mycobacterium sp.strain JC1 DSM 3803[J].Microbiology,2007,153:4174-4182.
[21] RO Y T,EOM C Y,SONG T,et al.Dihydroxyacetonesynthase from a methanol-utilizing carboxydobacterium,Acinetobacter sp.strain JC1 DSM 3803[J].J Bacteriol,1997,179:6041-6047.
(责任编辑 荀志金)
A computationally designed pathway for one carbon compounds utilization and its in vitro construction
CHEN Yang,YANG Xue,YUAN Qianqian,LUO Hao,LI Peishun,MA Hongwu
(Tianjin Institute of Industrial Biotechnology,Chinese Academy of Sciences,Tianjin 300308,China)
C1 compounds can be widely used in industrial biotechnology as low-cost alternative substrates to sugars.In normal pathways for the synthesis of acetyl coenzyme A derived products from C1 compound,one third of the carbon atoms from the substrate is wasted as CO2,and thus the product yield is low.Therefore,it is important to find new carbon conserved synthesis pathways for acetyl-CoA derived products from C1 compounds.In this study,we built a large metabolic network based on reactions from MetaCyc database and used the flux balance analysis method to find a new pathway from formaldehyde to acetyl coenzyme A derived products.The new pathway is carbon conserved and ATP independent.We screened and purified the enzymes in the pathway and tested the pathway in vitro using acetate as the target product.The concentration of acetate produced from the system is enhanced with the increased addition of formaldehyde substrate.The carbon molar yield can achieve 90%under certain conditions,exceeding the theoretical yield of 67%from naturally existing pathways.
metabolic networks;C1 compounds;acetyl coenzyme A;new pathway;in vitro experiment
Q815
A
1672-3678(2017)05-0086-07
10.3969/j.issn.1672-3678.2017.05.011
2017-06-14
天津科技支撑项目(14ZCZDSY00060);中国科学院重点研究项目(ZDRW-ZS-2016-3)
陈 阳(1989—),女,天津人,研究方向:体外酶催化;马红武(联系人),研究员,E-mail:ma_hw@tib.cas.cn

