白花草木樨EST-SSR标记的开发与筛选
剡转转,任 艳,吴 凡,骆 凯,张代玉,闫 启,张宇飞,赵玉凤,张吉宇
(草地农业生态系统国家重点实验室 兰州大学草地农业科技学院,甘肃 兰州 730020)
植物生产层
白花草木樨EST-SSR标记的开发与筛选
剡转转,任 艳,吴 凡,骆 凯,张代玉,闫 启,张宇飞,赵玉凤,张吉宇
(草地农业生态系统国家重点实验室 兰州大学草地农业科技学院,甘肃 兰州 730020)
草木樨(Melilotus)是重要的豆科牧草之一,然而草木樨分子标记匮乏,限制了草木樨种质资源的开发与利用。本研究以白花草木樨(M.albus)转录组数据为基础,设计了18 182对草木樨EST-SSR引物,并对所开发的EST-SSR引物进行筛选。通过PCR扩增从550对EST-SSR引物中筛选得到206对白花草木樨多态性引物,共检测出679个等位基因,平均每对引物检测出2.888个基因位点。多态信息含量PIC的分布范围为0.239~0.855,平均值为0.468。206对多态性引物Nei’s基因多样性指数(H)和Shannon信息指数(I)的平均值分别为0.169和0.239。本研究开发的EST-SSR丰富了草木樨属的分子标记,为研究种质资源的遗传多样性和分子辅助育种奠定了基础。
草木樨;EST-SSR;标记开发;多态信息含量;期望杂合度;遗传距离;遗传多样性
草木樨(Melilotusspp.)为豆科一年生或二年生自花授粉或异花授粉二倍体植物[1-2],全世界约有19种,主要分布于欧洲地中海区域、东欧和亚洲,在我国主要分布于东北、华南、西南。白花草木樨(M.albus)为该属生产利用常见种之一,自交亲和率约为49%[2],具有适应性广、抗逆性强[3]、繁殖速度快、种子产量高以及固氮能力强等优良特性[4],是仅次于苜蓿(Medicagosativa)的优良豆科牧草。作为豆科牧草,草木樨的固氮能力高,有利于草田轮作,因此,发展草木樨是提高农作物产量的一项有效措施[5]。近年来,我国越来越重视草牧业的发展,提出将粮食作物、经济作物的二元结构调整为粮食作物、经济作物、饲料作物的三元结构,通过种植优质高产饲草,推进粮食作物种植向饲草料作物种植方向转变[6],草木樨是粮改饲的主要草种之一。
简单序列重复(simple sequence repeats, SSR)标记是一类由几个核苷酸(一般为1~6个)为重复单位组成的长达几十个核苷酸的串联重复序列。因具有多态性高、位点特异性、突变率高、共显性、操作简单等优点[7-10],近年来已经快速发展成为国内外广泛应用的方法[11],利用分子标记技术来研究作物种质资源间的遗传变异也越来越受到人们重视,SSR标记还可以用来鉴别品种间的亲缘关系[12]。EST-SSR(express sequence taget-simple sequence repeat)是基于表达序列标签开发的微卫星分子标记,又称表达序列标签-简单序列重复。由于EST-SSR来源于基因组中的转录区域,且在同源基因中含有保守序列,因此,与基因组SSR相比,EST-SSR具有在植物物种之间可转移性的优点。目前,EST-SSR分子标记已在多个物种中得到了应用。Zhou等[13]利用EST-SSR对老芒麦(Elymussibiricus)的遗传多样性进行了研究,推动了披碱草属植物分子遗传多样性的研究。EST-SSR分子标记已经在油菜(Brassicacampestris)[14]、茶(Camelliasinensis)树[15]等作物和紫花苜蓿[16]、高丹草(Sorghumbicolor×S.sudanenes)[17]等牧草中得到了应用。
草木樨属的研究大多集中在栽培技术[18]、化学成分[19]等方面。骆凯等[20]对从国内外引进的19份二年生草木樨种质进行了农艺性状和品质的初步评价;Di等[21]对整个草木樨属的形态学以及分子系统进化进行了研究,证实草木樨属是单系统遗传,与苜蓿、三叶草(Trifoliumrepens)的亲缘关系最近。然而目前关于草木樨分子标记开发的研究报道较少,可用于研究草木樨属植物的遗传工具和资源非常有限。2007年Winton等[22]开发了9对草木樨基因组SSR标记研究了草木樨在阿拉斯加洲入侵的起源。随后Wu等[11]采用18对SSR引物研究了草木樨属18个种的遗传多样性。本研究利用生物信息学工具,从白花草木樨转录组数据中开发设计大规模的EST-SSR分子标记,进一步筛选多态性高标记,以期为草木樨属植物的遗传资源开发与利用奠定基础。
1 材料与方法
1.1试验材料
白花草木樨的5份种质来自我国国家牧草种质资源库(北京)(表1)。其中,ZXY06P-1732和 ZXY05P-983来自俄罗斯,HB2009-153来自中国信阳,中畜-1226和ZXY07P-3150来源地不详。
1.2试验方法
1.2.1EST-SSR引物的开发与设计 由诺和公司完成白花草木樨转录组测序数据已提交到NCBI(http://www.ncbi.nlm.nih.gov/Traces/sra),采用MISA(1.0版)对Unigene进行SSR检测,识别标准为:包含1、2、3、4、5、6个碱基重复单元的EST-SSR分子标记的最小重复数分别为10、6、5、5、5、5。通过两个引物之间不发生互补,特别是在引物3′端,即使无法避免,其3′端互补碱基也应不大于两个碱基的筛选原则,利用软件Primer3[23-24](默认参数)设计EST-SSR引物。

表1 供试材料及来源信息Table 1 Origin information of the tested Melilotus germplasm
注:“—”表示信息不详。
Note: “—”means the information is unknown.
1.2.2DNA的提取 每种材料取18个单株的新鲜叶片混合,用SDS法[25]提取叶片的总DNA,将提取出来的DNA用琼脂糖凝胶电泳检测其质量,对合格样品加ddH2O稀释到50 ng·μL-1,置于-20 ℃冰箱中保存。
1.2.3引物筛选、PCR扩增及电泳 转录组测序获得的18 182对EST-SSR引物,根据引物的碱基重复类型(除一碱基之外)、片段长度(150~200 bp)、退火温度(55~60 ℃)等条件筛选得到550对EST-SSR引物用于本试验。PCR反应体系总体积为10 μL,含1.0 μL模板DNA,4 μmol·L-1正反引物各1 μL,0.1 μLDNA聚合酶,4.9 μL 2 × Reaction Mix,2 μL ddH2O。初步筛选反应程序:94 ℃预变性3 min;94 ℃变性30 s,引物最适退火温度退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸7 min;4 ℃保存。扩增产物用6.0%的变性聚丙烯酰胺凝胶电泳分离,银染。本试验使用DL500DNA Marker。
1.3数据分析
通过人工读带,根据扩增条带在相对迁移位置的有无,记数为“1”或“0”,建立原始数据矩阵,统计每个EST-SSR位点的等位基因数,计算引物的多态信息含量PIC[26]以及期望杂合度(He)。
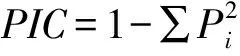
式中:Pi指第i个等位基因出现的频率。

用Popgen32软件统计分析Nei’s多样性指数和Shannon信息指数。
2 结果与分析
2.1EST-SSR引物的产生
通过SSR识别工具软件MISA(1.0版)对得到的功能基因进行位点识别后,从15 260个功能基因中共识别出了19 263个潜在的EST-SSR位点。EST-SSR引物的设计由Primer3软件完成,设计得到18 182对EST-SSR引物。其中单碱基的有11 398对,二碱基的有2 986对,三碱基的有3 461对,四碱基的有299对,五碱基的有29对,六碱基的有9对。进一步根据碱基重复类型、预期片段大小和退火温度等条件来挑选,共有符合要求的550对引物EST-SSR用于进一步筛选,其中二碱基的有136对,三碱基的有317对,四碱基的有84对,五碱基的有12对,六碱基的有1对(表2)。
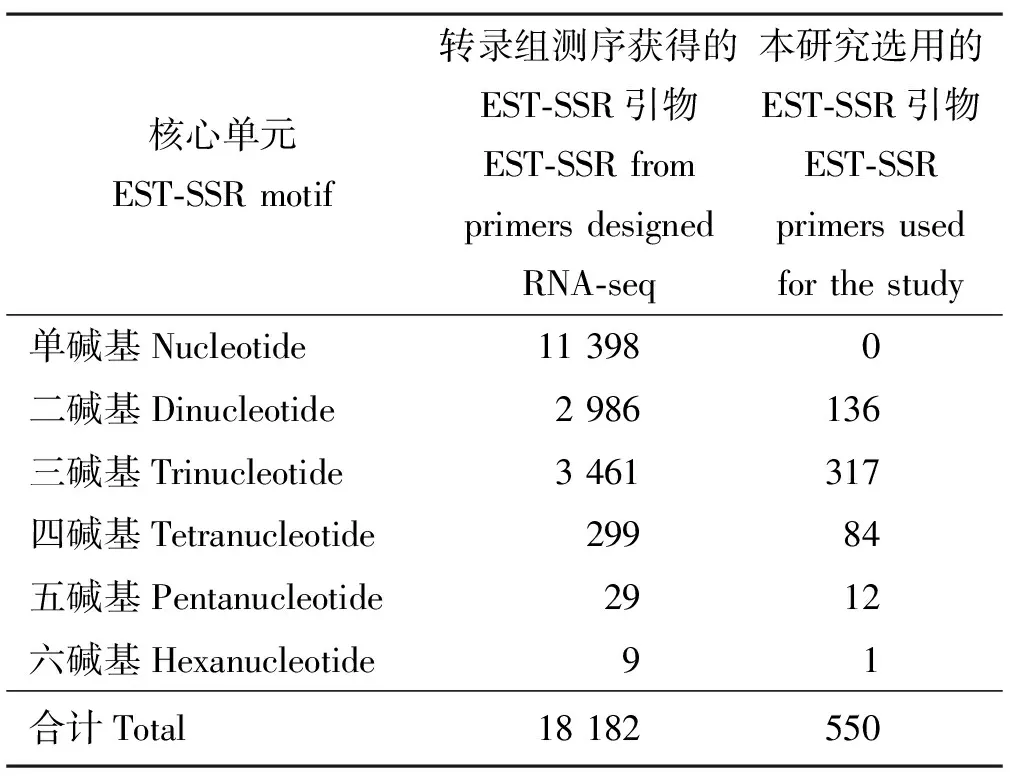
表2 EST-SSR引物的来源及碱基重复类型Table 2 The origin and variation type of the EST-SSR primers
2.2遗传多样性分析
利用白花草木樨材料对550对EST-SSR引物进行筛选验证,结果表明,共有351对引物能够扩增出条带,而剩余的199对引物在多个退火温度下都没有检测出PCR扩增产物。在能够成功扩增出条带的草木樨EST-SSR引物中,共有290对引物获得条带清晰、大小合适的扩增产物,而剩余的61对引物扩增出的PCR产物条带大小都大于或小于引物的预期片段大小。经过计算各个引物的多态性指标,在能够扩增出引物预期片段大小的草木樨EST-SSR引物中,206对引物表现出多态性(图1),而剩余的84对引物未表现出多态性(表3)。206对草木樨EST-SSR引物共检测出679个等位基因,变幅为2~6个,平均每对引物检测出2.888个基因位点,其中c9516_g1和c9897_g检测到的等位变异数最多,为6个。期望杂合度的变化范围为0.278~0.861,平均值为0.536。多态信息含量PIC的分布范围为0.239~0.855,平均值为0.468(表3)。
用550对EST-SSR引物对5份白花草木樨种质进行扩增,结果表明,多态性EST-SSR标记位点的观测等位基因数(Na)、期望等位基因数(Ne)、Nei’s基因多样性指数(H)和Shannon信息指数(I)的平均值分别为1.810、1.541、0.316和0.466。
2.3种质间遗传距离分析
白花草木樨5份种质的遗传距离介于0.455~0.580,Pop1、Pop3和Pop1与Pop4的遗传距离最大,均为0.580;Pop1与Pop2遗传距离最小,为0.455;5份种质的遗传一致度介于0.550~0.634,Pop1与Pop2之间的遗传一致度最大,为0.634(表4),表明这两份种质间的遗传相似性最高。Pop1、Pop3和Pop1与Pop4的遗传一致度最小,均为0.556,表明这两份种质间的遗传相似性最低。
3 讨论与结论
分子标记是作物分子遗传育种研究的重要工具之一。在众多的分子标记中,SSR已被广泛应用于种质遗传多样性的鉴定、分析以及种质遗传图谱的构建等研究中[27]。SSR分子标记已经在藜麦(Chenopodiumquinoa)[28]、小麦(Triticumaestivum)[29]等主要农作物的种质资源研究中得到了广泛应用,但在草木樨等豆科作物上,由于开发的分子标记较少,检测到的SSR分子水平的多态性较少,其应用受到了限制。近年来,通过生物信息学手段利用公共数据库中丰富的基因组和转录组序列发展分子标记成为开发SSR及其它类型分子标记的重要途径[30-32]。本研究通过NCBI数据库白花草木樨转录组测序设计出18 182对EST-SSR引物。并通过草木樨5份种质对550对EST-SSR引物进行筛选,共检测出679个等位基因,变幅为2~6个,平均每个位点2.888个。多态性信息含量PIC的分布范围为0.239~0.855,平均值为0.468。Nei’ s基因多样性指数、Shannon信息指数的平均值分别为0.169、0.239。陈立强和师尚礼[16]用15对SSR引物在42份紫花苜蓿材料中共检测到231条扩增带,其中163条具有多态性,Nei’ s基因多样性指数和Shannon信息指数(I)的平均值分别为0.210 0和0.326 2。Wu等[11]利用18对SSR引物在草木樨18个种共50份种质中共检测到287个等位基因,平均每个位点15.94个,多态信息含量PIC 的分布范围为0.71~0.93,平均值为0.87。Winton等[22]开发出的9对草木樨基因组SSR在白花草木樨29个种质中共检测到36个等位基因,平均每个位点4个。本研究所表现出的引物多态信指数相对较低,原因可能有:一是EST编码功能基因,序列保守性高,EST-SSR的多态性低于基因组SSR[33-34]。二是本研究所选的材料是草木樨属种内的材料,所选材料间的亲缘关系较近,不易得到多态性高的引物。
本研究从转录组测序获得18 182对EST-SSR引物,根据引物的碱基重复类型、片段长度、退火温度等条件筛选得到550对EST-SSR引物,再次筛选,共得到290对有扩增产物的引物,其中206对引物具有多态性。结果表明,白花草木樨EST-SSR含有丰富的SSR位点,本研究开发的EST-SSR丰富了草木樨属EST-SSR分子标记,为研究种质资源的遗传多样性和分子辅助育种奠定了基础。
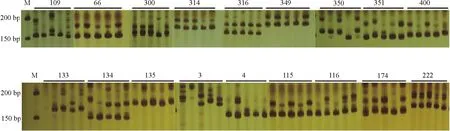
图1 部分引物对5份白花草木樨种质的扩增结果Fig. 1 Amplification products from five Melilotus albus accessions using select primer pairs
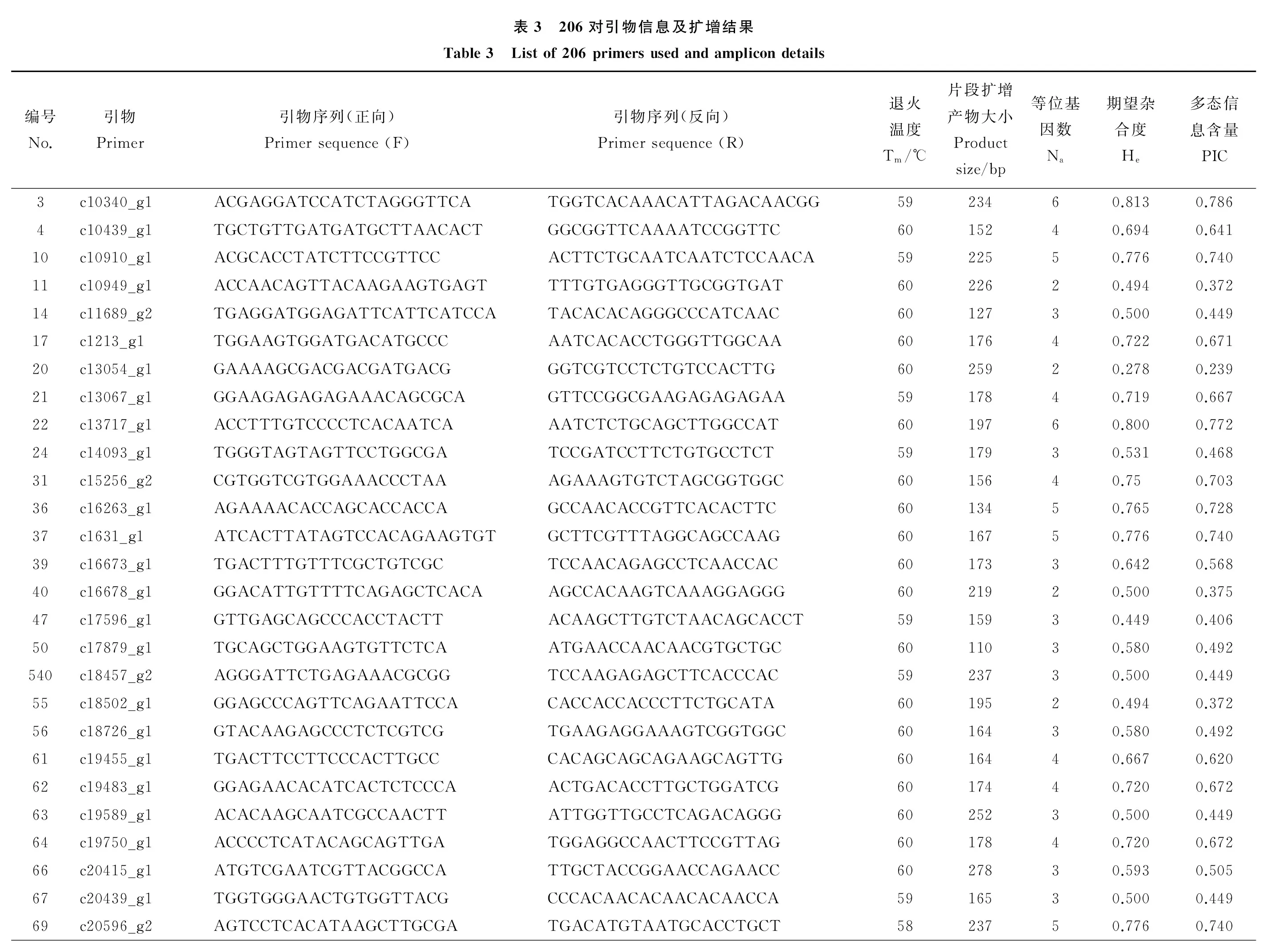
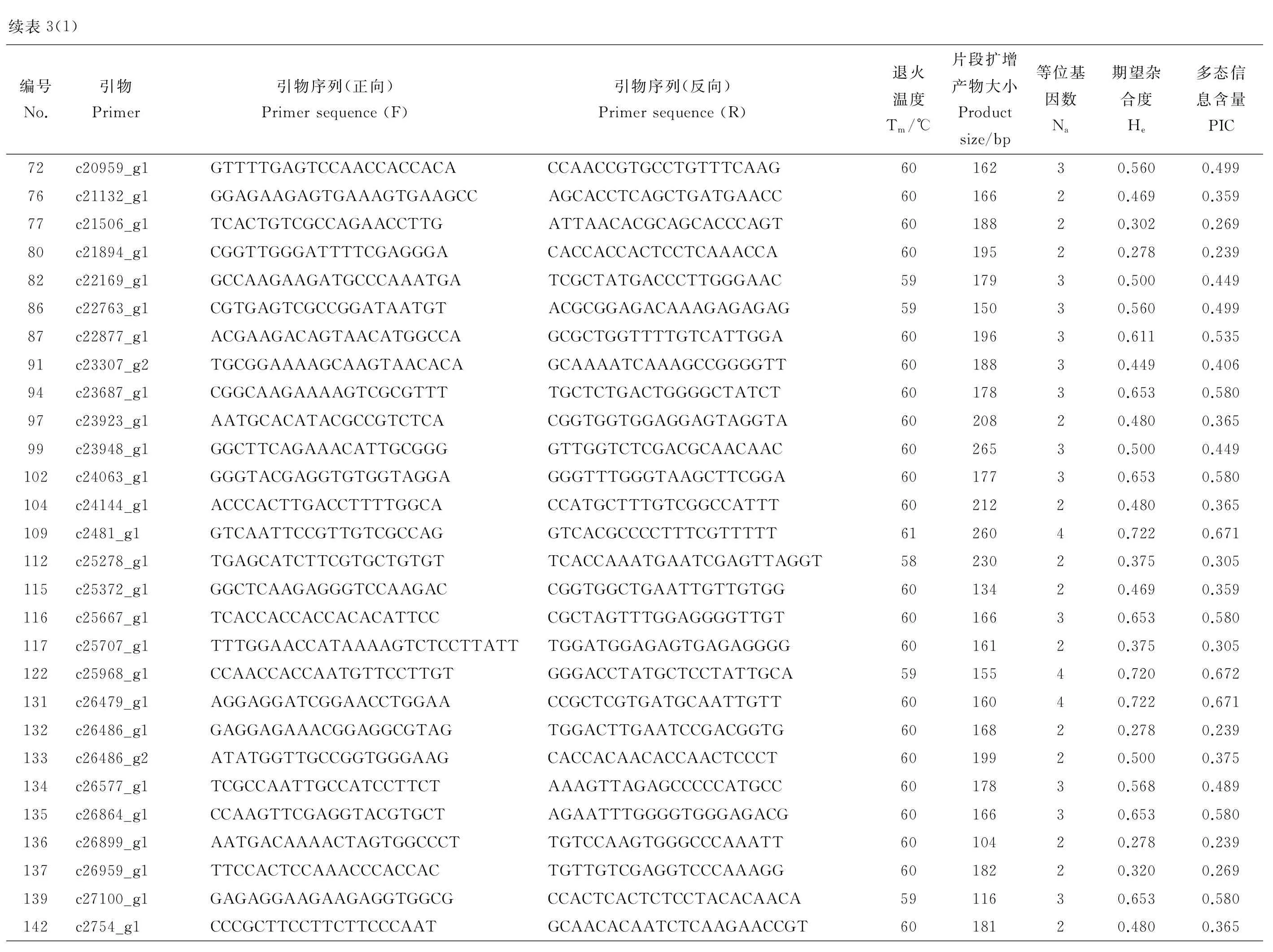

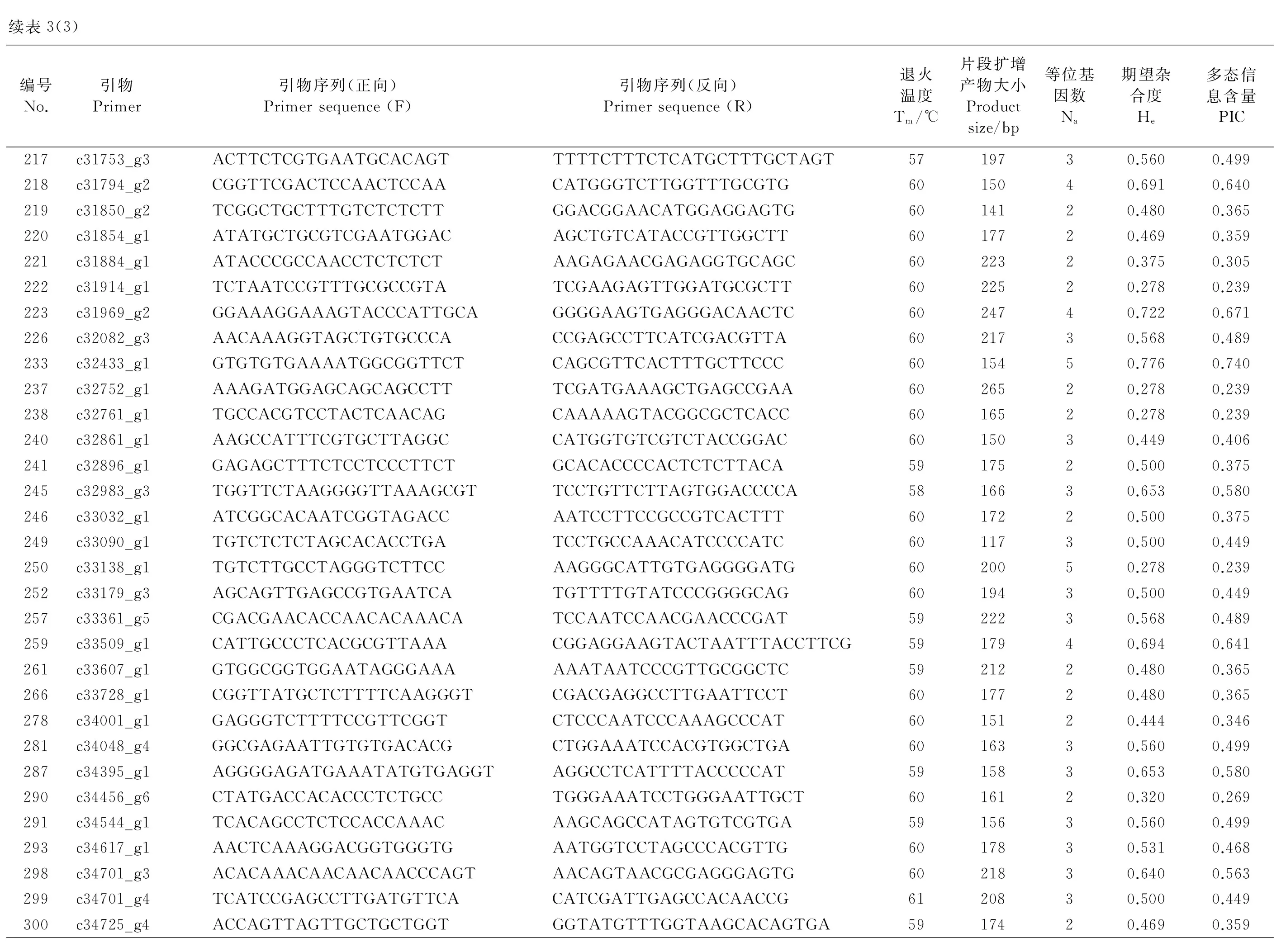


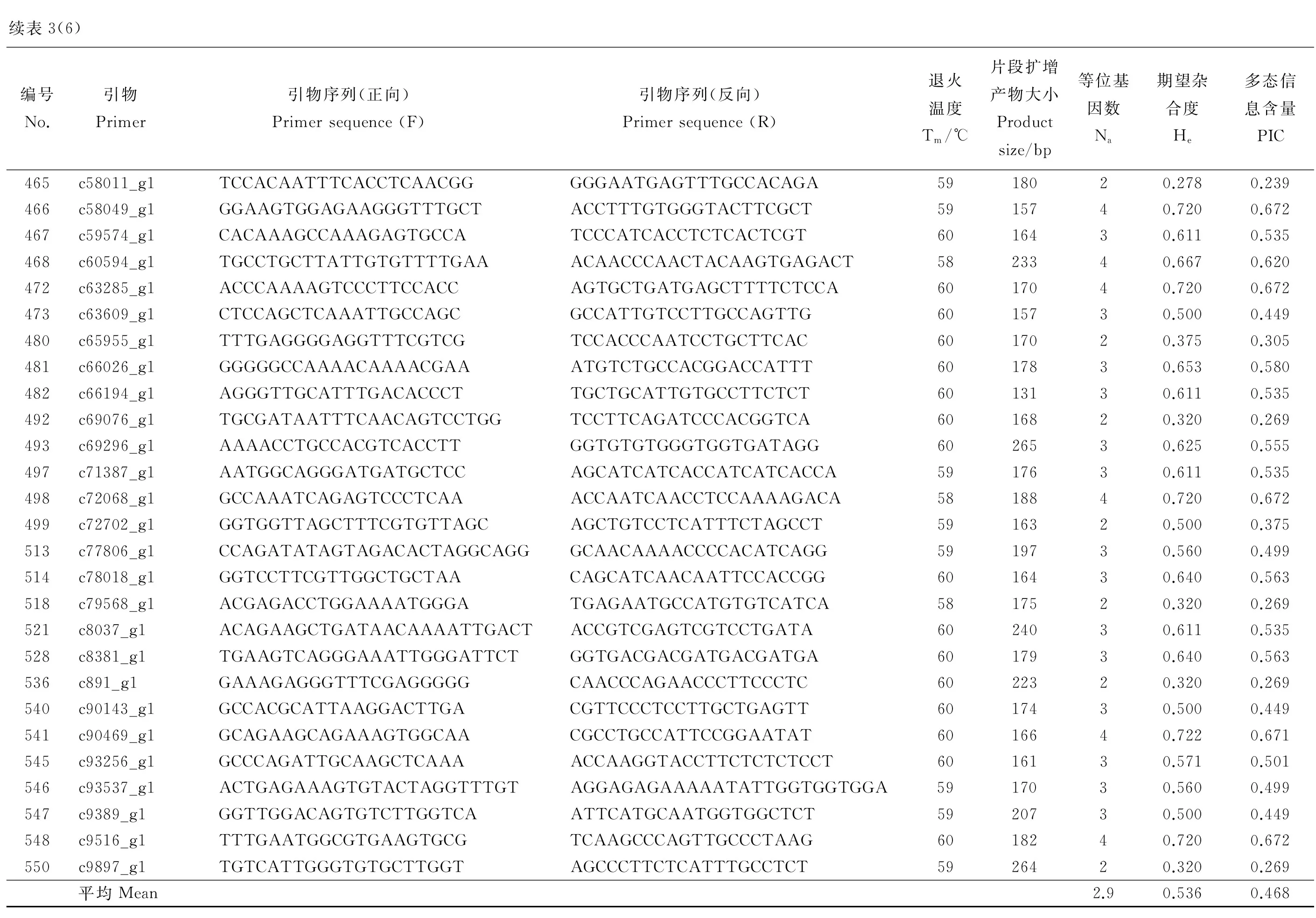

表4 5份种质的Nei’ s遗传一致度和遗传距离Table 4 Nei’ s genetic similarity coefficient and genetic distance between five accessions
注:对角线右上方为遗传一致度,对角线下方为遗传距离。
Note:The upper right of the diagonal is the genetic consistency, and below the diagonal is the genetic distance.
References:
[1] 汪永平,骆凯,胡小文,马福成,田小飞,张宝林,塔拉腾,刘晓燕,张吉宇.PEG和NaCl胁迫对草木樨种子萌发和幼苗生长的影响.草业科学,2016,33(6):1174-1182. Wang Y P,Luo K,Hu X W,Ma F C,Tian X F,Zhang B L,Talateng,Liu X Y,Zhang J Y.Effects of PEG and NaCl stress on seed germination,seedling growth ofMelilotusaccession.Pratacultural Science,2016,33(6):1174-1182.(in Chinese)
[2] Brenner D M.Methods forMelilotusgermplasm regeneration.Plant Genetic Resources Newsletter,2005,141:51-55.
[3] Luo K,Jahufer M Z Z,Wu F,Di H Y,Zhang D Y,Meng X C,Zhang J Y,Wang Y R.Genotypic variation in a breeding population of yellow sweet clover (Melilotusofficinalis).Frontiers in Plant Science,2016,7:972.
[4] 丛建民,陈凤清,孙春玲.草木樨综合开发研究.安徽农业科学,2012,40(5):2962-2963. Cong J M,Chen F Q,Sun C L.Study on comprehensive development ofMelilotussuaverolensL.Journal of Anhui Agricultural Science,2012,40(5):2962-2963.(in Chinese)
[5] 马丽.浅谈草木樨的综合利用.新疆畜牧业,2005(4):56-57.
[6] 陈志敏.“粮改饲”推进草食畜牧业发展.中国畜牧业,2016(18):71-72.
[7] 黄映萍.DNA分子标记研究进展.中山大学研究生学刊:自然科学·医学版,2010,31(2):27-36. Huang Y P.Progress of studies on the molecular marker.Journal of the Graduates:Natural Sciences·Medicine,2010,31(2):27-36.(in Chinese)
[8] 段永红,渠云芳.遗传标记在植物研究中的应用.北京:中国农业科学技术出版社,2010:210-221.
[9] Kaneko Y,Lian C,Watanabe S,Shimatani K,Sakio H,Noma N.Development of microsatellites inMachilusthunbergii(Lauraceae),a warm-temperate coastal tree species in Japan.American Journal of Botany,2012,99(7):265-267.
[10] Jun T H,Michel A P,Mian M A.Development of soybean aphid genomic SSR markers using next generation sequencing.Genome,2011,54(5):360-367.
[11] Wu F,Zhang D Y,Ma J X,Luo K,Di H Y,Liu Z P,Zhang J Y,Wang Y R.Analysis of genetic diversity and population structure in accessions of the genusMelilotus.Industrial Crops and Products,2016,85:84-92.
[12] 李为民,王宇超,黎斌,张燕,卢元,周元福,柏国清,丛晓峰,李思峰.陕西省野生大豆种质资源的SSR 遗传多样性研究.中国农学通报,2015,31(24):99-105. Li W M,Wang Y C,Li B,Zhang Y,Lu Y,Zhou Y F,Bai G Q,Cong X F,Li S F.Analysis of genetic diversity ofGlycinesojagermplasm resources in Shaanxi Province.Chinese Agricultural Science Bulletin,2015,31(24):99-105.(in Chinese)
[13] Zhou Q,Luo D,Ma L C,Xie W G,Wang Y,Wang Y R,Liu Z P.Development and cross-species transferability of SSR markers in Siberian wildrye (ElymussibiricusL.) using Illumina sequencing.Scientific Reports,2016,6:20549.
[14] 李小白,张明龙,崔海瑞.油菜EST-SSR标记的建立.分子细胞生物学报,2007,40(2):137-144. Li X B,Zhang M L,Cui H R.Data mining for SSRs in ESTs and development of EST-SSR marker in oilseed rape.Journal of Molecular Cell Biology,2007,40(2):137-144.(in Chinese)
[15] 陈熙,李佼,张羽,张颜青,徐凯明.基于EST-SSR的陕西茶树资源遗传多样性分析.四川农业大学学报,2016,34(3):322-327. Chen X,Li J,Zhang Y,Zhang Y Q,Xu K M.Genetic diversity analysis of tea in Shaanxi Province based on EST-SSR.Journal of Sichuan Agricultural University,2016,34(3):322-327.(in Chinese)
[16] 陈立强,师尚礼.42份紫花苜蓿种质资源遗传多样性的SSR分析.草业科学,2015,32(3):372-381. Chen L Q,Shi S L.Genetic diversity among 42 alfalfa accessions revealed by SSR makers.Pratacultural Science,2015,32(3):372-381.(in Chinese)
[17] 温莹,逯晓萍,任锐,米福贵,韩平安,薛春雷.高丹草EST-SSR标记的开发及其遗传多样性.遗传,2013,35(2):225-232. Wen Y,Lu X P,Ren R,Mi F G,Han P A,Xue C L.Development of EST-SSR marker and genetic diversity analysis inSorghumbicolor×Sorghumsudanenes.Hereditas,2013,35(2):225-232.(in Chinese)
[18] Conn J S,Seefeldt S S.Invasive white sweetclover(Melilotusofficinalis) control with herbicides,cutting,and flaming.Invasive Plant Science and Management,2009(2):270-277.
[19] Quijano-celis C E,Pino J A.Chemical composition of the leaves essential oil ofMelilotusofficinalis(L.) Pallas from Colombia.Journal of Essential Oil Bearing Plants,2010,13(3):313-315.
[20] 骆凯,狄红艳,张吉宇,王彦荣,李治钱.19份草木樨种质农艺学与品质性状初步评价.草业科学,2014,12(3):235-237. Luo K,Di H Y,Zhang J Y,Wang Y R,Li Z Q,Preliminary evaluation of agronomy and quality traits of nineteenMelilotusaccessions.Pratacultural Science,2014,12(3):235-237.(in Chinese)
[21] Di H Y,Duan Z,Luo K,Zhang D Y,Wu F,Zhang J Y,Liu W X,Wang Y R.Interspecific phylogenic relationships within genusMelilotusbased on nuclear and chloroplast DNA.PLoS One,2015,10(7):e0132596.
[22] Winton L M,Krohn A L,Conn J S.Microsatellite markers for the invasive plant species white sweetclover (Melilotusalba) and yellow sweetclover (Melilotusofficinalis).Molecular Ecology Notes,2007,7(6):1296-1298.
[23] Liu Z,Chen T,Ma L,Zhao Z,Zhao P X,Nan Z.Global transcriptome sequencing using the Illumina platform and the development of EST-SSR markers in autotetraploid alfalfa.PLoS One,2013,8(12):e83549.
[24] Zheng X,Pan C,Diao Y,You Y,Yang C,Hu Z.Development of microsatellite markers by transcriptome sequencing in two species ofAmorphophallus(Araceae).BMC Genomics,2013,14(1):490.
[25] 单志,吴宏亮,李成磊,陈惠,吴琦.改良SDS法提取多种植物基因组DNA研究.广东农业科学,2011,38(8):113-115. Shan Z,Wu H L,Li C L,Chen H,Wu Q.Improved SDS method for general plant genomic DNA extraction.Guangdong Agricultural Sciences,2011,38(8):113-115.
[26] 杨凯敏,李贵全,郭数进,乔玲.大豆自然群体SSR标记遗传多样性及其与农艺性状的关联分析.核农学报,2014,28(9):1576-1584. Yang K M,Li G Q,Guo S J,Qiao L.Genetic diversity and association analysis of agronomic traits with SSR in a natural population of soybean cultivars.Journal of Nuclear Agricultural Sciences,2014,28(9):1576-1584.(in Chinese)
[27] 吴硕,傅建敏,乌云塔娜,梁玉琴,李芳东.柿EST-SSR引物的开发及筛选.经济林研究,2012,30(3):27-31. Wu S,Fu J M,Wuyuntana,Liang Y Q,Li F D.Development and screening of EST-SSR makers inDiospyroskakiThunb.Nonwood Forest Research,2012,30(3):27-31.(in Chinese)
[28] 张体付,戚维聪,顾闽峰,张晓林,李坦,赵涵.藜麦EST-SSR的开发及通用性分析.作物学报,2016,42(4):492-500. Zhang T F,Qi W C,Gu M F,Zhang X L,Li T,Zhao H.Exploration and transferability evaluation of EST-SSRs in quinoa.Acta Agronomica Sinica,2016,42(4):492-500.(in Chinese)
[29] 朱振东,贾继增.小麦SSR标记的发展及应用.遗传,2003,25(3):355-360. Zhu Z D,Jia J Z.Microsatellite marker development and application in wheat genetics and breeding.Hereditas,2003,25(3):355-360.(in Chinese)
[30] 张体付,葛敏,韦玉才,赵涵.玉米功能性Insertion/Deletion(InDel)分子标记的挖掘及其在杂交种纯度鉴定中的应用.玉米科学,2012,20(2):64-68. Zhang T F,Ge M,Wei Y C,Zhao H.Discovery for maize function Insertion/Deletion (InDel) polymorphic marker and its implication in purity identification of maize hybrid seeds.Journal of Maize Sciences,2012,20(2):64-68.(in Chinese)
[31] 戚维聪,程计华,黄邦全,李坦,林峰.基于海甘蓝RNA-Seq序列开发EST-SSR分子标记.江苏农业学报,2014,30:997-1002. Qi W C,Cheng J H,Huang B Q,Li T,Lin F.Development and characterization of EST-SSR markers derived from RNA-Seq inCrambeabyssinica.Jiangsu Journal of Agricultural Sciences,2014,30:997-1002.(in Chinese)
[32] 束永俊,李勇,吴娜拉胡,柏锡,才华,纪巍,朱延明.大豆EST-SNP的挖掘、鉴定及其CAPS标记的开发.作物学报,2010,36:574-79. Shu Y J,Li Y,Wu N L H,Bai X,Cai H,Ji W,Zhu Y M.Mining and identification of SNP from EST sequences and conversion of CAPS markers in soybean.Acta Agronomica Sinica,2010,36:574-579.(in Chinese)
[33] Chabane K,Ablett G A,Cordeiro G M,Valkoun J,Henry R J.EST versus genomic derived microsatellite markers for genotyping wild and cultivated barley.Genetic Resources and Crop Evolution,2005,52:903-909.
[34] Cho Y G,Ishii T,Temnykh S,Chen X,Lipovich L,McCouch S R,Park W D,Ayres N,Cartinhour S.Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice (OryzasativaL.).Theoretical and Applied Genetics,2000,100:713-722.
(责任编辑 王芳)
ThedevelopmentandscreeningofEST-SSRmarkersinMelilotusalbus
Yan Zhuan-zhuan, Ren Yan, Wu Fan, Luo Kai, Zhang Dai-yu, Yan Qi, Zhang Yu-fei, Zhao Yu-feng, Zhang Ji-yu
(State Key Laborotary of Grassland Agro-ecosystems, College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020, China)
Melilotusis one of the most important legume forages, however, the lack of molecular markers has limited the development and utilization ofMelilotusgermplasm resources in breeding programs. In this study, 18 182 EST-SSR primers were developed and screened based on transcriptome data fromM.albus. A total of 206 primer pairs fromM.albuswere found to be polymorphic from a total of 550 pairs of EST-SSR primers screened. A total of 679 alleles were detected with an average of 2.888 alleles per locus. The polymorphism information content (PIC) values ranged from 0.239 to 0.855, with an average of 0.468. Nei’s gene diversity and Shannon’s information index for 206 pairs of polymorphic primers were 0.169, and 0.239, respectively. The EST-SSRs developed in this study enriches the EST-SSR molecular markers ofMelilotus, which will serve as the foundation for exploring the genetic diversity of germplasm resources for marker-assisted breeding.
Melilotus; EST-SSR; marker development; polymorphism information content; expected heterozygosity; genetic distance; genetic diversity
Zhang Ji-yu E-mail:zhangjy@lzu.edu.cn
S541+.903;Q943.2
:A
:1001-0629(2017)09-1802-13
10.11829/j.issn.1001-0629.2016-0643
剡转转,任艳,吴凡,骆凯,张代玉,闫启,张宇飞,赵玉凤,张吉宇.白花草木樨EST-SSR标记的开发与筛选.草业科学,2017,34(9):1802-1814.
Yan Z Z,Ren Y,Wu F,Luo K,Zhang D Y,Yan Q,Zhang Y F,Zhao Y F,Zhang J Y.The development and screening of EST-SSR markers inMelilotusalbus.Pratacultural Science,2017,34(9):1802-1814.
2016-12-28接受日期:2017-06-06
科技部“973”课题(2014CB138704);国家自然科学基金(31572453);教育部长江学者和创新团队发展计划(IRT_17R50);草业科学国家级实验教学示范中心(兰州大学);草地农业生态系统国家重点实验室开放课题(SKLGAE201702)
剡转转(1994-),女,甘肃静宁人,在读硕士生,主要从事牧草种质资源创新与利用。E-mail:yanzhzh16@lzu.edu.cn
张吉宇(1977-),男,甘肃民乐人,副教授,博导,博士,主要从事牧草育种与分子生物学研究。E-mail:zhangjy@lzu.edu.cn

