甘蓝型油菜脯氨酸合成相关同源基因的进化和差异表达分析
王翠平华学军林 彬刘爱华宁夏林业研究院种苗生物工程国家重点实验室, 宁夏银川 75000;中国科学院植物研究所, 北京 0009;中国科学院大学, 北京0009;Department of Plant Science, University of Manitoba, Winnipeg, RTN, MB, Canada
甘蓝型油菜脯氨酸合成相关同源基因的进化和差异表达分析
王翠平1华学军2,*林 彬3刘爱华4
1宁夏林业研究院种苗生物工程国家重点实验室, 宁夏银川 750004;2中国科学院植物研究所, 北京 100093;3中国科学院大学, 北京100049;4Department of Plant Science, University of Manitoba, Winnipeg, R3T2N2, MB, Canada
以异源四倍体甘蓝型油菜(Brassica napus)及其二倍体祖先白菜(B. rapa)和甘蓝(B. oleracea)为对象, 研究了脯氨酸合成途径关键酶基因P5CS和OAT的进化命运以及各自不同祖先来源的同源基因的差异表达情况。序列比对和进化分析表明, 甘蓝型油菜中P5CS基因和OAT基因和其二倍体祖先的相对应基因高度同源; 进化上, 和二倍体亲本相比甘蓝型油菜P5CS2基因发生了1个拷贝的丢失, 而OAT基因没有基因丢失现象; 半定量RT-PCR结果表明, 甘蓝型油菜中来自二倍体亲本白菜和甘蓝的P5CS2和OAT同源基因在所有检测器官中均表达, 没有发生基因沉默; 但是它们可能发生了亚功能化, 不同祖先来源的2个P5CS2同源基因存在较弱的偏向性表达, 不同器官的同源基因表达模式稍有不同; 而OAT基因明显偏向于表达来自于甘蓝祖先的同源基因, OAT的2个同源基因的不同器官表达模式基本一致; 盐胁迫处理后, 来自于甘蓝的BnaC.P5CS1.d表达量显著高于来自于白菜的BnaA.P5CS1.a, 表明盐处理条件下甘蓝型油菜偏向性表达 BnaC.P5CS1.d。甘蓝型油菜的脯氨酸合成基因 BnaA.P5CS1.a、BnaC.P5CS1.d及BnaA.P5CS2.a、BnaC.P5CS2.c的盐诱导表达模式均基本保持了亲本来源基因的特征。以上结果表明, 与二倍体祖先相比, 甘蓝型油菜中脯氨酸合成基因序列和表达模式均存在高度保守性, 这可能说明了脯氨酸积累在进化上对植物的有利性。
甘蓝型油菜; 脯氨酸合成; 多倍化; 偏向性表达; 亚功能化
在环境胁迫, 如干旱、盐、重金属、紫外线等条件下, 高等植物可以通过短时间内迅速大量地积累脯氨酸等渗透调节物质, 对自身起到保护作用,以此来增强植物对渗透胁迫的抵抗能力[1-5]。在胁迫条件下, 脯氨酸可以作为兼容性渗透调节剂、蛋白质和亚细胞结构稳定剂、活性氧清除剂和氧化还原平衡剂[6-7]。并且近年来有研究发现, 脯氨酸参与了开花及胚胎发育过程[8-11]。高等植物中, 脯氨酸由吡咯啉-5-羧酸(P5C)经吡咯啉-5-羧酸还原酶(P5CR)催化合成, 根据 P5C来源的不同, 脯氨酸合成分为谷氨酸途径和鸟氨酸途径。谷氨酸途径主要负责胁迫条件下脯氨酸的积累, 而鸟氨酸途径主要在氮充足条件下起作用, 不参与胁迫情况下脯氨酸的积累[4,12]。吡咯啉-5-羧酸合成酶(P5CS)催化谷氨酸途径的第一步,是脯氨酸合成途径的关键酶[13-14]。拟南芥中吡咯啉-5-羧酸合成酶(P5CS)由 2个基因编码(AtP5CS1, At2g39800; AtP5CS2, At3g55610), 其中AtP5CS1基因在大多数器官中表达, 受高盐、干旱和ABA胁迫的诱导, 但是在快速分裂的细胞组织中不表达[15]。而 AtP5CS2是看家基因, 在快速分裂的细胞系、分生组织和繁殖器官中表达[16]。鸟氨酸途径的前体是鸟氨酸, 通过鸟氨酸转氨酶(δ-OAT)转氨基作用催化鸟氨酸生成谷氨酸半醛(GSA), GSA自发环化成Δ1-二氢吡咯-5-羧酸(P5C), 然后通过P5C还原酶(P5CR)的作用生成脯氨酸[4]。
多倍化是植物进化过程中重要的动力, 许多作物经历了多倍化过程, 而多倍体能够为新表型的出现提供原材料, 具有进化上的优势[17]。由于染色体重组和基因重排, 多倍化之后一些基因表达模式会发生变化, 某些基因甚至会丢失[18-19]。研究发现人工合成的拟南芥四倍体的开花时间与重复基因FLOWERING LOCUS C (FLC) 的表达变化相关。拟南芥四倍体中来自Arabidopsis thaliana祖先的FLC表达上调, 来自另一个祖先亲本A. arenosa的FLC的表达下调, 这种基因表达模式的变化导致拟南芥四倍体出现比2个亲本开花时间都晚的晚花表型[19]。多倍化还可能导致基因沉默和同源基因的偏向表达。甘蓝型油菜中, 来源于二倍体祖先甘蓝的 BnPOX1能够被核盘菌(Sclerotinia sclerotiorum)的感染所诱导, 而来源于白菜的同源基因则未被诱导[20]。
芸薹属(Brassica)植物作为一种非常好的模式植物体系, 常用于研究由多倍化引起的基因丢失、沉默和偏向表达, 我们利用禹氏三角模型来解释芸薹属植物之间的进化关系[21]。甘蓝型油菜(Bassica napus)是最新形成的异源四倍体, 来源于二倍体白菜(B. rapa)和甘蓝(B. oleracea)的种间杂交, 并且经历了染色体加倍的过程[22]。而白菜、甘蓝和拟南芥起源于一个共同的祖先, 大概在 14.5~20.4百万年之前, 这个祖先分化为拟南芥和白菜、甘蓝的祖先[23]。比较物理图谱研究表明, 白菜和甘蓝的祖先经历了三倍化和基因重排的过程[24]。而白菜基因组测序数据也表明, 白菜中大多数基因具有 2~3个同源基因[25]。那么, 我们推测甘蓝型油菜中同源基因拷贝数最多应该多至6个。有报道称, 甘蓝型油菜中基因拷贝数目可能由于进化过程中基因丢失而减少, 从而使甘蓝型油菜中基因平均拷贝数为4个[26]。
胁迫条件下脯氨酸积累是一个非常保守且有效的过程, 然而迄今为止我们仍不清楚四倍体中脯氨酸代谢相关基因的进化模式及其和二倍体祖先脯氨酸代谢相关基因的关系。我们前期研究发现, 甘蓝型油菜中含有6个脯氨酸合成途径关键基因P5CS1,即该基因没有发生丢失, 并且不同器官中和胁迫诱导情况下不同来源的P5CS1基因表达模式不同[27]。本文中, 我们集中研究了甘蓝型油菜中脯氨酸合成代谢相关基因进化命运, 包括与二倍体亲本物种的基因序列差异和重复同源基因的表达模式差异。研究结果能够加深多倍化对脯氨酸代谢相关基因影响的理解, 为多倍体表型和重复基因调控的关系提供理论依据。
1 材料与方法
1.1 试验材料和无菌苗培养
甘蓝型油菜 Brassica napus L. cv. Westar, 为加拿大的双低春油菜品种。甘蓝型油菜种子由加拿大卡尔加里大学(University of Calgary)的 Maurice MOLONEY博士, Ed YEUNG博士和唐明娟博士惠赠。白菜(B. rapa cv. Zhongbai 60)和甘蓝(B. oleracea cv. Zhonggan 21)种子均购买自中国农业科学院蔬菜花卉研究所, 经实验室自交纯化。选取饱满种子, 经70%乙醇浸泡30 s, 10%次氯酸钠处理15 min, 无菌水洗涤7~8次, 转至MS培养基, 培养箱生长条件为25℃, 16/8 h光周期。
1.2 植物材料的盐胁迫处理
于不含 NaCl的 MS培养基上播种, 生长3周,转移到新的烧杯中, 在含250 mmol L–1NaCl的MS液体培养基中培养, 处理0、6、12、24和48 h后整株取样, 每种处理 3个生物学重复, 每个重复至少包含 6株幼苗。取样后, 将幼苗立即以液氮速冻保存于–80℃冰箱。
1.3 基因序列分析
白菜和甘蓝的相应基因序列由中国农业科学院蔬菜花卉研究所王晓武研究员提供, 并且通过数据库(http://www.brassica.info/; http://www.ocri-genomics. org/bolbase/index.html)检索和比对确认。甘蓝型油菜基因序列来自于甘蓝型油菜基因组序列数据库网站(http://www.genoscope.cns.fr/brassicanapus/)。应用DNAMAN 8软件(Lynnon Biosoft, Vaudreuil, Quebec, Canada)进行核酸序列比对分析。通过同源基因的基因组DNA序列比对和序列分析, 用MEGA4软件中邻位相连法构建系统进化树[28]。
1.4 基因差异表达分析
采用LiCl-SDS方法提取总RNA[29], 用DNase I (TaKaRa, 中国大连)去除总RNA中可能存在的基因组 DNA 污染, 通过 Superscript II (Invitrogen, Carlsbad, CA, USA)反转录合成单链cDNA。
半定量 RT-PCR采用 10 ng cDNA作为模板, 1×PCR缓冲液, 0.25 mmol L–1dNTPs, 0.25 μmol L–1正向和反向引物(表 1), 0.5 U的 Taq DNA聚合酶(Qiagen, Valencia, CA, USA), 用 ddH2O补足至 20 μL。PCR扩增程序为94℃预变性4 min; 94℃变性30 s, 55℃退火30 s, 72℃延伸60 s, 28个循环; 72℃延伸10 min。PCR结束后, 经1.0%琼脂糖凝胶电泳分离, 并用嗅化乙锭(EB)染色观察。
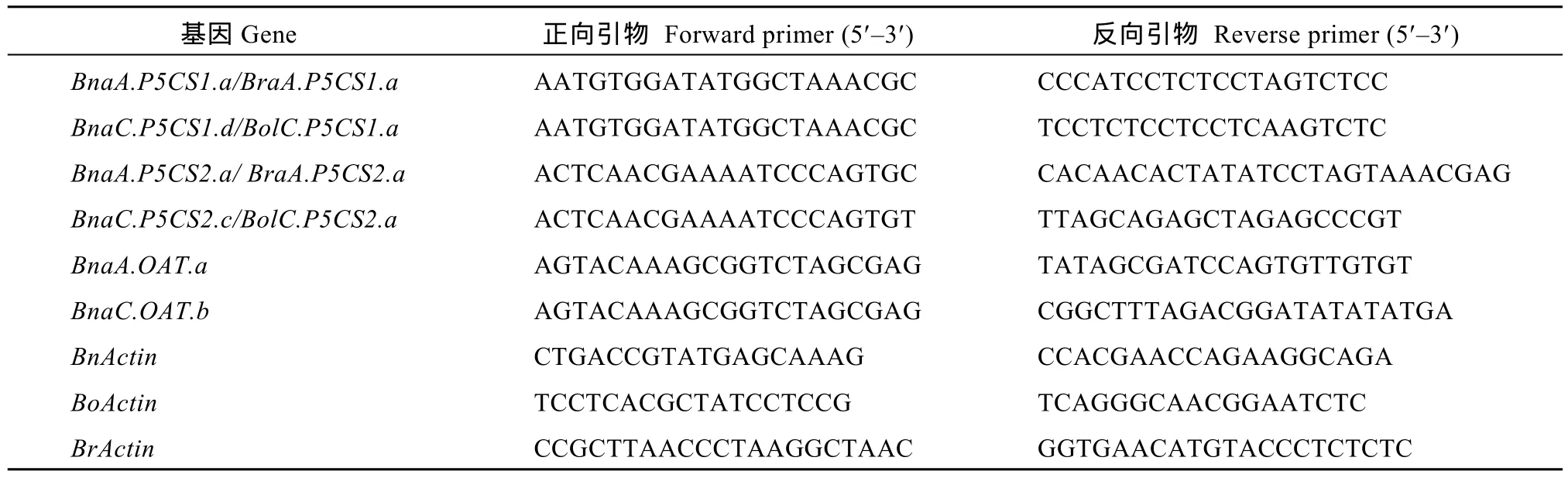
表1 半定量RT-PCR引物序列Table 1 Primers for semi-quantitive RT-PCR
2 结果与分析
2.1 甘蓝型油菜中脯氨酸合成代谢相关重复基因
2.1.1 白菜、甘蓝、油菜中的 P5CS基因 通过数据库序列比对和分析表明, 白菜中存在 2个P5CS2的同源基因(http://www.brassica.info/) (ChrA9, Bra007179; ChrA4 Bra014742), 甘蓝中存在 2个P5CS2基因 (http://www.ocri-genomics.org/bolbase/ index.html) (Bol044364, Bol044229), 甘蓝型油菜中存在 3个 P5CS2基因(ChrA8, NM_001315755.1; ChrA4, XM_013886902.1; ChrC8, XM_013800452.1) (http://www.genoscope.cns.fr/brassicanapus/)。根据系统命名法则[30], 将白菜的2个同源P5CS2基因分别命名为BraA.P5CS2.a和BraA.P5CS2.b, 这2个基因的cDNA编码区长度分别为2184 bp和2181 bp, 与BraA.P5CS2.a的cDNA编码区相比, BraA.P5CS2.b的起始密码子后缺失了3个碱基(图1-A)。将甘蓝中的2个BoP5CS2基因分别命名为BolC.P5CS2.a和BolC.P5CS2.b, 它们的 cDNA 编码区长度分别为2184 bp和2181 bp, 与BolC.P5CS2.b的编码区比较, BolC.P5CS2.a在起始密码子后发生了碱基序列缺失(图1-A)。将甘蓝型油菜的3个BnP5CS2基因分别命名为 BnaA.P5CS2.a、BnaA.P5CS2.b和 BnaC. P5CS2.c, cDNA编码区长度分别为 2184、2181和2184 bp, 与 BnaA.P5CS2.a和 BnaC.P5CS2.c相比, BnaA.P5CS2.b起始密码子之后发生了3个碱基的缺失(图1-A)。
2.1.2 白菜、甘蓝和甘蓝型油菜中OAT基因 基因组测序结果表明, 白菜中存在 1个 OAT基因(Bra025033), 位于白菜第 6染色体上, 被命名为BraA.OAT.a, 该基因的 cDNA编码区长度为 1434bp。甘蓝中存在 1个 OAT基因, 被命名为BolC.OAT.a, 该基因的编码区长度为 1431 bp。与BolC.OAT.a相比, 基因BolC.OAT.b的编码区出现3个碱基的缺失。甘蓝型油菜中存在2个BnOAT基因, 分别被命名为 BnaA.OAT.a和 BnaC.OAT.b, cDNA编码区长度分别为 1434 bp和 1431 bp, 与BnaC.OAT.b相比, BnaA.OAT.a同样出现了3个碱基的缺失(图1-B)。
2.2 脯氨酸合成酶基因的同源性和进化关系
2.2.1 P5CS基因的同源性和进化关系 甘蓝型油菜基因组中含有的3个P5CS2基因, 为研究其祖先来源, 即这 3个基因在甘蓝型油菜染色体加倍过程中来自白菜基因组还是甘蓝基因组, 我们将 3个基因分别和白菜及甘蓝的P5CS2基因的基因组序列和编码区(CDS)序列两两比对(表 2)。同时, 将甘蓝型油菜、白菜、甘蓝和拟南芥的同源P5CS2基因的基因组DNA序列作多重比对, 构建了该基因的系统发生树(图2)。

图1 白菜、甘蓝和甘蓝型油菜的同源基因部分差异序列比对Fig. 1 Alignment of partial sequence of homologous genes in B. rapa, B. oleracea, and B. napus
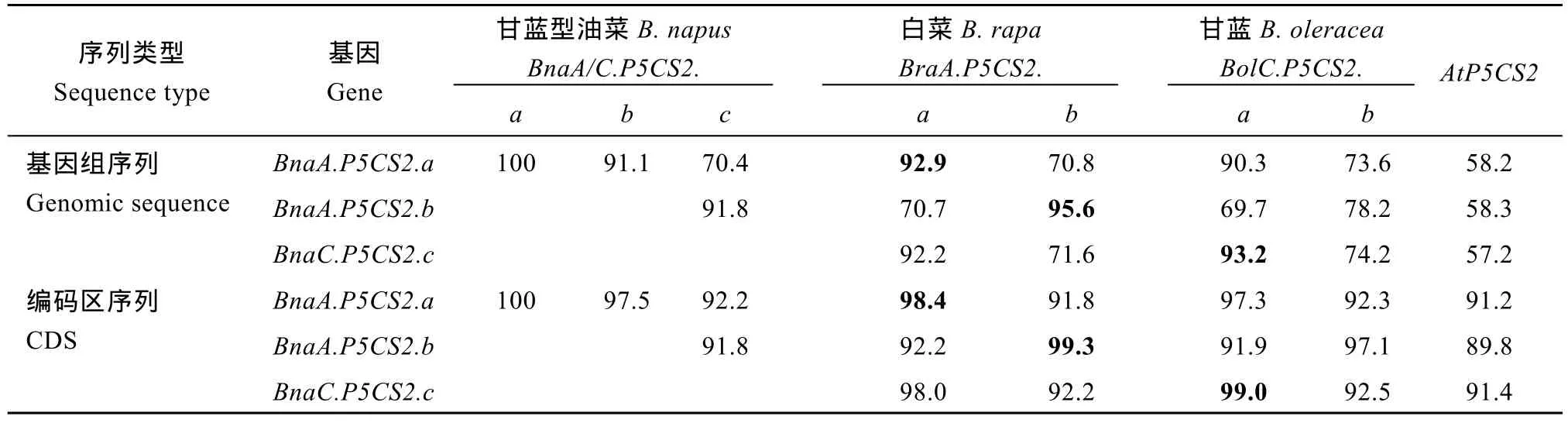
表2 甘蓝型油菜BnP5CS2基因与二倍体亲本白菜和甘蓝P5CS2基因序列相似性Table 2 Sequence identities of P5CS2 genes among B. napus, diploid ancestors B. rapa and B. oleracea (%)
BnaA.P5CS2.a与BraA.P5CS2.a, BnaA.P5CS2.b与BraA.P5CS2.b, BnaC.P5CS2.c与BolC.P5CS2.a之间的基因组DNA相似性分别为92.9、95.6和93.2,而他们之间编码区相似性更高, 分别为 98.4、99.3和 99.0, 无论是基因组序列还是编码区序列均高于同系列其他比对(表 2)。因此, 我们推断 BnaA. P5CS2.a、BnaA.P5CS2.b和BnaC.P5CS2.c可能分别来源于BraA.P5CS2.a、BraA.P5CS2.b和BolC.P5CS2.a。
而基于相对应的基因组序列的进化关系分析表明 BnaA.P5CS2.a、BnaA.P5CS2.b和 BnaC.P5CS2.c分别为白菜 BraA.P5CS2.a、BraA.P5CS2.b和甘蓝BolC.P5CS2.a的直向同源基因(图2)。
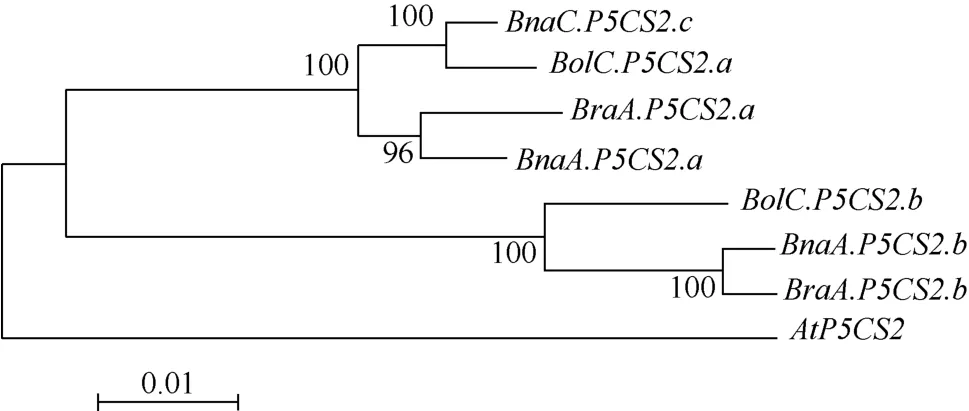
图2 芸薹属植物P5CS2基因进化关系分析Fig. 2 Phylogenetic analysis of Brassica P5CS2 genes
2.2.2 OAT基因的同源性和进化关系 甘蓝型油菜基因组中含有2个同源的OAT基因, 为了研究其祖先来源, 将其分别和白菜及甘蓝的OAT基因基于基因组序列和编码区(CDS)序列两两比对(表 3)。同时, 将甘蓝型油菜、白菜、甘蓝和拟南芥的所有已知序列的同源OAT基因的基因组DNA序列作多重比对, 构建了该基因的系统发生树(图3)。
BnaA.OAT.a和 BraA.OAT.a, BnaC.OAT.b 和BolC.OAT.a基因组 DNA序列相似性高达 95.3%和 99.6% (表3), 而他们之间编码区相似性分别为98.2和99.7 (表3), 无论是基因组序列还是编码区序列均高于同系列其他比对, 我们推测 BnaA.OAT.a和BnaC.OAT.b 可能分别来源于 BraA.OAT.a 和BolC.OAT.a。而基于相对应的基因组序列的进化关系分析也表明BnaA.OAT.a和BnaC.OAT.b分别为白菜 BraA.OAT.a和甘蓝 BolC.OAT.a的直向同源基因(图3)。
2.3 甘蓝型油菜不同器官中脯氨酸合成相关同源基因的差异表达
半定量 RT-PCR结果表明, 甘蓝型油菜的P5CS2和OAT同源基因在所有器官中均表达(图4)。说明在不同发育阶段中, 所检测的不同祖先来源的4个同源基因都没有发生基因沉默。
图4表明, 甘蓝型油菜中2个不同祖先来源的P5CS2同源基因存在较弱的偏向性表达, 甘蓝型油菜幼叶中来自白菜的拷贝(BnaA.P5CS2.a)表达较高,其他器官中2个拷贝表达量相近。同时, 这2个同源基因器官特异性表达的模式也稍有不同, BnaA.P5CS2.a在幼叶和花苞中表达量最高, 而BnaC.P5CS2.c在花和花苞中表达量最高。而甘蓝型油菜的不同祖先来源的2个OAT同源基因存在明显的偏向表达, 在所有器官中, 来自甘蓝的基因表达量高于来自白菜的拷贝。但是, 不同祖先来源的OAT的 2个同源基因的表达模式基本一致, 都是在花器官中表达量最高(图4)。
2.4 盐胁迫下甘蓝型油菜、白菜和甘蓝的脯氨酸代谢相关同源基因差异表达情况

表3 甘蓝型油菜中OAT基因的基因组片段与二倍体祖先中相对应的基因核苷酸序列相似性Table 3 Sequence identities of OAT genes between B. napus and its diploid ancestors B. rapa and B. oleracea (%)
甘蓝型油菜中 P5CS1的表达情况如图 5所示,在250 mmol L–1NaCl处理条件下, BnaA.P5CS1.a和BnaC.P5CS1.d的表达均受盐胁迫诱导, 但是BnaC.P5CS1.d的诱导更快, 基因表达量在6 h达到最大值。通过比较不同时间点的表达量还发现, 盐胁迫处理后, BnaC.P5CS1.d表达量显著高于BnaA.P5CS1.a, 表明盐处理条件下甘蓝型油菜偏向性表达BnaC.P5CS1.d。
之前的研究结果表明, 甘蓝型油菜基因BnaA.P5CS1.a来源于 BraA.P5CS1.a, 而 BnaC. P5CS1.d来源于 BolC.P5CS1.a, 所以分别检测了BraA.P5CS1.a和BolC.P5CS1.a在二倍体白菜和甘蓝中的盐胁迫诱导表达情况。如图 5所示, 白菜的BraA.P5CS1.a的表达受盐胁迫诱导, 表达量在12 h达到最大值。甘蓝的 BolC.P5CS1.a表达受盐胁迫诱导, 表达量在 6 h达到最大值。因此, BolC. P5CS1.a受盐胁迫诱导的速率快于BraA.P5CS1.a。而BnaC.P5CS1.d受盐胁迫诱导的速率也快于BnaA. P5CS1.a (图 5), 我们认为甘蓝型油菜的 BnaA. P5CS1.a和 BnaC.P5CS1.d在盐诱导基因表达速率方面均保持了其二倍体亲本来源基因的特征。
甘蓝型油菜 Bna.P5CS2.a的表达量在盐处理后2 h后开始上调, 表达水平一直保持到24 h, BnaC. P5CS2.c的表达量不受盐诱导。而二倍体祖先亲本基因中, 甘蓝的BolC.P5CS2.b表达不受盐处理的影响,白菜的BraA.P5CS2.a在盐处理后6 h和12 h微弱上调(图 5)。因此, 盐处理条件下, 甘蓝型油菜 BnaA. P5CS2.a和BnaC.P5CS2.c的盐诱导表达模式均基本保持了亲本来源基因的特征。
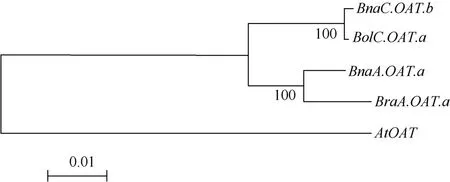
图3 芸薹属植物OAT基因进化关系分析Fig. 3 Phylogenetic analysis of Brassica OAT genes
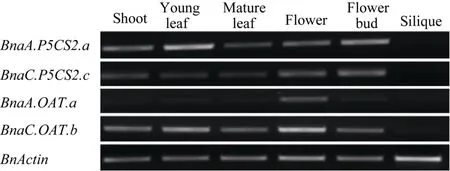
图4 甘蓝型油菜不同祖先来源的P5CS2和OAT基因在不同器官中的表达模式Fig. 4 Expression pattern for P5CS2 and OAT from different diploid ancestors in different organs of B. napus
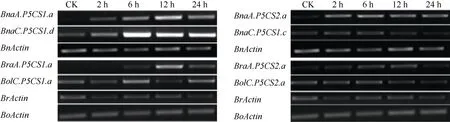
图5 250 mmol L–1NaCl胁迫下甘蓝型油菜、白菜和甘蓝的P5CS1和P5CS2的表达模式Fig. 5 Expression pattern of P5CS1 and P5CS2 in B. napus, B. rapa, and B. oleracea under 250 mmol L–1NaCl
3 讨论
3.1 甘蓝型油菜的 P5CS2基因发生了一个拷贝的丢失
甘蓝型油菜(B. napus)起源于白菜(B. rapa)和甘蓝(B. oleracea)的杂交, 并且经过了染色体加倍, 而其祖先白菜和甘蓝自身也经历了基因组三倍化和重排[22], 这样我们推测甘蓝型油菜中每个基因最多可以达到6个。然而研究发现, 由于基因丢失现象的存在, 甘蓝型油菜中同源基因的拷贝数平均为4个[26]。研究表明, 甘蓝型油菜中存在3个ALCATRAZ (ALC)基因[32], 3个 GPAT4 (sn-glycerol-3-phosphate acyltransferase 4)基因, 以及 3个来源于白菜另外 3个来源于甘蓝的6个PSY (phytoene synthase)基因[32],而我们之前的研究发现, 甘蓝型油菜中存在 6个P5CS1基因[27]。本研究中, 甘蓝型油菜中存在 3个P5CS2基因, 而其祖先白菜和甘蓝中各存在 2个P5CS2基因, 进化过程中甘蓝型油菜发生了 P5CS2基因一个拷贝的丢失, 根据序列比对和进化分析结果, 我们推测来自于二倍体祖先甘蓝的P5CS2基因的一个拷贝发生丢失。甘蓝型油菜中存在2个OAT基因, 其祖先白菜和甘蓝中各存在 1个 OAT基因,甘蓝型油菜中OAT基因没有发生丢失。
3.2 甘蓝型油菜中脯氨酸代谢相关基因与二倍体亲本来源基因高度同源
根据白菜和甘蓝全基因组测序结果, 白菜和甘蓝中P5CS1、P5CS2和OAT同源基因拷贝数都分别为3、2和1, 而甘蓝型油菜中拷贝数为6、3和2。序列比对结果表明, 甘蓝型油菜脯氨酸代谢途径相关基因均和其亲本白菜和甘蓝来源的基因高度同源, P5CS1、P5CS2和OAT同源基因和亲本来源基因的基因组 DNA同源性分别为 96%~100%、92.9%~95.6%和95.3%~99.6%。其中BnaC.P5CS1.d基因组DNA序列和亲本基因序列完全相同, 说明这些基因在甘蓝型油菜进化过程中高度保守, 即使进化上相对不保守的内含子序列也没有发生变异。这和之前的甘蓝型油菜基因克隆的相关研究报道一致, 甘蓝型油菜中克隆到3个PISTILLATA基因, 两两序列相似性为 96.49%~98.72%[33]。脯氨酸合成途径上不同基因拷贝数的不同或许说明这些基因在进化上的重要性不同。
3.3 甘蓝型油菜中脯氨酸代谢同源基因表达模式发生分化
研究认为, 重复基因在进化中会有功能丧失(non-functionalization)、亚功能化(subfunctionalization)和新功能化(neo-functionalization) 3种命运, 而亚功能化是重复基因在进化过程中的重要模式[35]。研究发现棉花(Gossypium hirsutum)中来源于不同亲本的2个乙醇脱氢酶A (adhA)在非生物胁迫处理和不同器官中均表现出表达模式的分化[35]。本研究发现, 甘蓝型油菜脯氨酸合成途径基因 BnP5CS1(BnaA.P5CS1.a 和 BnaC.P5CS1.d)和 BnP5CS2 (BnaA.P5CS2.a和BnaC.P5CS2.c)的表达模式出现分化, 表现在基因对盐胁迫的响应和在不同器官中的偏向表达。盐胁迫下, BnaA.P5CS1.a的诱导速率比BnaC.P5CS1.d快; 正常生长条件下, BnaC.P5CS1.d在幼叶中表达量最高, 而BnaA.P5CS1.a在茎中表达量较高。盐处理下, BnaA.P5CS2.a表达上调而BnaC.P5CS2.c表达不变; 正常条件下, BnaA. P5CS2.a在幼叶中表达量最高, 而BnaC.P5CS2.c在花和花苞中表达量较高。
盐处理条件下, 甘蓝型油菜中P5CS2同源基因来自于白菜的一个拷贝被诱导, 另一个来自于甘蓝的拷贝不被诱导。这与之前的过氧化物酶(POX1)的研究结果类似, 感染病菌后, 甘蓝型油菜中来自甘蓝(B. oleracea)的过氧化物酶基因表达量增加, 而来自白菜(B. rapa)的过氧化物酶基因几乎不受影响[20]。胁迫条件下这种表达模式在同源基因之间分化, 可能有利于基因功能特异性和多样性的维持, 从而提高多倍体的适应环境的能力。
与基因P5CS1和P5CS2相比, 脯氨酸合成途径的OAT基因(BnaA.OAT.a和BnaC.OAT.b)在不同器官中的表达模式没有分化, 均在花器官中表达量最高。BnaA.OAT.a和 BnaC.OAT.b在甘蓝型油菜中可能相互协调, 在发育过程中发挥相似的功能。
3.4 甘蓝型油菜BnP5CS1和BnP5CS2同源基因保留亲本来源基因的表达模式
甘蓝型油菜 BnaA.P5CS1.d受盐胁迫诱导表达上调的速率快于 BnaA.P5CS1.a, 2个基因的表达量分别在盐处理后6 h和12 h最高, 盐诱导基因表达的速率均保持了其二倍体亲本来源基因的特征。甘蓝型油菜 BnaA.P5CS2.a基因表达量受盐胁迫诱导微弱上调, 而BnaC.P5CS2.c表达不受盐处理的影响,这 2个基因均保持了亲本来源基因对盐胁迫的响应。这种现象在文献中曾有报道, Zhao等[20]比较研究了甘蓝型油菜中的两种过氧化物酶(POX1和POX2)和亲本白菜及甘蓝中基因的表达模式, 结果表明在生物胁迫条件下, 2个POX2基因在不同器官中的表达模式都和亲本中的基因一样, 而2个POX1基因在不同器官中的表达模式和亲本来源基因不一致, 发生了变化。甘蓝型油菜中的P5CS1同源基因(BnaA.P5CS1.a和BnaC.P5CS1.d)和P5CS2同源基因(BnaA.P5CS2.a和BnaC.P5CS2.c)都保持了亲本来源基因盐胁迫下的诱导模式, 说明了这些基因在多倍化过程中的功能保守性。
4 结论
甘蓝型油菜中脯氨酸代谢途径基因(P5CS和OAT)存在多个拷贝, 它们和其二倍体亲本白菜及甘蓝的对应基因序列高度相似; 和亲本相比, 甘蓝型油菜的P5CS2基因来自二倍体祖先甘蓝的一个拷贝丢失, 而 OAT基因没有发生丢失; 盐胁迫下, 甘蓝型油菜P5CS1和P5CS2基因的诱导模式都和亲本中的祖先基因诱导模式一致, 说明甘蓝型油菜进化中该基因盐诱导模式的保守性。
致谢: 感谢Calgary大学的Maurice MOLONEY博士,Ed YEUNG博士和唐明娟博士提供甘蓝型油菜(B. napus L. cv. Westar)种子, 感谢中国农业科学院蔬菜花卉研究所王晓武研究员提供白菜和甘蓝的 P5CS基因和OAT基因的相关序列信息。
[1] Kishor P B K, Hong Z L, Miao G H, Hu C A A, Verma D P S. Overexpression of Δ1-pyrroline-5-carboxylate synthetase increases proline production and confers osmotolerance in transgenic plants. Plant Physiol, 1995, 108: 1387–1394
[2] Schat H, Sharma S S, Vooijs R. Heavy metal-induced accumulation of free proline in a metal-tolerant and a nontolerant ecotype of Silene vulgaris. Physiol Plant, 1997, 101: 477–482
[3] Yang S L, Lan S S, Gong M. Hydrogen peroxide-induced proline and metabolic pathway of its accumulation in maize seedlings. J Plant Physiol, 2009, 166: 1694–1699
[4] Tavakoli M, Poustini K, Alizadeh H. Proline accumulation and related genes in wheat leaves under salinity stress. J Agric Sci Tech, 2016, 18: 707–716
[5] Dar M I, Naikoo M I, Rehman F, Naushin F, Khan F A. Proline accumulation in plants: roles in stress tolerance and plant development. In: Iqbal N, Nazar R, Khan N A, eds. Osmolytes and Plants Acclimation to Changing Environment: Emerging Omics Technologies. New Delhi: Springer India Press, 2016. pp 155–166
[6] Chaves M M, Flexas J, Pinheiro C. Photosynthesis under drought and salt stress: regulation mechanisms from whole plant to cell. Ann Bot, 2009, 103: 551–560
[7] Wu H H, Zou Y N, Rahman M M, Ni Q D, Wu Q S. Mycorrhizas alter sucrose and proline metabolism in trifoliate orange exposed to drought stress. Sci Rep, 2017, 7: 42389
[8] Mattioli R, Marchese D, D’Angeli S, Altamura M M, Costantino P, Trovato M. Modulation of intracellular proline levels affects flowering time and inflorescence architecture in Arabidopsis. Plant Mol Biol, 2008, 66: 277–288
[9] Mattioli R, Costantino P, Trovato M. Proline accumulation in plants: not only stress. Plant Signal Behav, 2009, 4: 1016–1018
[10] Mattioli R, Falasca G, Sabatini S, Altamura M M, Costantino P, Trovato M. The proline biosynthetic genes P5CS1 and P5CS2 play overlapping roles in Arabidopsis flower transition but not in embryo development. Physiol Plant, 2009, 137: 72–85
[11] Funck D, Winter G, Baumgarten L, Forlani G. Requirement of proline synthesis during Arabidopsis reproductive development. BMC Plant Biol, 2012, 12: 191
[12] Delauney A J, Hu C A A, Kishor P B K, Verma D P S. Cloning of ornithine delta-aminotransferase cDNA from Vigna-Aconitifolia by transcomplementation in Escherichia coli and regulation of proline biosynthesis. J Biol Chem, 1993, 268: 18673–18678
[13] Kishor P B K, Sangam S, Amrutha R N, Laxmi P S, Naidu K R, Rao K R S S, Rao S, Reddy K J, Theriappan P, Sreenivasulu N. Regulation of proline biosynthesis, degradation, uptake and transport in higher plants: its implications in plant growth and abiotic stress tolerance. Curr Sci India, 2005, 88: 424–438
[14] Wang L, Guo Z, Zhang Y, Wang Y, Yang G, Yang L, Wang R, Xie Z. Characterization of LhSorP5CS, a gene catalyzing proline synthesis in Oriental hybrid lily Sorbonne: molecular modelling and expression analysis. Bot Stud, 2017, 58(1): 10
[15] Yoshiba Y, Kiyosue T, Katagiri T, Ueda H, Mizoguchi T, Yamaguchishinozaki K, Wada K, Harada Y, Shinozaki K. Correlation between the induction of a gene for Δ1-pyrroline-5-carboxylate synthetase and the accumulation of proline in Arabidopsis thaliana under osmotic-stress. Plant J, 1995, 7: 751–760
[16] Fabro G, Kovacs I, Pavet V, Szabados L, Alvarez M E. Proline accumulation and AtP5CS2 gene activation are induced by plant-pathogen incompatible interactions in Arabidopsis. Mol Plant Microbe In, 2004, 17: 343–350
[17] Soltis P S, Soltis D E. The role of genetic and genomic attributes in the success of polyploids. Proc Nati Acad Sci USA, 2000, 97: 7051–7057
[18] Buggs R J, Doust A N, Tate J A, Koh J, Soltis K, Feltus F A, Paterson A H, Soltis P S, Soltis D E. Gene loss and silencing in Tragopogon miscellus (Asteraceae): comparison of natural and synthetic allotetraploids. Heredity, 2009, 103: 73–81
[19] Wang J, Tian L, Lee H S, Chen Z J. Nonadditive regulation of FRI and FLC loci mediates flowering-time variation in Arabidopsis allopolyploids. Genetics, 2006, 173: 965–974
[20] Zhao J W, Buchwaldt L, Rimmer S R, Brkic M, Bekkaoui D, Hegedus D. Differential expression of duplicated peroxidase genes in the allotetraploid Brassica napus. Plant Physiol Bioch, 2009, 47: 653–656
[21] Nagahara U. Genome analysis in Brassica with special reference to the experimental formation of B. napus and peculiar mode of fertilisation. J Jpn Bon, 1935, 7: 389–452
[22] Blanc G, Wolfe K H. Functional divergence of duplicated genes formed by polyploidy during Arabidopsis evolution. Plant Cell, 2004, 16: 1679–1691
[23] Yang Y W, Lai K N, Tai P Y, Li W H. Rates of nucleotide substitution in angiosperm mitochondrial DNA sequences and dates of divergence between Brassica and other angiosperm lineages. J Mol Evol, 1999, 48: 597–604
[24] Park J Y, Koo D H, Hong C P, Lee S J, Jeon J W, Lee S H, Yun P Y, Park B S, Kim H R, Bang J W, Plaha P, Bancroft I, Lim Y P. Physical mapping and microsynteny of Brassica rapa ssp. pekinensis genome corresponding to a 222 kbp gene-rich region of Arabidopsis chromosome 4 and partially duplicated on chromosome 5. Mol Genet Genom, 2005, 274: 579–588
[25] Wang X, Wang H, Wang J, Sun R, Wu J, Liu S, Bai Y, Mun J H, Bancroft I, Cheng F, Huang S, Li X, Hua W, Freeling M, Pires J C, Paterson A H, Chalhoub B, Wang B, Hayward A, Sharpe A G, Park B S, Weisshaar B, Liu B, Li B, Tong C, Song C, Duran C, Peng C, Geng C, Koh C, Lin C, Edwards D, Mu D, Shen D, Soumpourou E, Li F, Fraser F, Conant G, Lassalle G, King G J, Bonnema G, Tang H, Belcram H, Zhou H, Hirakawa H, Abe H, Guo H, Jin H, Parkin I A, Batley J, Kim J S, Just J, Li J, Xu J, Deng J, Kim J A, Yu J, Meng J, Min J, Poulain J, Hatakeyama K, Wu K, Wang L, Fang L, Trick M, Links M G, Zhao M, Jin M, Ramchiary N, Drou N, Berkman P J, Cai Q, Huang Q, Li R, Tabata S, Cheng S, Zhang S, Sato S, Sun S, Kwon S J, Choi S R, Lee T H, Fan W, Zhao X, Tan X, Xu X, Wang Y, Qiu Y, Yin Y, Li Y, Du Y, Liao Y, Lim Y, Narusaka Y, Wang Z, Li Z, Xiong Z, Zhang Z. The genome of the mesopolyploid crop speciesBrassica rapa. Nat Genet, 2011, 43: 1035–1039
[26] Udall J A, Wendel J F. Polyploidy and crop improvement. Crop Sci, 2006, 46: S3–S14
[27] Wang C P, Lin B, Zhang Y Q, Lin Y H, Liu A H, Hua X J. The evolutionary fate of Δ1-pyrroline-5-carboxylate synthetase 1 (P5CS1) genes in allotetraploid Brassica napus. J Syst Evol, 2014, 52: 566–579
[28] Tamura K, Dudley J, Nei M, Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol, 2007, 24: 1596–1599
[29] Hua X J, van de Cotte B, Van Montagu M, Verbruggen N. The 5 ' untranslated region of the At-P5R gene is involved in both transcriptional and post-transcriptional regulation. Plant J, 2001, 26: 157–169
[30] Østergaard L, King G J. Standardized gene nomenclature for the Brassica genus. Plant Methods, 2008, 4: 10
[31] Hua S, Shamsi I H, Guo Y, Pak H, Chen M, Shi C, Meng H, Jiang L. Sequence, expression divergence, and complementation of homologous ALCATRAZ loci in Brassica napus. Planta, 2009, 230: 493–503
[32] Cardenas P D, Gajardo H A, Huebert T, Parkin I A, Iniguez-Luy F L, Federico M L. Retention of triplicated phytoene synthase (PSY) genes in Brassica napus L. and its diploid progenitors during the evolution of the Brassiceae. Theor Appl Genet, 2012, 124: 1215–1228
[33] Deng W, Zhou L, Zhou Y T, Wang Y J, Wang M L, Zhao Y. Isolation and characterization of three duplicated PISTILLATA genes in Brassica napus. Mol Biol Rep, 2011, 38: 3113–3120
[34] Force A, Lynch M, Pickett F B, Amores A, Yan Y L, Postlethwait J. Preservation of duplicate genes by complementary, degenerative mutations. Genetics, 1999, 151: 1531–1545
[35] Adams K L, Liu Z L. Expression partitioning between genes duplicated by polyploidy under abiotic stress and during organ development. Curr Biol, 2007, 17: 1669–1674
Evolutionary Fate and Expression Pattern of Genes Related to Proline Biosynthesis in Brassica napus
WANG Cui-Ping1, HUA Xue-Jun2,*, LIN Bin3, and LIU Ai-Hua4
1State Key Laboratory of Seedling Bioengineering, Ningxia Forestry Institute, Yinchuan 750004, China;2Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China;3University of Chinese Academy of Sciences, Beijing 100049, China;4Department of Plant Science, University of Manitoba, Winnipeg, R3T2N2, MB, Canada
Proline accumulation is a widespread metabolic adaptation in many organisms in response to various environmental stresses, and it was proved to play protective roles for plants under adverse conditions. Polyploidization is a prominent driving force during plant evolution and many important crops have experienced this process during their evolutionary history. Brassica napus (AACC) is believed to be a newly formed allotetraploid, evolving from the inter-specific hybridization of two diploids, B. rapa (AA) and B. oleracea (CC) followed by chromosome doubling. In this research, we studied the evolutionary fate of genes related to proline synthesis in allotetraploid (B. napus), and its diploids ancestors (B. rapa and B. oleracea), to explore the effect of polyploidition on homologous gene evolution. First, we obtained the genes for proline biosynthesis (P5CSs, OAT) by database searching, and studied the similarities and the expression regulation pattern of homologous genes in allotetraploid (B. napus), in comparison with its diploids progenitors (B. rapa and B. oleracea), in different organs and in response to salt stress. Sequence analysis and phylogenetic analysis revealed that BnaA.P5CS2.a, BnaA.P5CS2.b, and BnaC.P5CS2.c originated from BraA.P5CS2.a, BraA.P5CS2.b, and BolC.P5CS2.a, respectively; BnaA.OAT.a, BnaC.OAT.b originated from BraA.OAT.a and BolC.OAT.a, respectively. And, one copy of gene loss from B. oleracea occurred for BnP5CS2 but not for BnOAT. Expressionpatterns of these homologous genes in response to salt stress in different organs were also characterized by semi-quantitive RT-PCR. In B. napus, two homologous gene pairs with different origins, BnaA.P5CS2.a and BnaC.P5CS2.c, BnaA.OAT.a and BnaC.OAT.b exhibited biased expression in different organs, implying possible sub-functionalization of P5CS2 and OAT. The genes BnaA.P5CS1.a and BnaC.P5CS1.d with different diploid ancestors were induced by NaCl treatment, and the expression of BnaC.P5CS1.d was higher than that of BnaA.P5CS1.a, showing a biased expression. RT-PCR manifested that preservation of expression pattern of original genes in diploid was found for P5CS1 (BnaA.P5CS1.a and BnaC.P5CS1.d), P5CS2 (BnaA.P5CS2.a and BnaC.P5CS2.c). These results suggest that the gene sequence and expression pattern existing in allotetraploid (B. napus) were conserved, which is benefit to proline accumulation for plant adaptation to environmental stresses.
Polyploidization; Proline biosynthesis; Biased expression; Salt stress; Sub-functionalization
(
): 2017-01-17; Accepted(接受日期): 2017-05-10; Published online(网络出版日期): 2017-05-22.
10.3724/SP.J.1006.2017.01480
本研究由宁夏自然科学基金项目(NZ16215)资助。
The study was supported by the Ningxia Natural Science Foundation of China (NZ16215).
*通讯作者(Corresponding author): 华学军, E-mail: xjhua@ibcas.ac.cn
联系方式: E-mail: wangcuipingcas@163.com
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170522.0916.006.html

