用CRISPR/CAS9系统构建原位表达dPit:GFP的果蝇品系
李博,胡君诚,金珊
(湖北大学生命科学学院,湖北 武汉 430062)
用CRISPR/CAS9系统构建原位表达dPit:GFP的果蝇品系
李博,胡君诚,金珊
(湖北大学生命科学学院,湖北 武汉 430062)
SLC20A2基因是Ⅲ型纳磷转运蛋白2(PiT2)的编码基因,PiT2可将无机磷从细胞外运送至细胞内.SLC20A2基因突变导致PiT2不能正常地将无机磷运送到细胞内,血液中的无机磷含量升高,磷和钙离子结合形成沉淀,堵塞血管.SLC20A2基因在果蝇中的同源基因是CG42575(dpit).本实验使用CRISPR/CAS9系统,构建原位表达dPiT:GFP的果蝇品系,将绿色荧光蛋白(green fluorescent protein, GFP)编码序列插入dpit基因末端,使果蝇表达dPiT:GFP融合蛋白.该融合蛋白可以被dPiT及GFP的抗体同时识别,通过荧光直接观察dPiT:GFP的位置,从而准确定位dpit,进而为dpit基因的功能研究提供基础.
果蝇;CRISPR/CAS9系统;SLC20A2;dpit
0 引言
Ⅲ型纳磷转运蛋白2(PiT2)是SLC20A2基因的表达产物,其主要功能有两个:从细胞外转运无机磷和作为逆转录病毒的受体.SLC20A2基因突变后,会导致PiT2合成受阻,血液中的无机磷酸盐含量升高.多余的磷酸盐可以和钙离子结合,形成沉淀,该过程是人类家族性基底节钙化发病的原因之一[1].目前,在哺乳动物中,PiT2的分布未能精确认知,这阻碍了PiT2的功能分析的进展.哺乳动物实验,周期较长,成本较高,不能快捷地获取结果.根据序列比对,SLC20A2基因在果蝇体内的同源基因为CG42575(dpit),该基因位于三号染色体右臂上的68A7到68A8区域,编码 667个氨基酸,它编码的氨基酸与SLC20A2的一致性与相似性分别为39%和55%,dpit的表达位置对该蛋白的功能研究具有重要意义.本实验用CRISPR/ CAS9技术构建了内源性表达的dPiT:GFP复合蛋白的果蝇,成功地对果蝇基因进行高效快速编辑.该果蝇的获得可以使我们精确定位dpit,为后续结构及功能的研究提供基础.
CRISPR/CAS9(clustered regulary interspaced short palindromic repeat system)系统是一种定向的靶序列基因修饰技术,最初发现于细菌的防御系统中,用来阻止病毒或外源质粒的入侵[2-5].Cas9核酸内切酶通过crRNA(CRISPR RNA)与外源DNA配对的20个碱基识别靶序列位点[6-8],后与tracer RNA(trans-acting crRNA)结合共同引导Cas9核酸内切酶工作.后续实验中,研究者将crRNA和tracrRNA融合成一条sgRNA(single guide RNA)[8-9],简化后该系统设计靶位点依赖于sgRNA5′端的20个碱基,唯一的限制是引导序列的3′端必须要有一个PAM(protospacer adjacent motif)序列.针对靶序列进行切割后,会形成DSB(double strand break),DSB的修复方式有两种:NHEJ(nonhomologous end joining)和HDR(homology-directed).NHEJ是一种倾向错配的修复方式,修复后通常会产生碱基的插入或缺失,这种方式有利于构建null突变体或氨基酸缺失的果蝇品系.HDR是一种精确的修复方式,修复过程依赖于同源臂的交换.通过这这种方式,在修复过程中提供供体序列(Donor),可以用来构建定点突变或对基因中较短的序列进行编辑[8,10].作为一种新兴的基因编辑技术,CRISPR/CAS9系统具有靶位点广泛,突变效率高,操作简便,应用广泛等优点.相比于传统的基因编辑技术如锌指酶和TALEN等技术,虽然都是依赖切割DNA双链产生DSB后,生物体内自身的修复系统对其进行修复,但CRISPR/CAS9系统只有两个工作元件,却更加准确地识别靶位点,这就节省了大量的工作.相比于TALEN技术,CRISPR/CAS9系统识别位点更为广泛,其使用范围更广,常见的PAM区域多为NGG,但也有研究表明NAG或GAA也可作为PAM区域[11-13].
随着CRISPR/CAS9系统的成熟,部分果蝇的研究者将该系统引入果蝇系统中实现果蝇的基因编辑.2013年,Gratz等第一次将表达Cas9酶的质粒与表达sgRNA的质粒注射到果蝇的卵中,实现了CRISPR/ CAS9系统在果蝇系统中的基因编辑[14].随后,Bassett和Yu等将表达Cas9的mRNA和sgRNA同时注射,大大提高了突变的效率.但是,由于RNA的不稳定性,该方法操作难度较高.还有一些研究者将两种质粒分别注射,得到能够分别表达Cas9酶和sgRNA的果蝇,然后将二者杂交.该方法提高了突变的效率,但实验周期较长,且得到的目的果蝇必须经过纯化,去掉带有Cas9和sgRNA的染色体[10,15].本实验参考了Ren等的方法,将sgRNA的表达载体注射到只在生殖腺表达Cas9的果蝇卵中,注射成功的果蝇后代为目的转基因果蝇[16].该方法具有较高的突变效率,同时实验操作简便,实验周期较短.
本实验通过CRISPR/CAS9系统构建出可以表达dPiT:GFP复合蛋白的果蝇,利用CRISPR/CAS9系统切割后,果蝇自身发生的HDR修复.在Cas9核酸内切酶切割后,提供外源Donor,将eGFP编码序列整合到dpit基因终止子之前.相比传统方法,该方法更为准确,方便,缩短了实验周期,且突变效率高,易于操作.
1 材料与方法
1.1 果蝇品系 购自Bloomington果蝇中心,用于二号染色体定点插入的BL25709(attp40)、用于在果蝇生殖腺内表达Cas9酶的BL54591(nos-Cas9)和BL51323(vasa-Cas9)及balancer的果蝇MKRS/TM6B、Sp/CyO(v-).
1.2 载体 本实验所用载体参见表1.
1.3 抗体 本实验所用抗体参见表2所示.

表1 本实验所用载体
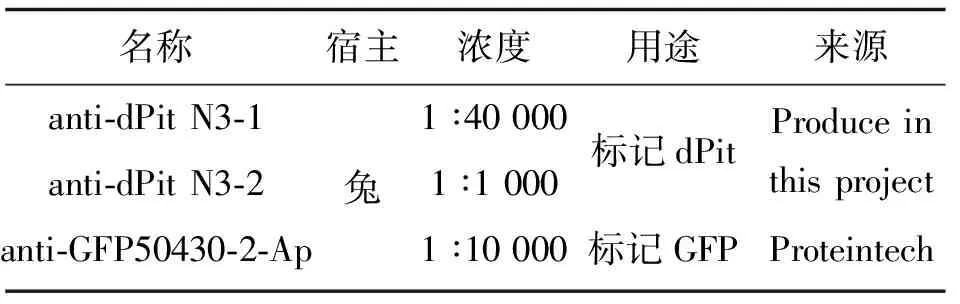
表2 本实验所用抗体
1.4 实验方法
1.4.1 sgRNA表达载体的构建 本实验预计将eGFP插入到dpit终止子TAA的前面,sgRNA序列在CRISPR optimal target finder(http://tools.flycrispr.molbio.wisc.edu/targetFinder/)设计后交由生物公司合成两条带有BbsⅠ酶切位点的单链.通过PCR将两条单链合成短片段双链DNA后,连入事先酶切过的pCDF-3质粒中.转化入大肠杆菌中扩大培养,抽取质粒.
1.4.2 donor载体的构建 提取野生型果蝇的总DNA后,使用引物扩增出用于同源重组修复的同源臂;以pUAST-GFP质粒为模板,扩增出没有终止子的GFP片段.使用overlap PCR技术将3个片段连接在一起,使其成为左同源臂-GFP-右同源臂的一个片段.将所得片段与PMD18-T连接后,转化入大肠杆菌中扩大培养,抽取质粒.

图1 显微注射流程图
1.4.3 显微注射 显微注射方法参考Yu等[15].将sgRNA表达载体注射到attp40果蝇的卵中,获得的sgRNA转基因果蝇与表达Cas9酶的果蝇杂交,在杂交后代的卵中注射GFP-donor质粒.将所得成虫与MKRS/TM6B杂交,然后使用PCR鉴定后代,得到转基因果蝇(见图1).

1.4.5 免疫染色 将三龄幼虫固定在解剖盘上,将幼虫尾部横向剪开,展开果蝇的表皮和肌肉层,保证大脑处于头部中间.用4%的PFA固定1 h后用0.2%的PBST(tritonX-100)清洗幼虫皮,室温封闭0.5 h.4 ℃孵育一抗2 h,清洗后室温孵育二抗2 h,清洗后封片.
2 结果
2.1 sgRNA表达载体的构建 为了保证sgRNA的效率,设计了3条sgRNA(来自CRISPR optimal target finder).将 sgRNA插入表达载体pCDF3中,测序(见图2).

图2 sgRNA的序列及表达载体的构建 A)sgRNA的序列及靶位点; B)pCDF3-sgRNA表达载体与pCDF3载体琼脂糖电泳图示,由于使用BbsⅠ切割pCDF3质粒,切下的片段与sgRNA大小一致,故sgRNA表达载体大小与原pCDF3载体大小一致;C)sgRNA表达载体测序结果及峰图.
2.2 获得能够稳定表达sgRNA的转基因果蝇 将得到的sgRNA表达载体注射到attp40果蝇的卵中.由于attp40果蝇的眼色背景为w+,v-眼色为鲜红色;sgRNA的表达载体中带有vermilion基因,故转基因成功的果蝇的眼色基因会成为w+,v+从鲜红变成深红.提取转基因果蝇的基因组,以其为模板,引物为pCDF3载体上的一段,PCR鉴定测序.将所得的转基因果蝇杂交后得到纯合的后代(见图3).

图3 sgRNA的转基因果蝇鉴定 A)转基因果蝇与原始果蝇眼色对比. B)转基因果蝇PCR结果电泳图. C)PCR测序结果显示8-5为sgRNA1的转基因果蝇,8-6为sgRNA2的转基因果蝇,8-8为sgRNA3的转基因果蝇.
2.3 获得带有donor的载体 本实验同源臂长度为1 kb.提取果蝇基因组DNA后,以其为模板扩增出左右同源臂;以载体pUAST-GFP为模板扩增出GFP片段;使用overlap PCR技术将3个片段连接在一起后,连入克隆载体PMD18-T中,测序(见图4).
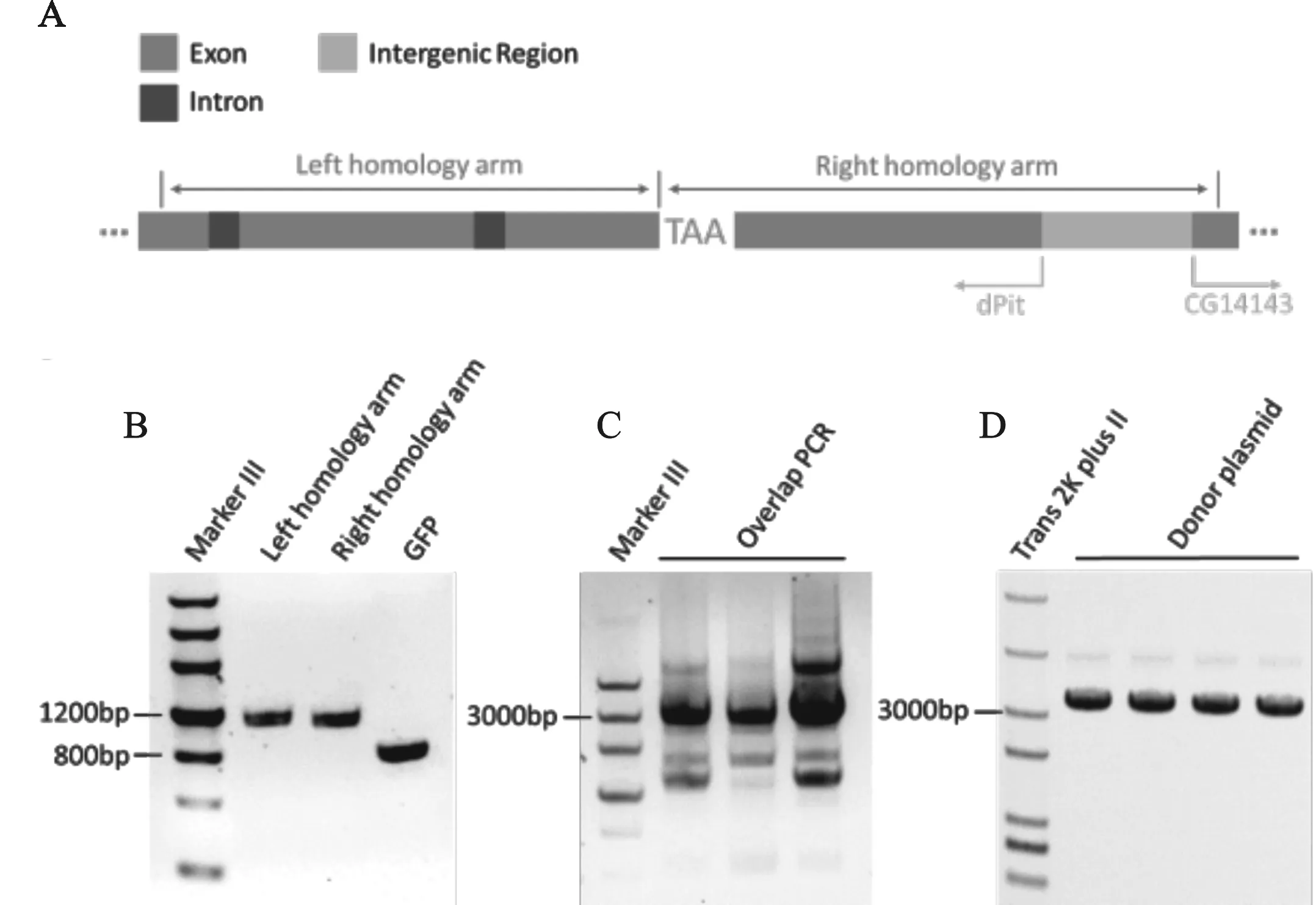
图4 带有donor载体的构建 A)左右同源臂的位置示意图.B)获取的左右同源臂及GFP片段的电泳图;C)overlap PCR后电泳图.D)overlap PCR后胶回收的目的片段.
2.4 能够稳定遗传dPiT:GFP果蝇品系的鉴定 将所得的sgRNA果蝇与Cas9果蝇杂交后收集后代的卵,注射donor载体.注射完毕共收集21只成虫,与MKRS/TM6B杂交后取子代果蝇PCR鉴定,第13号果蝇中插入了GFP序列,后续中还有两只成功鉴定到突变.对13号果蝇子代进行单独检测,分别设计引物扩增左右同源臂及GFP序列,均得到目的片段(见图5).

图5 dPiT:GFP果蝇品系的鉴定 A)注射成功后杂交后代的检测.上游引物位于dPiT最后一个外显子上,下游引物位于3′-UTR上,扩增长度为452 bp.当GFP序列成功插入后,片断长度为1 166 bp.B)对13号果蝇子代单独鉴定结果的电泳图.
2.5 获得的dPiT:GFP果蝇的鉴定 得到目的果蝇后,需确定插入的GFP是否能够正常工作.通过Western blot检测蛋白质的分子量,结果显示,融合蛋白与预期一致,融合蛋白可以被dPiT及GFP的抗体同时识别.通过共聚焦显微镜观察dPiT:GFP果蝇的三龄幼虫大脑,能够看到GFP受激发产生的荧光,相比于抗体染色,直接观察时背景较少.同时使用dPiT的抗体N3-2对野生型果蝇大脑免疫染色,结果与直接观察结果一致(见图6).

图6 融合蛋白的检测 A)dPiT:GFP的Western blot结果.分别使用dPiT和GFP抗体,二者均能和融合蛋白结合,且分子量大小和预期一致.但是在对照的泳道中只有N3-1能够与dPiT结合.B)使用N3-2抗体对野生型果蝇三龄幼虫的大脑进行免疫组织染色及共聚焦显微镜观察融合蛋白.
3 讨论
本实验使用的CRISPR/Cas9系统是目前广泛应用于基因编辑的一种技术,近年来被广泛应用于生物学的各个领域.实验中,我们将sgRNA注射到只有生殖腺表达Cas9酶的果蝇卵中,提高突变效率,同时降低了工作量.本实验注射了500个左右的卵,最终成活的18只幼虫中有4只为转基因果蝇,大大降低了实验耗时和工作量,操作更加简便快捷.基于CRISPR/Cas9系统的这些优点,产生了更多的改进与优化技术,这些技术的出现和成熟为我们的后续实验提供了帮助,缩短了实验周期,但仍旧有更多的新技术和新方法等待我们去探索和发现.
PiT2作为参与生物体磷酸盐代谢的重要组成部分,在哺乳动物的肾脏、大脑和小肠中都有分布.然而,到目前为止,PiT2的在生物体内的完整分布尚未明确.确定PiT2的表达位置,对于该蛋白功能相关信号通路的研究具有重要意义.SLC20A2基因与dpit的高度相似性和一致性,使得在果蝇体内模拟研究PiT2成为可能.在果蝇体内,PiT2分布相对简单,且果蝇生活周期短,后代多,易培养.在初步的观察和统计中,果蝇体内PiT2的表达位置能够与哺乳动物对照参考.本实验构建的能够原位表达的dPiT:GFP融合蛋白,可以直观地显示PiT2的分布.
通过对果蝇三龄幼虫大脑的观察,dPiT:GFP融合蛋白在果蝇大脑中的分布与Karsai等研究中所示区域相似[17],主要存在于大脑中.相比于抗体染色,直接观察时背景较少,减少了实验背景的干扰因素,使实验结果更加准确.dpit基因功能的相关研究还在进行,能够原位表达dPiT:GFP融合蛋白的果蝇品系是后续实验中需要的工具果蝇之一,后续研究我们将继续探索.
[1] Wang C, Li Y, Shi L, et al. Mutations in SLC20A2 link familial idiopathic basal ganglia calcification with phosphate homeostasis[J]. Nature Genetics, 2012, 44(3): 254-256.
[2] Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion inEscherichiacoli, and identification of the gene product[J]. Journal of Bacteriology, 1987,169(12):5429-5433.
[3] Jansen R, Embden J, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575.
[4] Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712.
[5] Garneau J E, Dupuis M, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468(7320): 67-71.
[6] Brouns S J J, Jore M M, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J].Science, 2008, 321(5891): 960-964.
[7] Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proceedings of the National Academy of Sciences, 2012, 109(39):E2579-E2586.
[8] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821.
[9] Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013,339(6121): 819-823.
[10] Bassett A R, Tibbit C, Ponting C P, et al. Mutagenesis and homologous recombination inDrosophilacell lines using CRISPR/Cas9[J]. Biology Open, 2013: BIO20137120.
[11] Mali P, Aach J, Stranges P B, et al. CAS9 transcriptional activators for target specificity screening and pairednickases for co-operative genome engineering[J]. Nature Biotechnology, 2013, 31(9): 833-838.
[12] Mojica F J M, Diez-Villasenor C, Garcia-Martinez J, et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system[J]. Microbiology, 2009, 155(3): 733-740.
[13] Esvelt K M, Esvelt K M, Mali P, et al. Orthogonal Cas9 Proteins for RNA-Guided Gene Regulation and Editing[J]. Nature Methods, 2013, 10(11):1116-1121.
[14] Gratz S J, Cummings A M, Nguyen J N, et al. Genome engineering ofDrosophilawith the CRISPR RNAguided Cas9 nuclease[J]. Genetics, 2013, 194(4): 1029-1035.
[15] Yu Z, Ren M, Wang Z, et al. Highly efficient genome modifications mediated by CRISPR/Cas9 inDrosophila[J]. Genetics, 2013, 195(1):289.
[16] Ren X, Sun J, Housden B E, et al. Optimized gene editing technology forDrosophilamelanogasterusing germ line-specific Cas9[J]. Proceedings of the National Academy of Sciences, 2013,110(47): 19012-19017.
[17] Karak S, Jacobs J S, Kittelmann M, et al. Diverse roles of axonemal dyneins inDrosophilaauditory neuron function and mechanical amplification in hearing[J]. Scientific Reports, 2015, 5(2):17085.
(责任编辑 游俊)
Generate a dPit:GFP fly stock with the CRISPR/ Cas9 system
LI Bo, HU Juncheng,JIN Shan
(College of Life Sciences, Hubei University, Wuhan 430062, China)
Phosphate can be transported to intracellular by type Ⅲ Na+/Pi co-transporters (PiT2), which is coded bySLC20A2. In theSLC20A2 mutants, phosphate precipitates with Ca2+in the blood and block blood vessels. The homologous gene of humanSLC20A2 is theCG42575 (dpit) in Drosophila. In our study, we used CRISPR/CAS9 system to insert the green fluorescent protein (GFP) in the carbon end ofdpitto get aDrosophila, which expresses the dPiT:GFP fusion protein. The dPiT:GFP fusion protein could be observed by GFP fluorescence directly and stained by the antibody of dPiTor GFP. It is a useful tool to locate thedpitexactly and to investigate thedpitfunction.
Drosophila; CRISPR/CAS9;SLC20A2;dpit
2016-11-16
国家自然科学基金(31230045、81471154)资助
李博(1991-),男,硕士生;金珊,通信作者,教授,E-mail: jinshan@hubu.edu.cn
1000-2375(2017)05-0462-06
Q789
A
10.3969/j.issn.1000-2375.2017.05.005

