无义mRNA降解途径的机制与进化
柴宝峰,王美,石文鑫,柴杨丽,吕佳
(山西大学 黄土高原研究所,山西 太原 030006)
无义mRNA降解途径的机制与进化
柴宝峰,王美,石文鑫,柴杨丽,吕佳
(山西大学 黄土高原研究所,山西 太原 030006)
含有无义突变的mRNA在细胞中被无义介导的mRNA降解(nonsense-mediated mRNA decay,NMD)途径选择性降解,同时该途径还能调控细胞中一些生理性RNA的丰度。NMD途径的核心是含有无义突变的mRNA的识别和降解机制,其中关键因子上游移码蛋白(up-frameshift,UPF)和生殖器形态效应抑制因子(suppressor with morphological effect on genitalia,SMG)的功能及其调控机制是NMD途径研究的热点问题。NMD途径核心因子的种类和功能在不同进化地位的生物中既有一定的保守性,也有一定的差异,说明NMD途径具有一定的进化学特征。高等真核生物NMD途径机制的研究取得了一定的进展,而在原生生物中尚无机制层面的研究。利用原生动物作为模式生物开展NMD途径关键因子的功能和进化研究,有助于从进化学角度揭示NMD途径的功能和调控机制,为建立人类细胞中NMD途径的调控网络提供数据支持,为人类遗传学疾病和肿瘤的预防和治疗提供理论基础。
NMD途径;无义mRNA识别;核心因子;功能调控;进化
基因的表达受到转录和翻译水平多重机制的调控,各种核酸和蛋白质因子协调作用,对基因表达的特定步骤或者特定基因的精确表达进行调控。在不同层次对靶基因进行实时调控的特异因子和具体机制是现代分子生物学研究的核心课题[1]。转录后RNA的质量控制是基因精确表达的关键环节之一。DNA的突变、转录和转录后加工过程中的任何错误,都可能导致mRNA中产生提前密码子(premature termination codon,PTC)。这些含有PTC的mRNA的翻译会使细胞中积累截短型蛋白质,对细胞产生毒害。有趣的是,在真核细胞中存在一种RNA质控机制,即无义介导的mRNA降解途径(nonsense-mediated mRNA decay,NMD),可以降解含有PTC的mRNA[2-3]。此外,NMD途径还可以调控细胞中10%~20%的正常的生理性mRNA和非编码RNA,确保细胞的内平衡(homeostasis),与细胞免疫、神经系统发育,如轴突导向、突触强度和神经元表达[3-5]以及胚胎发育等密切相关[6]。大约有三分之一的遗传性疾病和多种癌症的发生与NMD途径有关[7-9]。NMD途径机制的研究成果对于人类遗传性疾病和肿瘤的预防和治疗具有广阔的应用前景[7,10-11]。因此,近年来无义突变基因的表达调控机制的研究成为分子细胞生物学的热点之一[4,12-13]。
1 NMD 途径的机制
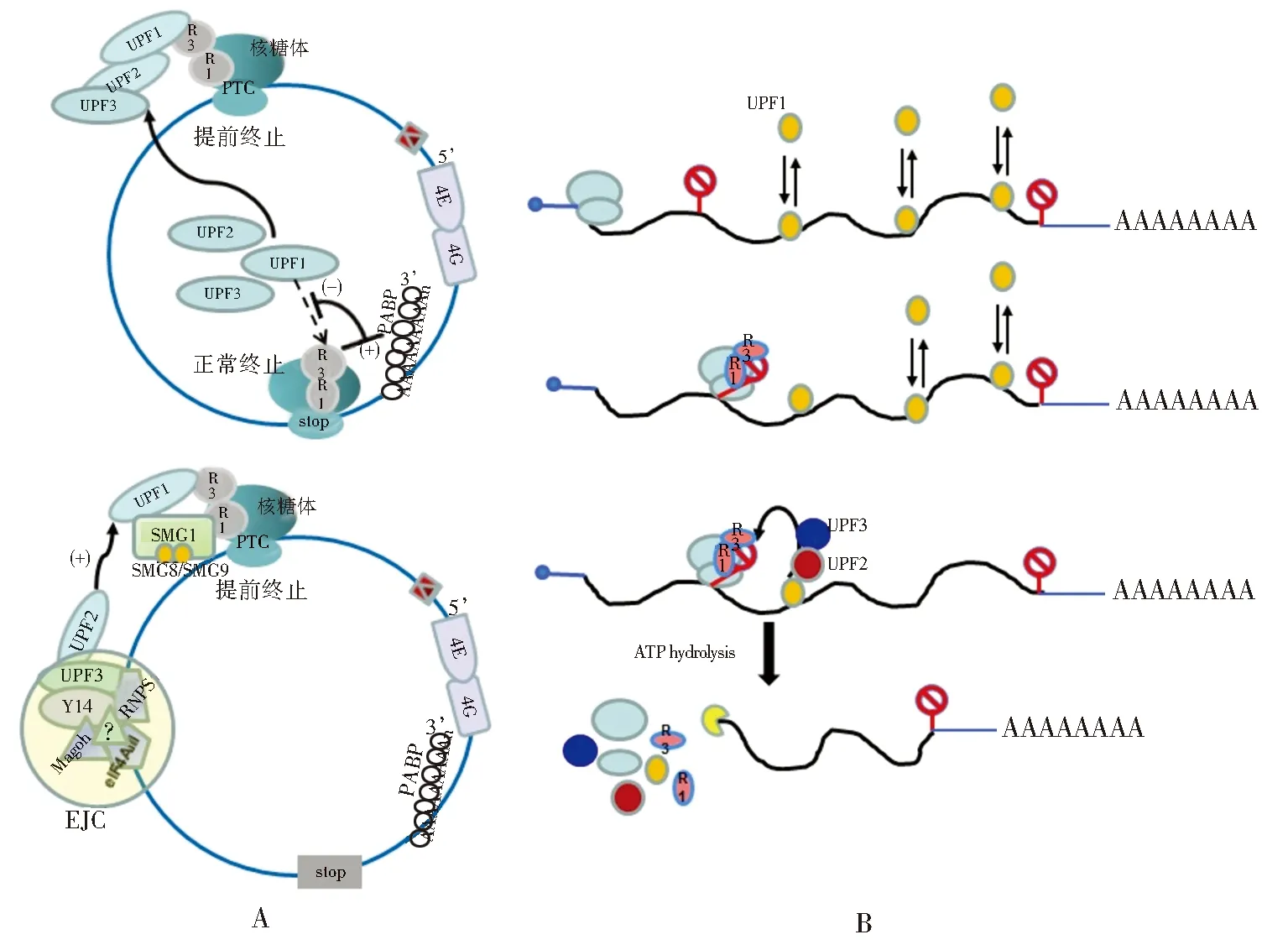
Fig.1 The model of NMD pathway and the degradation of mRNAA:the faux 3’-UTR model (up) and exon-exon junction complex model (down) dependent on translation termination;B:the UPF1 3’-UTR sensing and potentiation modelA:依赖于翻译终止过程的伪3’-UTR模型(上)和EJC模型(下);B不依赖于翻译终止的UPF1 3’-UTR感应和增效模型(Serdar,2016[31])图1 NMD 途径模型及mRNA的降解
NMD途径研究的核心是含有无义密码子的mRNA的识别和降解机制。即NMD途径如何区分含有无义密码子的mRNA(PTC-mRNA)与含有正常终止密码子的mRNA。目前为止有三个主要模型(图1):以酵母和果蝇细胞为代表的“伪3’-UTR模型(the faux 3’-UTR model)”[14-16];以哺乳动物细胞为代表的“外显子连接复合体模型(exon-exon junction complex,the EJC model)”[16];UPF1 3’-UTR 感应和增效模型(UPF1 3’-UTR sensing and potentiation model)[17]。伪3’-UTR模型中(图1A上),当翻译终止复合体停滞在提前终止密码子(PTC)位置时,mRNA 3’端的多聚腺苷酸A结合蛋白PABP(poly(A) binding protein)与第二类肽链释放因子eRF3 (eukaryotic polypeptide release factor) 距离较远而不能相互作用时,NMD途径核心因子UPF (up-frameshift suppressor)与eRF3相互作用,启动了NMD途径。EJC模型中(图1A下),翻译终止复合体在提前终止密码子处停滞,肽链释放因子与NMD途径核心因子UPF1和SMG1形成SMG1-UPF1-eRF3-eRF1复合体结构(SURF),其中UPF1通过UPF2与下游(>50-55nt)的外显子拼接复合体EJC核心因子相互作用[18],调控SMG1 (suppressor with morphological effect on genitalia) 激酶活性,磷酸化修饰UPF1,启动NMD途径[19]。以上两个模型均依赖于翻译终止过程。UPF1 3’-UTR 感应和增效模型中(图1B),UPF1因子优先结合到mRNA的PTC下游3’-UTR区,而且UPF1与mRNA的结合与3’-UTR的长度有关,而与序列特征无关。UPF1可以感知3’-UTR的长度,启动NMD途径,该过程与翻译过程无关。
2 NMD途径的核心因子及其进化
三个模型中,UPF1作为核心因子与多种调控因子 (regulator) 和效应子(effector)相互作用,包括UPF2、UPF3、SMG1,以及核糖体和肽链释放因子eRF1和eRF3等,但各因子间相互作用的功能和机制有待阐明[2]。UPF1具有ATPase和RNA helicase 活性,同时作为磷酸蛋白质被SMG1修饰,为招募mRNA的降解因子提供平台。UPF1结合ATP后,与翻译终止复合体结合,形成SURF结构,促进无义mRNA的翻译终止和截短型肽链的释放。ATP被UPF1水解后,将翻译终止复合体从mRNA解离,进而招募mRNA降解因子,启动NMD途径[20];在SURF复合体中,eRF1和eRF3可能抑制UPF1的ATPase活性,而UPF2和UPF3激活UPF1的ATPase活性和解旋酶活性[20]。在后生动物中,UPF1的N端和C端包含多个保守的丝氨酸和苏氨酸磷酸化位点,被SMG1或者其它激酶超磷酸化修饰后,招募SMG6和SMG5-SMG7因子,以及脱帽酶Dcp2(decapping enzyme),直接或间接降解mRNA(图1B)[21]。而在酵母(Saccharomycescerevisiae)细胞中,UPF1则没有富含磷酸化位点的N端和C端结构域,尽管酵母的UPF1也被磷酸化修饰,但UPF1因子上却没有SMG1的结合位点[22],目前为止,酵母细胞中还没有克隆到SMG1基因。原生动物贾第虫(Giardialamblia)[23]和草履虫(Parameciumtetraurelia)、锥虫(Trypanosomabrucei)[24]细胞与后生动物线虫(Caenorhabditiselegans)、果蝇(Drosophilamelanogaster)、拟南芥(Arabidopsisthaliana)、人类细胞相比,NMD途径因子的种类(表1)和一级结构序列和高级结构上也存在一定差异[23,25-26],所以NMD途径机制的保守性和进化特征是一个值得关注的问题。
表1 不同进化地位的真核生物中基因中内含子含量、外显子复合体和NMD途径核心因子[27]
Table 1 Intron content,exon-exon complex and core factors in genes of eukaryotic organisms at different evolutionary position
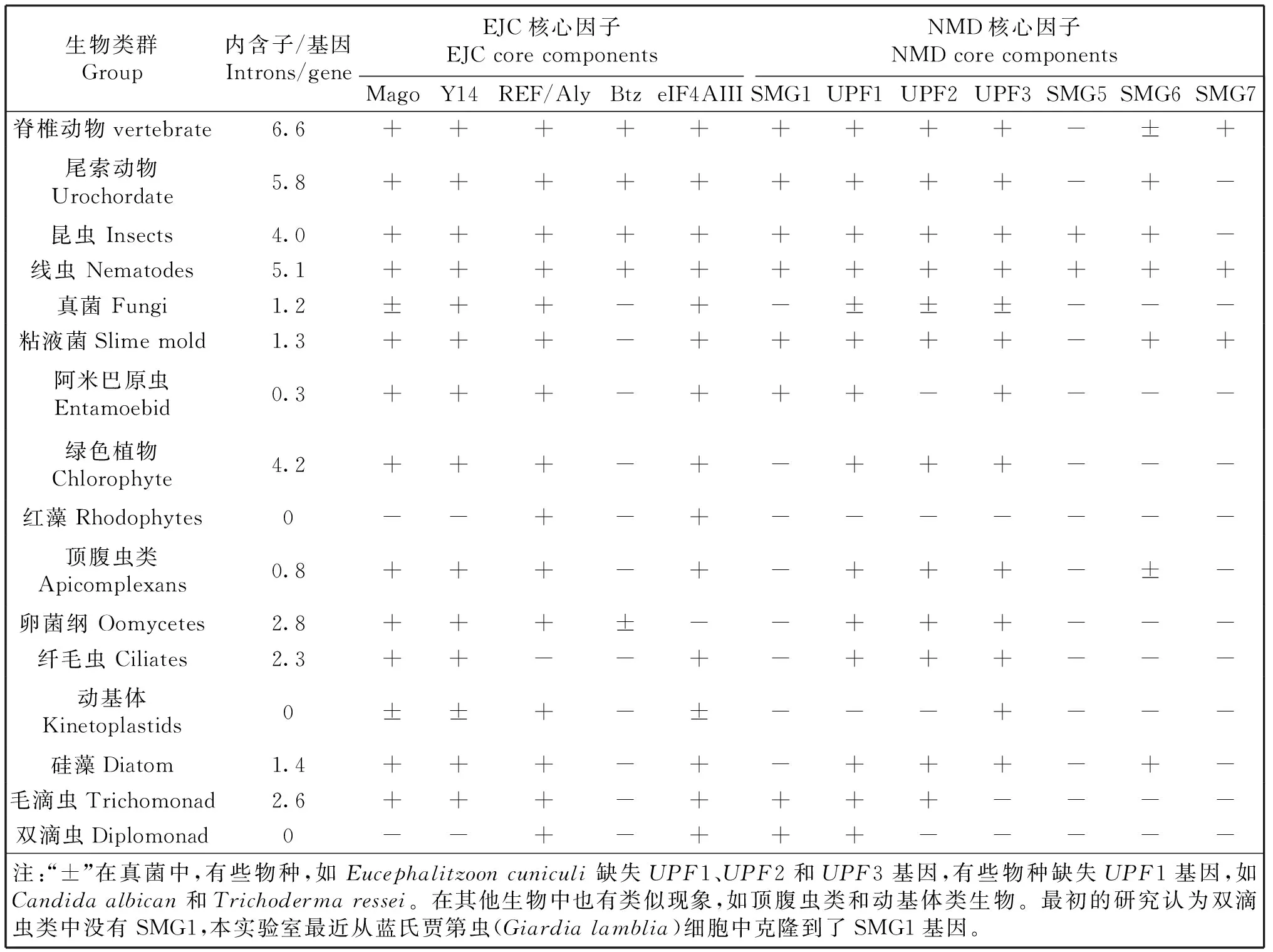
生物类群Group内含子/基因Introns/geneEJC核心因子EJCcorecomponentsNMD核心因子NMDcorecomponentsMagoY14REF/AlyBtzeIF4AIIISMG1UPF1UPF2UPF3SMG5SMG6SMG7脊椎动物vertebrate6.6+++++++++-±+尾索动物Urochordate5.8+++++++++-+-昆虫Insects4.0+++++++++++-线虫Nematodes5.1++++++++++++真菌Fungi1.2±++-+-±±±---粘液菌Slimemold1.3+++-+++++-++阿米巴原虫Entamoebid0.3+++-+++-+---绿色植物Chlorophyte4.2+++-+-+++---红藻Rhodophytes0--+-+-------顶腹虫类Apicomplexans0.8+++-+-+++-±-卵菌纲Oomycetes2.8+++±--+++---纤毛虫Ciliates2.3++--+-+++---动基体Kinetoplastids0±±+-±---+---硅藻Diatom1.4+++-+-+++-+-毛滴虫Trichomonad2.6+++-++++----双滴虫Diplomonad0--+-+++-----注:“±”在真菌中,有些物种,如Eucephalitzooncuniculi缺失UPF1、UPF2和UPF3基因,有些物种缺失UPF1基因,如Candidaalbican和Trichodermaressei。在其他生物中也有类似现象,如顶腹虫类和动基体类生物。最初的研究认为双滴虫类中没有SMG1,本实验室最近从蓝氏贾第虫(Giardialamblia)细胞中克隆到了SMG1基因。
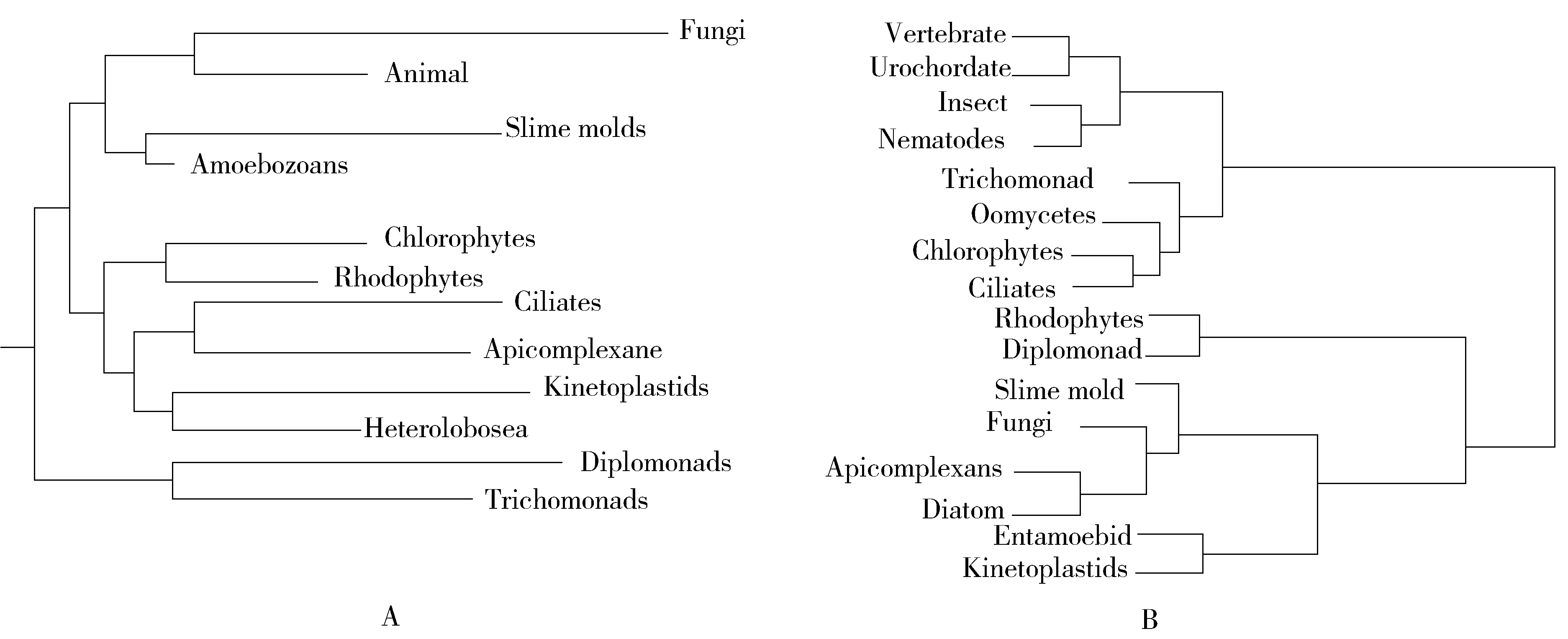
Fig.2 Comparison of the eukaryotic phylogenetic trees between that constructed using ribosome RNA and sequences of multiple proteins and that using sequences of NMD core factors listed in table 1图2 真核生物门水平上根据核糖体RNA和多蛋白序列构建的系统树[28](A)与NMD途径核心因子(表1)构建的系统树(B)比较
根据Baldauf等利用多种蛋白质序列数据在门水平构建的真核生物系统树[28],简化后如图2A所示。用NMD途径核心因子(表1)构建的系统树如图2B所示。两个系统树之间具有一定的相似性,图2A中真核生物进化关系明确,而图2B中真菌的地位与图2A中的真菌所处的位置差异较大。现在的问题是在动基体门(kinetoplastids)的锥虫(Trypanosoma)和利什曼虫(Leishmania)、红藻门(Rhodophyte)的红藻(Cyanidioschyzon)、真菌门(Fungi)的微孢子虫(Encephalitozoon)、双滴虫门(Diplomonad)的贾第虫(Giardia)这些生物中,缺少NMD途径的一些关键的因子UPF或SMG(表1),是NMD途径在这些生物中刚显雏形,还是NMD途径功能的退化?那么,尚存的那些NMD途径因子的功能又是什么呢?目前为止尚无机制层面的研究,没有确定的结论。用NMD途径因子构建的系统树不能反映真核生物的进化关系,很可能是我们还没有完全阐明NMD途径的起源和进化,还没有完全弄清NMD途径因子来龙去脉。
3 NMD途径研究的热点问题
目前关于NMD途径分子机制研究的热点问题是:无义mRNA的分子特征是什么?NMD途径因子如何感知到的这些特征;翻译正常终止和提前终止过程中终止复合体结构和功能差异;NMD途径核心因子(UPF1) 是何时何处结合到mRNA上的,即在细胞核内的转录过程中,还是在细胞质mRNA的翻译过程中,是如何结合到mRNA上的;底物mRNA是如何降解的等。三个模型对这些问题进行了初步阐述,但仍然存在许多机制性的问题,一些机制上的矛盾仍然没有解决,致使NMD途径受到一些学者质疑[29]。
有些研究认为无义mRNA上的核苷酸序列元件是NMD途径识别该mRNA的主要特征。如在酿酒酵母细胞中,无义突变位点PTC的下游存在一种下游序列元件DSE (downstream sequence element),可以结合Hrp1p因子,激活NMD途径[15],但在其它生物中没有找到类似的DSE序列。而最近的一项研究显示,PTC的上下游核苷酸序列对于无义mRNA的识别发挥重要作用[30]。而主流研究都认为,无论伪3’-UTR模型还是EJC模型,对于无义mRNA的识别过程都与翻译终止过程相关,即依赖于翻译终止复合体(核糖体和肽链释放因子复合体)。在两种模型中,是翻译终止复合体招募了UPF1,形成SURF结构,接着SURF复合体中的UPF1被SMG1磷酸化,招募了RNA酶等相关因子,开启无义mRNA的降解过程。不过在PTC处的翻译终止效率要低于正常的终止过程,为NMD途径因子的招募提供了机会,此时终止复合体中肽链释放因子 (eRF1、eRF3) 与PABP、UPF1以及ATP结合盒式蛋白ABCE1 (ATP-binding cassette protein E1) 等因子相互作用关系的动力学特征发生变化[31]。正常终止过程中,eRF3水解GTP激活eRF1的活性,释放新生肽链,招募ABCE1,其作为一种ATPase,水解ATP,解离mRNA上终止复合体[32],而在PTC处,是UPF1水解ATP,促进终止复合体的解离[33]。因此,翻译终止过程和翻译终止复合体结构是NMD途径机制的主要组成部分,二者之间的耦联关系有待详细研究。但是,最近的研究认为,UPF1在细胞核内mRNA加工过程中作为RNP结合到mRNA上,这种结合是一种动态过程[34]。在正常翻译过程中翻译复合体将UPF1从mRNA上驱离,但当翻译意外终止时,终止复合体中的eRF3与(结合在mRNA上)UPF1相互作用,UPF1水解ATP使终止复合体解离,引发NMD途径[33]。在无义mRNA的PTC的下游结合大量的UPF1,UPF1的N端和C端中的[S/T]Q模体结构被超磷酸化修饰,招募各种RNA酶,降解无义mRNA[1]。这意味着UPF1是无义mRNA的先行识别者,为终止复合体与UPF1的相互作用奠定基础,与之前的结果矛盾。
目前为止,所有的无义mRNA识别模型中,UPF1都是核心因子,对于UPF1的研究焦点在于(1)UPF1如何被招募到mRNA上的,是直接与mRNA相互作用,还是由RNA结合蛋白介导;(2)UPF1的ATPase和解旋酶活性在无义mRNA识别和降解中的作用[20];(3)UPF1的ATPase和解旋酶活性的调控机制;(4)UPF1的磷酸化和超磷酸化的功能和调控机制[35]。这些问题的解决将有助于阐明NMD途径的分子机制,建立人类细胞中NMD途径的调控网络,为人类遗传疾病和肿瘤的预防和治疗提供理论基础。
[1] Durand S,Franks T M,Lykke-Andersen J.Hyperphosphorylation Amplifies UPF1 Activity to Resolve Stalls in Nonsense-mediated mRNA Decay[J].NatCommun,2016,7:12434.DOI:10.1038/ncomms12434.
[2] Kervestin S,Jacobson A.NMD:a Multifaceted Response to Premature Translational Termination[J].NatRevMolCellBiol,2012,13(11):700-712.DOI:10.1038/nrm3454.
[3] Smith J E,Baker K E.Nonsense-mediated RNA Decay-a Switch and Dial for Regulating Gene Expression[J].Bioessays,2015,37(6):612-623.DOI:10.1002/bies.201500007.
[4] Nicholson P,Yepiskoposyan H,Metze S,etal.Nonsense-mediated mRNA Decay in Human Cells:Mechanistic Insights,Functions Beyond Quality Control and the Double-life of NMD Factors[J].CellMolLifeSci,2010,67(5):677-700.DOI:10.1007/s00018-009-0177-1.
[5] Karam R,Wengrod J,Gardner L B,etal.Regulation of Nonsense-mediated mRNA Decay:Implications for Physiology and Disease[J].BiochimBiophysActa,2013,1829(6-7):624-633.DOI:10.1016/j.bbagrm.2013.03.002.
[6] Hwang J,Maquat L E.Nonsense-mediated mRNA Decay (NMD) in Animal Embryogenesis:to Die or not to Die,That is the Question[J].CurrOpinGenetDev,2011,21(4):422-430.DOI:10.1016/j.gde.2011.03.008.
[7] Welch E M,Barton E R,Zhuo J,etal.PTC124 Targets Genetic Disorders Caused by Nonsense Mutations[J].Nature,2007,447:87-91.DOI:10.1038/nature05756.
[8] Holbrook J A,Neu-Yilik G,Hentze M W,etal.Nonsense-mediated Decay Approaches the Clinic[J].NatGenet,2004,36(8):801-808.DOI:10.1038/ng1403.
[9] Bhuvanagiri M,Schlitter A M,Hentze M W,etal.NMD:RNA Biology Meets Human Genetic Medicine[J].BiochemJ,2010,430(3):365-377.DOI:10.1042/BJ20100699.
[10] Ainsworth C.Nonsense Mutations:Running the Red Light[J].Nature,2005,438(7069):726-728.DOI:10.1038/438726a.
[11] Wang X,Gregory-Evans C Y.Nonsense Suppression Therapies in Ocular Genetic Diseases[J].CellMolLifeSci,2015,72(10):1931-1938.DOI:10.1007/s00018-015-1843-0.
[12] Fatscher T,Boehm V,Gehring N H.Mechanism,Factors,and Physiological Role of Nonsense-mediated mRNA Decay[J].CellMolLifeSci,2015,72(23):4523-4544.DOI:10.1007/s00018-015-2017-9.
[13] Durand S,Lykke-Andersen J.SnapShot:Nonsense-Mediated mRNA Decay[J].Cell,2011,145(2):324-324 e322.DOI:10.1016/j.cell.2011.03.038.
[14] Zhang S,Ruiz-Echevarria M J,Quan Y,etal.Identification and Characterization of a Sequence Motif Involved in Nonsense-mediated mRNA Decay[J].MolCellBiol,1995,15(4):2231-2244.
[15] Gonzalez C I,Ruiz-Echevarria M J,Vasudevan S,etal.The Yeast hnRNP-like Protein Hrp1/Nab4 Marks a Transcript for Nonsense-mediated mRNA Decay[J].MolCell,2000,5(3):489-499.
[16] Brogna S,Wen J.Nonsense-mediated mRNA Decay (NMD) Mechanisms[J].NatStructMolBiol,2009,16(2):107-113.DOI:10.1038/nsmb.1550.
[17] Hogg J R,Goff S P.Upf1 senses 3′UTR Length to Potentiate mRNA Decay[J].Cell,2010,143(3):379-389.DOI:10.1016/j.cell.2010.10.005.
[18] Lopez-Perrote A,Castano R,Melero R,etal.Human Nonsense-mediated mRNA Decay Factor UPF2 Interacts Directly with eRF3 and the SURF Complex[J].NucleicAcidsRes,2016,44(4):1909-1923.DOI:10.1093/nar/gkv1527.
[19] Yamashita A,Izumi N,Kashima I,etal.SMG-8 and SMG-9,Two Novel Subunits of the SMG-1 Complex,Regulate Remodeling of the mRNA Surveillance Complex During Nonsense-mediated mRNA Decay[J].GenesDev,2009,23(9):1091-1105.DOI:10.1101/gad.1767209.
[20] Lee S R,Pratt G A,Martinez F J,etal.Target Discrimination in Nonsense-Mediated mRNA Decay Requires Upf1 ATPase Activity[J].MolCell,2015,59(3):413-425.DOI:10.1016/j.molcel.2015.06.036.
[21] Kurosaki T,Li W,Hoque M,etal.A Post-translational Regulatory Switch on UPF1 Controls Targeted mRNA Degradation[J].GenesDev,2014,28(17):1900-1916.DOI:10.1101/gad.245506.114.
[22] Lasalde C,Rivera A V,Leon A J,etal.Identification and Functional Analysis of Novel Phosphorylation Sites in the RNA Surveillance Protein Upf1[J].NucleicAcidsRes,2014,42(3):1916-1929.DOI:10.1093/nar/gkt1049.
[23] Chen Y H,Su L H,Sun C H.Incomplete Nonsense-mediated mRNA Decay in Giardia Lamblia[J].InternationalJournalforParasitology,2008,38(11):1305-1317.DOI:10.1371/journal.pone.0003609.
[24] Delhi P,Queiroz R,Inchaustegui D,etal.Is There a Classical Nonsense-mediated Decay Pathway in Trypanosomes?[J].PLoSOne,2011,6(9):e25112.DOI:10.1371/journal.pone.0025112.
[25] Jaillon O,Bouhouche K,Gout J F,etal.Translational Control of Intron Splicing in Eukaryotes[J].Nature,2008,451(7176):359-362.DOI:10.1038/nature06495.
[26] Saul H,Elharrar E,Gaash R,etal.The Upstream Open Reading Frame of the Arabidopsis AtMHX Gene Has a Strong Impact on Transcript Accumulation Through the Nonsense-mediated mRNA Decay Pathway[J].PlantJ,2009,60(6):1031-1042.DOI:10.1111/j.1365-313X.2009.04021.x.
[27] Lynch M,Hong X,Scofield D G.NMD and the Evolution of Eukaryotic Gene Structure.In:Nonsense-MediatedmRNADecay,Edited by Maquat LE.Georgetown,Texas:Landes Bioscience;2006:197-211.
[28] Baldauf S L,Roger A J,Wenk-Siefert I,etal.A Kingdom-level Phylogeny of Eukaryotes based on Combined Protein Data[J].Science,2000,290(5493):972-977.
[29] Brogna S,McLeod T,Petric M.The Meaning of NMD:Translate or Perish[J].TrendsGenet,2016,32(7):395-407.DOI:10.1016/j.tig.2016.04.007.
[30] Zahdeh F,Carmel L.The Role of Nucleotide Composition in Premature Termination codon Recognition[J].BMCBioinformatics,2016,17(1):519.DOI:10.1186/s12859-016-1384-z.
[31] He F,Jacobson A.Nonsense-Mediated mRNA Decay:Degradation of Defective Transcripts Is Only Part of the Story[J].AnnuRevGenet,2015,49:339-366.DOI:10.1146/annurev-genet-112414-054639.
[32] Shoemaker C J,Green R.Kinetic Analysis Reveals the Ordered Coupling of Translation Termination and Ribosome Recycling in Yeast[J].ProcNatlAcadSciUSA,2011,108(51):E1392-1398.DOI:10.1073/pnas.1113956108.
[33] Serdar L D,Whiteside D L,Baker K E.ATP Hydrolysis by UPF1 is Required for Efficient Translation Termination at Premature Stop Codons[J].NatCommun, 2016,7:14021.DOI:10.1038/ncomms14021.
[34] Hug N,Longman D,Caceres J F.Mechanism and Regulation of the Nonsense-mediated Decay Pathway[J].NucleicAcidsRes,2016,44(4):1483-1495.DOI:10.1093/nar/gkw010.
[35] Melero R,Hug N,Lopez-Perrote A,etal.The RNA Helicase DHX34 Functions as a Scaffold for SMG1-mediated UPF1 Phosphorylation[J].NatCommun,2016,7:10585.DOI:10.1038/ncomms10585.
Mechanism and Evolution of Nonsense-mediated mRNA Decay
CHAI Baofeng,WANG Mei,SHI Wenxin,CHAI Yangli,LÜ Jia
(Institute of Loess Plateau,Shanxi University,Taiyuan 030006,China)
The nonsense-mediated mRNA decay (NMD) pathway selectively degrades mRNAs harboring premature termination codons (PTCs),but also regulates the abundance of a large number of cellular physiological mRNAs. The core of NMD pathway is the recognition and degradation of nonsense mRNA,and the function and regulation mechanism of the key factors UPFs and SMGs becomes the focus of NMD research area. The mechanism of NMD is conserved in organisms,while have differences in organisms at different evolutionary status,indicating evolutionary characteristics of the NMD pathway. The most research progresses of NMD mechanism were made from high eukaryotes,but little is known in protozoan. The researches on the function and evolution of core components of NMD using model organisms will help us unveil the function and regulatory mechanism of NMD,and establish the NMD network in humans to provide the theoretical basis for prevention and treatment of genetic diseases and cancers.
NMD pathway;nonsense-mRNA recognition;core component;functional regulation;evolution
10.13451/j.cnki.shanxi.univ(nat.sci.).2017.03.032
2017-04-17;
2017-05-18
山西省回国留学人员科研资助项目(2014-重点1)
柴宝峰(1967-),男,博士,教授,从事分子生物学方面的研究。E-mail:bfchai@sxu.edu.cn
Q756
A
0253-2395(2017)03-0639-06

