刨花润楠SRAP-PCR体系建立与优化*
周 鹏林 玮周祥斌陈晓阳
(1.广东生态工程职业学院,广东 广州510520;2.华南农业大学林学与风景园林学院/广东省森林植物种质创新与利用重点实验室,广东 广州510642)
刨花润楠SRAP-PCR体系建立与优化*
周 鹏1,2林 玮2周祥斌2陈晓阳2
(1.广东生态工程职业学院,广东 广州510520;2.华南农业大学林学与风景园林学院/广东省森林植物种质创新与利用重点实验室,广东 广州510642)
以刨花润楠(Machilus pauhoi)1.5 a生小苗幼嫩叶片为试材,对影响刨花润楠SRAP-PCR扩增的模板DNA量、引物、dNTP和Mg2+体积摩尔浓度、Taq DNA聚合酶、退火温度6个主要因素进行优化。结果表明,SRAP-PCR的最佳反应体系为:25 μL的SRAP-PCR反应体系中,2.5 μL 10×PCR buffer、模板DNA量60 ng、Mg2+2.0 mmol/L、dNTP 0.225 mmol/L、引物0.3 μmol/L和Taq DNA聚合酶1.25 U。对优化的反应体系和扩增程序的验证结果表明,优化的刨花润楠SRAP-PCR反应体系和扩增程序是稳定可行的。
刨花润楠;SRAP-PCR;体系优化
刨花润楠(Machilus pauhoi),又名刨花楠、粘柴(福州市)、刨花(广东省),属樟科(Lauraceae)润楠属植物,为常绿大乔木,产于长江以南各省,主要分布在海拔300~1 200 m的山坡及沟谷地带。刨花润楠全身是宝,是一种极具经济价值和开发前景的阔叶树种,其树干通直,生长迅速,出材量大,6 a生林木可进行采伐利用,且其砍伐后萌芽力强,更新快。从该树种叶片中提取的精油是优良的天然香料,具有独特药用价值,而树皮富含树脂和橡胶,种子含油率高达50%,可作为优良的工业润滑油,或供制皂及制蜡用[1-2]。此外,刨花润楠树形高大美观,枝繁叶茂,四季常青,嫩叶呈粉红色或红棕色,也是优良的园林绿化树种[3]。目前,对刨花润楠的研究主要集中在生物量结构、生长规律、育苗技术和观赏性等方面[4-7],有关刨花润楠种质资源保存与开发利用、种质分子鉴别等方面尚未见报道。
SRAP分子标记是由美国加州大学Li与Quiros博士提出的一种研究DNA多态性的有效新型分子标记技术[8]。该分子标记稳定可靠,信息量大,多态性及共显性高,引物具有通用性,且该技术操作简单,成本低廉[9]。目前在作物和蔬菜的遗传图谱构建、遗传多样性分析、比较基因组学和品种分子鉴定等研究领域已得到广泛应用[10-15]。近年来,逐步应用于林木的指纹图谱构建、遗传多样性分析及指纹检索等研究中[16-20]。本试验通过摸索适于刨花润楠SRAP-PCR扩增的技术条件,以期为SRAP分子标记技术应用于樟科润楠属植物的鉴别及遗传多样性的研究提供技术参考。
1 材料与方法
1.1 试验材料与试剂
供试的刨花润楠种子采于广西壮族自治区,在华南农业大学启林北苗圃播种育苗。随机选取10个家系(分别为广西贺州4个家系,广西兴安4个家系,广西昭平2个家系),每个家系选取1株生长良好的幼苗,苗龄为1.5 a生,取幼嫩叶片,在–80 ℃下保存备用,用于提取总DNA。电泳检测时选用DL2000 DNA Marker(M)进行比对参照物,所选用SRAP引物由上海生工生物工程技术服务有限公司合成。
1.2 试验方法
1.2.1 DNA提取及检测 李荣华等[21]认为,在提取DNA过程中加入预冷异丙醇和24 : 1的氯仿:异戊醇混合液,可有效去除叶片中所含的多酚、蛋白质、脂类、多酯的角质等不利于DNA提取的干扰物质,同时适当加大β-巯基乙醇的用量,可进一步减少褐变。本实验对OMEGA试剂盒(EasyPure Plant Genomic DNA Kit)加以调整改进(在提取DNA过程中加入预冷异丙醇和24 : 1的氯仿:异戊醇混合液)后,提取基因组总DNA。经1%琼脂糖凝胶电泳检测DNA质量后,稀释至所需质量体积浓度(25 ng /μL),在-20 ℃下保存备用。
1.2.2 引物的筛选 按照随机抽样方式从100对SRAP引物中选取10对进行PCR扩增,选择条带较为丰富的2对引物组合Me3(5'-TGAGTCCAAACCGGAAT-3')/Em4(5'-GACTGCGTACGAATTTGA-3')和Me4(5'-TGAGTCCAAACCGGACC-3')/Em4(5'-GACTGCGTACGAATTTGA-3')进行优化实验[22]。
1.2.3 SRAP-PCR的基础扩增程序 PCR反应在多功能PCR仪中进行,循环程序为:94 ℃预变性5 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,35个循环;最后再72 ℃延伸10 min;10 ℃保存。扩增结果用2%琼脂糖凝胶电泳检测,在紫外凝胶成像系统上观察和记录。
1.2.4 SRAP-PCR反应体系的建立与优化验证选择5个25 μL的PCR 扩增体系,采用模板2、4号样本DNA(DNA样品点样点为2 μL),选用引物组合(Me3/Em4、Me4/Em4)进行扩增,从中筛选出1个较好的反应体系;在此体系的基础上,针对影响PCR 反应的6个因素,分别对DNA模板用量、引物、dNTP和Mg2+摩尔体积浓度、Taq DNA聚合酶和退火温度设置不同的梯度(表1)进行单因素试验,在PCR扩增仪上进行扩增,对所产生的结果进行直观分析,以确定最佳反应体系[23]。并运用不同引物组合(Me18/Em7、Me18/Em6和Me24/Em9)分别对刨花润楠8个不同家系样品(选取了上述10个家系中的8个,分别是广西贺州4个家系,广西兴安中的2个家系,广西昭平2个家系)进行反应体系的验证。
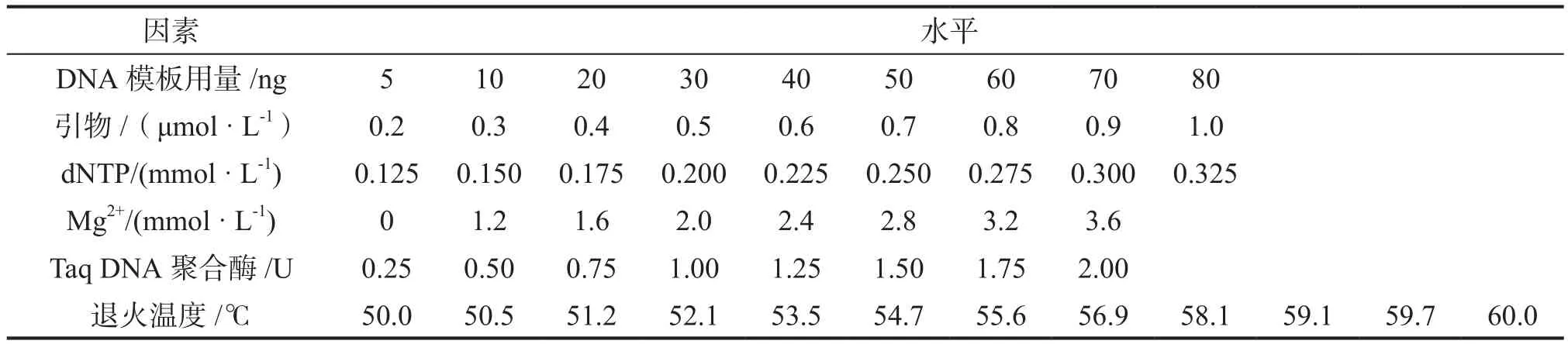
表1 SRAP反应体系梯度设置
2 结果与分析
2.1 刨花润楠叶片总DNA提取
由图1可以看出,刨花润楠总DNA的电泳图谱清晰完整,片段大于20 kb,无降解拖尾现象产生,无明显RNA存在,点样孔附近无滞留物。经估算,蛋白快速检测仪检测所提取DNA的OD260/ OD280值均为1.73~1.92,质量体积浓度为20~80 ng/μL,符合SRAP-PCR模板要求。
2.2 刨花润楠SRAP-PCR反应体系的优化
2.2.1 DNA模板用量 模板DNA用量直接影响SRPA-PCR扩增结果的条带。对刨花润楠总DNA进行PCR扩增,电泳检测扩增结果见图2。结果显示,DNA模板量对反应的影响相对较大,但DNA用量在10~80 ng时均可扩增出条带,而以模板用量为60~80 ng时最为稳定;模板用量低于60 ng时,扩增条带较少。结合本试验的结果,确定60 ng为最佳DNA模板用量。

图1 刨花润楠10株不同幼苗基因组总DNA提取电泳图

图 2 基因组DNA模板量对刨花润楠SRAP-PCR反应的影响
2.2.2 Taq DNA聚合酶浓度 Taq DNA聚合酶的活性和用量对SRAP-PCR反应影响较大。由图3可以看出,0.25~2.00 U都可以得到扩增条带,聚合酶用量过低时,PCR反应无法完全进行,条带较为模糊;1.25~1.50 U时效果最好,扩增结果稳定性好、多态性高;随着酶用量增大,易产生部分非特异性产物且出现拖尾现象。据此,本试验最终采用的Taq酶适宜用量为1.25 U。
2.2.3 Mg2+浓度 Mg2+摩尔体积浓度是影响SRAPPCR反应的主要因素之一。本研究中对不同摩尔体积浓度的Mg2+进行PCR 扩增,结果(图4)表明,Mg2+摩尔体积浓度对反应的影响较为明显,当Mg2+为0时无扩增条带出现;随着Mg2+的增加,扩增条带亮度明显增大;当增至2.0 mmol/L时,条带清晰,此时效果最佳;当Mg2+为3.6 mmol/L,产生的扩增条带模糊且特异性差。故选择2.0 mmol/L为最佳。

图 3 TaqDNA聚合酶量对刨花润楠SRAP-PCR反应的影响
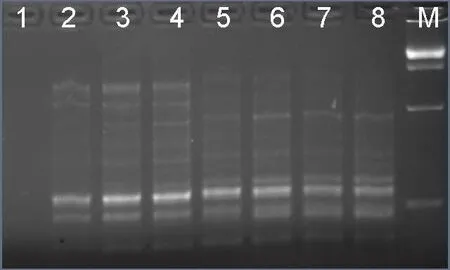
图 4 Mg2+摩尔体积对刨花润楠SRAP-PCR反应的影响
2.2.4 dNTP浓度 dNTP是SRAP-PCR扩增反应的原料。由图5 可知,当dNTP为0.125~0.325 mmol/ L时,都能扩增出条带, dNTP介于0.125~0.225 mmol/L之间时,扩增条带相对较少且较为模糊,当dNTP为0.225 mmol/L时,此时扩增条带最多且最为清晰;dNTP大于0.225 mmol/L,随着摩尔体积浓度增加,扩增条带逐渐减弱。据此,本试验最终确定dNTP的摩尔体积浓度为0.225 mmol/L。
2.2.5 引物浓度 引物的浓度直接影响PCR扩增效果。由图6可知,当引物低于0.2 μmol/L,扩增产物较少;当引物在0.3~0.5 μmol/L时,扩增产物条带多且条带清晰稳定。之后,随着引物增加,扩增条带清晰度降低。根据该试验结果,将适宜的引物摩尔体积确定为0.3 μmol/L。

图 5 dNTP摩尔体积浓度对刨花润楠SRAP-PCR反应的影响
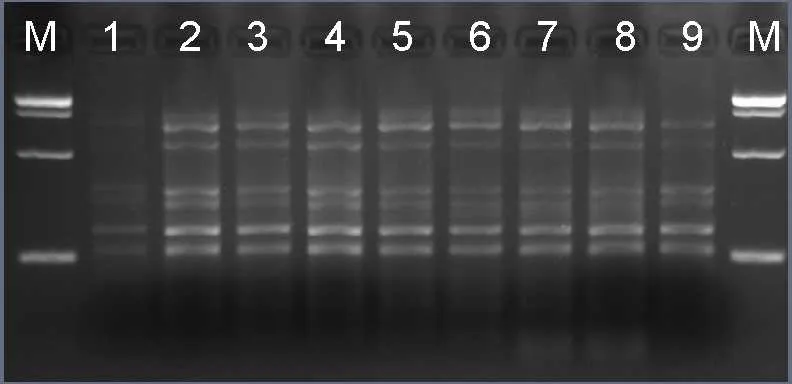
图 6 引物摩尔体积对刨花润楠SRAP-PCR反应的影响
2.2.6 退火温度 退火温度直接影响引物与模板DNA的特异性结合能力。由图7可知,本试验中,当退火温度为54.7~56.9 ℃时,扩增出的谱带多且清晰明亮,故确定PCR反应体系中退火温度为56 ℃。在引物筛选时,针对不同引物退火温度可做适当的调整。
2.2.7 SRAP-PCR最佳反应体系的验证 综合以上各试验及所获结果,从扩增产物条带稳定性和节约成本角度考虑,刨花润楠SRAP-PCR最佳反应体系为:25 μL的SRAP-PCR反应体系中,2.5 μL 10×PCR buffer、模板DNA量60 ng、Mg2+2.0 mmol/L、dNTP 0.225 mmol/L、引物0.3 μmol/L和Taq DNA聚合酶1.25 U。运用不同引物组合(Me18/Em7、Me18/Em6和Me24/Em9)分别对刨花润楠8个家系样品进行验证(图8),检验结果表明引物能对绝大多数的个体扩增出清晰谱带,说明优化的刨花润楠SRAP-PCR反应体系和扩增程序是稳定可行的。
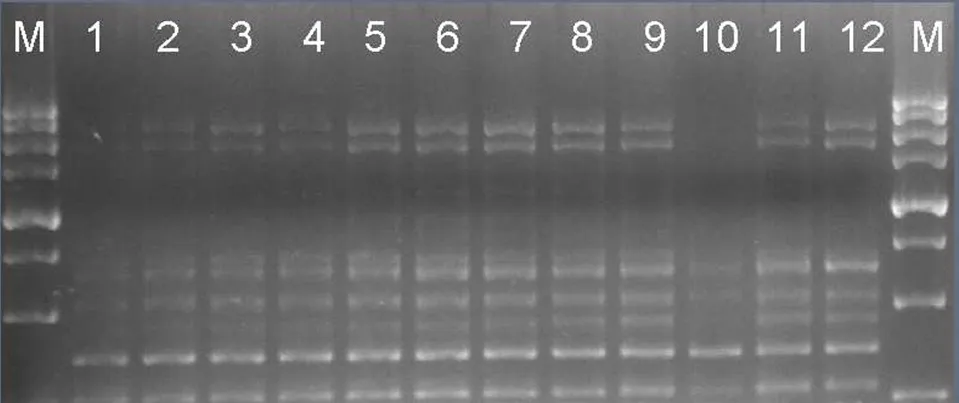
图 7 退火温度对刨花润楠SRAP-PCR反应的影响
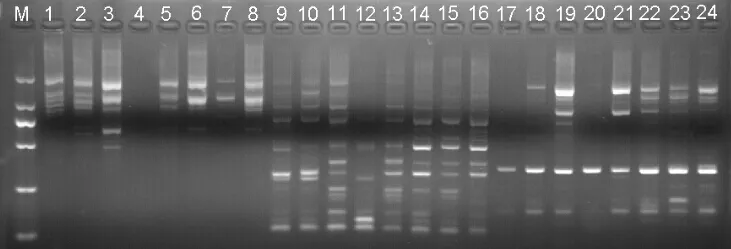
图8 应用SRAP-PCR优化体系对刨花润楠不同样本进行多态性检测
3 结论与讨论
本试验使用omega试剂盒,采用改良CTAB法提取刨花润楠的基因组DNA,试验中发现,在提取过程中,加入24 : 1的氯仿:异戊醇混合液充分振荡混匀后,在18~20 ℃环境中离心,可有效提高DNA得率及质量。
对于不同的林木材料,扩增体系所用的DNA模板量、Taq 聚合酶、Mg2+、dNTP、引物摩尔体积浓度差异均很大,本试验所得到的SRAP-PCR扩增体系与麻疯树(Jatropha carcas)[24]、石斛(Dendrobium nobile)[25]、杨树(Populussp.)[26]、油茶(Camellia oleifera)[27]等的扩增体系及循环条件都有所不同,这可能与不同材料的基因组和材料本身的特殊性以及对试验结果条带的判断具有主观性等有关,因而需要对PCR体系进行不断优化。PCR循环条件中的退火温度对反应影响较大,该反应中退火温度相比其他已报道麻疯树、石斛、油茶、杨树等植物偏高,筛选出的适宜退火温度(56 ℃)与理论退火温度(50 ℃)[28]存在较大差异。但同时退火温度过高或过低都不能得到良好的扩增产物;扩增体系中DNA模板量和Mg2+浓度对反应的影响相对较大,微量变化就有可能导致SRAP-PCR的结果发生较大变化,合理调整DNA模板和Mg2+摩尔体积浓度对反应的扩增很重要。
利用本试验优化所得的SRAP-PCR反应体系,对刨花润楠23个种源进行SRAP扩增,扩增结果表明在不同种源的刨花润楠中均能获得清晰稳定的条带,不同种源间表现出明显的多态性[29]。由此可见,该反应体系可用于刨花润楠的遗传多样性分析。
[1] 胡希华. 刨花润楠的优良特性及育苗栽培技术[J]. 湖南林业科技, 2006, 33(1): 65-66.
[2] 吴振伙, 吴兆平, 全尚龙. 刨花润楠综合利用价值及其育苗技术[J]. 现代农业科技, 2008(20): 55.
[3] 钟智群, 谭梓峰, 杨志玲, 等. 刨花楠生长发育特点及开发前景分析[J]. 湖南林业科技, 1997, 24(2): 50-51.
[4] 程栋梁, 靳冰洁, 徐朝斌, 等. 年龄对刨花楠胸径生长速率的影响[J]. 安徽农业大学学报, 2013, 40(1) : 28 -31.
[5] 徐朝斌, 钟全林, 程栋梁, 等. 江西4刨花楠种源苗木叶片表型性状与生物量分配的比较[J]. 安徽农业大学学报, 2012, 39(6) : 920 -924.
[6] 张冬生, 谢金兰, 叶雪兰, 等. 基于层次分析法与熵技术法的野生刨花润楠观赏性评价[J]. 林业与环境科学, 2016, 32(1): 23-30.
[7] 罗阿水.刨花楠容器育苗技术研究[J].福建林业科技, 2012, 39(3) : 126-129.
[8] LI G, QUIROS C F. Sequence-related amplifed polymorpgism (SRAP) a new marker system based on a simple PCR reaction: its application to mapping and genetagging in Brassica[J]. TAG Teoretical and Applied Genetics, 2001, 103: 455-461.
[9] 王国泽, 左福元, 曾兵. SRAP分子标记的研究进展及在植物上的应用[J]. 畜牧与兽医, 2013, 45(9): 92-95.
[10] LI G, GAO M, YANG B, et al. Gene for genealignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J]. TAG Theoretical and Applied Genetics, 2003, 107(1): 168-180.
[11] 井赵斌, 徐明, 雷玉山. 猕猴桃SRAP-PCR体系的建立及品种资源亲缘关系研究[J]. 园艺学报, 2016, 43(2): 337-346.
[12] 李达, 熊兴耀, 于晓英, 等. 红花檵木SRAP反应体系的建立及优化[J]. 中南林业科技大学学报, 2008, 28 (5) : 22-27.
[13] 刘君, 李东, 曾林, 等. SRAP分子标记分析辣椒种质资源遗传多样性研究[J]. 中国调味品, 2015, 12(40): 48-51.
[14] 王燕, 龚义勤, 赵统敏, 等. 番茄SRAP-PCR体系优化与品种分子鉴定[J]. 南京农业大学学报, 2007, 30(1): 23-29.
[15] 李怀志, 张峻, 李翔, 等. 应用SRAP标记对茄子品种进行遗传多样性分析与指纹图谱构建[J]. 南京农业大学学报, 2011, 34(4): 18-22.
[16] 艾鹏飞, 苏姗, 靳占忠. 仁用杏品种SRAP遗传多样性分析及指纹检索系统的开发[J]. 园艺学报, 2014, 41(6): 1191-1197.
[17] 廖柏勇, 王芳, 陈丽君, 等. 基于SRAP分子标记的苦楝种质资源遗传多样性分析[J]. 林业科学, 2016, 52(4): 48-58.
[18] 李培, 阙青敏, 欧阳昆唏, 等. 不同种源红椿SRAP标记的遗传多样性分析[J]. 林业科学, 2016, 52(1): 62-70.
[19] 杨会肖, 刘天颐, 罗锐, 等. 用SRAP标记构建松树良种指纹图谱方法的研究[J]. 广东林业科技, 2012, 28(5): 1-8.
[20] 刘振, 赵洋, 杨培迪, 等. SSR、SRAP、ISSR分子标记在茶树品种亲本鉴定上的比较分析[J]. 茶叶科学, 2014(6): 617-624.
[21] 李荣华, 夏岩石, 刘顺枝, 等. 改进的CTAB法提取植物DNA的方法[J]. 实验室研究与探索, 2009, 28(9): 14-16.
[22] 李建军, 刘志坚, 肖层林, 等. SRAP技术在遗传的研究进展[J]. 现代生物医学进展, 2007, 7(5): 783-786.
[23] 刘玉香, 宋晓琛, 江香梅. 润楠ISSR-PCR优化反应体系建立及引物筛选[J]. 林业科技开发, 2013, 27(5): 24-28.
[24] 仲丛来, 丁贵杰, 沈凌. 麻疯树SRAP-PCR反应体系的优化[J]. 贵州农业科学, 2010, 38(4): 19-22.
[25] 樊洪泓, 李廷春, 邱婧, 等. 石斛属植物SRAP反应体系的建立与优化[J]. 分子植物育种, 2006, 4(6S): 153-156.
[26] 谭碧玥, 王源秀, 徐立安. 杨树基因组SRAP扩增体系的建立与优化[J]. 林业科技开发, 2009, 23(2): 25-29.
[27] 祝全东, 张党权, 李晓云, 等. 油茶SRAP标记的PCR体系建立与优化[J]. 中南林业科技大学学报, 2010, 30(3): 57-62.
[28] 卢盛栋. 现代分子生物学实验技术[M].北京: 中国协和医科大学出版社, 1999: 458-463.
[29] 周鹏, 林玮, 朱芹, 等. 基于SRAP分子标记的刨花润楠遗传多样性分析[J] . 北京林业大学学报, 2016, 38(9): 16-24.
Establishment and Optimization of SRAP-PCR System forMachilus pauhoi
ZHOU Peng1,2LIN Wei2ZHOU Xiangbin2CHEN Xiaoyang2
(1.Guangdong Eco-engineering Polytechnic, Guangzhou,Guangdong 510520, China; 2. College of Forestry and Landscape Architecture, Sourth China Agricultural University/Guangdong Key Laboratory for Innovative Development and Utilization of Forest Plant Germplasm, Guangzhou, Guangdong 510642, China)
Machilus pauhoiis a tree specie with variety of economic value and development prospects. This study aimed to establish an optimized SRAP-PCR system forM. pauhoi,and the young leaves of the 1.5 years old seedlings were used as test materials. Six quality factors including the template DNA, primer concentration, dNTP concentration, Mg2+concentration, Taq DNA polymerase, and annealing temperature were optimized forM. pauhoiSRAP-PCR assay. The obtained results suggested a optimized reaction system of SRAP-PCR (total 25 μL) involving 2.5 μL 10×PCR buffer, 60 ng DNA, 2.0 mmol/ L Mg2+, 0.225 mmol/L dNTP, 0.3 μmol/L primer, 1.25 U Taq DNA polymerase. The verification results showed that the optimized SRAP-PCR reaction system and amplifcation program were stable and feasible.
Machilus pauhoi;SRAP-PCR;system optimization
S718.43,S792.23
:A
:2096-2053(2017)04-0029-05
“十二五”国家科技支撑项目“刨花润楠和黄樟良种选育研究”(2012BAD01B04);广东省林业科技创新项目“楝科、樟科优质速生树种良种选育和高效栽培技术研究与示范”(2011KJCX002)。
周鹏(1989— ),男,助教,主要从事林木遗传育种研究,E-mail:zhoupeng_0119@163.com。
陈晓阳(1958— ),男,教授,主要从事林木良种选育与生物技术研究,E-mail:xychen@scau.edu.cn。

