七里峪三裂绣线菊种群的遗传分化研究
李 佳,陈 伟,樊泽璐,武艳虹,郭晋宏,郭雅坤,王祎玲
(山西师范大学 生命科学学院,山西 临汾 041000)
七里峪三裂绣线菊种群的遗传分化研究
李 佳*,陈 伟*,樊泽璐,武艳虹,郭晋宏,郭雅坤,王祎玲
(山西师范大学 生命科学学院,山西 临汾 041000)
采用Simple Sequence Repeats(SSR)分子标记对三裂绣线菊(Spiraea trilobata L.)8个海拔的种群进行遗传多样性和遗传结构分析. 结果表明,17对具有稳定多态性的SSR引物,共被检测出380个多态性位点,多态位点百分率(PPB)为59.61%;观察等位基因平均数(Na)、有效等位基因数(Ne)、Shannon多样性指数(I)、Nei's基因多样性指数(H)分别为1.596 1,1.309 3,0.287 4,0.187 7. 三裂绣线菊种群间的遗传多样性较高;种群内的遗传变异高于种群间(GST=0.309 7),说明种群内变异是其主要变异来源. 8个种群明显聚为两支,海拔最高的两个种群(种群7、8)聚为一支,与PCoA和Structure分析结果相吻合. 本文结果可为三裂绣线菊种质资源保护和可持续开发利用提供科学的理论依据.
三裂绣线菊;SSR;遗传多样性;遗传差异
1 引言
三裂绣线菊(Spiraea trilobata L.)为蔷薇科(Rosaceae)绣线菊属(Spiraea L.)落叶灌木,主要分布于华北、华东、西北、西南、华中及东北等地,常生长于海拔450~2 400 m的多岩石向阳坡、林缘、路边和沟旁等地的灌木丛中,具有很高的观赏价值和药用价值[1-2]. 目前对三裂绣线菊的研究主要集中在内部成分、生物学特性等方面[3-8],如三裂绣线菊的引种实验和抗旱性研究、土壤成分对叶结构的影响、身份证的构建和区分.
Simple Sequence Repeats(SSR)又叫微卫星DNA(Microsatellite DNA),在真核生物基因组中大量存在,分布在结构基因之间[7]. SSR是由2~6个碱基构成的基本重复单元串联重复而产生的长度达几十个碱基的序列,不同材料在同一重复区段的重复次数不完全相同,当选用与重复区域两侧高度保守的序列(一般是单拷贝)互补的特异引物进行PCR扩增,就可用电泳检测出该重复区域的多型性[8]. 目前,SSR己广泛地应用于遗传连锁图谱的构建、基因定位、品种鉴定、种质分类及遗传多样性研究[9-13].
本文采用SSR分子标记对三裂绣线菊不同海拔种群进行遗传差异分析,旨在揭示三裂绣线菊不同种群的遗传多样性及遗传结构,分析遗传变异与环境变化之间的关系,为进一步分析其进化历史、进化潜力和未来命运提供重要的资料.
2 材料与方法
2.1 材料采集
根据三裂绣线菊在山西七里峪的分布,从1 000 m海拔开始采集,海拔之间相隔100 m,每个海拔各采集20个个体,共采集8个海拔种群,分别记为1(1 000 m),2(1 100 m),3(1 200 m),4(1 300 m),5(1 400 m),6(1 600 m),7(1 700 m),8(1 900 m). 每个海拔收集新鲜健康的幼嫩叶片,用变色硅胶迅速干燥,变色后立即更换直至叶片变脆,带回实验室,置于冰箱低温保存并进行实验分析.
2.2 实验方法
2.2.1 DNA的提取与检测
运用改良的2×CTAB法[14]提取三裂绣线菊基因组的DNA. 用紫外分光光度计和0.8%琼脂糖凝胶电泳检测DNA质量,在-20 ℃保存备用.
2.2.2 SSR-PCR扩增引物筛选与扩增
从28对SSR通用引物中筛选出扩增产物条带清晰,重复性好的17对引物(见表1),对所有的DNA个体样本进行扩增. 实验根据不同引物的Tm值设计12个温度梯度,确定不同引物的退火温度.
反应体系及程序:15 μL反应体系(4.5 μL引物,0.8 μg DNA,7.5 μL 2×Mix)中,95 ℃ 4 min;35个循环(94 ℃ 50 s,(Tm梯度)1 min,72 ℃1 min);72 ℃ 10 min;10 ℃保存.
用浓度为12%的聚丙烯酰氨凝胶电泳分离PCR扩增产物. 缓冲液为1×TBE,取6 μL PCR产物进行点样,2 μL Marker作为标尺,电压200 V、电流30 mA,电泳时间约为3 h. 电泳结束后,小心取下胶并放
置在倒有固定液的摇胶盒中,摇至蓝色条带消失(摇动过程中尽量保持匀速);再用双蒸水清洗2 min,倒掉并控水,加入AgNO3染色液染色10~15 min;染色结束后,用双蒸水冲洗凝胶2~3次,加入含1.5% NaOH和0.4% HCHO的显影液,摇至所有条带显现为止;取出凝胶,拍照保存并记录数据.

表1 三裂绣线菊SSR扩增引物
2.3 数据统计与分析
根据所得到的凝胶电泳图像,对每个位点是否存在条带分别进行统计,有条带的计数为“1”,无条带的计数为“0”,最终得到原始的“0,1”数据矩阵.
运用POPGENE V1.32软件对每个矩阵位点进行统计分析,分析得到等位基因数(Na)、有效等位基因数(Ne)、多态位点百分率(PPB)、Shannon多样性指数(I)、Nei's基因多样性指数(H)等相关参数.
根据Nei's基因遗传距离,采用NTSYS软件聚类分析不同海拔种群的亲缘关系,Treeplot模块生成树状聚类图. 运用GenAlEx V6.1软件进行PCoA分析,揭示不同种群个体间的相互关系;运用Structure V2.2软件分析其遗传结构,估计其最佳群体组群数K,设定取值范围为2~8,根据Structure harvester软件分析得到最佳K值.
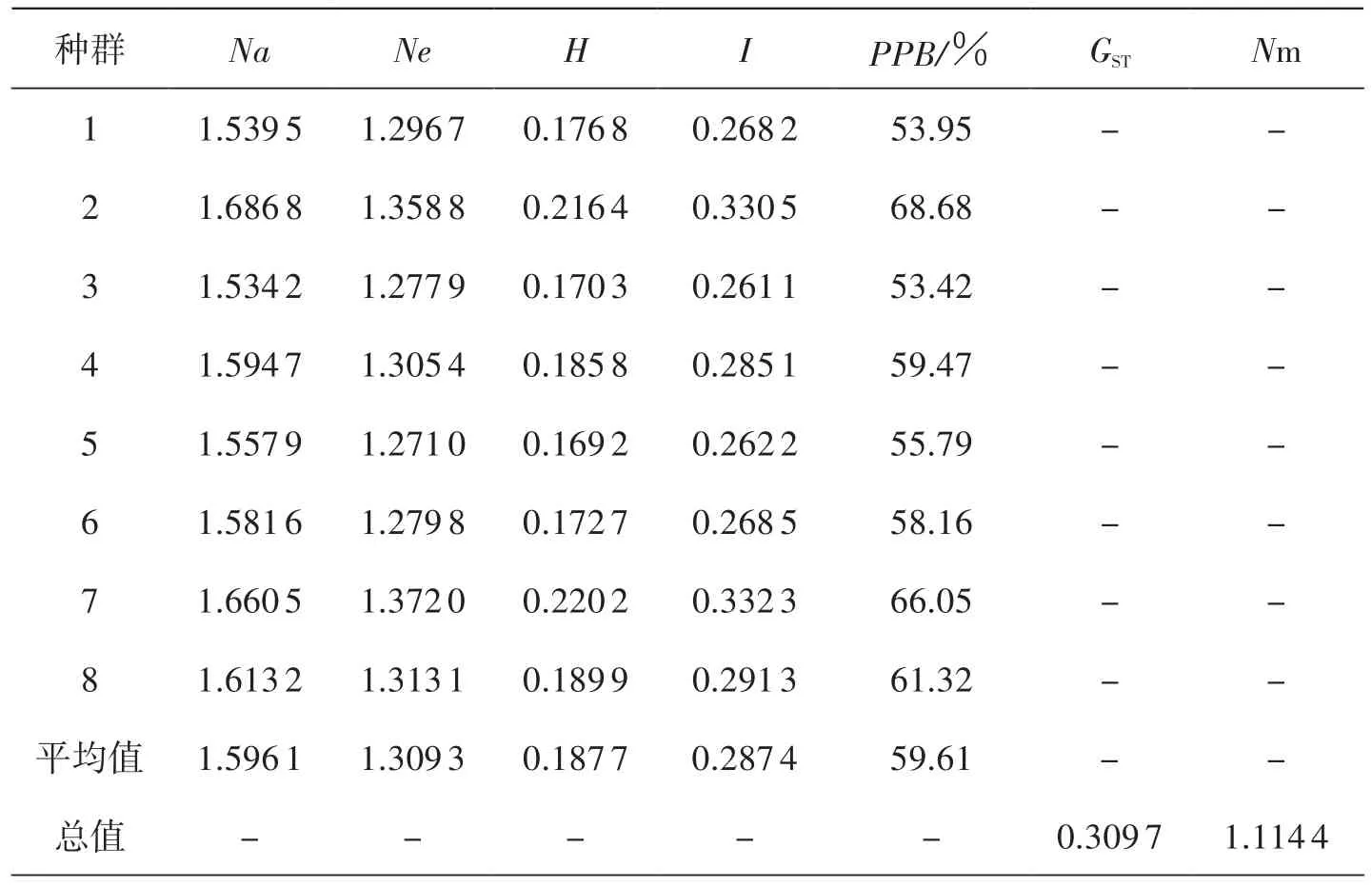
表2 三裂绣线菊遗传多样性指数
3 结果与分析
3.1 三裂绣线菊的遗传多样性
不同海拔种群的等位基因数(Na)的波动范围在1.534 2~1.686 8之 间,平 均值为1.596 1;有效等位基因数(Ne)处于1.271 0~1.372 0之 间,平 均 值 为1.309 3;Shannon多样性指数(I)变动范围为0.261 1~0.332 3,平均值为0.287 4;Nei's基因多样性指数(H)在0.169 2~0.220 2之间,平均值为0.187 7;多态位点百分率(PPB)介于53.42%~68.68%之间,平均值为59.61%(见表2). H、I和PPB均表明三裂绣线菊种群的遗传多样性较高.
在各项遗传多样性指数中,种群2的遗传多样性最高(Na=1.686 8;Ne=1.3588;H=0.216 4;I=0.330 5;PPB=68.68%);种群3的遗传多样性最低(Na=1.534 2;Ne=1.277 9;H=0.170 3;I=0.261 1;PPB=53.42%),三裂绣线菊的种群遗传多样性从大到小依次为种群2>7>8>4>6>5>1>3.
3.2 三裂绣线菊种群的遗传结构
三裂绣线菊8个不同海拔种群的遗传分化系数GST为0.309 7,Nm值为1.114 4,说 明 不同海拔三裂绣线菊种群的遗传多样性主要存在于种群内. 种群之间的遗传一致度介于0.835 1~0.968 2之间,遗传距离介于0.032 3~0.180 6之间,种群7和8之间的遗传一致度最高,种群1和8之间的遗传一致度最低,表明种群7和8之间亲缘关系最近,种群1和8之间亲缘关系最远(见表3).
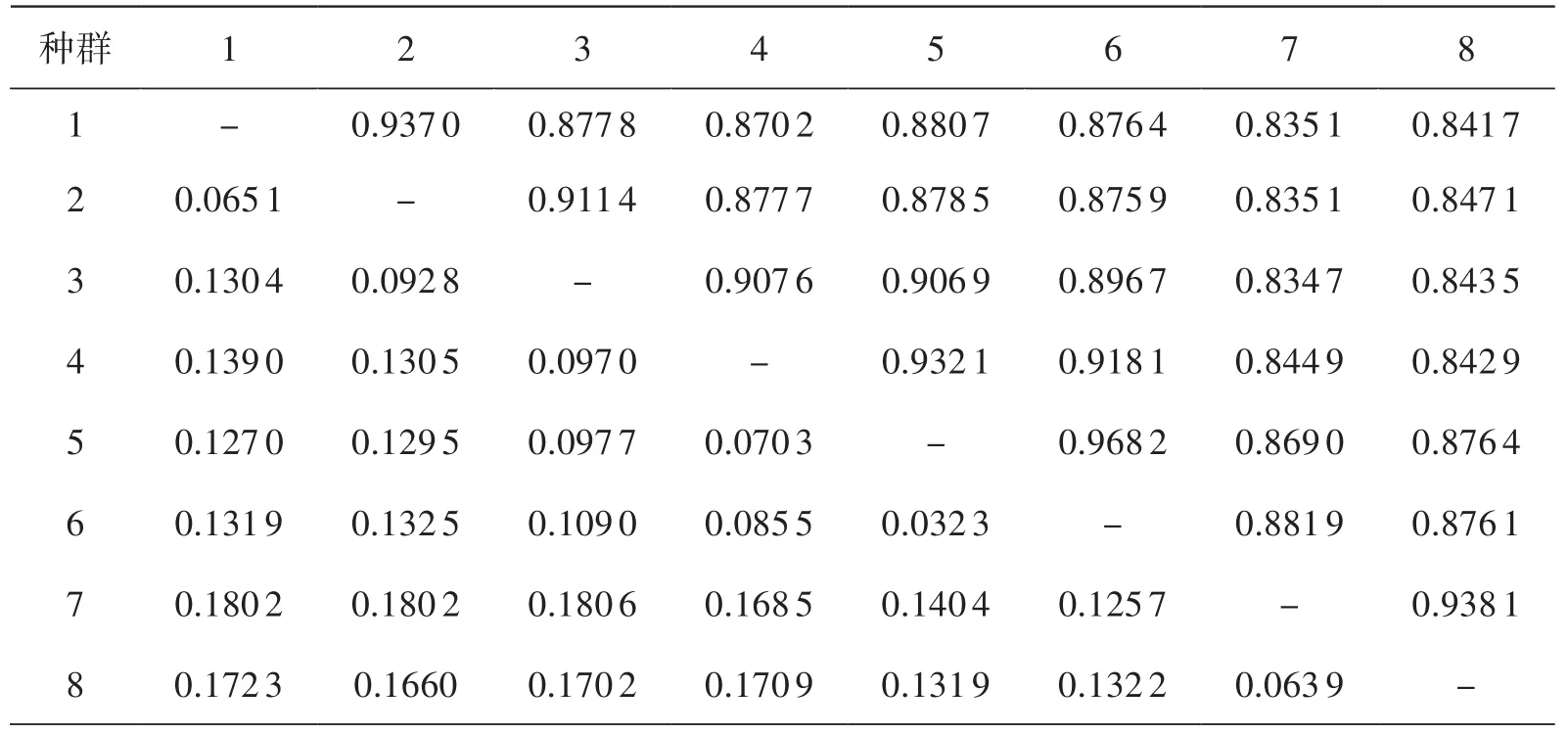
表3 三裂绣线菊种群基于Nei's指数的遗传距离(对角线下方)和遗传一致度(对角线上方)
NTSYS聚类分析中,三裂绣线菊8个种群明确分为两支:种群1,2,3,4,5和6聚为一支,种群7和8聚为一支,这个结果与PCoA分析结果(图1)相吻合. Structure分析中,K=2时,△K达到最高值(图2),与PCoA分析、NTSYS聚类(图3)基本一致.
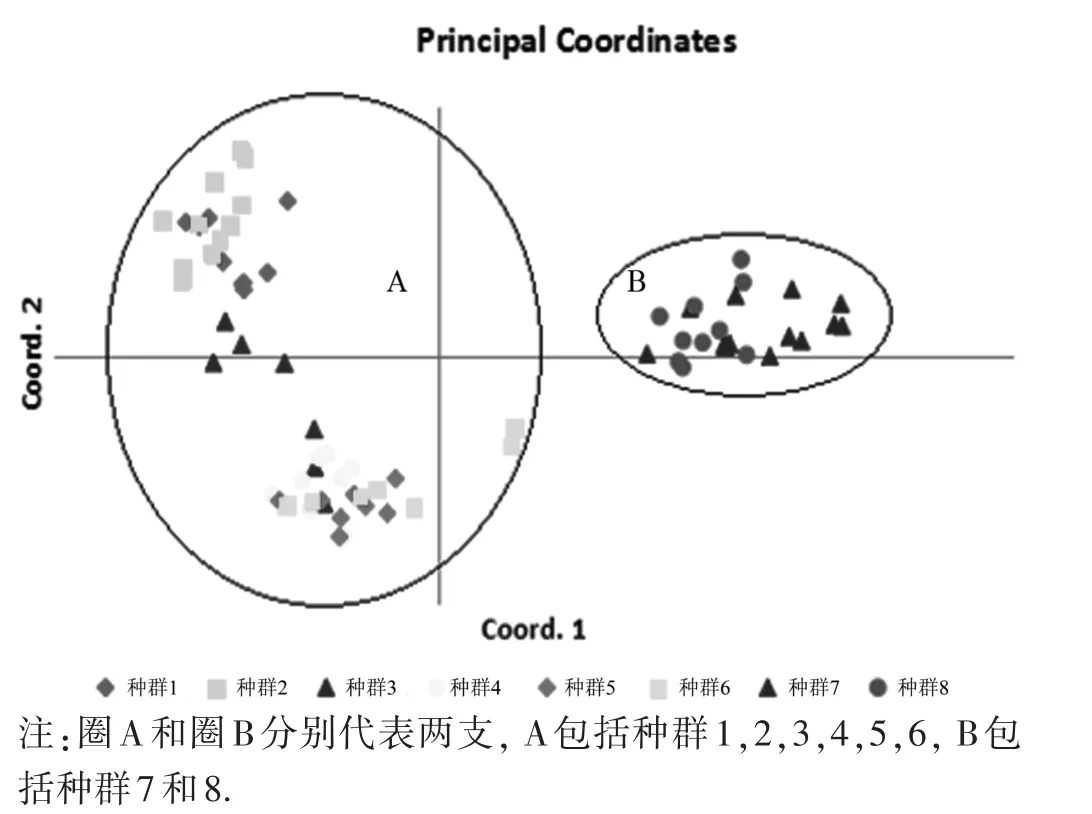
图1 三裂绣线菊8个种群的PCoA分析图
4 讨论
遗传多样性是指生物所携带的遗传信息的总和,是种群生存、进化和发展的基石,反映了物种对于其生存环境的适应能力和对非正常环境影响下的生存能力,同时作为生物多样性的基础和重要组成部分,是学者们长期研究的热点[15-18].一般来说,遗传多样性受很多因素的影响,如繁育体系、生物学特性、地理分布和种群大小等[19].三裂绣线菊是多年生灌木植物,繁殖速度快且易成活,在长期的进化过程中积累了一定量的遗传变异. 另外,广布种的遗传多样性水平要比分布狭窄的物种高,三裂绣线菊广泛分布在中国地区,也是其遗传多样性较高的一个原因[20]. 而七里峪三裂绣线菊主要分布在海拔1 000~1 900 m左右的路边向阳岩石上,这是该风景区放牧和游玩的主要区域,因此我们采集材料的种群大小受到了一定的限制.
8个不同海拔三裂绣线菊种群的遗传多样性差异较为明显,遗传变异随着海拔高度的升高呈低-高-低-高的分布,这种变异情况的产生可能来自于生存环境的异质性. 七里峪采样地属于太岳山国家森林公园的重要组成部分,高海拔三裂绣线菊种群所处的生境多为阳坡,分布的灌木树种比较少,主要以三裂绣线菊为亚优势种的群落居多,群落结构单一,相对于低海拔生境更适合于其生长;而低海拔的群落正处于与红柄白鹃梅、连翘等物种的交错区,并受到放牧、砍伐等人为干扰,导致三裂绣线菊种群间群落结构及种类组成趋于单一[21].
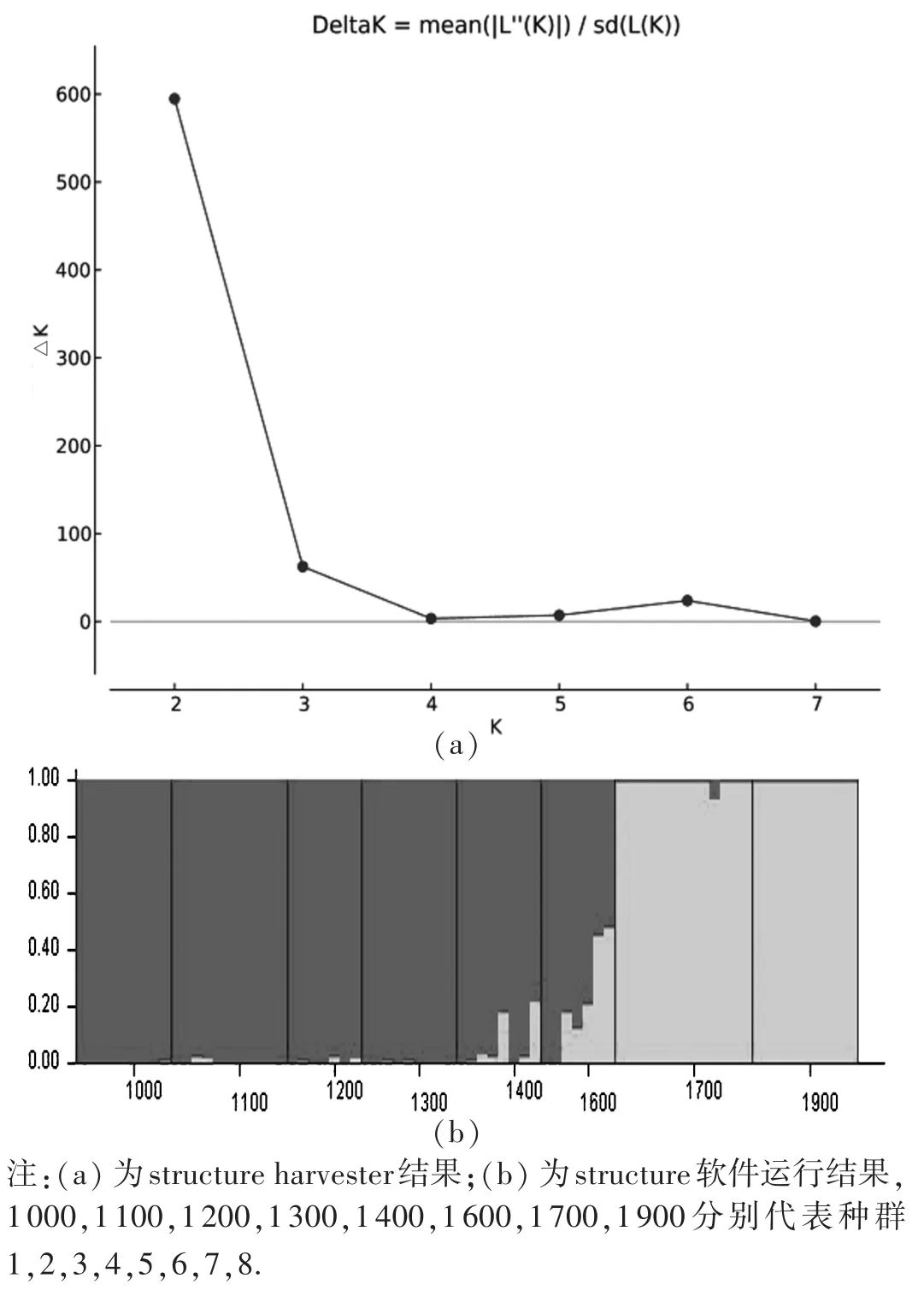
图2 用Structure分析得到的8个三裂绣线菊种群遗传结构(K=2)
种群遗传结构是指遗传多样性在种群间和种群内的分化[22],本文中三裂绣线菊8个不同海拔种群的遗传变异主要存在于种群内. 由于基因流在一定程度上能反映种群间遗传物质的交流,Wright以1作为评价基因流高低的临界点,认为基因流大于1能发挥其均质化的作用;而小于1则说明基因流成为遗传分化的主要原因[23]. 本文中三裂绣线菊种群间的Nm值为1.114 4,大于1,说明种群间存在基因交流,在一定程度上阻碍了种群间发生遗传分化. 通过野外调查采样发现,三裂绣线菊种子很容易萌发为幼苗,幼苗在向成株转化阶段的死亡率较低,增加了种群内的遗传多样性. 三裂绣线菊为喜光物种,以种子繁殖,种群呈较强的集群分布,林隙和光斑对其种群结构和功能起着重要作用[24-25].另外,不同海拔高度的环境因子不同,海拔1 700~1 900 m土壤多为棕色森林土和山地草甸土,三裂绣线菊种群由于成年数量较多,对光照、水分等空间环境因子竞争激烈,产生自疏作用,个体空间分布相对均匀,增加了种内遗传变异度. 低海拔多为褐土带,受人为干扰和破坏严重,形成的林缘、林隙相对较多,因其喜光以寻求光照,呈现较高程度的集群分布,降低了种内的遗传分化.
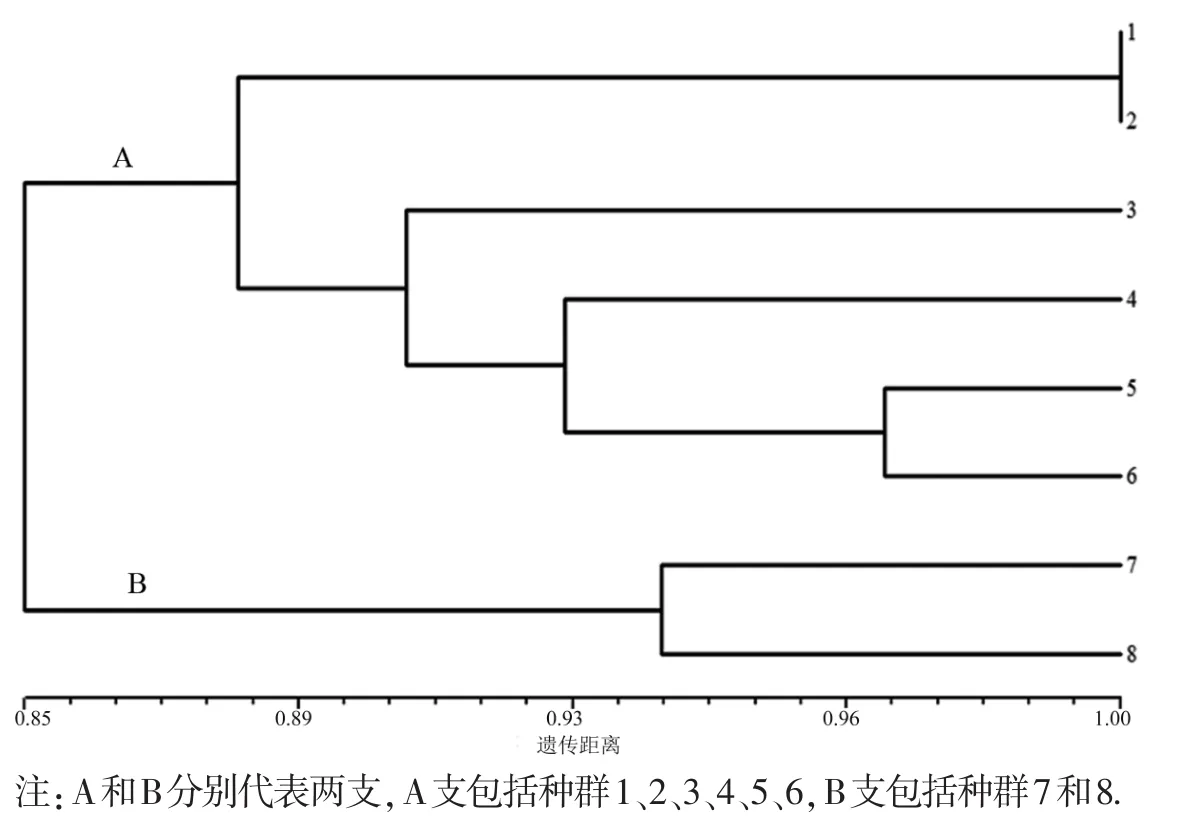
图3 NTSYS三裂绣线菊种群之间遗传距离聚类分析图
[1]陆玲娣. 中国蔷薇科绣线菊亚科的演化、分布:兼述世界绣线菊亚科植物的分布[J]. 植物分类学报,1996(4):361-375.
[2]桂炳中,陈东青,高惠茹. 华北地区三裂绣线菊栽培养护[J]. 中国花卉园艺,2015(14):44-45.
[3]肖芳. 三种绣线菊引种及抗旱适应性研究[D]. 保定:河北农业大学,2006.
[4]王芳,刘宁. 不同坡位野生三裂绣线菊和黄刺玫成年植株的叶特性[J]. 中国水土保持科学,2015(3):117-123.
[5]李春红. 几种绣线菊生物学特性、地上生物量及繁殖技术研究[D]. 呼和浩特:内蒙古农业大学,2008.
[6]邢燕红,燕玲,岳秀贤. 五种绣线菊属植物的化学成分分析及其资源评价[J]. 干旱区资源与环境,2010(6):143-148.
[7]李春红. 几种绣线菊生物学特性、地上生物量及繁殖技术研究[D]. 呼和浩特:内蒙古农业大学,2008.
[8]王芳,刘宁. 不同坡位野生三裂绣线菊和黄刺玫成年植株的叶特性[J]. 中国水土保持科学,2015,13(3):117-123.
[9]谢云. 浙江红山茶遗传多样性分析及观赏价值评价[D]. 北京:中国林业科学研究院,2011.
[10]郑常祥. 玉米二环系源杂交种亲本选择及其相关系的SSR分子标记分析[D]. 雅安:四川农业大学,2004.
[11]于海萍. 牡丹SSR分子标记的开发及其在亲缘关系分析中的应用[D]. 北京:北京林业大学,2013.
[12]李明芳. 荔枝SSR标记的研究及其对部分荔枝种质的遗传多样性分析[D]. 海口:华南热带农业大学,2003.
[13] 林丽丽,张翠琴,王祎玲. 太白山独叶草6个海拔种群的遗传多样性SSR分析[J]. 西北植物学报,2014(6):1112-1118.
[14]淦国英,漆艳香,蒲金基,等. 改良CTAB法提取高质量香蕉叶片总RNA[J]. 广东农业科学,2009(7):192-195.
[15]陈灵芝. 中国的生物多样性现状及其保护对策[M]. 北京:科学出版社,1993:93-113.
[16] KERR JT. Species Richness,Endemism and the Choice of Areas for Conservation[J]. Conservation Biology,1997,11:1094-1100.
[17] 李丹,彭少麟. 三个不同海拔梯度马尾松种群的遗传多样性及其与生态因子的相关性[J]. 生态学报,2001(3):415-421.
[18] 金则新,李钧敏. 浙江天台山不同海拔高度七子花种群遗传多样性及其与环境因子的相关性分析[J]. 浙江大学学报(理学版),2005,32(4):452-458.
[19] HAMRIEKE JL,GODT MJ W,SHERMAN-BROYES L. Factors in fluencing levels diversity in two plant species[J]. New Forest,1992(6): 95-124.
[20] TATE JA,SIMPSON BB. Paraphyly of Tarasa (Malvaceae) and diverse origins of the polyploidy species[J]. Systematic Botany, 2003, 28: 723-737.
[21]姚雪芹. 山西土庄绣线菊和三裂绣线菊群落特征研究[D]. 临汾:山西师范大学,2016.
[22] 黄玮,孙平,张文生,等. 北京东灵山地区不同海拔柴胡种群的遗传多样性[J]. 植物遗传资源学报,2008(4):453-457.
[23]WRIGHT S. Evolution in Mendelian population[J]. Genetics,1931,16:97-159.
[24]武小钢,郭晋平. 关帝山华北落叶松天然更新种群结构与空间格局研究[J]. 武汉植物学研究,2009,27(2):165-170.
[25] 李庆康,马克平. 植物群落演替过程中植物生理生态学特性及其主要环境因子的变化[J]. 植物生态学报,2002,26(9):109-116.
Abstract:In this study, simple sequence repeats were used to analyze the genetic diversity and genetic structure of Spiraea trilobata L. in eight populations. The results showed that 17 pairs had SSR primers with stable polymorphism, that a total of 380 polymorphic loci were detected, that the percentage of polymorphic band (PPB) was 59.61%, that the observed number of alleles (Na), the effective number of alleles (Ne), Shannon's information index (I) and Nei’s gene diversity (H) were 1.5961, 1.3093, 0.2874 and 0.1877 respectively, and that a higher level of genetic diversity among the populations of Spiraea trilobata L. The genetic diversity within populations was higher than among populations (GST=0.3097), indicating that variation within population was the main source of variation for its population. The eight populations were clustered into two groups, and the two highest altitude populations (7 and 8) were clustered into one group, which was consistent with the results of PCA analysis and structure. Not only can the result provide scientific theory basis for germplasm resources protection of Spiraea trilobata L., but it can also be used for its sustainable development and utilization.
Key words:SSR maker; Spiraea trilobata L; genetic diversity; genetic difference
Genetic Differentiation of Spiraea trilobata L. in Qiliyu
LI Jia , CHEN Wei , FAN Zelu, WU Yanhong, GUO Jinhong, GUO Yakun, WANG Yiling
(College of Life Sciences, Shanxi Normal University, Linfen 041000, China)
Q941
A
1008-2794(2017)04-0080-06
2017-05-20
王祎玲,教授,博士,研究方向:分子生态学,E-mail:ylwangbj@hotmail.com.
*李佳、陈伟为共同第一作者.

