基于GenBank数据探讨兜兰属植物DNA条形码
杨 鹏,关 萍,陈兴银,张凯凯,彭昌琴,成 宇,石建明
(贵州大学 生命科学学院,贵州 贵阳 550025)
基于GenBank数据探讨兜兰属植物DNA条形码
杨 鹏,关 萍,陈兴银,张凯凯,彭昌琴,成 宇,石建明*
(贵州大学 生命科学学院,贵州 贵阳 550025)
为筛选出适合兜兰属植物的DNA条形码序列。本研究基于GenBank中下载的29种兜兰属植物的253条序列,利用遗传距离法及分子系统学方法分析兜兰属植物候选条形码。结果表明,在选取的3个候选序列片段中对兜兰属植物鉴定的成功率最高的是核糖体ITS(nrITS)序列,分辨率为62.96%,其次是叶绿体matK序列,分辨率为50%,最后是叶绿体rbcL序列,分辨率仅为21.43%,因此,本研究推荐nrITS序列作为兜兰属植物的DNA条形码候选序列,叶绿体matK序列作为补充序列。
兜兰;DNA条形码;nrITS序列
兜兰是兰科(Orchidaceae)兜兰属(PaphiopedilumP)植物的统称,多生长于热带及亚热带林下,是兰科植物中最具特色的一个类群,也是最具特色魅力的观赏性花卉植物[1,2]。全世界兜兰属植物有96~100个种,主要分布在印度东部至我国南部、东南亚、菲律宾及马来西亚群岛地区[3],由于具有极高的观赏价值和经济价值,导致人们对兜兰野生资源进行了毁灭性的采挖,加之生境破坏等原因,野生兜兰的数量急剧减少,分布区逐渐萎缩,使得兜兰野生种均被列入《濒危野生动植物种国际贸易公约》[4]。近年来,人们为了保护兜兰种植资源,兜兰的杂交种也随之增加,加之相互引种,导致人们对兜兰属植物鉴定增加了一大难度[5]。
DNA条形码技术(DNA barcoding)是用DNA保守片段对物种进行快速准确鉴定的一种新兴技术[6],该技术由加拿大圭尔夫大学(University of Guelph)教授Hebert等[7]于2003年首次提出后并受到广泛的关注,现已成为生物分类和鉴定的研究热点,在物种鉴定方面显示了广阔的应用前景。目前国内外有关兜兰属植物DNA条形码的研究尚处于探索阶段,Parven等[8]通过对印度8种濒危兜兰植物的rpoB、rpoC1、rbcL、matK、和nrITS6个序列进行对比分析,结果表明matK基因对印度濒危兜兰植物的条形码研究具有较高的分辨率;Guo等[9]对金星兜兰(OrchidaceaeVenusSlipper)植物的DNA条形码研究表明,matK+ atpF-atpH能较好的区分该植物;Min等[10]等通过对韩国89种兰科植物的rbcL、matK、atpF-atpH IGS,psbK-psbI IGS和trnH-psbA 5个序列进行研究,结果表明trnH-psbA IGS 基因对韩国兰科植物的分辨率最高。本研究基于Genebank中的数据对兜兰属部分植物的nrITS序列、叶绿体matK序列及rbcL编码基因进行对比分析,旨在选出鉴别兜兰属植物的最适DNA条形码候选片段,为解决兜兰属植物的分子鉴定提供参考依据。
1 材料与方法
1.1 材料来源
从GenBank数据库中下载的27种兜兰属植物的117条核糖体ITS序列、22种兜兰属植物的78条叶绿体matK序列及14种兜兰属植物的58条叶绿体rbcL编码序列,其物种名称及GenBank登录号见表1。
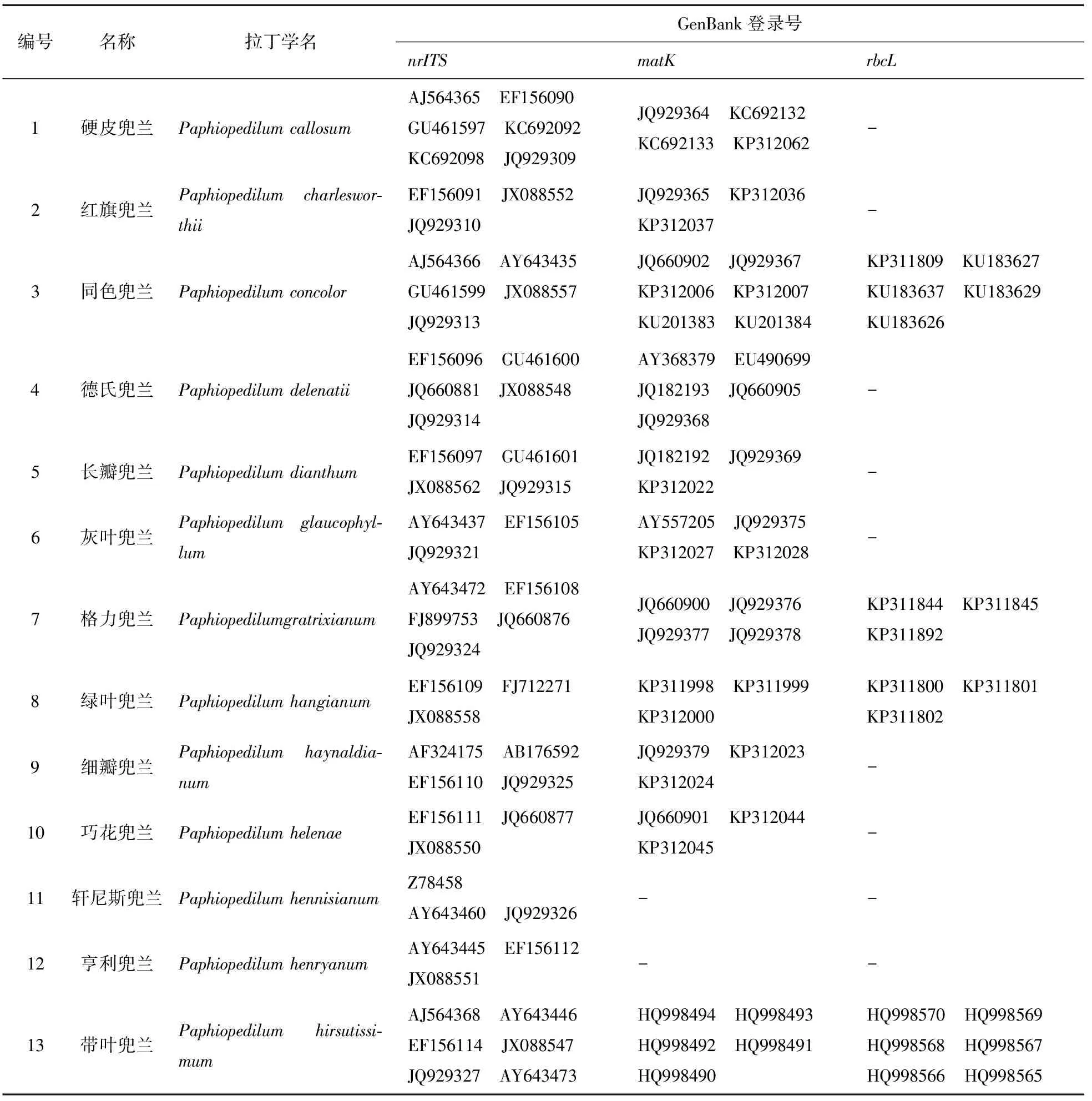
表1 不同物种及序列的GenBank登录号
续表1

编号名称拉丁学名GenBank登录号nrITSmatKrbcL14波瓣兜兰PaphiopediluminsigneAJ564369 AY643448JQ929329 AY643449JQ660898 JQ929381KP312049 KP312050HQ998574 HQ998573HQ998572 HQ99857115麻栗坡兜兰PaphiopedilummalipoenseEF156125 FJ712272JX088554 JQ929336HQ123427JQ929388 KF361653KP311996-16硬叶兜兰PaphiopedilummicranthumAY643432 EF156128FJ712270 JX088561JQ929338-HQ998561 HQ998562HQ998563 HQ99856417飘带兜兰PaphiopedilumparishiiEF156140 JQ660868JX088555 JQ929340JQ660891 JQ929392KP312026-18菲律宾兜兰PaphiopedilumphilippinenseAY643475 EF156141EF156142 JQ929341AJ564356JQ929393 KP312014KP312015HQ998557 HQ998558HQ99855919黄花兜兰PaphiopedilumprimulinumAY643438 EF156143JQ929342 JQ929343JQ929394 KP312030JQ929395-20白旗兜兰PaphiopedilumspicerianumAY643450 EF156145JX088545 JQ929347HQ998509 HQ998507HQ998506 HQ998505HQ998586 HQ998584HQ998583 HQ998582HQ998581 HQ99858021天伦兜兰Paphiopedilumtranlienia⁃numEF156151 FJ712267GU461602 JX088556-KP311810 KU183630KU183630 KU18363222越南兜兰PaphiopedilumvietnamenseAY643433 EF156158GU461603--23紫毛兜兰PaphiopedilumvillosumEF156159 FJ712268JQ660875 JX088563JQ929354JQ660899 KP312057JQ929405HQ998595 HQ998594HQ998593 HQ99859224彩云兜兰PaphiopedilumwardiiAY643469 EF156161JX088546 JQ929356--25秀丽兜兰PaphiopedilumvenustumHQ998471 HQ998472HQ998473 HQ998474HQ998475HQ998513 HQ998512HQ998511 HQ998510HQ998591 HQ998590HQ998589 HQ998588HQ99858726卷萼兜兰Paphiopedilumappletonia⁃numKC692113 KC692112KC692111 JQ929306JQ929362 KC692115KC692116 KC692140KP312059KP311833 KP311834KP311836 KP31183527杏黄兜兰PaphiopedilumarmeniacumJX088560 JX088560JX088560 AY643431EU490698 JQ660906KP311994-28虎斑兜兰Paphiopedilumtigrinum-KP312033 KP312054KP312055KP311835 KP311856KP31185729巨瓣兜兰Paphiopedilumbellatulum--KP311803 KP311804KU183624
1.2 数据分析
采用Clustal X软件对下载的序列进行比对[11],采用MEGA 7软件的K2P模型分别计算种内及种间遗传距离[12],并对序列信息进行分析,用office excel 2007软件对数据进行构建遗传距离间隔(barcoding gap)柱状图,并用MEGA 7软件采用邻接法(Neighbor Joining)对序列构建系统发育树。
2 结果与分析
2.1 候选序列特征及信息位点
通过MEGA 7软件对兜兰属植物的核糖体ITS、叶绿体matK及rbcL编码序列的信息分析结果见表2,可以看出,G+C含量比例最大的是核糖体ITS序列,rbcL编码序列次之,matK序列中G+C含量比例最小;从变异位点及信息位点来看,其大小依次为:nrITS>matK>rbcL;而碱基转换与颠换比最大是nrITS序列,最小的是matK序列。

表2 候选序列碱基组成及变异信息率
2.2 候选序列的Barcoding Gap评估
从不同候选序列种内与种间距离频率分布图可以看出(图1、图2、图3),三个候选序列都没有明显的Barcoding Gap。在nrITS序列片段中,种内遗传距离大部分集中在0.00~0.01区域,而种间遗传距离多集中于0.03以上区域,且种内与种间的遗传距离重叠区域不大,在matK序列片段中,种内遗传距离大部分集中在0.00~0.01区域,种间的遗传距离主要集中在0.02以下区域,而rbcL片段中,种内和种间的遗传距离都主要集中在0.01以内,几乎不存在Barcoding Gap,由此可见,nrITS序列符合DNA条形码候选序列要求。

图1 基于nrITS序列种内及种间的遗传距离频率分布
2.3 系统聚类分析
通过邻接法及k2-p模型对三个候选片段构建的聚类树图可以看出(图4、图5、图6),基于nrITS构建的聚类树能准确鉴定17种兜兰属植物,鉴定成功率高达62.96%。基于matK序列构建的聚类树中,能准确将11种兜兰属植物鉴定出来,鉴定成功率为50%。而基于14种兜兰属植物的rbcL基因构建的聚类结果显示,仅有卷萼兜兰(P.appletonianum)、带叶兜兰(P.hirsutissimum)和绿叶兜兰(P.hangianum)能准确鉴定,其分辨率仅为21.42%,由此可见,三个序列的鉴别能力为nrITS>matK>rbcL。
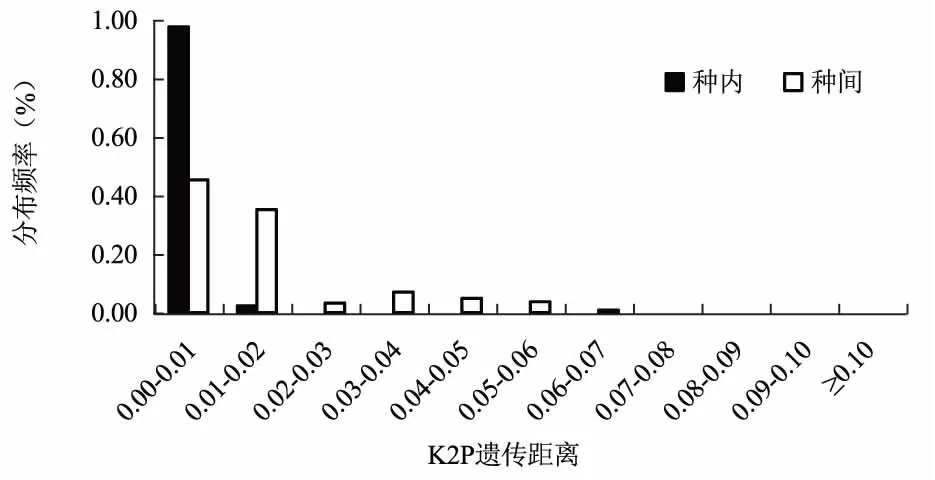
图2 基于matK序列种内及种间的遗传距离频率分布
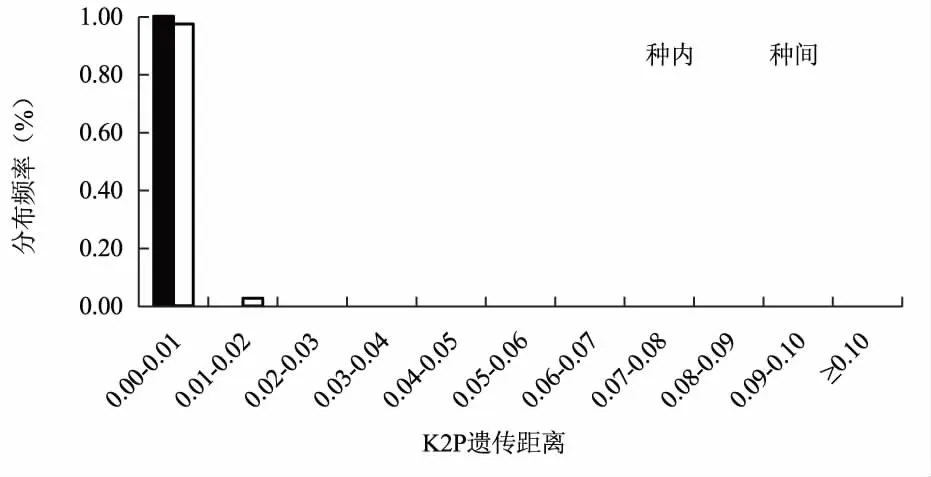
图3 基于rbcL序列种内及种间的遗传距离频率分布
3 结论与讨论
DNA条形码作为一种分子诊断技术现在已被普遍应用到物种鉴定[13,14],其结果客观性强且操作较简便,大大提高了非专业人员进行物种鉴定的效率和准确性,成为分类学家非常有力的工具[15]。理想的条形码基因片段应该满足以下几个特点:种间变异明显大于种内变异;一个或少数基因片段即可准确鉴定物种;重复性好;稳定性高;操作简便,可实现自动化;有统一的管理平台等[16,17]。本研究结果表明,在选择的3个候选序列中,nrITS序列对兜兰属植物的鉴定成功率最好,且满足理想的DNA条形码要求,故在候选序列中推荐nrITS序列为兜兰属植物首选的DNA条形码序列,这与Parven等[8]研究不一致,究其原因可能与Parven选取的兜兰物种数量不足有一定关系。

图4 基于nrITS序列构建的系统发育树
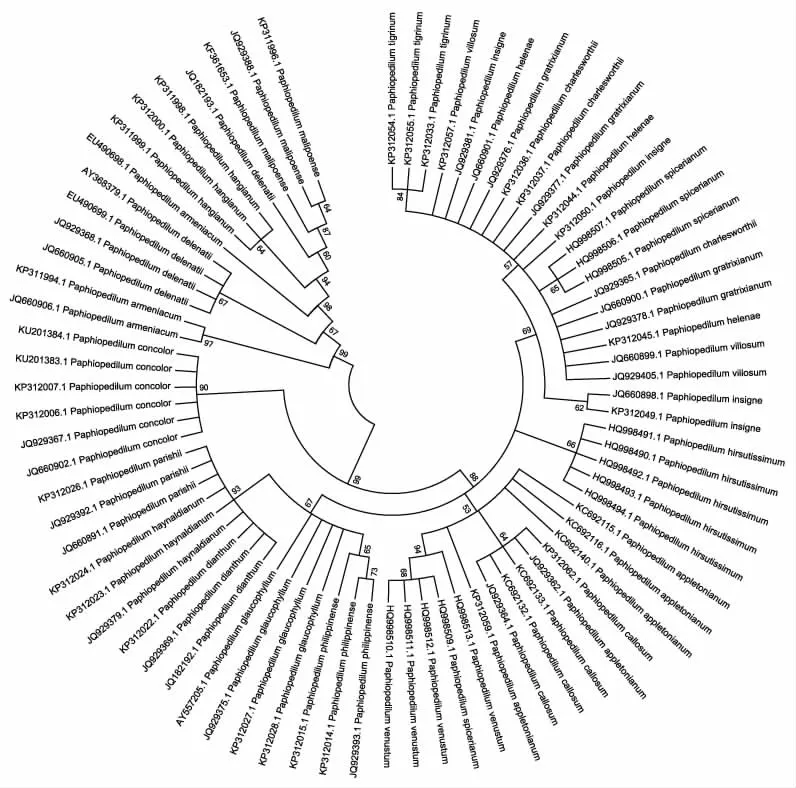
图5 基于matK序列构建的系统发育树
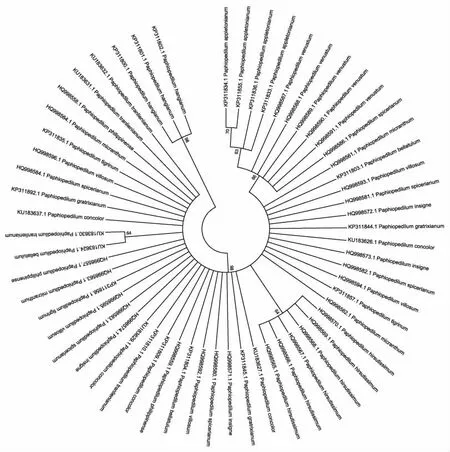
图6 基于rbcL序列构建的系统发育树
叶绿体matK保守性好,但其通用性差,且部分植物中对matK基因的扩增率极低[18,19]。据报道[20,21],matK序列因进化速率低等特点可以适用于属级水平DNA条形码研究,郑司浩等[22]对半夏属及其伪品进行鉴定研究表明,matK序列能较好的鉴别半夏及其伪品;生书晶等[23]利用matK序列成功鉴定出何首乌及其伪品,本研究结果表明,基于matK序列对兜兰属植物的鉴定率为61.5%,虽然matK序列在种内的变异最小,但种间的变异不大,且在DNA barcoding图中出现种内及种间的遗传距离频率分布重叠区域太大,综合其考虑,本研究推荐matK序列作为兜兰属植物的DNA条形码候选序列的补充序列。
rbcL序列被作为DNA条形码的候选序列以来,备受学者的关注,但Kress等[24]研究表明,在有花植物中,rbcL序列进化速率较慢,不适合作为种水平上的物种鉴定标记。本研究结果表明,rbcL序列在兜兰属植物中的种间及种内的遗传距离差异均不大,表明兜兰属植物中rbcL序列存在相对稳定,很难通过rbcL序列将其鉴别,由此可见,rbcL序列也不适用于兜兰属植物候选DNA条形码。
关于植物DNA条形码技术的研究一直处于探索之中,植物DNA条形码不像动物那样,COⅠ基因作为动物的DNA条形码序列已经得到了广泛的认可[25]。近年来,很多学者提出了以序列片段组合的方式作为候选片段对植物DNA条形码的研究[26],Xie等[27]对我国22个属74种有毒植物的DNA条形码研究结果表明,推荐用nrITS+matK+rbcL组合作为其DNA条形码的候选片段;Yang等[28]基于matK、rbcL及trnH-psbA 3个序列对菖蒲(Acoruscalamus)的DNA条形码研究,结果表明,matK+rbcL+trnH-psbA 3个片段组合对菖蒲的鉴定成功率最高; Saarela等[29]对490种维管束植物DNA条形码的研究结果表明,rbcL序列与matK序列组合研究对维管束植物的鉴定效果最好,由于本研究中所采用的网上数据每条序列对应的种类数和样本数均不相同,不是平行数据,所以未将nrITS、matK及rbcL序列的数据联合起来进行分析,许多研究结果表明[30-31],nrITS、matK及rbcL等序列的组合对兰科植物的鉴定成功率较高,因此预计这一组合在兜兰属植物的鉴定中将会发挥较好的作用。
[1] Braemg,Chiron G. Paphiopedilum[M]. Paris:Tropicalia,1988.
[2] Guo YY,Huang LQ,Liu ZJ,etal.Promise and Challenge of DNA Barcoding inVenus Slipper (Paphiopedilum)[J].PlosOne,2016,11(1):1-13.
[3] Zeng S,Huang W,Wu K,etal.In vitro propagation of Paphiopedilum orchids[J].CriticalReviewsinBiotechnology,2016,36(3):521-534.
[4] Luo Y,Jia JS,Wang CL. A general review of the conservation status of Chinese orchids [J].ChineseBiodiversity,2003,11(1):70-77.
[5] 曾宋君,田瑞雪,陈之林,等.兜兰属植物杂交育种研究进展[J].热带亚热带植物学报,2010,18(2):459-468.
[6] 李永青,焦 婷,吴建平,等.禾本科8种牧草DNA条形码通用序列筛选[J].草业科学,2016,33(9):1702-1710.
[7] Hebert PDN,Ratnasingham S,Waard JR.Barcoding animal life: cytochrome coxidase subunit 1 divergences among closely related species[J].ProcBiolSci,2003(270):96-99.
[8] Parveen I,Singh HK,Raghuvanshi S,etal.DNAbarcoding of endangered IndianPaphiopedilumspecies[J].MolecularEcologyResources,2011,12(1):82-90.
[9] Guo YY,Huang LQ,Liu ZJ,etal.Promise and Challenge of DNA Barcoding inVenus Slipper (Paphiopedilum)[J].PlosOne,2016,11(1):1-13.
[10] Min KH,Sang-Hun O,Shankar BG,etal.DNA barcoding of Orchidaceae in Korea[J].MolecularEcologyResources,2014,14(3):499-507.
[11] Tamura K,Dudley J,Nei M,etal.MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]MolecularBiologyandEvolution,2007,24(8):1596-1599.
[12] Thompson JD,Gibson TJ,Plewniak F,etal. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J].NucleicAcidsResearch,1997,25(24):4876-82.
[13] TaberletP,CoissacE,Pompanon F,etal.Power and limitations of the chloroplasttrnL (UAA) intron for plant DNA barcoding [J].NucleicAcidsRes,2007,35(3):e14.
[14] Kress WJ,Erickson DL.A two-locus global DNAbarcode for land plants: the codingrbcLgene complementsthe non-codingtrnH-psbAspacer region[J].PlosOne2007,2(6):e508.
[15] 王 柯,陈科力,刘 震,等.锦葵科植物DNA条形码通用序列的筛选[J]. 植物学报,2011,46(3):276-284.
[16] Slabbinck B,Dawyndt P,Martens M,etal.TaxonGap: a visualization tool for intra-and inter-species variation amongindividual biomarkers[J].Bioinformatics,2008,24(6):1464-75.
[17] Chase MW,Salamin N,Wilkinson M,etal.Land plants and DNAbarcodes: short-term and long-term goals[J].PhilosTransRSocLondBiolSci,2005,360(1462):1889-95.
[18] Fazekas AJ,Burgess KS,Kesanakurti PR,etal.Multiple multilocus DNA barcodes fromthe plastid genome discriminate plant species equally well[J].PLoSOne2007,3(7):e2802.
[19] Yu J,Xue JH,Zhou SL. New universalmatKprimers for DNA barcoding angiosperms[J].JournalofSystematicsandEvolution,2011,49(3):176-181.
[20] 任保青,陈之端.植物DNA条形码技术[J].植物学报,2010,45(1):1-12.
[21] Tallei TE,Kolondam B J.DNA Barcoding of Sangihe Nutmeg ( Myristica fragrans ) usingmatKGene[J]HayatiJournalofBiosciences,2015,22(1):1-7.
[22] 郑司浩,魏文龙,任伟光,等. 基于DNA条形码的半夏属及其伪品分子鉴别[J].植物学报,2014,49(6):710-719.
[23] 生书晶,严 萍,郑传进,等. 何首乌及其常见混淆品的matK基因序列分析及鉴别[J].中药材,2010,33(11):1707-1711.
[24] Kress WJ,Wurdack KJ,Zimmer EA,etal.Use of DNA barcodes to identify flowering plants[J].ProceedingsoftheNationalAcademyofScience,2005,102(23),8369-8374.
[25] IftikharR,Ashfaq M,Rasool A,etal.DNA Barcode Analysis of Thrips(Thysanoptera) Diversity in Pakistan RevealsCryptic Species Complexes[J].PlosOne,2016,11(1),e0146014.
[26] Li DZ,Liu JQ,Chen ZD.Plant DNA barcoding in China[J].JournalofSystematicsandEvolution,2011(49):165-168.
[27] Xie L,Wang YW,Guan SY,etal.Prospects and Problems for Identification of PoisonousPlants in China using DNA Barcodes[J].BiomedEnvironSci,2014,27(10):794-806.
[28] Yang HQ,Dong YR,Gu ZJ,etal.A Preliminary Assessment ofmatK,rbcLandtrnH-psbA as DNA Barcodes for Calamus (Arecaceae) Species in China with a Note on ITS[J].AnnalesBotaniciFennici,2012,49(5-6):319-330.
[29] Saarela J M,Sokoloff P C,Gillespie L J,etal.DNA barcoding the Canadian Arctic flora: core plastid barcodes (rbcL+matK) for 490 vascular plant species[J].PlosOne,2013,8(10):e77982-e77982.
[30] 黄明忠. 基于matK基因和ITS序列的海南野生兰科植物DNA条形码探讨[D].海口:海南大学,2010.
[31] Min KH,Sang-Hun O,Shankar B G,etal.DNA barcoding of Orchidaceae in Korea[J].MolecularEcologyResources,2014,14(3):499-507.
Study on the DNA Barcode Sequence ofPaphiopedilumBased on Data from GenBank
YANGPeng,GUANPing,CHENXing-yin,ZHANGKai-kai,PENGChang-qing,CHENGYu,SHIJian-ming*
(CollegeoflifeSciences,GuizhouUniversity,Guiyang,Guizhou550025China)
In order to screen the DNA barcode sequence of Paphiopedilum [Asparagales: Orchidaceae], we downloaded 253 sequences of 29 species of Paphiopedilum from GenBank. The candidate barcodes of Paphiopedilum were analysed by genetic distance method and molecular phylogenetic method. The results showed that the highest success rate is by the nrITS sequence with a resolution of 62.96%, followed by the matK sequence with a resolution of 50%, and lastly by the rbcL sequence with a resolution of only 21.43%. Therefore, we recommend the nrITS sequence be used as Paphiopedilum DNA barcoding sequence, and the matK sequence as a supplement sequence.
Paphiopedilum;DNA barcode;nrITSsequence
2017-03-03;
2017-05-15
贵州省自然科学基金资助项目([2013]2124)。
Q949.71+8.43
A
1008-0457(2017)04-0021-06 国际
10.15958/j.cnki.sdnyswxb.2017.04.004
*通讯作者:石建明(1962-),男,博士,副教授,主要研究方向:植物学及植物生物技术研究;E-mail:guanp508@163.com。

