舟山海域梭子蟹科重要种类DNA条形码建立及其系统发育研究
王秀亮,沈 璐,韩志强
(浙江海洋大学水产学院,浙江舟山 316022)
舟山海域梭子蟹科重要种类DNA条形码建立及其系统发育研究
王秀亮,沈 璐,韩志强
(浙江海洋大学水产学院,浙江舟山 316022)
舟山海域梭子蟹科蟹类资源丰富,种类较多,具有及高的经济价值。然而,传统分类学中对其分类主要采用形态观察和行为学的技术手段,表型变异受环境因子影响显著,导致表型性状的不确定性。本研究采用蟹类DNA条形码通用的引物来扩增舟山海域梭子蟹科6种常见蟹类的COI基因序列并构建系统发育树,结果显示,所有分析蟹类COI基因长度为658 bp。种内变异较小,遗传距离范围0.000 0~0.008 2,平均0.002 9;种间变异较大,遗传距离介于0.140 0~0.241 7,平均0.195 1。NJ系统发育树支持蟳属、梭子蟹属和圆趾蟹属具为三个独立的系群,而与圆趾蟹属的亲缘关系较为疏远,这为梭子蟹科的物种鉴定和系统发育重建研究等方面提供重要参考。
DNA条形码;梭子蟹科;系统发育;COI基因
Key words:DNA barcode;Portunidae;molecular phylogeny;COI gene
梭子蟹科Portunidae隶属节肢动物门Arthropoda、甲壳纲Crustacea、十足目Decapoda,又名蝤蛑科,是短尾次目Brachyura梭子蟹总科Portunoidea的其中一科[1]。梭子蟹科的蟹类均生活于海洋中,主要栖息于热带、亚热带潮间带及大陆架浅海区(极少数栖息于深海),少数种类也出现在河口半咸水域。梭子蟹类骨骼轻、眼大足细,螯足细长且相当强壮,末对步足呈浆状,完全适合游泳运动。它们依靠迅速游泳的能力,可以捕获运动迅速的猎物,也可依此避开较大的猎食者。绝大部分的梭子种类为杂食性,只有一部分蟹类特化为肉食性,少部分的蟹是植食性或腐食性的。
梭子蟹科约35个属共计300余种,我国大约有60种。其中,三疣梭子蟹Portunus trituberculatus、日本蟳Charybdisjaponica、双斑蟳Charybdis bimaculata、细点圆趾蟹Ovalipes punctatus、锈斑蟳Charybdis feriatus、红星梭子蟹Portunus sanguinolentus,肉质鲜美、营养丰富具有较高的经济价值,是我国重要的海洋捕捞蟹,在海洋渔业中占有重要的经济地位[2]。虽然梭子蟹科的经济意义重大,但是其系统发育及进化起源研究仍然没有太大发展,并且很多种类至今没有分清[3]。准确的物种鉴定和了解其系统发育关系对蟹类资源的可持续利用至关重要[4]。
近年来,分子标记手段为蟹类系统发育提供了一个新的方法[5]。目前用于蟹类系统发育研究的分子标记方法有同工酶(allozyme)、随机扩增多态性DNA(RAPD)、限制性内切酶片段长度多态性(RFLP)和DNA序列分析等[6]。DNA序列分析是目前使用得越来越多的一类分子标记,它的准确性受到众多科研工作者的青睐,在甲壳动物的物种鉴定及遗传结构的研究中,这一标记已被广泛采用。
蟹类的分子分类研究运用的线粒体序列主要为12S rRNA,16S rRNA,COI等基因片段。COI基因较12S rRNA,16S rRNA等基因变异高,突变率约为1.4%/Ma[7],更适于对亲缘关系较近的物种的研究和群体间的遗传变异研究。线粒体COI基因目前一般作为DAN条形码(DNA Barcode)用于区分种间差异,进行物种鉴定[8],也常联合其他线粒体基因共同分析[9-12],并且在蟹类的科属分类阶元下进行系统发育分析具有较好的潜力。本研究通过对梭子蟹科舟山海域常见的6种经济蟹类的COI基因片段序列进行扩增,并构建系统发育树来分析梭子蟹科蟹类种内种间的变异程度和亲缘关系,为梭子蟹科的物种鉴定和系统发育提供理论依据。
1 材料与方法
1.1 实验采样
于2011年9月至2016年3月,在舟山近海海域采集梭子蟹科中蟳属、梭子蟹属和圆趾蟹属共6个种的33个样本。采样种类经形态鉴定分别为蟳属:日本蟳、双斑蟳、锈斑蟳;梭子蟹属:红星梭子蟹、三疣梭子蟹;圆趾蟹属:细点圆趾蟹。将新鲜样品带回实验室,取肌肉保存于无水酒精中。每种采样信息和分析样品数见表1。

表1 梭子蟹科采样信息,包括样本量、采样地点、采样时间Tab.1 The sampling information for Portunidae species(sample size,sample location and collection time)
1.2基因组DNA提取与PCR扩增
按照酚-氯仿方法用蛋白酶K消化肌肉从中提取DNA基因组。
COI基因用无脊椎动物通用引物L1490和H2198进行扩增。引物序列如下:
L1490:(5'-GGTCAACAAATCATAAAGATATTGG-3')(正向);
H2198:(5'-TAAACTTCAGGGTGACCAAAAAATCA-3')(反向)
PCR 反应体积为 25 μL (50 mmol/L KCl,10 mmol/L Tris·HCl,pH 为 8.3,MgCl21.5mmol/L,各种dNTP200 μmol/L,Ex Taq 酶 1.25 units,每个引物各 0.2 mmol/L,模板 DNA1 μL,加灭菌蒸馏水至 25 μL。PCR反应用扩增仪(Eppendorf Mastercycler 5333)进行。反应的条件是:94℃预变性3 min,接着进行40个循环,每循环包括94℃ 45 s,50℃ 45 s,72℃1 min,最后在72℃下延伸10 min。以上反应都使用阴性对照检查有否DNA污染。取1.5 μL扩增产物通过1.5%琼脂糖凝胶电泳检测。产物送上海桑尼公司进行双向测序。
1.3 数据分析
所得序列经SeqMan软件(DNAstar软件包)处理,去除引物并结合人工校对,确定每一序列位点准确无误。使用MEGA6.0软件对6种蟹类的COI基因片段碱基组成进行分析。遗传距离的计算和NJ系统发育树的构建也采用MEGA软件。利用Kimura两参数模型(Kimura-2-parameter,K2P)[13]计算样品间两两遗传距离(pairwise distances),并且计算出碱基差异数。采用邻接距离法(Neighbor-Joining)构建系统发育树,进行1 000次自检,计算各分支的支持率。
2 结果
2.1 COI基因碱基组成
对采自浙江舟山海域的梭子蟹科的6种重要种类的线粒体COI基因片段进行PCR扩增和序列测定。对其比对后,得到658 bp的同源序列,其碱基组成(表2)。A、T、G、C的平均含量分别为36.8%、20.0%、25.7%和17.5%,A+T的平均含量为62.6%。同时由表可知,虽然6种蟹类的A、T、G、C组成上存在差异,但A+T的含量均明显高于G+C,具有AT偏向性。
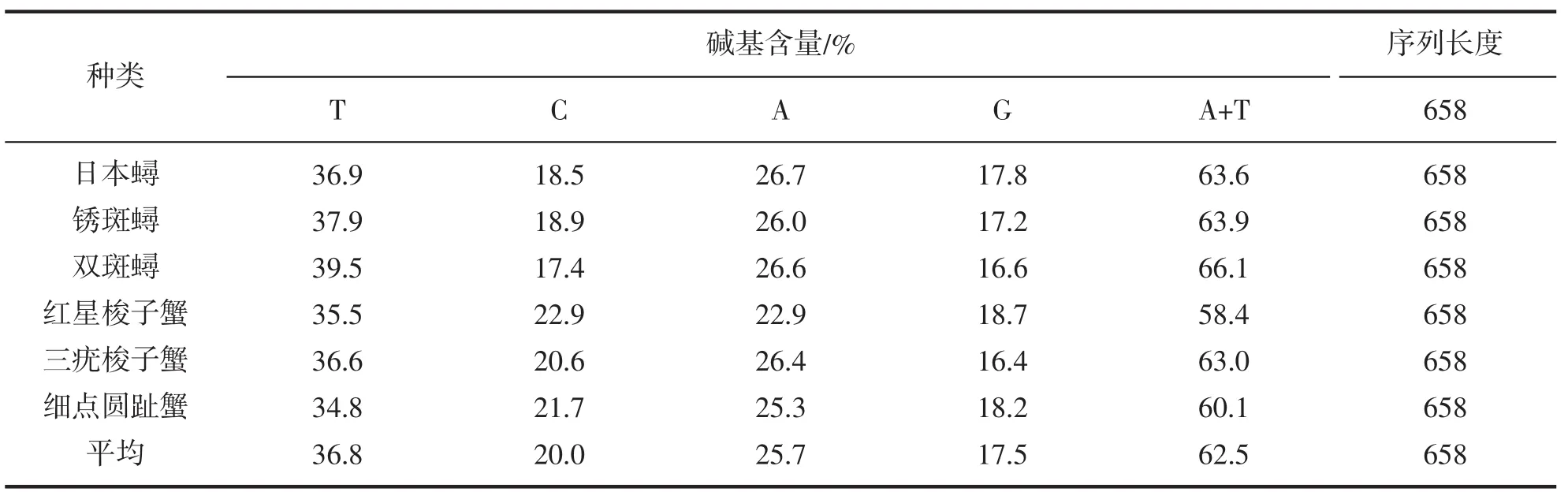
表2 梭子蟹科的6种重要种类的COI基因片段碱基组成Tab.2 The base composition for COI gene in six Portunidae species
2.2 种内的COI基因变异
6种蟹类种内COI基因变异度(表3),有表可知,6种蟹类种内变异较小,种内遗传距离范围0.000 0~0.008 2,平均值为0.002 9;种内个体间碱基平均差异数范围0.000 0~5.333 3,平均值为1.866 7。日本蟳种内6个个体遗传距离最小为0.000 0,6个个体共享一个单倍型;细点圆趾蟹种内遗传距离最大为0.002 9,在细点圆趾蟹中检测到3个单倍型,个体间平均碱基差异数为1.866 7。

表3 梭子蟹科的6种重要种类的种内COI基因变异Tab.3 The intraspecific COI gene variations in six Portunidae species
2.3 种间的遗传差异及系统发育
2.3.1 遗传差异
6种蟹类COI的遗传距离(表4),由表4可知,6种蟹类的种间遗传距离为0.140 0~0.241 7,其中,红星梭子蟹和细点圆趾蟹的遗传距离最大,为0.241 7,双斑蟳与日本蟳的遗传距离最小,为0.140 0。6种蟹类的种间平均碱基差异数为83.00~133.70,其中,红星梭子蟹和细点圆趾蟹的碱基差异最大,为133.70,双斑蟳与日本蟳的碱基差异最小,为83.00。这表明遗传距离和碱基差异数显示出来的遗传差异具有一致性。

表4 梭子蟹科的6种重要种类的遗传距离(下对角线)和碱基差异数(上对角线)Tab.4 The genetic distance(below the diagonal)and base number of differences(above the diagonal)between species in Portunidae
2.3.2 系统发育
6梭子蟹科种类的分子系统发育树如图1,线长表示的是分歧度,线上的数值表示的经过1 000次重复抽样所获得的支持率。系统发育树显示这6种蟹类分为三支,日本蟳、锈斑蟳和双斑蟳首先聚为一支为蟳属,红星梭子蟹和三疣梭子蟹聚为一支,是梭子蟹属,细点圆趾蟹单独聚为一支,是圆趾蟹属。根据系统树表明蟳属、梭子蟹属和圆趾蟹属3属间具有较相近的亲缘关系。
3 讨论

图1 梭子蟹科的6种重要种类的NJ系统发育树Fig.1 The neighbor-joining tree for Portunidae species
本实验结果显示,所有分析蟹类COI基因长度为658 bp,无插入缺失。HEBERT等[14]推荐的生物条形码种间遗传距离阈值为0.02,多数物种间的遗传距离远大于该值。在本研究中6种蟹类种内平均遗传距离0.002 9;种间平均遗传距离0.195 1。种间和种内的遗传距离相差约65倍,说明COI基因用于物种间鉴定时,具有很好的种间间断性,符合HEBER有关的种间阈值范围,但细点圆趾蟹种内遗传距离较大,种内个体间平均遗传距离为0.008 2,说明在该种内COI基因进化较快。此外,COI基因在5种蟹类种内多样性较低,6个日本蟳个体共享一个单倍型,双斑蟳5个个体中只检测到2个单倍型,表明COI基因在群体水平上遗传变异较小,种内较稳定。
DNA条形码的应用以标准基因的确立为基础,自HERBERT[14]提出以COI基因作为物种的条形码,越来越多的研究对该基因在不同生物类群中应用的有效性进行了验证,证明COI基因在动物条形码应用中的优势。而COI基因的这种优势之一就在于COI通用引物的广泛适用性。本研究采用无脊椎动物的通用引物对舟山海域的6种梭子蟹科的重要种类进行扩增,均顺利获得相应序列,表明该通用引物在蟹类物种中具有较普遍的适用性。采用COI基因对6种蟹类系统发育研究表明,6种蟹类按照属分别进行聚类,支持形态学的分类结果,蟳属、梭子蟹属和圆趾蟹属互为单系群。本研究结果同金珊等[15]基于RAPD标记构建的6种梭子蟹科种类系统发育树基本一致。
通过基于线粒体COI基因的DNA条形码技术在蟹类物种的鉴定识别中区分度高。目前多数学者认为,基于COI基因的生物条形码可以较好实现物种鉴定工作,可望成为物种鉴定的重要工具[16]。但DNA条形码存在一定的“错误标签”,对于杂交种类和存在种间渐渗的种类,难以完全取代形态学分类方法在分类学上的地位。所以应用DNA条形码鉴定物种仍需结合形态学或辅以其他基因进行验证。
[1]BOWMAN TE,ABELE LG.Classification of recent Crustacea[M].New York:Academic Press,1982:1-92.
[2]杨思谅.中国动物志无脊椎动物甲壳动物亚门十足目梭子蟹科[J].北京:科学出版社,2012:1-50.
[3]HARRISON MK,CRESPI BJ.Phylogenetics of cancer crabs(Crustacea;Decapoda;Brach-yura)[J].Molecular Phylogenetics and Evolution,1999,12(2):186-199.
[4]高天翔,王玉江,刘进贤,等.基于线粒体12S rRNA序列探讨4种青蟹系统发育关系及中国沿海青蟹的分类地位[J].中国海洋大学学报:自然科学版,2007,37(1):57-60.
[5]IGUCHI A,ITO H,UENO M,et al.Molecular phylogeny of the deep-sea Buccinum species(Gastropoda:Buccinidae)around Japan:Inter-and intraspecific relationships inferred frommitochondrial 16S rRNA Sequences[J].Molecular Phylogenetics and Evolution,2007,44(3):1 342-1 345.
[6]廖秀珍,林荣澄.多毛类18S rDNA和COI基因序列片段及其分子系统发育研究[J].台湾海峡,2006,25(4):490-497.
[7]KNOWLTON N,WEIGT L A.New dates and new rates for divergence across the Isthmus of Panama[J].Proceeding of the Royal Society London Series B,1998,265:2 257-2 263.
[8]孔晓瑜,喻子牛,刘亚军,等.中华绒螫蟹与日本绒螯蟹线粒体COI基因片段的序列比较研究[J].青岛海洋大学学报,2001,31(6):861-866.
[9]SCHUBART C D,DIESEL R,HEDGES S B.Rapid evolution to terrestrial life in Jamaican crabs[J].Nature,1998,393:363-365.
[10]SCHUBART C D,NEIGEL J E,FELDER D L.Molecular phylogeny of mud crabs(Braehyura:Panopeidae)from the northwestern Atlantic and the role of morphological stasis and convergence[J].Marine Biology,2000,137:11-18.
[11]SHIH H T,NG P K L,SCHUBART C D,et al.Phylogeny and phylogeography of the genus Geothelphusa(Crustacea:Decapoda,Brachyura,Potamidae)in southwestern Taiwan based on two mitochondrial genes)[J].Zoological Science,2007,24(1):57-66.
[12]TANG B P,ZHOU K Y,SONG D X,et al.Molecular systematics of the Asian mitten crabs,genus Eriocheir(Crustacea:Brachyura)[J].Molecular Phylogenetics and Evolution,2003,29(2):309-316.
[13]Kimura M.A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16:111-120.
[14]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B Biological Sciences,2003,270:313-321.
[15]金 珊,赵青松,王春琳,等.梭子蟹科六种海产蟹的RAPD标记[J].动物学研究,2004,25(2):172-176.
[16]王 鹤,林 琳,柳淑芳,等.中国近海习见头足类DNA条形码及其分子系统进化[J].中国水产科学,2011,18(2):245-255.
DNA Barcode and Molecular Phylogeny of the Important Species in Family Portunidae
WANG Xiu-liang,SHEN Lu,HAN Zhi-qiang
(Fishery School of Zhejiang Ocean University,Zhoushan 316022,China)
The family Portunidae contains many economic crab species in Zhoushan sea area.However,the traditional taxonomy methods mainly used the morphology and behavioral characters.The phenotypic variation was significantly affected by environmental factors,resulting in some uncertainty.In this study,the mtDNA cytochrome oxidase subunit I(COI)gene was amplified from six economic crab species to construct the phylogenetic tree for family Portunidae.The results showed that the fragment of COI gene amplified in this study was 658 bp.The intraspecific genetic distances within species were small,ranging from 0.000 0 to 0.008 2 with an average of 0.002 9;the interspecific genetic distances between species were large,ranging from 0.140 0 to 0.247 0 with an average of 0.195 1.Based on the NJ tree,the separation of three genus Portunus,Charybdis and Ovalipes have been supported.The present study provided an important reference for the reconstruction of the system and the crab species identification.
Q959.223+.63
:A
2016-12-02
国家自然科学基金(32170527)
王秀亮(1990-),男,山东临沂人,硕士研究生,研究方向:种群遗传学.
韩志强,教授.E-mail:d6339124@163.com
1008-830X(2017)01-0014-05

