基于SSR和ISSR标记对江苏大竹蛏野生群体和人工增殖群体的联合分析
曹 奕,陈爱华,吴杨平,张 雨,姚国兴
(江苏省海洋水产研究所,江苏省海洋经济贝类研究开发中心,南通 226007)
基于SSR和ISSR标记对江苏大竹蛏野生群体和人工增殖群体的联合分析
曹 奕,陈爱华,吴杨平,张 雨,姚国兴
(江苏省海洋水产研究所,江苏省海洋经济贝类研究开发中心,南通 226007)
为探索人工增殖活动对本地大竹蛏(Solen grandis)种质资源带来的影响,采用SSR及ISSR分子标记手段对江苏大竹蛏野生群体与人工增殖群体的遗传多样性及遗传结构进行了联合分析。筛选的14对SSR引物扩增获得多态性位点150个(PPB=93.16%),Nei′s基因多样性指数和平均Shannon信息指数分别为0.134 2和0.253 5;筛选的10条ISSR引物获得多态性位点85个(PPB=91.93%),Nei′s基因多样性指数和平均Shannon信息指数分别为0.131 0和0.230 2。两种方法分析得到野生群体的遗传多样性参数均显著高于人工增殖群体,但两群体间并未产生明显的遗传分化。实验结果表明,江苏沿海大竹蛏群体的遗传多样性水平较高,人工增殖群体的遗传多样性虽有所下降,但并未导致该地区大竹蛏种质资源遗传结构的显著改变。因此人工增养殖活动仍然能够作为补充大竹蛏资源的主要手段,但应探索采用高效生态的增养殖模式,对大竹蛏的种质资源进行合理的开发与利用。
大竹蛏;SSR;ISSR;增殖群体;遗传多样性
大竹蛏(Solen grandis)隶属瓣鳃纲,帘蛤目,竹蛏科,竹蛏属,在我国各海域均有分布。其个大壳薄,肉味鲜美,是我国重要的经济贝类[1]。但由于自然资源量较少,而且主要通过采捕方式获得,所以产量较低。开展大竹蛏人工增殖,既可以对野生资源进行保护,也可以提高产量,带动贝类产业发展[2]。江苏省文蛤良种场自2007年来持续进行人工增殖活动,主要地点为江苏如东蒋家沙海域,使得大竹蛏资源量得到了部分恢复[3]。但增殖活动是否会改变江苏海域大竹蛏的遗传多样性和遗传结构,从而对生长、抗病、抗逆等生长性状造成影响,是一个需要关注的问题。
简单重复序列(simple sequence repeats,SSR)和简单重复序列区间(inter-simple sequence reapeats,ISSR)标记技术都是目前广泛应用于辅助遗传育种的分子标记技术[4-6],均具有多态性丰富、重复性好、稳定性高、操作简单等优点[7-8]。本研究采用这两种分子标记技术,对江苏大竹蛏野生群体与增殖群体的遗传多样性进行分析,揭示增殖群体的遗传分化状况,以期评估出大竹蛏进行良种选育工作的可行性,也为保护原有种质资源提供理论数据。
1 材料与方法
1.1 样品采集
2015年8月,野外采样获得江苏如东蒋家沙增殖群体(JJ)、吕四野生群体(LS)大竹蛏(表1)。从两个群体中各选取25 ind平均长度约10 cm、质量约35 g的三龄健康个体,取其斧足用于实验。

表1 大竹蛏样品采集地点、时间、数量Tab.1 Sampling coordinate,time,and number for different S.grandis populations
1.2 DNA提取
采用酚-氯仿法进行提取。紫外分光光度计测定样品DNA质量,并进行琼脂糖电泳验证,提取所得的DNA-20℃保存。
1.3 引物序列
筛选得到大竹蛏SSR引物14对、ISSR引物10条[9-10],由上海生工生物工程公司合成,引物序列及PCR反应条件见表2。
1.4 SSR-PCR及ISSR-PCR反应体系及程序
SSR-PCR反应体系为25μL,包括1x PCR Buffer,Mg2+浓度1.5 mM,0.2 mmol·L-1dNTPs,1.0μmol·L-1引物,Taq酶1.0 U,模板约100 ng。程序为:首先进行94℃变性5 min;然后94℃变性40 s,Tm复性30 s,72℃延伸1 min,反应进行35个循环,最后72℃延伸10 min。扩增产物用ABI3730进行毛细管电泳,确定个体的等位基因大小。
ISSR-PCR反应体系含50 ng·μL-1模板DNA 1.0μL,10×PCR buffer(含Mg2+)2.5μL,10 mmol·L-1dNTP 1.0μL,10μmol·L-1引物1 μL,5 U·μL-1Taq酶0.2μL,加ddH2O至25 μL;反应条件如下:反应前94℃预变性4 min,94℃变性45 s,38~55℃退火45 s,72℃延伸60 s,共35个循环,反应后72℃延伸7min。产物进行2%琼脂糖电泳电泳,染色后用Bio-Rad凝胶成像系统拍照。对所得电泳图进行分析。
1.5 数据分析
获得各微卫星位点上等位基因的条带大小后,将所得结果进行归类;ISSR电泳图谱通过Quantity One软件进行分析。采用Popgene 32软件进行SSR和ISSR检测所得结果遗传参数分析[11-12],并根据各群体遗传多样性参数计算各群体间的遗传分化系数、遗传距离。基于遗传距离,用Mega 5.0软件构建所有50个样品的UPGMA聚类树。

表2 筛选获得引物序列Tab.2 Nucleotide sequences of selected primer
2 结果与分析
2.1 野生群体与增殖群体的遗传多样性
14对SSR引物在野生群体和增殖群体中分别检测到多态性位点143、138个,总群体等位基因数为6~18个,共检测到161个等位基因。各位点的多态信息含量PIC介于0.611~0.712之间,均表现出高度的多态性(表3)。表明野生群体和增殖群体均具有相对丰富的遗传多样性。
10条ISSR引物从野生群体及增殖群体中分别扩增得到多态性位点83、76个,共检测得到93个等位基因。H值和I值分别为0.1311和0.2410(表4),两种分子方法分析得到蒋家沙增殖群体的遗传多样性参数均低于吕四野生群体,但差异不显著。
2.2 野生群体与增殖群体的遗传变异
LS群体的大竹蛏样品平均等位基因数、等位基因丰富度、观察杂合度、期望杂合度均略高于JJ群体,分别为10.2、10.13、0.809、0.815。LS群体近交系数0.007>0,而JJ增殖群体的近交系数则为0.008,两者间无显著差异(表5)。
2.3 遗传分化状况
分化系数可表示遗传分化状况。分化系数在0~0.05之间,群体间分化很弱;0.05~0.15之间表示中等分化;0.15~0.25之间表示分化大;大于0.25表示分化极大[13]。
14个位点在大竹蛏群体内的遗传分化系数Fst为0.029~0.046,表明大竹蛏增殖群体与野生群体在这些位点上并无明显的遗传分化。各群体位点组合均符合Hardy-Weinberg平衡。
而通过10条ISSR引物分析得到JJ群体与LS群体的遗传分化系数Gst<0.05(表6),验证了SSR分析得到的结果。
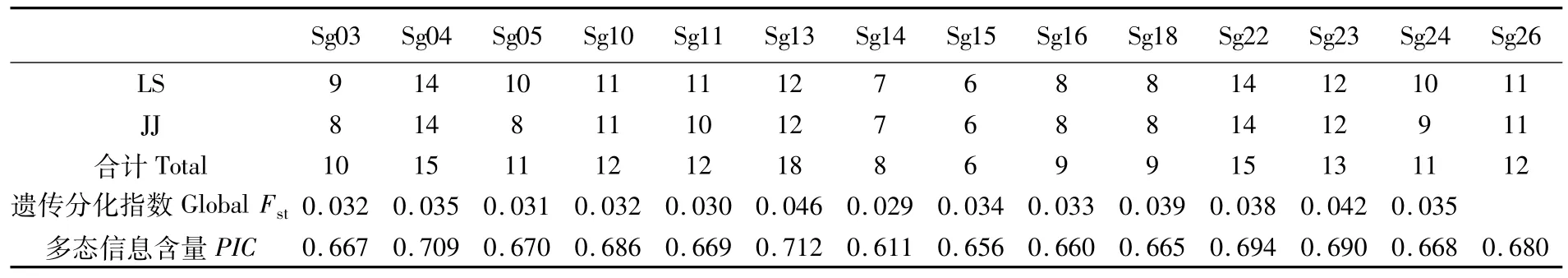
表3 14个微卫星位点在大竹蛏野生群体及增殖群体中扩增得到等位基因数、遗传分化指数、多态信息含量Tab.3 Allelenumber,GlobalFst,PICfrom14microsatelliteloci withinwildandstockpopulationofSolengrandis
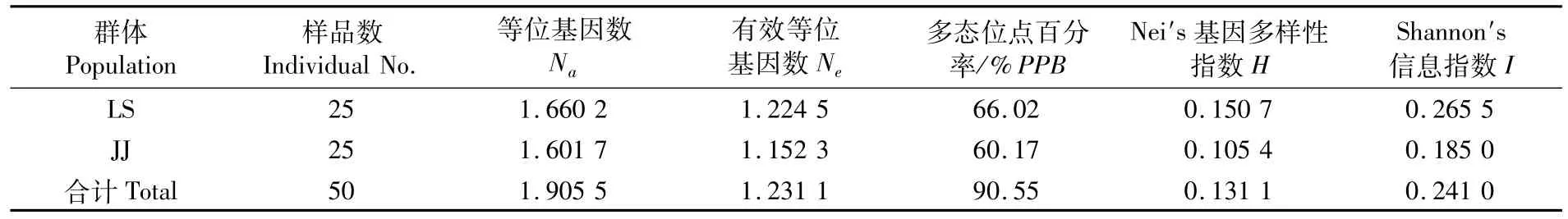
表4 ISSR分析得到的大竹蛏野生群体与增殖群体遗传多样性参数Tab.4 GeneticdiversityparametersofthewildandstockS.grandispopulationsbyISSR

表5 野生与增殖大竹蛏群体在14个微卫星位点的遗传变异情况Tab.5 Geneticvariationat14microsatellitelociforthewildandstockSolengrandispopulations

表6 大竹蛏野生群体与增殖群体间遗传距离和遗传分化系数Tab.6 Genetic distance and genetic diversity coefficient of the 6 S.grandis populations
2.4 聚类分析
基于SSR得到的UPGMA聚类分析结果显示(图1),JJ4、JJ5、JJ9、JJ20号个体与大部分LS群体个体聚为一支,LS13、LS14、LS19、LS21号个体与大部分JJ个体聚为一支。而ISSR的结果则显示(图2),JJ3、JJ14、JJ17号个体与大部分LS群体个体聚为一支,LS4、LS9、LS20号个体聚在一起后,与大部分JJ个体聚为一支。
本研究的聚类结果显示,SSR与ISSR分析所得结果基本一致,而野生群体与增殖群体中存在个体与对方群体中的个体聚为一支,证明了相互之间存在着一定的亲缘关系。
3 讨论
3.1 大竹蛏群体的遗传多样性
多态信息含量PIC>0.5属于高度多态性位点,0.25<PIC<0.5为中度多态位点,PIC<0.25为低度多态位点[14]。而本研究所得的微卫星位点的PIC值均在0.5以上,说明野生群体和增殖群体均具有较高的遗传多样性水平。而两种方法分析得到JJ群体Nei′s基因多样性、Shannon′s信息指数和多态位点百分率均低于LS群体,但差异并不显著。该结果说明:增殖群体仍然保持着较高的遗传多样性水平,多样性较野生群体下降程度较缓,其下降原因主要可能是人工育苗进行增殖时,亲本数量和品质带来的影响。该情况可以通过育苗过程中增加亲本数量等措施加以改善。
3.2 大竹蛏群体的遗传分化状况
Hardy-Weinberg平衡检测结果表明,供试大竹蛏各群体位点组合均处于Hardy-Weinberg平衡状态,等位基因分布频率相对稳定,观测杂合度和期望杂合度之间没有显著差异,说明大竹蛏野生群体及增殖群体尚未受到选择、突变等外界自然环境因素影响。
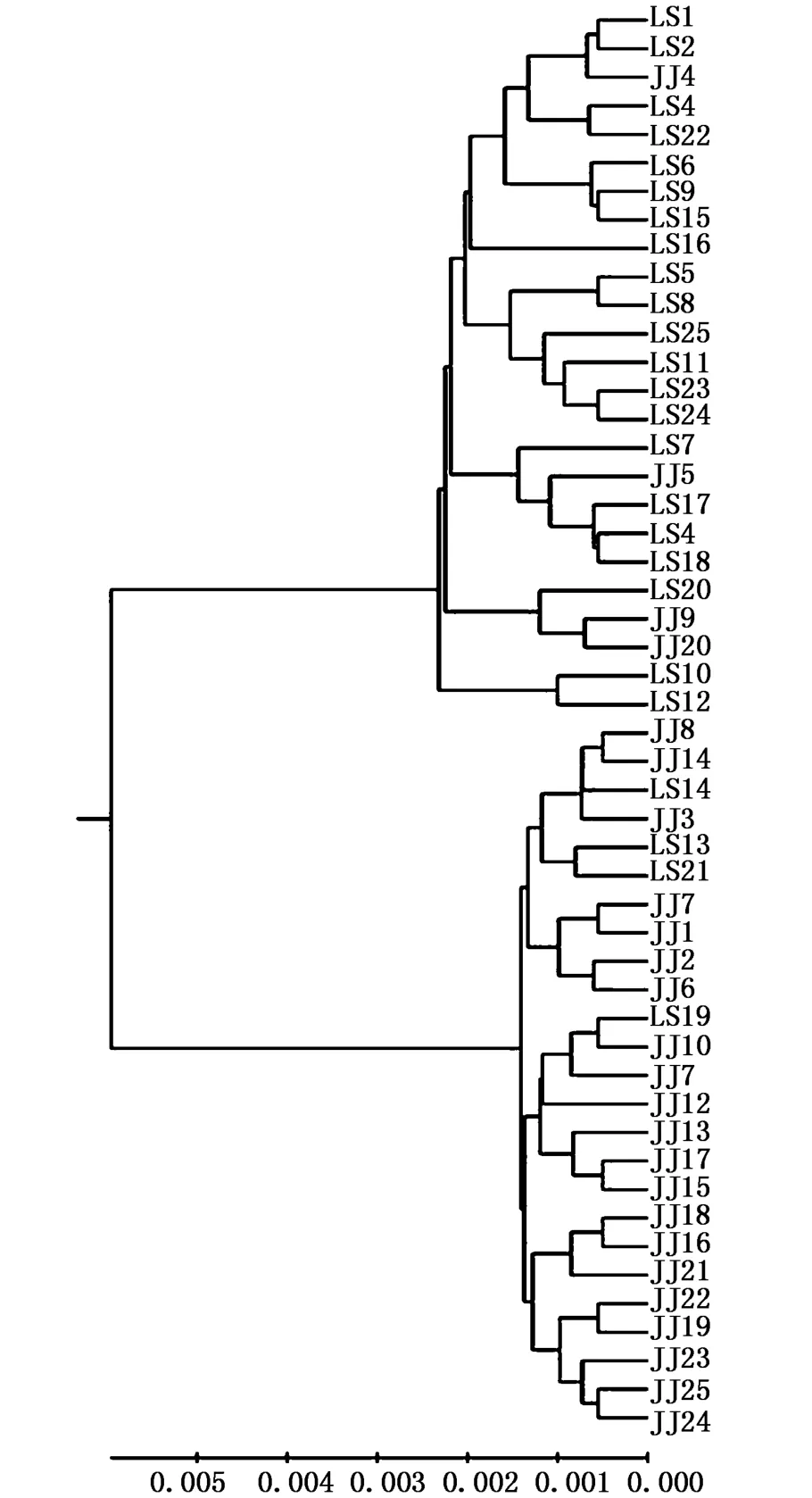
图1 基于微卫星标记的大竹蛏野生群体及增殖群体个体的UPGMA聚类图Fig.1 UPGMA dendrogram of individuals from the w ild and stock populations of S.grandis by SSR

图2 基于ISSR分子标记的大竹蛏野生群体及增殖群体个体的UPGMA聚类图Fig.2 UPGM A dendrogram of individuals from the w ild and stock population of S.grandis by ISSR
JJ群体与LS群体的Fst及Gst均小于0.05,表明增殖群体与野生群体并没有产生明显的遗传分化。而二者的近交系数分别为0.007、0.008,并未出现显著差异。说明数年来的增殖活动没有使增殖群体相对野生群体产生明显的遗传变异,大竹蛏进行良种选育工作是可行的。
3.3 遗传多样性分析对大竹蛏增殖活动带来的启示
江苏南通地区野生群体与增殖群体大竹蛏的遗传多样性和遗传分化状况分析结果表明,该地大竹蛏增殖群体在保持着高遗传多样性水平的同时,遗传多样性略有下降,但未产生明显的遗传变异。因此继续开展大竹蛏人工育苗,进行增殖活动是十分必要且可行的,同时也可以探索大竹蛏的养殖技术与模式,为大竹蛏资源的恢复利用扩展渠道。但必须持续对增殖群体进行相关的监测工作,同时严格把关人工育苗的各个环节,防止出现种质退化情况。
[1] 戴聪杰.大竹蛏软体部分营养成分分析及其评价[J].集美大学学报(自然科学版),2002,7(4):304-308.DAIC J.Evaluation on nutrition components of the soft part in Solen gradis[J].Journal of Jimei University(Natural Science),2002,7(4):304-308.
[2] 闫喜武,赵生旭,张 澎,等.培育密度及饵料种类对大竹蛏幼虫生长、存活及变态的影响[J].大连水产学院学报,2010,25(5):386-390.YAN XW,ZHAO SX,ZHANG P,et al.Effects of stocking density and alga species on larval growth,survival and metamorphosis in bamboo clam Solen grandis[J].Journal of Dalian Ocean University,2010,25(5):386-390.
[3] 陈爱华,姚国兴,张志伟,等.温度、盐度和底质对大竹蛏稚贝生长及存活的影响[J].热带海洋学报,2010,29(5):94-97.CHEN A H,YAO G X,ZHANG Z W,et al.Effects of temperature,salinity and sediment on the growth and survival of Solen grandis Dunker juveniles[J].Journal of Tropical Oceanography,2010,29(5):94-97.
[4] 李 超,侯吉伦,王桂兴,等.基于牙鲆RNA-seq数据中SSR标记的信息分析[J].海洋渔业,2015,37(2):122-127.LIC,HOU J L,WANG G X,et al.Bioinformatic analysis of SSR markers in transcriptomic sequenceing Paralichthys olivaceus[J].Marine Fisheries,2015,37(2):122-127.
[5] 牛东红,李家乐,冯冰冰,等.缢蛏6个群体遗传结构的ISSR分析[J].应用与环境生物学报,2009,15(3):332-336.NIU D H,LIJL,FENG B B,et al.ISSR analysis on genetic structure of six Sinonovacula constricta populations[J].Chinese Journal of Applied andEnvironmental Biology,2009,15(3):332-336.
[6] 张 滔.大竹蛏群体遗传多样性研究[D].山东:鲁东大学.2012.ZHANG T.Study on the genetic diversity of Solen gradis populations[D].Shangdong:Ludong University,2012.
[7] HOU L,LV H L,ZOU X Y,et al.Genetic characterizations of Mactra veneriformis(Bivalve)along the Chinese coast using ISSR-PCR markers[J].Aquaculture,2006,261(3):865-871.
[8] 乔洪金,刘相全,孙国华,等.大竹蛏(Solen grandis)cDNA文库中微卫星标记的筛选[J].海洋与湖沼,2012,43(6):1128-1133.QIAO H J,LIU X Q,SUN G H,et al.Isolation and characterization of microsatellite markers from cDNA library of Solen grandis[J].Oceanologia Et Limnologia Sinica,2012,43(6):1128-1133.
[9] YUAN Y,LIQ,KONG L F,et al.Isolation and characterization of polymorphic microsatellite loci in the grand jackknife clam Solen grandis(Bivalvia:Veneroida)[J].Genes and Genomics,2010,32(3):191-197.
[10] YUAN Y,LIQ,KONG L F,et al.The complete mitochondrial genome of the grand jackknife clam,Solen grandis(Bivalvia:Solenidae):A novel gene order and unusual non-coding region[J].Molecular Biology Reports,2012,39(2):1287-1292.
[11] NEI M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89(3):583-590.
[12] SLATKIN M,BARTON N H.A comparison of three indirectmethods for estimating average levels of gene flow[J].Evolution,1989,43(7):1349-1368.
[13] WRIGHT S.Evolution and the genetics of population variability within and among natural populations[M].Chicago:University of Chicago Press,1978:4.
[14] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
Joint analysis of w ild and proliferative Solen grandis populations from Jiangsu based on SSR and ISSR analysis
CAO Yi,CHEN Ai-hua,WU Yang-ping,ZHANG Yu,YAO Guo-xing
(Jiangsu Marine Economic Shellfish Research and Development Center,Jiangsu Marine Fisheries Research Institute,Nantong 226007,China)
To explore the impact of artificial proliferative activities to the local Solen grandis germplasm resources,a joint analysis including genetic diversity and genetic structure of wild and artificial proliferative Solen grandis populations from Jiangsu based on SSR and ISSR markers were carried out.150 polymorphic lociwere amplified from 14 pairs of SSR primers(PPB=93.16%).Nei′s genetic diversity index(He)and average Shannon information index(I)were 0.134 2 and 0.253 5,respectively.85 polymorphic loci were amplified from 10 ISSR primers(PPB=91.93%),and Nei′s genetic diversity index and average Shannon information index were 0.131 0 and 0.230 2,respectively.Genetic diversity parameters of the wild populations obtained by the two methods were significantly higher than those of the artificial proliferative population(P<0.05),but genetic differentiation was not significant between two populations(P>0.05).The results showed that a high genetic diversity of Solen grandis populations from the coastal area of Jiangsu,though the genetic diversity of the artificial proliferative population decreasing,were not leading to a significant change of the genetic structure of Solen grandis germplasm resources in this region.Therefore,vigorous artificial breeding activities could still serve as the primary measure for supplementing Solen grandis resources.But ecological aquaculture modes should be developed for conducting rational development and utilization for Solen grandis germplasm resources.
Solen grandis;SSR;ISSR;proliferative population;genetic diversity
S 917
A
1004-2490(2017)03-0249-07
2016-02-16
江苏省农业科技自主创新资金项目〔CX(14)2083号〕;南通市农村科技创新及产业化项目(HL2014018号);江苏省属公益类科研院所能力提升项目(BM2015017-2号)
曹 奕,研究实习员,主要从事分子生物学研究。E-mail:caoyi19881123@126.com
陈爱华,研究员。E-mail:chenah540540@aliyun.com

