SCoT与EST-SSR标记检测甘草属(GlycyrrhizaL.)植物遗传多样性的比较研究
宋凤,陆嘉惠,*,韩春,牛清东,陈超南,李学禹
(1石河子大学生命科学学院/新疆生产建设兵团绿洲生态农业重点实验室,新疆 石河子832003;2石河子大学甘草研究所,新疆 石河子832003)
SCoT与EST-SSR标记检测甘草属(GlycyrrhizaL.)植物遗传多样性的比较研究
宋凤1,陆嘉惠1,2*,韩春1,牛清东1,陈超南1,李学禹2
(1石河子大学生命科学学院/新疆生产建设兵团绿洲生态农业重点实验室,新疆 石河子832003;2石河子大学甘草研究所,新疆 石河子832003)
为探讨SCoT、EST-SSR标记技术在甘草属植物遗传多样性研究的适用性,采用SCoT、EST-SSR分子标记对甘草属8自然居群的4种1变种共计14个个体进行基因组DNA的多态性检测。实验筛选出的18对SCoT通用引物共扩增出313条带,其中312条为多态性条带,平均多态率(PPB)为99.68%,平均有效等位基因数(Ne)和位点品均期望杂合度(He)等遗传参数与EST-SSR标记无显著差异。多态性检测效率高的标记为SCoT,E=17.333,Ai=21.145,高于EST-SSR,E=6.909,Ai=8.719。SCoT与EST-SSR标记聚类分析结果趋势相似,均将14份材料划分为3组,与基于形态学分类结果一致。2种标记相比,EST-SSR(0.820)产生的平均相似系数范围大于SCoT(0.785),EST-SSR标记个体间遗传差异检测能力略高于SCoT。结果表明SCoT和EST-SSR两种标记均可揭示甘草属种间与种内的遗传多样性水平以及亲缘关系,是进行甘草属植物自然群体的遗传结构、种间基因渐渗等研究的有效分子标记。其中,SCoT标记多态性高,信息量大,更适于甘草属植物种质资源的遗传多样性分析;个体间遗传差异检测上EST-SSR标记效果更佳,更利于疑难种鉴定及亲本分析。
甘草属;SCoT;EST-SSR;遗传多样性
豆科(Leguminosae)甘草属(GlycyrrhizaL.)植物在全世界分布有21种[1],其中乌拉尔甘草(Glycyrrhiza uralensisL.)、光果甘草(G.glabraL.)、胀果甘草(G.inflataBat.)作为药用甘草植物收录于《中国药典(第一部)》[2]。然而野生环境下甘草属植物群体间存在自然杂交现象,种内、种间遗传变异复杂[3-6],给甘草属植物野外物种识别、鉴定及种质资源的利用和保护带来很大的困扰,因此采用合理的分析方法研究野生甘草属植物遗传多样性、种质资源是非常重要的。
目标起始密码子多态性(Start codon targeted polymorphism,SCoT)标记是 Collard 和 Mackill(2009)在水稻中提出的一种基于单引物扩增反应(single primer amplification reaction,SPAR)的新型分子标记方法,这种标记能获得与性状联系紧密的目的基因,并能对性状进行跟踪,具有操作简单、多态性高、遗传信息丰富、成本低廉、引物通用性强等优点,其中以多态性检测效率高,遗传信息量丰富最为突出[7-10]。目前已广泛应用于植物种质资源遗传多样性、群体遗传结构、种质鉴定、基因差异表达和分子遗传图谱等研究中,如鸭茅[11]、桃[12]、鹰嘴豆[13]、牛鞭草[14]、穿心莲[15]和甜椒[16]等,但在甘草属植物中的应用还未见报道。表达序列标签的微卫星标记(expressed sequence tags-simple sequence repeats,EST-SSR)自开发以来,广泛应用于植物遗传图谱构建、遗传多样性、性状关联分析等研究[17-19]。目前EST-SSR标记在甘草属植物种间亲缘关系和遗传多样性研究显示,该标记具有较好的适用性 。
在对野生植物种质资源的研究中,研究目的对种质材料的信息要求不同,如物种亲缘关系分析中,样本的组分、样本间遗传距离和相似系数十分重要;以遗传多样性评估和群体结构分析为目的,则要求大量的遗传信息;若以育种为目的则需要更多与性状相关的信息。针对不同的研究目的,选择适宜的标记显得十分重要。
因此,本实验在前人开发的EST-SSR标记的基础上,结合SCoT标记对甘草属8个自然居群的4个种1个变种共计14个个体,进行遗传多样性分析,通过比较两种分子标记的相关遗传参数及聚类结果,探讨2种标记在甘草属植物遗传多样性研究的适用性,进一步为甘草属植自然群体的遗传结构及种间杂交基因渐渗等方向的研究提供新方法,避免单一标记方法对研究结果造成偏差,更为全面合理的评估甘草属的遗传多样性水平,更好的为我国甘草野生种质资源保护及可持续开发提供理论基础。
1 材料与方法
1.1 样品
供试甘草于2015年10月采集自8个自然居群,共14个个体。材料及凭证标本存放于石河子大学甘草标本室。材料详情见表1。每株采集其绿色叶片,使用变色硅胶室温干燥后,置于4℃冰箱保存备用。
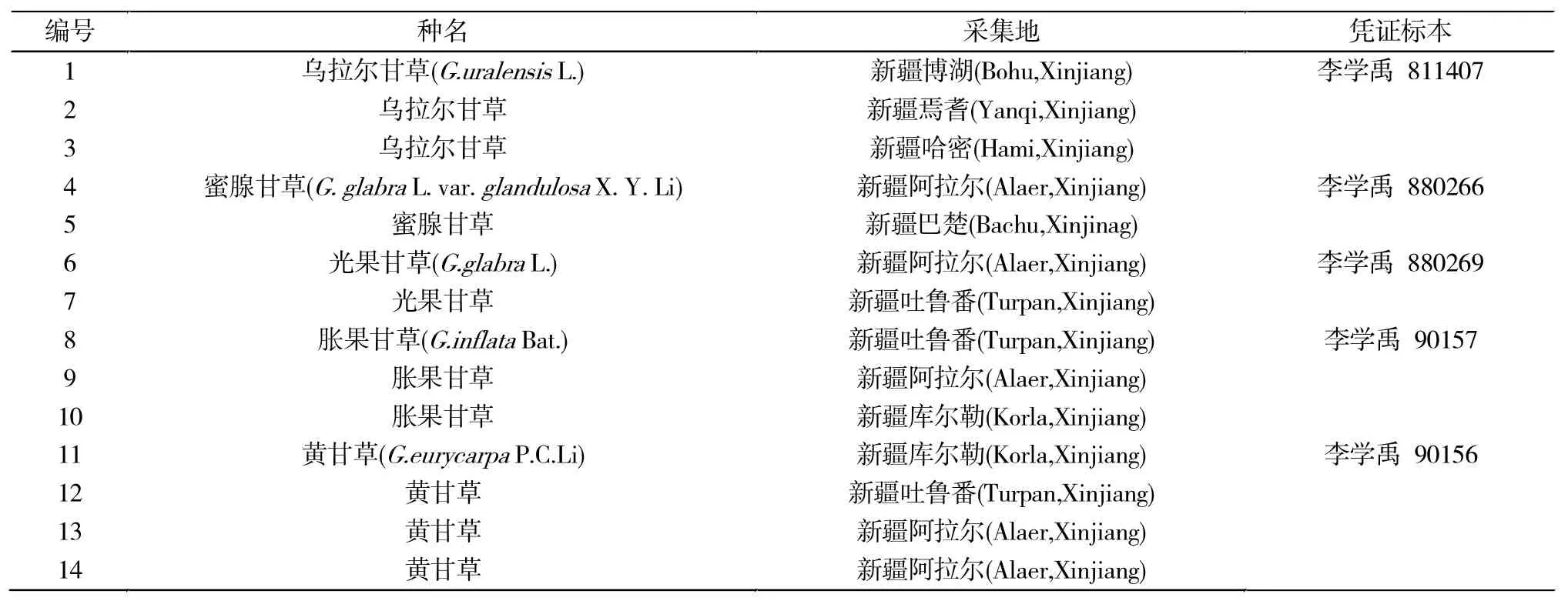
表1 实验材料及其采集地Tab.1 Experimental materials and their collecting localities
1.2 仪器与试剂
ProFlexTMPCR System PCR热循环仪(Applied Biosystems),MultifugeX1R 高速冷冻离心机(Thermo Fisher Scientific),Gel DocTMEZ凝胶成像系统(BIORAD),JY600C型电泳槽(北京君意东方电泳设备有限公司),Thermo Scientific Nano-Drop 2000 Spectrophotometers分光光度计(Thermo Fisher Scientific)。
EST-SSR、SCoT引物(上海生工生物工程有限公司)、D2000DNAMarker(北京天根)、植物全基因组DNA提取试剂盒(北京天根)、2×Taq PCR MasterMix(北京天根),溴化乙锭(EB)核酸染料(北京索莱宝)、琼脂糖(Biowest)、其余为国产分析纯试剂。
1.3 总DNA提取及检测
使用天根植物全基因组DNA提取试剂盒提取甘草总DNA。1%琼脂糖凝胶电泳检测DNA质量,并用紫外分光光度计测定其浓度和纯度,并稀释至25 ng/μL,置于-20℃冰箱保存备用。
1.4 SCoT-PCR扩增及检测
SCoT引物采用Collard和Mackill开发的引物[7],由上海生工生物工程有限公司合成,共计36条。选取5种甘草 DNA模板进行引物筛选,最终确定18条扩增产物条带清晰、重复性高、稳定性好、具有差异的引物用于后续实验。具体引物信息见表2。
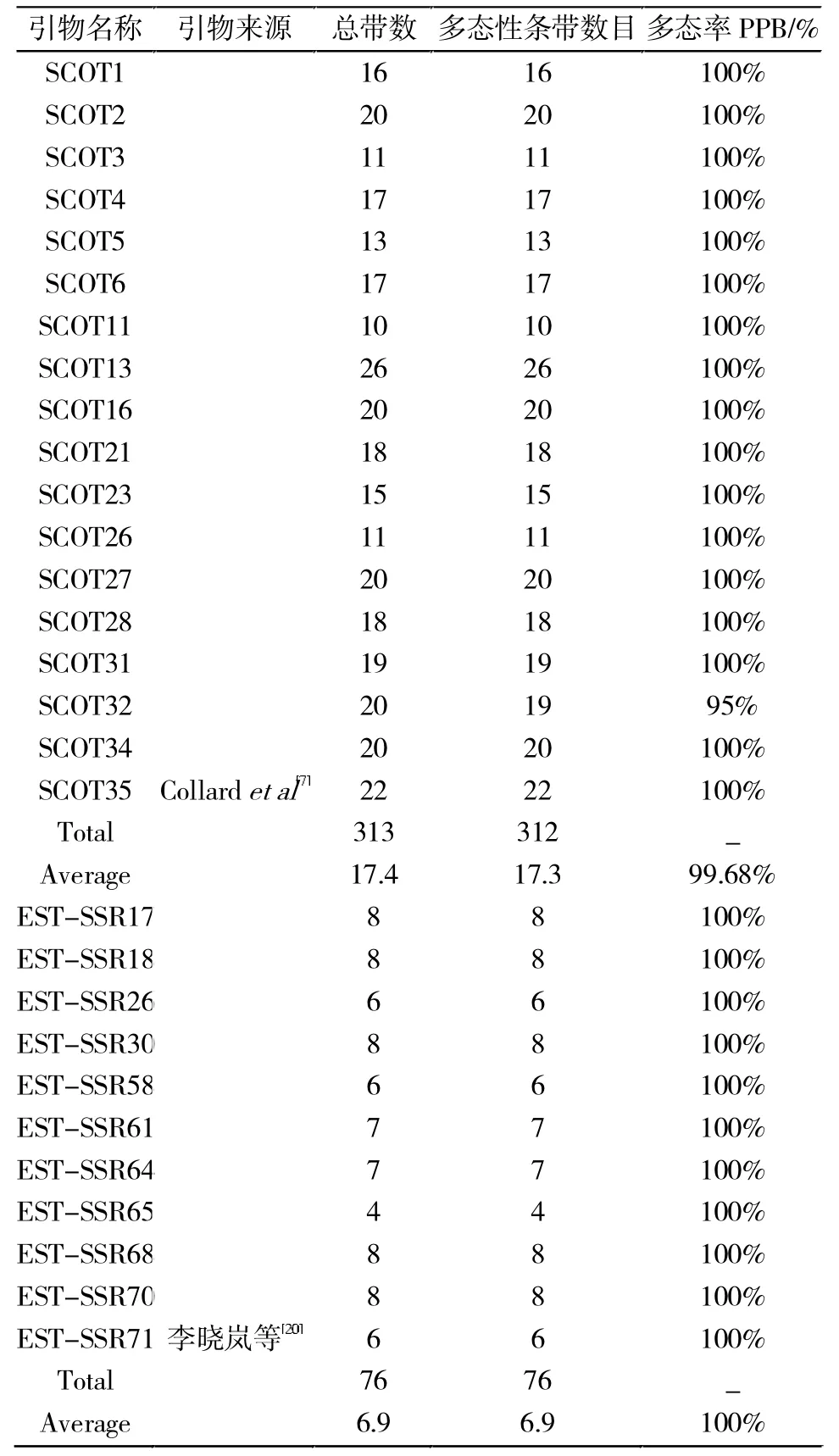
表2 EST-SSR、SCoT引物信息Tab.2 Information of EST-SSR、ScoT primers
SCoT-PCR 反应体系:总体积 20 μL,内含2×Taq PCR Master Mix 10 μL 引物(10 μmol/L)0.8 μL,模板 DNA(25 ng/μL)1.0 μL,ddH2O 补至 20 μL。扩增程序为94℃预变性3 min,然后进行36个循环:94℃变性 50 s,52℃退火 1 min,72 ℃延伸2 min,循环结束后72℃延伸5 min,4℃保存。PCR结束后各取产物5 μL,在3.0%的琼脂糖凝胶上电泳检测(1×TBE缓冲液,电压100V,1 h)。电泳结束后在凝胶成像系统中观察并拍照保存。
1.5 EST-SSR PCR扩增及检测
EST-SSR引物采用李晓岚[20]设计引物(表2),分析使用 PCR反应总体积为20体积 20 μL,内含2×TaqPCRMasterMix10 μL正 反 向 引 物(10 μmol/L)0.8 μL,模板 DNA (25 ng/μL)1.0 μL,ddH2O补至20 μL。反应程序94℃预变性4 min,94 ℃变性30 s,55 ℃退火 30 s,72 ℃延伸 45 s,35个循环结束后72℃延伸5 min,4℃保存。PCR结束后各取产物5 μL,在5.0%的琼脂糖凝胶上电泳检测(1×TBE缓冲液,电压100 V,1 h)。电泳结束后在凝胶成像系统中观察并拍照保存。
1.6 数据处理
用软件Image Lab(BIO-RAD)分析胶片,统计每个处理扩增得到的DNA条带数,经人工校正后以D2000 DNA Marker作为参照标准分子量,对扩增条带进行分子量预测,将分子量在±3 bp范围内视为同一位置,后根据每个样品的扩增片段大小和位点进行统计,同一位点出现的为“1”,同一位置无条带或不易分辨的弱带计为“0”,建立原始“0,1”矩阵。采用GenAlEx6.502软件计算参数包括多态位点百分率(PPB)、平均有效等位基因数(Ne)、Shannon’s多样性信息指数(I)和位点品均期望杂合度(He),ANOVA方差分析比较两种标记位点多态性揭示效果。依据Belaj[22]的方法计算标记指数(MI)和有效复合比(E)样本间遗传相似系数分析使用NTSYS-pc2.1,并按非加权算数平均数聚类(UPGMA)方法构建聚类图。
2 结果与分析
2.1 EST-SSR与SCoT多态性分析
筛选出的18条SCoT引物共扩增313条清晰条带,312条多态性条带,平均多态率为99.68%,扩增片段长度变化范围在2000 bp到500 bp之间。11对EST-SSR引物多态性检测共得到76条清晰条带,其中76条多态性条带,扩增多态性比率为100%,扩增片段长度在100-250 bp以内。2种标记在5种甘草样本间均能产生有效的多态性条带,多态位点百分率(PPB)值十分接近。
2.2 遗传信息比较
利用GenAlEx6.502软件计算了基于两种分子标分析样本所得相关遗传参数,结果见表3。
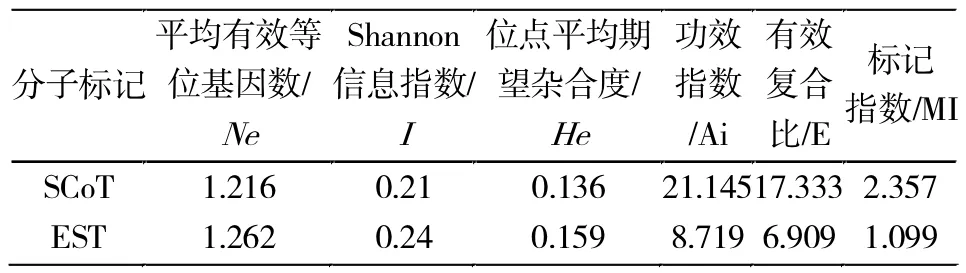
表3 SCoT和EST-SSR标记遗传多样性分析比较Tab.3 Comparison of genetic diversity based on SCoT and EST-SSR
EST-SSR和SCoT标记检测出的平均有效等位基因数(Ne)分别为 1.262和 1.216、Shannon’s多样性信息指数(I)分别为0.240和0.210,所揭示的位点平均期望杂合度(He)分别为0.136和0.159。其中Ne和He的ANOVA方差分析(表4)显示 2种标记无显著差异(PNe=0.1579,>0.05;PHe=0.2632,>0.05)。这表明2种标记揭示位点多态性的效果相似,即评估物种遗传多样性水平的能力没有显著差异(表4)。

表4 Ne和He的ANOVA方差分析Tab.4 ANOVA analysis ofNeandHe
2.3 标记效率分析
有效复合比(E)和功效指数(Ai)能够评价标记多态位点产生效率和位点多态性效率,2种标记中,ScoT的 E和 Ai(17.333,21.145) 均高于 EST-SSR(6.909,8.719),相比之下SCoT标记具有更高的多态性检测效率。标记指数(MI)能够综合的反映位点平均的预期杂合度和有效复合比,SCoT标记为2.357,高于 EST-SSR 的 1.099。
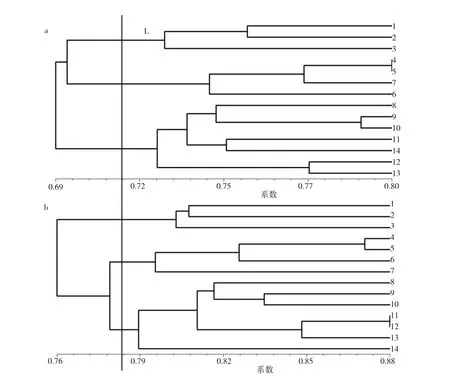
图1 基于SCoT(a)和EST-SSR(b)标记的5种甘草属植物UPGMA聚类图Fig.1 UPGMA dendrogram of fiveGlycyrrhizaspecies based on SCoT(a)and EST-SSR(b)markers
2.4 聚类分析比较
按非加权算数平均数聚类(UPGMA)方法构建聚类图,结果显示2种标记聚类趋势大致相同,结合线为L时,14份材料均被划分为 3组(图1a,b):乌拉尔甘草组(1~3),组内相似系数范围分别为;光果、蜜腺甘草组(4~7);胀、黄甘草组(8~14)。不同物种分支,说明基于SCoT、EST-SSR分子标记的五种甘草属植物遗传分化结果与基于形态学分类结果是一致的,两种标记均可用来分析甘草属植物亲缘关系分析。此外,基于SCoT标记建树(图1a)的相似系数范围为0.690-0.800,平均相似系数0.785;基于EST-SSR标记建树(图1b)的相似系数范围为0.760-0.880,平均相似系数0.820。2种标记相比,EST-SSR产生的平均相似系数范围最大,因此该标记比SCoT可检测到更高的个体间遗传差异。
3 讨论
甘草属植物存在自然杂交现象[3-6]野生环境下的杂交种及种内不同变异型的存在使得根据形态学的分类鉴定变得复杂。有的野生甘草的荚果形态是介于2种或几种甘草间的过度类型,或偏向于某一种,如黄甘草类群,物种分类鉴定较为困难,因此通过分子标记手段对物种进行分类鉴定是十分必要的。
本实验首次将SCoT分子标记应用到甘草属植物种质资源研究中,筛选的18对引物共扩增312条多态性条带,多态率为99.68%,高于该标记在重楼[23]、杜仲[24]、桃儿七[25]、川续断[26]等中的多态性百分率。EST-SSR标记11对引物多态性为100%,而RAPD[27]和AFLP[28-29]标记在甘草属植物中的多态性百分率仅别为81.7%和69.43%,说明SCoT、EST-SSR标记能够为甘草属植物研究提供更为丰富的多态信息。ANOVA方差分析(表 4)显示2种标记的有效等位基因数(Ne)和位点平均期望杂合度(He)没有显著差异,但 SCoT标记的标记指数(MI)为 2.357大于EST-SSR标记(1.099),说明 SCoT标记多态性检测效率高于EST-SSR标记,这与以上2种标记应用于鸭茅锈病的遗传多样性研究的结果相同[30]。此外,Hajibarat等[10]应用SSR、SCoT及CDDP标记分析鹰嘴豆遗传多样性时表明SCoT和CDDP标记较SSR标记具有更多的多态引物及更高的多态性。因而,SCoT标记可能更适合用于评估群体遗传多样性,或指纹图谱分析[31-32]。
聚类分析显示,2种标记相比,EST-SSR(0.820)产生的平均相似系数范围大于 SCoT(0.785),EST-SSR标记个体间遗传差异检测能力略高于SCoT。而在群体划分上,2种标记的趋势相近,在结合线为L时14份材料划分为3组,其中4、5号蜜腺甘草与6、7号光果甘草聚为一支,符合经典分类中将蜜腺甘草作为光果甘草变种的处理。而黄甘草类群荚果形态复杂多变,于1960年首次提出时,便认为是胀果甘草与乌拉尔甘草的杂交种[1],本实验中两种标记均将11-14号黄甘草与胀果甘草聚为一组,表明黄甘草与与胀果甘草亲缘关系较近,胀果甘草为黄甘草的杂交亲本之一,这与陆嘉惠等[27]以及李晓岚等[20]的研究结果相同。此外,根据EST-SSR标记聚类结果可知,光果、蜜腺甘草组与胀、黄甘草组聚为一支,四者之间亲缘关系较近,且这几种甘草采集地重叠,根据李晓岚等[20]提出黄甘草遗传特性可能会因同域分布亲本种不同的观点,可以推测本实验中黄甘草的另一可能杂交亲本为光果甘草或者蜜腺甘草。以上结果表明2种标记均能够揭示甘草属植物种间、种内亲缘关系,而在个体间遗传差异检测上EST-SSR标记效果更佳,更利于疑难种鉴定及亲本分析。
4 结论
SCoT分子标记初步应用于甘草属植物的研究中,引物多态性及聚类分析均显示出该分子标记在甘草属植物遗传多样性研究中的巨大潜能。在与EST-SSR标记比较中,发现两种标均能作为遗传标记,用于甘草属遗传多样性分析。其中,SCoT标记多态性高于EST-SSR,能够提供丰富的遗传信息,更适于甘草属种质资源的遗传多样性分析。而EST-SSR标记更有利于疑难种鉴定及亲本分析。因此结合两种标记能够对甘草属植物做出更加全面合理的分析,为我国甘草野生种质资源保护及可持续开发提供理论基础。
[1] 李学禹,陆嘉惠.甘草属(GlycyrrhizaL.)分类系统与实验生物学研究[M].上海:复旦大学出版社,2015:3-50.
[2] 中国药典委员会.中华人民共和国药典.一部[M].北京:中国医药科技出版社,2015:148.
[3] 张新玲,李学禹,魏凌基.新疆甘草属的种间杂交[J].西北植物学报,1998,18(1):132-136.Zhang X L,Li X Y,Wei L J,et al.Th e inters pecific hybridization ofGlycyrrhizain Xinjiang[J].Acta Boreal.-Occident.Sin.,1998,18(1):132-136.
[4] 廖云海.两种药用甘草繁育系统的研究[D].新疆石河子:石河子大学,2011.
[5] 田润炜,陆嘉惠,谢良碧,等.光果甘草与乌拉尔甘草开花与传粉方式对生殖及种间关系的影响[J].西北植物学报,2012,32(10):2004-2008.Tian R W,Lu J H,Xie L B,et al.Effect of flowering mode and pollination on reproductive success and the relationship betweenGlycyrrhiza glabraL.andGlycyrrhiza uralensis Fisch[J].Acta Boreal.-Occident.Sin.,2012,32(10):2004-2008.
[6] 谢良碧,陆嘉惠,李晓岚,等.三种甘草属植物的种间杂交亲和性及杂交种子活力[J].植物分类与资源学报,2014,36(3):342-348.Xie L B,Lu J H,Li X L,et al.The cross compatibility and hybrid seed vigor among threeGlycyrrhizaspecies[J].Plant Diversity and Resources,2014,36(3):342-348.
[7] Collard B C Y,Mackill D J.Start codon targeted(SCoT)polymor-phism:a simple,novel DNA marker technique for generatinggene-targeted markers in plants[J].Plant Mol Biol Rep,2009,27(1):86-93.
[8] Xiong F,Zhong R,Han Z,et al.Start codon targeted polymorphism for evaluation of functional genetic variation and relationships in cultivated peanut(Arachis hypogaeaL.)genotypes.[J].Molecular Biology Reports,2010,38(5):3487-3494.
[9] 龙治坚,范理璋,徐刚,等.SCoT分子标记在植物研究中的应用进展[J].植物遗传资源学报,2015,16(2):336-343.Long Z J,Fan L Z,Xu G,et al.Application advance of SCoT molecular markers in plants[J].Journal of Plant Genetic Resources,2015,16(2):336-343.
[10] Hajibarat Z,Saidi A,Hajibarat Z,et al.Characterization of genetic diversity inchickpeausing SSR markers,Start Codon Targeted Polymorphism(SCoT)and conserved DNADerived Polymorphism(CDDP)[J].Physiology and Molecular Biology of Plants,2015,21(3):365-373.
[11] 蒋林峰,张新全,黄琳凯,等.鸭茅品种的SCoT遗传变异分析[J].草业学报,2014,23(1):229-238.Jiang L F,Zhang X Q,Huang L K,et al.Analysis of genetic diversity in a cocksfoot(Dactylis glomerata)variety using SCoT markers[J].Acta Pratacult Sin,2014,23(1):229-238.
[12] 陈红,杨鑫,安华明.贵州桃种质资源遗传多样性的SCoT分析[J].西北植物学报,2014,34(8):1559-1564.Chen H,Yang X,AN H M.Genetic diversity of Peach accessions in Guizhou analysed by SCoT markers[J].Acta Boreal.-Occident.Sin.2014,34(8):1559-1564.
[13] Amirmoradi B,Talebi R,Karami E.Comparison of genetic variation and differentiation among annualCicerspecies using start codon targeted(SCoT)polymorphism,DAMDPCR,and ISSR markers[J].Plant Systematics&Evolution,2012,298(298):1679-1688.
[14]Huang L,Huang X,Yan H,et al.Constructing DNA fingerprinting ofHemarthriacultivarsusing EST-SSR and SCoT markers[J].Genetic resources and crop evolution,2014,61(6):1047-1055.
[15]Tiwari G,Singh R,Singh N,et al.Study of arbitrarly amplified(RAPD and ISSR)and gene targeted(SCoT and CBDP)markers for genetic diversity and population structure in Kalmegh[Andrographis paniculata(Burm.f.)Nees][J].Industrial Crops&Products,2016,19(86):1-11.
[16] Tsaballa A,Ganopoulos I,Timplalexi A,et al.Molecular characterization of Greek pepper(Capsicum annuum,L)landraces with neutral(ISSR)and gene-based(SCoT and ESTSSR)molecular markers[J].Biochemical Systematics&Ecology,2015,2(59):256-263.
[17] 姜春芽,廖娇,徐小彪,等.植物EST-SSR技术及其应用[J].分子植物育种,2009,7(1):125-129.Jiang C Y,Liao J,Xu B,et al.Plant EST-SSR technology and its application[J].Molecular Plant Breeding,2009,7(1):125-129.
[18] 董清华,王西成,赵密珍,等.草莓EST-SSR标记开发及在品种遗传多样性分析中的应用[J].中国农业科学,2011,44(17):3603-3612.Dong Q H,Wang X C,Zhao M Z,et al.Development of EST-Derived SSR markers and their application in strawberry genetic diversity analysis[J].Scientia Agricultura Sinica,2011,44(17):3603-3612.
[19] 苏会,刘建军,贺巍,等.豫南茶区茶品质相关性状与ESTSSR标记的关联分析[J].农业生物技术学报,2016,24(9):1328-1336.Su H,Liu J J,He W,et al.Association analysis of traits related to tea(Camellia sinensis)quality with EST-SSRs in southern Henan area[J].Journal of Agricultural Biotechnology,2016,24(9):1328-1336
[20] 李晓岚,陆嘉惠,谢良碧,等.4种甘草属植物EST-SSR引物开发及其亲缘关系分析[J].西北植物学报,2015,35(3):480-485.Li X L,Lu J H,Xie L B,et al.Development of EST-SSR primers and genetic relationship analysis in fourGlycyrrhizaL.species[J].Acta Boreal.-Occident.Sin.,2015,35(3):480-485.
[21] Um Y,Jin M L,Lee Y,et al.Genetic diversity analysis of Glycyrrhiza uralensisusing 8 novel polymorphic microsatellite markers[J].J Plant Biotechnol,2016,43:174-180
[22] Belaj A,Satovic Z,Cipriani G,et al.Comparative study of the discriminating capacity of RAPD,AFLP and SSR markers and of their effectiveness in establishing genetic relationships in olive[J].Theoretical and Applied Genetics,2003,107(4):736-744.
[23] 李 壮,辛本华,杨 华,等.重楼属植物遗传多样性的SCoT标记[J].广西植物,2014,34(3):315-319.Li Z,Xin B H,Yang H,et al.SCOT genetic diversity of plants inParis[J].Guihaia,2014,34(3):315-319.
[24] 肖承鸿,周 涛,江维克,等.贵州栽培杜仲表型性状与SCoT分子标记的遗传多样性分析[J].中药材,2014,37(8):1343-1349.Xiao C H,Zhou T,Jiang W K,et al.Analysis of genetic diversity of cultivated(eucommia ulmoides) from Guizhou province by phenotypic traits and SCoT markers[J].Journal of Chinese Medicinal Materials,2014,37(8):1343-1349.
[25] 陈大霞,赵纪峰,刘 翔,等.濒危药用植物桃儿七野生居群遗传多样性与遗传结构的SCoT分析 [J].中国中药杂志,2013,38(2):278-283.Chen D X,Zhao J F,Lou X,et al.Genetic diversity and genetic structure of endangered wildSinopodophyllum emodiby start codon targeted polymorphism[J].China Journal of Chinese Materia Medica,2013,38(2):278-283.
[26] 陈大霞,张 雪,崔广林,等.川续断野生种质资源遗传多样性的SCoT分析[J].中国中药杂志,2015,40(10):1898-1903.Chen D X,Zhang X,Cui G L,et al.Analysis on genetic diversity among wildDipsacus asperoidesby SCoT[J].China Journal of Chinese Materia Medica,2015,40(10):1898-1903.
[27] 陆嘉惠,李学禹,马 淼,等.国产甘草属植物的RAPD分析及其分类学研究[J].西北植物学报,2006,26(3):527-531.Lu J H,Li X Y,Ma M,et al.Analysis and classification of GlycyrrhizaL.plants in China by RAPD[J].Acta Boreal.-O-ccident.Sin.,2006,26(3):527-531.
[28] 葛淑俊,李广敏,马峙英,等.甘草野生种群遗传多样性的AFLP 分析[J].中国农业科学,2009,42(1):47-54.Ge S J,Li G M,Ma Z Y,et al.Analysis on genetic diversity of wild populations of Licorice (Glycyrrhiza uralensis Fisch.)with AFLP markers[J].Scientia Agricultura Sinica,2009,42(1):47-54.
[29] Hakimi A,Zolfaghari M,Sorkheh K.Genetic structure and diversity analysis revealed by AFLP markers on different Glycyrrhiza glabra,L.an endangered medicinal species from South of Iran and implications for conservation[J].Biochemical Genetics,2016(3):1-17.
[30]Yan H,Zhang Y,Zeng B,et al.Genetic diversity and association of EST-SSR and SCoT markers with rust traits in Orchardgrass(Dactylis glomerataL.).[J].Molecules,2016,21(1):1-13.
[31] Gorji A M,Poczai P,Polgar Z,et al.Efficiency of arbitrarily amplified dominant markers (SCOT,ISSR and RAPD)for diagnostic fingerprinting in tetraploid Potato[J].American Journal of Potato Research,2011,88(3):226-237.
[32] 赵梦然,陈强,张金霞,等.IGS2-RFLP、SCoT和ISSR在白灵侧耳遗传多样性分析中的比较研究[J].菌物学报,2013,32(4):682-689.Zhao M R,Chen Q,Zhang J X,et al.Comparison studies of genetic diversity ofPleurotus eryngivar.tuoliensis by IGS2-RFLP,SCoT and ISSR markers[J].Mycosystemma,2013,32(4):682-689.
Comparison between SCoT and EST-SSR markers in detection of genetic diversity of wildGlycyrrhizaL.species
Song Feng1,Lu Jiahui1,2*,Han Chun1,Niu Qingdong1,Chen Chaonan1,Li Xueyu2
(1 College of Life Science,Shihezi University/The Key Oasis Eco-Agriculture Laboratory of Xinjiang Production and Construction Group,Shihezi,Xinjiang 832003,China;2 Institute of Licorice in Shihezi University,Shihezi,Xinjiang 832003,China)
To applied researches in germplasm resources and genetic diversity of medicinal plant inGlycyrrhizawere carried out based on SCoT and EST-SSR molecular markers.Genomic DNA polymorphic comparative analysis by SCoT and EST-SSR markers in,a total of 14 individuals,are collected from 8 natural populations,including four species and one variant of Glycyrrhiza.The results showed that total of 313 bands were detected using 18 pairs of primers,among which 312 were polymorphic bands.The average percentage of polymorphic bands was 99.68%,and there were no significant difference in the average effective number of alleles(Ne)and the expected heterozygosity(He)between EST-SSR and SCoT markers.SCoT marker(E=17.333,Ai=21.145)was higher than EST-SSR (E=6.909,Ai=8.719)in assay efficiency.The systematic diagram of genetic relationship indicated that SCoT and EST-SSR markers grouped the 14 individuals into three major groups of the same.This cluster was in accord with the classical taxonomy,indicating that both methods were efficient in revealing interspecific or intraspecific genetic difference and relationship.These results indicates that SCoT markers bring a rich in polymorphism and informativeness and it would be applied more efficient than EST-SSR to germplasm analysis of the genetic diversty for Glycyrrhiza L..Meanwhile,EST-SSR is good at detecting differences among individuals,and better for identification and parentage analysis.
GlycyrrhizaL.;SCoT;EST-SSR;Genetic diversity
Q949.4
A
10.13880/j.cnki.65-1174/n.2017.02.014
1007-7383(2017)02-0213-07
2016-11-30
国家自然科学基金项目(31260042)
宋凤(1991-),女,硕士研究生,专业方向为植物分类与多样性。
*通信作者:陆嘉惠(1974-),女,副教授,从事药用植物分类与资源研究,e-mail:jiahuil@shzu.edu.cn。

