大叶落地生根细胞周期蛋白依赖激酶KdCDK基因克隆及表达分析
朱敏群,梁小红,黄慧青,欧素荣,葛永强,张利娟,钟天秀
(1.深圳市日昇园林绿化有限公司,广东 深圳 518040;2.北京林业大学 林学院 草坪研究所,北京 100083;3.暨南大学 深圳旅游学院,广东 深圳 518053;4.华南农业大学 林学与风景园林学院,广东省草业工程技术研究中心,广东 广州 510642)
大叶落地生根细胞周期蛋白依赖激酶KdCDK基因克隆及表达分析
朱敏群1,梁小红2,黄慧青1,欧素荣1,葛永强1,张利娟3,钟天秀4
(1.深圳市日昇园林绿化有限公司,广东 深圳 518040;2.北京林业大学 林学院 草坪研究所,北京 100083;3.暨南大学 深圳旅游学院,广东 深圳 518053;4.华南农业大学 林学与风景园林学院,广东省草业工程技术研究中心,广东 广州 510642)
为了更好理解大叶落地生根中细胞周期调控的分子机制,利用RACE-PCR技术从大叶落地生根中克隆了1个新的基因KdCDK。该基因编码295个氨基酸残基,分子量为34.12 kDa,等电点为6.15,其开放阅读框长度为888 bp。其蛋白与笋瓜CDK蛋白的同源性最高,属于CDKA类蛋白家族。实时荧光定量PCR分析表明,该蛋白基因在大叶落地生根的根中表达量最高,且受渗透胁迫(甘露醇)的诱导上调表达。从大叶落地生根中克隆出KdCDK基因,为将来该基因的功能研究打下了基础。
大叶落地生根;细胞周期蛋白依赖激酶;基因表达
细胞周期是受到严格调控的过程,通过一系列不连续的关键点来确保DNA正确复制和细胞的成功分裂。真核生物中的细胞周期由丝氨酸/苏氨酸蛋白激酶家族调控,最常见的就是细胞周期蛋白依赖激酶(CDKs)[1-3]。CDKs活性调节主要有以下3种方式:磷酸化和去磷酸化、结合相互作用蛋白如细胞周期蛋白依赖激酶抑制蛋白(ICK)或细胞周期蛋白依赖激酶活化蛋白(CAK)、结合细胞周期蛋白[4-5]。到目前为止,从41种植物中克隆出152个CDK基因,根据与细胞周期蛋白结合位点基序的差异把它们分为8个亚家族,包括CDKA~G和CDKL(类细胞周期蛋白依赖激酶)[6-7]。CDKA类蛋白家族含有PSTAIRE特征的序列,CDKB类蛋白家族含有PPTALRE或PPTTLRE特征序列,CDKC类蛋白家族含有具有PITAIRE特征序列,CDKD、CDKE和CDKF类蛋白家族调控CDK活化子,含有SPTAIRE特征序列,CDKG类蛋白家族含有PLTSLRE特征序列,CDKL类蛋白家族含有(V、I、L、K、R)FMAREI特征序列,根据基序中氨基酸残基的变化将CDKL分为CDKL1~CDKL15[6]。
第一个CDK激酶是在酵母中发现的,酵母中cdc2/cdc28突变体的表型能够完全被CDKA修复,说明单一的CDK基因就能完全调控酵母的细胞周期[8]。到现在为止,已有大量的研究表明CDKA在受精胚发育早期和体细胞胚发育过程中起到了至关重要的作用[9-10]。过表达CDKA∶1的植株没有出现表型上的改变,但是该基因的显性失活突变体抑制转基因烟草中细胞周期过程,使细胞分裂速度下降,细胞变大,植株矮小[11]。在拟南芥中,cdc2At基因的显性突变体(与CDKA∶1是同源基因)中细胞周期蛋白依赖激酶失活,细胞周期活动受到抑制,植株无法存活[11];而该基因的显性失活突变体影响胚胎形成过程,C13株系不能形成种子,其他株系C1、C5、C12中胚胎上下轴模式(Apical-basal embryo pattern)出现畸形[11]。Nowack等[12]的研究都表明CDKA∶1基因完全被敲掉之后会导致胚胎死亡,花粉粒中出现双细胞或者三细胞的精细胞。最近一项研究表明,CDKA∶1基因不仅在细胞进入有丝分裂间期中的S期中有非常重要的作用,而且具有促进根中的细胞增殖使根发育完整并维持根中分生组织的功能[12]。
大叶落地生根胎生苗的发育过程兼具胚胎发生途径和器官发生途径,是研究植物发生发育的理想模型[13]。为揭示大叶落地生根胎生苗发育机制,采用抑制消减杂交技术(SSH)构建了大叶落地生根胎生苗和普通叶片的SSH-cDNA文库[14],并从该差减文库中筛选出一条与细胞周期蛋白依赖激酶基因片段同源的表达序列标签(EST)。本研究以该EST片段为基础,采用RACE-PCR技术克隆出大叶落地生根细胞周期蛋白依赖激酶的cDNA基因全长(命名为KdCDK),利用生物信息学方法分析KdCDK基因的序列、蛋白结构、系统发育,并研究该基因在大叶落地生根不同组织和渗透胁迫诱导下的表达变化,为该基因的进一步研究利用打下了基础。
1 材料和方法
1.1 材料的培养
大叶落地生根(Kalanchoedaigremontiana)种植于北京林业大学草坪研究所温室,培养基质为腐叶土、蛭石和珍珠岩(按4∶2∶1混合),在16 h光照/8 h黑暗,光照强度为25 μmol/(m2·s),温度为(30±3)℃下培养。采集叶片后快速在液氮中固定,置于-80 ℃超低温冰箱保存。
1.2 总RNA的提取以及反转录
使用TaKaRa MiniBEST universal RNA Extraction Kit(Code No.9769)提取落地生根叶片的总RNA,并在-80 ℃下保存。
1.3KdCDK基因的克隆和测序
根据差减文库的测序结果,分别设计上游引物F1和下游引物R1(表1)。以总RNA为模板,按照PrimeScriptTMⅡ High Fidelity RT-PCR Kit(TaKaRa Code No.RO23A)的操作说明合成第一链cDNA,使用TaKaRa Tks Gflex DNA polymerase(Code No.R060A)进行PCR扩增。PCR反应体系为50 μL,包括1 μL cDNA模板,1 μL Tks Gflex DNA Polymerase(1.25 U/μL),1 μLF1引物(20 μmol/L),1 μLR1引物(20 μmol/L),25 μL 2×Gflex Buffer(Mg2+,dNTP plus),21 μL ddH2O 。反应程序为: 98 ℃变性 10 s,55 ℃退火30 s,68 ℃ 延伸1 min,共30个循环。以上述PCR产物为模板,进行2次PCR,反应体系及条件同上。取5 μL PCR产物进行1%琼脂糖凝胶电泳。用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.3.0(Code No.9762)回收,用F1/R1为特异引物对回收后的DNA测序。
根据获得的CDK基因cDNA片段序列设计5′RACE Outer PCR特异引物R01和Inner PCRR02(表1)。使用SMARTerTMRACE cDNA Amplification Kit(Clontech Cat.No.634923)对反转录总RNA合成cDNA。使用TaKaRa Tks Gflex DNA polymerase(Code No.R060A)进行PCR扩增。Outer PCR反应完成后,立即取反应液1 μL进行Inner PCR反应。PCR产物经1%琼脂糖凝胶电泳后,用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver.3.0(Code No.9762)回收。接着用TaKaRa DNA Ligation Kit Ver.2.1(Code No.6022)中的连接酶,将PCR产物与T-Vector pMDTM18(Code No.3271)进行连接,热转化至大肠杆菌(Escherichiacoli)感受态细胞JM109(Code No.9052)中,涂布平板,37 ℃过夜培养。挑选阳性菌落,提取质粒,用M13-47引物对进行测序。由于第1次5′RACE 结果不理想,重新设计引物,进行再次5′RACE。5′RACE Outer PCR特异引物R06和Inner PCRR07见表1。具体操作同第1次5′RACE过程。
根据获得的CDK基因cDNA片段序列设计3′RACE Outer PCR特异引物F01和Inner PCR特异引物F02(表1)。以1 μL的RNA为模板,使用3′-Full RACE Core Set with PrimeScriptTMRTase(Code No.6106)反转录合成cDNA。使用TaKaRa Tks Gflex DNA polymerase(Code No.R060A)进行PCR反应。2轮PCR反应完成后,PCR产物经1%琼脂糖凝胶电泳。经切胶回收和连接克隆,用M13-47引物对质粒进行测序分析。具体步骤同5′RACE。
对CDK基因cDNA片段的5′端和3′序列进行拼接,获得KdCDK基因的全长序列。并分别设计上游引物YZF1和YZF2及下游引物YZR1(表1),以验证试验中使用的cDNA为模板,进行2轮PCR反应。第1轮PCR反应使用引物YZF1和YZR1。第2轮PCR反应使用YZF2和YZR1。使用TaKaRa Tks Gflex DNA polymerase(Code No.R060A)进行PCR扩增。取5 μL PCR产物进行1%琼脂糖凝胶电泳。用YZR1进行测序分析。
1.4KdCDK基因的生物信息学分析
应用NCBI数据库中ORF Finder软件寻找KdCDK序列中的ORF,推导编码的氨基酸序列。应用Expasy的ScanProstie、ProtParam、TMpred、Protscale程序分析KdCDK蛋白的保守结构域、分子量和等电点、跨膜结构域和亲疏水性[15]。用Blast程序检索相似蛋白,利用ClustalX 2.1 软件对不同物种的氨基酸序列进行多重比对[16];利用MEGA 6.0进行氨基酸序列分析,用邻接法构建进化树,并用Bootstrap校正[17];根据NPS@ web server网站中的PHD方法预测蛋白的二级结构[18]。应用在线分析工具Phyre分析CDK蛋白三级结构[19]。
1.5KdCDK表达的荧光定量分析
以大叶落地生根actin作为内参基因(上下游引物见表1),根据KdCDK的序列信息设计设计荧光定量的上游引物F2和下游引物R2(表1)。以大叶落地生根中的根、茎、叶、叶柄为总RNA材料分析KdCDK基因在不同组织中的表达情况。对大叶落地生根幼苗进行渗透胁迫处理(300 mmol/L甘露醇),分别在处理的0,3,6,9,12,24 h采集叶位相同叶片,每个处理设3个重复,3个重复混合取样,提取总RNA分析KdCDK基因在渗透胁迫下的表达情况。按照Ist Strand cDNA Synthesis Kit(TaKaRa)试剂盒的操作说明合成cDNA,根据SYBR FAST qPCR Kit Master Mix(2×)Universal(KAPA Biosystems)试剂盒进行PCR扩增。荧光定量反应体系为:SYBR FAST qPCR Kit Master Mix(2×)Universal 5 μL,上下游引物(10 μmol/L)0.2 μL,cDNA 1 μL,ROX校正染料 0.2 μL,dH2O 3.4 μL,共10 μL。反应在ABI7900HT实时定量PCR仪上进行,反应条件为:95 ℃预变性 5 min;95 ℃变性30 s,60 ℃退火30 s。采用2-ΔΔCT法分析结果。

表1 KdCDK基因克隆和鉴定中所用的引物序列
2 结果与分析
2.1KdCDKcDNA克隆与测序
以大叶落地生根叶片第一链cDNA为模板,并用引物F1和R1进行PCR扩增,获得约150 bp的目的片段(图1-A)。根据获得的SAHH基因cDNA片段序列分别设计3′RACE和5′RACE特异引物,获得约600,200 bp的目的片段(图1-B、C)。由于测序结果不理想,再次设计引物进行5′RACE试验,获得约1 000 bp的目标片段,电泳检测结果如图1-D。将获得的cDNA片段的5′端和3′端序列进行拼接,以大叶落地生根叶片第一链cDNA为模板,设计验证引物,获得约1 200 bp的目的片段(图1-E)。因此,大叶落地生根CDK基因的cDNA全长为1 437 bp(GenBank登录号:KU740360),命名为KdCDK,含有888 bp完整开放阅读框(ORF),编码295个氨基酸(图2),不包括PolyA尾的长度为1 426 bp,5′UTR(非编码区)361 bp,3′UTR 176 bp。
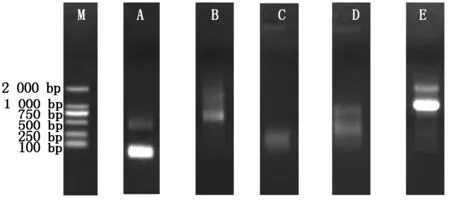
M.D2000 DNA Marker;A.根据差减文库的结果设计引物验证KdCDK基因;B.3′ RACE 结果;C.5′ RACE 结果;D.再次5′RACE结果;E.RACE结果验证。
M.D2000 DNA Marker;A.Validation fragments ofKdCDKgene according to the results of subtractive library;B.The result of 3′ RACE;C.The result of 5′ RACE;D.The result of second 5′ RACE;E.The verification of RACE results.
图 1 大叶落地生根KdCDK同源扩增和RACE 克隆以及序列验证
Fig.1 RT-PCR amplification,RACE cloning and sequence verification ofKdCDKgene in
Kalanchoedaigremontiana

下划线ATG为起始密码子;方框TGA为终止密码子;灰色阴影部分为蛋白激酶ATP结合信号区域;虚线部分为CDKA类蛋白的特征序列;双下划线为丝氨酸/苏氨酸蛋白激酶活性位点信号序列。
2.2 KdCDK 序列分析及系统进化树的构建
KdCDK与笋瓜(CBJ18166.1)、陆地棉(ABV64386.1)、毛果杨(XP 002306004.1)、金鱼草(CAA66233.1)的相似性分别为68%,67%,66%,65%。通过ScanProsite在线软件分析,发现该蛋白具有2个保守的结构域:蛋白激酶ATP结合区域信号序列(10-33)、丝氨酸/苏氨酸蛋白激酶活性位点信号序列(123-135)。含有保守的基序(motif)“PSFALRE”,与CDKA蛋白激酶类似,是细胞周期蛋白的结合位点。通过SIB中的Motif Scan,预测得到一些不同的基序,包括1个SANT 保守域(1-12)、2个蛋白酪氨酸激酶(4-20、49-152)、2个酪氨酸激酶磷酸化位点(8-15、280-287)、3个MYRISTYL N-豆蔻酰化位点(114-119、148-153、191-196)、4个酪蛋白激酶Ⅱ磷酸化位点(70-73、93-96、183-186、222-225)、5个蛋白激酶C磷酸化位点(25-27、120-122、141-143、274-276、278-280)。
将大叶落地生根KdCDK与其他25种植物中的CDKA进行了氨基酸序列比对,可以看出,不同植物CDK氨基酸序列在几个重要功能位点的序列保守性程序很高,如蛋白激酶ATP-结合区域、丝氨酸/苏氨酸蛋白激酶活性位点、T-Loop区域(图3)。利用Mega 6.0中的相邻连接法构建系统进化树(图4),采用默认参数,自检举1 000次,对生成的系统进化树进行Bootstrap校正。结果表明,KdCDK与CDKB类蛋白完全分开,与其他植物中的CDKA类蛋白同属于一类。双子叶、单子叶、裸子植物中的CDKA类聚为一类,而大叶落地生根的CDK蛋白单独聚为一类。
2.3 KdCDK的理化性质及三级结构预测
KdCDK蛋白的理论分子量为34.12 kDa,理论等电点为6.15。脂溶指数为92.51,总平均亲水性(GRAVY)为-0.188,不稳定系数为34.51,表明该蛋白是一个稳定的蛋白。二级结构预测结果表明,KdCDK含有48.47%的α-螺旋,20.68%的β-折叠和30.85%的随机卷曲(图5,6),没有信号肽及其剪切位点,是水溶性的非分泌型蛋白,有1个明显的跨膜区(表2),被定位于细胞质中。

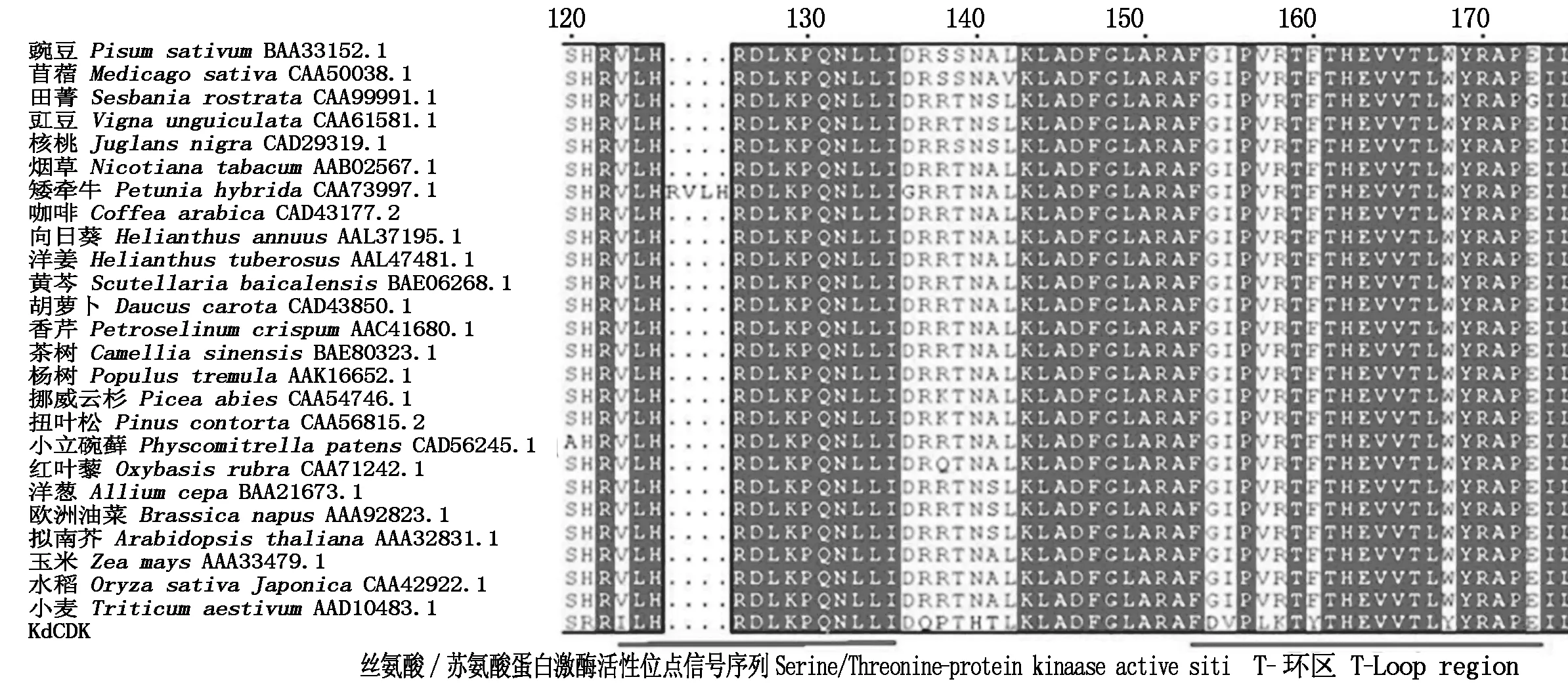
图3 大叶落地生根细胞周期蛋白依赖激酶与其他植物的细胞周期蛋白依赖激酶氨基酸序列比对
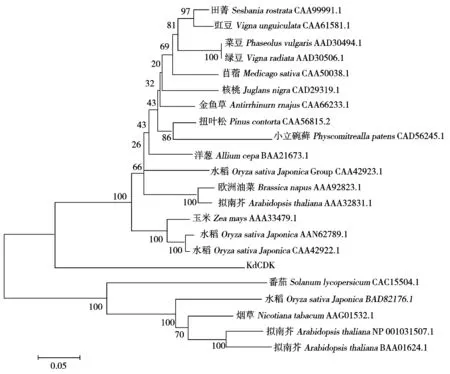
图4 大叶落地生根细胞周期蛋白依赖激酶与其他植物细胞周期蛋白依赖激酶氨基酸序列的进化分析
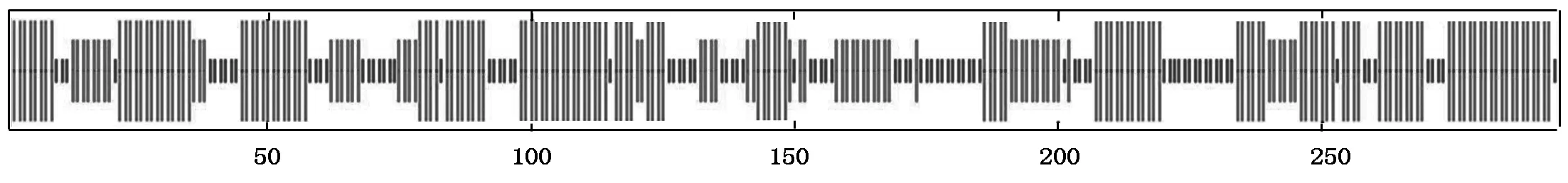
数字代表氨基酸;最长的细线代表α-螺旋;中等长度的细线代表β-折叠。
通过Phyre在线预测网站预测的三维结构表明,KdCDK具有细胞周期蛋白依赖激酶典型的三维结构,包括2个小叶结构,一个小叶主要由α-螺旋构成,另一个小叶由α-螺旋和β-折叠构成。共有11个α-螺旋和8个β-折叠(图6)。

表2 KdCDK 跨膜区域
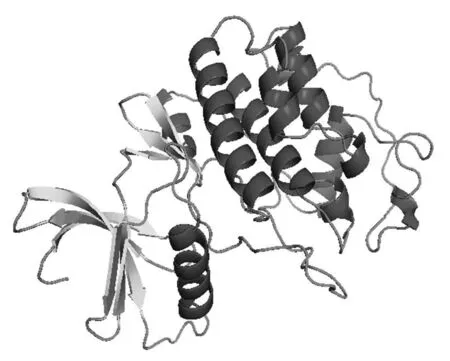
螺旋为α-螺旋;箭头为β-折叠;曲线为无规则卷曲。
2.4KdCDK的组织表达分析
利用荧光实时定量PCR对KdCDK在大叶落地生根中的表达情况进行检测,结果如图7所示,KdCDK在根中表达量最高,大约是叶中的1.6倍。根、叶、叶柄、茎中KdCDK的表达量没有明显差异。这些结果表明,KdCDK在各组织中均有表达,但不具有组织特异性。
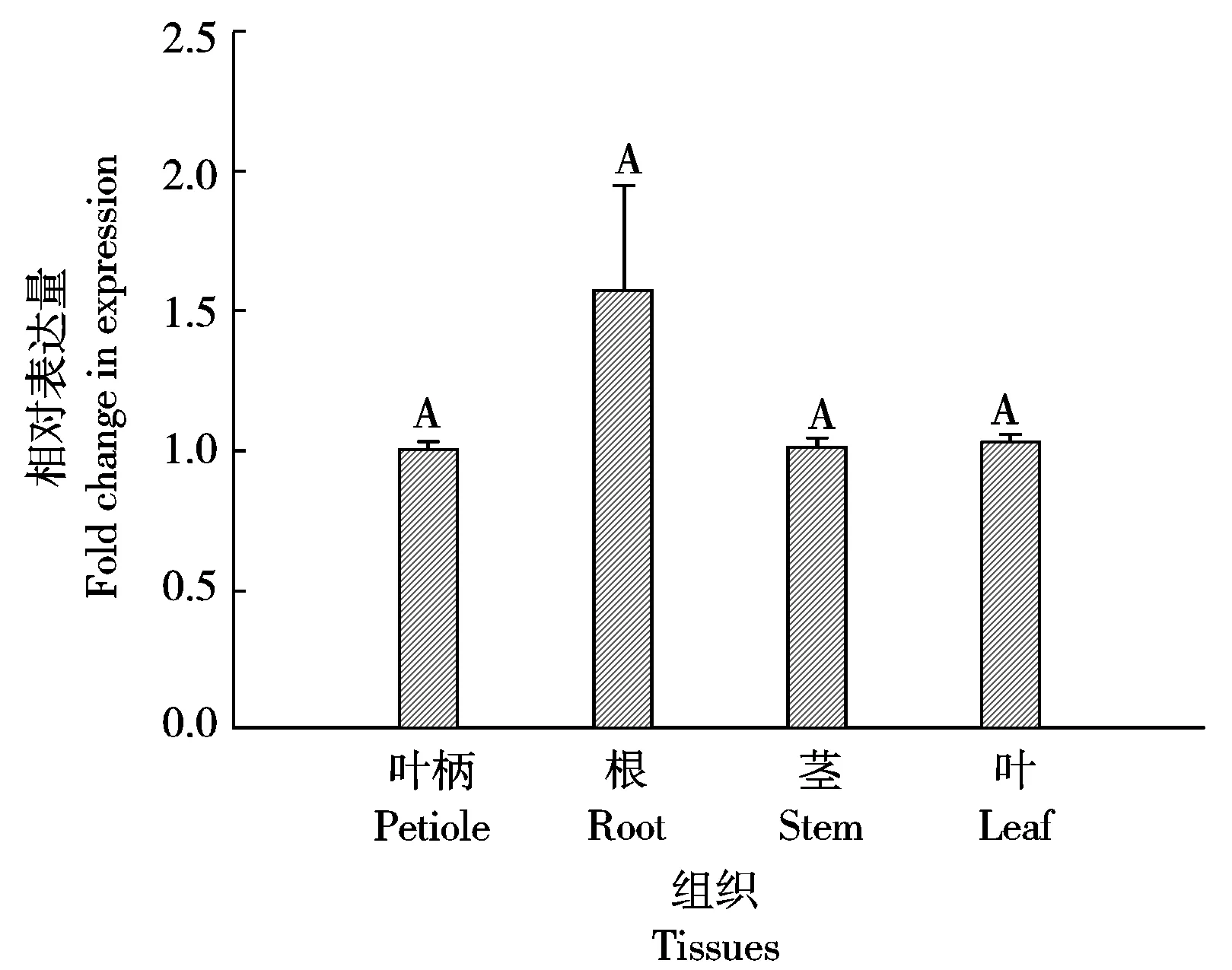
图7 KdCDK在大叶落地生根中不同组织中的表达
2.5KdCDK的诱导表达分析
用300 mmol/L甘露醇对大叶落地生根进行渗透胁迫,以实时荧光定量PCR法分析其表达动态(图8)。结果显示,KdCDK在甘露醇处理下存在上调表达的趋势。在处理 24 h后,KdCDK的表达量达到最大值。
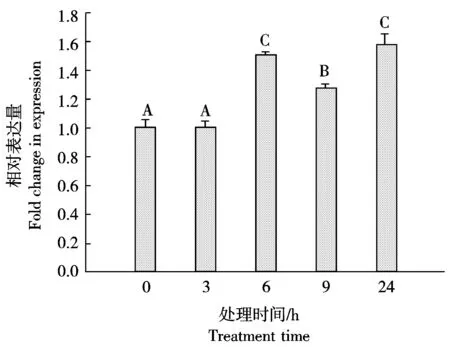
图8 KdCDK在渗透胁迫下的表达
3 讨论
目前,已经从不同的植物中克隆出大量的CDK类基因。本研究从大叶落地生根中克隆出CDK基因序列含有888 bp完整开放阅读框,编码的蛋白质与笋瓜、陆地苗、毛白杨等植物中的CDKA类蛋白相似性都很高[20]。对CDKA类蛋白进行比对分析发现该类蛋白5′端相对保守,而在3′端差异比较大,说明A类CDK蛋白家族的氨基酸序列相对保守。而且,进化树比对分析结果表明大叶落地生根细胞周期依赖激酶属于CDKA类蛋白聚在一起,与CDKB类蛋白完全分开,与Zhang等[21]的研究结果一致。所有这些结果说明KdCDK是一个新的CDKA类基因,与之前报道的植物CDKA类基因在序列信息上有诸多相似之处。
一般情况下,根据不同的基序推测蛋白的生物学功能。在KdCDK中,含有“PSFALRE”,与植物中A型CDK类的典型基序“PSTAIRE”十分相似,推测KdCDK属于CDKA类蛋白,并具有相应的功能。KdCDK特征序列中存在2个氨基酸突变,可能是因为大叶落地生根特殊的繁殖方式。在KdCDK中,也存在其他的保守区域,如蛋白激酶ATP结合区域、丝氨酸/苏氨酸蛋白激酶活性位点,这些都与维持CDK的功能密切相关。总的来说,通过对KdCDK保守区域的预测可以推测,它的功能应该类似于植物中其他CDKA类蛋白[22-23]。但是,KdCDK蛋白的具体功能还需从生物化学和基因角度去探究。
通过蛋白三维结构建模分析蛋白结构有利于蛋白的功能分析。目前,CDKs家族蛋白结构建模主要集中在酵母和人上[23-25]。KdCDK蛋白的三维结构是在人类CDK结构(PDB code:d1unla)的基础上进行建模,与其他植物的CDK蛋白结构相似,2个小叶结构都以α-螺旋和β-折叠为主要构件,无规则卷曲为连接方式,围绕着一个中心激活区域。这说明大部分CDK,特别是CDKA类蛋白进化出类似的三维结构[26]。
植物中的CDKA类基因转录本大部分是在组织分裂细胞中表达量较高,比如顶芽、根尖、发育的叶片和花器官组织[27]。非生物胁迫如机械损伤能提高叶中CDKA的表达量[27]。本研究中,KdCDK的表达量在根中最高,说明组织中KdCDK的表达量与细胞分裂和分化的类型有关[28]。在渗透胁迫下,KdCDK的表达量逐渐升高,说明KdCDK的表达量与非生物胁迫有关[29-32]。
本研究对大叶落地生根细胞周期蛋白依赖激酶进行克隆和生物信息学分析,证明该基因在序列结构和功能上与植物CDKA类蛋白类似,在分裂组织(根)中表达量最高,并且受渗透胁迫的诱导,说明KdCDK可能在大叶落地生根根发育和抵抗非生物胁迫的过程中起到一定的作用,关于KdCDK在大叶落地生根中的具体功能还需要进一步的试验进行验证。
[1] Malumbres M. Cyclin-dependent kinases[J]. Genome biology,2014, 6(15):122.
[2] Schmitz M L, Kracht M. Cyclin-dependent kinases as coregulators of inflammatory gene expression[J]. Trends in Pharmacological Sciences,2016, 37(2): 101-113.
[3] Asghar U, Witkiewicz A K, Turner N C, et al. The history and future of targeting cyclin-dependent kinases in cancer therapy[J]. Nature Reviews Drug Discovery,2015, 14(2): 130-146.
[4] Yang K, Wang H, Xue S, et al. Requirement for a-type cyclin-dependent kinase and cyclins for the terminal division in the stomatal lineage ofArabidopsis[J]. Journal of Experimental Botany,2014, 65(9):2449-2461.
[5] Francis D. The plant cell cycle-15 years on[J]. The New Phytologist,2007,174(2): 261-278.
[6] Tank J G, Thaker V S. Cyclin dependent kinases and their role in regulation of plant cell cycle[J]. Biologia Plantarum,2011, 55(2): 201-212.
[7] Vandepoele K. Genome-wide analysis of core cell cycle genes inArabidopsis[J].The Plant Cell Online,2002, 14(4): 903-916.
[8] Nasmyth K. At the heart of the budding yeast cell cycle[J]. Trends Genet,1996, 12(10): 405-412.
[9] Hemerly A S, Ferreira P C, Van Montagu M, et al. Cell division events are essential for embryo patterning and morphogenesis: studies on dominant-negative cdc2aAt mutants ofArabidopsis[J]. The Plant Journal,2000, 23(1): 123-130.
[10] Hemerly A, Engler J A, Bergounioux C, et al. Dominant negative mutants of the Cdc2 kinase uncouple cell division from iterative plant development[J]. EMBO J,1995, 14(16): 3925-3936.
[11] van den Heuvel S, Harlow E. Distinct roles for cyclin-dependent kinases in cell cycle control[J]. Science,1993, 262(5142): 2050-2054.
[12] Nowack M K, Harashima H, Dissmeyer N, et al. Genetic framework of cyclin-dependent kinase function inArabidopsis[J]. Developmental Cell,2012, 22(5): 1030-1040.
[13] Garces H, Sinha N. The ‘mother of thousands’(Kalanchoedaigremontiana): a plant model for asexual reproduction and CAM studies[J]. Cold Spring Harb Protoc,2009, 2009(10): 133.
[14] Zhong T, Zhu C, Zeng H, et al. Analysis of gene expression inKalanchoedaigremontianaleaves during plantlet formation under drought stress[J]. Electron J Biotech,2013, 16(6): 4.
[15] Gasteiger E, Hoogland C, Gattiker A, et al. Protein identification and analysis tools on the ExPASy Server[J].Humana Press, 2005, 112(112),571-607.
[16] Larkin M A, Blackshields G, Brown N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics,2007, 23(21): 2947-2948.
[17] Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics asnalysis (MEGA) software Version 4.0[J]. Molecular Biology and Evolution,2007, 24(8): 1596-1599.
[18] Combet C, Blanchet C, Geourjon C, et al. NPS@: network protein sequence analysis[J]. Trends Biochem Sci,2000, 25(3): 147-150.
[19] Kelley L A, Sternberg M J E. Protein structure prediction on the Web: a case study using the Phyre server[J]. Nature Protocols,2009, 4(3): 363-371.
[20] Joubès J, Phan T, Just D, et al. Molecular and biochemical characterization of the involvement of cyclin-dependent kinase a during the early development of tomato fruit[J]. Plant Physiology,1999,121(3): 857-869.[21] Zhang G, Song C, Zhao M, et al. Characterization of an A-type cyclin-dependent kinase gene fromDendrobiumcandidum[J]. Biologia,2012, 67(2):360-368.
[22] Montero-Cortés M, Rodríguez-Paredes F, Burgeff C, et al. Characterisation of a cyclin-dependent kinase (CDKA) gene expressed during somatic embryogenesis of coconut palm[J]. Plant Cell, Tissue and Organ Culture (PCTOC),2010, 102(2): 251-258.
[23] Brown N R, Noble M E M, Lawrie A M, et al. Effects of phosphorylation of threonine 160 on cyclin-dependent kinase 2 structure and activity[J]. Journal of Biological Chemistry,1999, 274(13): 8746-8756.
[24] Yang M, Ge Y, Wu J, et al. Coevolution study of mitochondria respiratory chain proteins: Toward the understanding of protein-protein interaction[J]. Journal of Genetics and Genomics,2011, 38(5): 201-207.
[25] Mcgrath C F, Pattabiraman N, Kellogg G E, et al. Homology model of the CDK1/cyclin B complex[J].Journal of Biomolecular Structure & Dynamics,2005, 22(5): 493-502.
[26] Endicott J A, Noble M E. Structural characterization of the cyclin-dependent protein kinase family[J]. Biochem Soc Trans,2013, 41(4): 1008-1016.
[27] Hemerly A S, Ferreira P, de Almeida E J, et al. cdc2a expression inArabidopsisis linked with competence for cell division[J]. Plant Cell,1993, 5(12):1711-1723.
[28] Malumbres M. Cyclin-dependent kinases[J]. Genome biology, 2014, 15(6): 122.
[29] Ma X, Qiao Z, Chen D, et al. CYCLIN-DEPENDENT KINASE G2 regulates salinity stress response and salt mediated flowering inArabidopsisthaliana[J]. Plant molecular Biology, 2015, 88(3): 287-299.
[30] Ohama N, Sato H, Shinozaki K, et al. Transcriptional regulatory Network of plant heat stress[J].Trands in Plant Science,2016, 22(1):53.
[31] Guan C, Ji J, Guan W, et al. LcKRP, a cyclin-dependent kinase inhibitor from Lyciumchinense, is involved in an ABA-dependent drought stress-signaling pathway[J]. Plant and soil, 2014, 382(1-2): 43-59.
[32] Nadal-Ribelles M, Solé C, Xu Z, et al. Control of Cdc28 CDK1 by a stress-induced lncRNA[J]. Molecular cell, 2014, 53(4): 549-561.
cDNA Cloning and Expression Analysis of Cyclin-dependent Kinase (KdCDK)Gene inKalanchoedaigremontiana
ZHU Minqun1,LIANG Xiaohong2,HUANG Huiqing1,OU Surong1,GE Yongqiang1,ZHANG Lijuan3,ZHONG Tianxiu4
(1.Shenzhen Risheng Landscape Company Limited,Shenzhen 518040,China;2.Turfgrass Research Institute,The College of Forestry,Beijing Forestry University,Beijing 100083,China;3.Shenzhen Tourism College of Ji′nan University,Shenzhen 518053,China;4.College of Forestry and Landscape Architecture,South China Agricultural University,Guangdong Engineering Research Center of Grassland Science,Guangzhou 510642,China)
To better understand the molecular mechanisms of CDKs involved inKalanchoedaigremontiana,an A-Type CDK gene,KdCDK,was identified using rapid amplification of cDNA end(RACE)PCR.KdCDKgene consists of an ORF of 888 bp that was predicted to encode a 295 amino acid residue-long protein of 34.12 kDa with an isoelectric point of 6.15.KdCDK was related most closely to CmCDK and was clustered to CDKA subfamily.Real-time PCR analysis revealed thatKdCDKtranscript was expressed highly in root and upregulated under osmotic stress.This study characterized the novelKdCDKgene fromKalanchoedaigremontianafor the first time and the results would be useful for further functional determination of the gene.
Kalanchoedaigremontiana;Cyclin-dependent kinase;Gene expression
2017-04-12
深圳市科技计划项目(CXZZ20140418110342522);北京林业大学与厦门日懋城建园林建设股份有限公司产学研合作项目
朱敏群(1978-),女,江西赣州人,工程师,主要从事园林绿化工作。
钟天秀(1986-),女,四川内江人,讲师,博士,主要从事草业生物技术研究。
Q78
A
1000-7091(2017)03-0033-09
10.7668/hbnxb.2017.03.006

