基于Minimum Cuts的蛋白质交互识别
张 景,吴红梅,牛 耘
(南京航空航天大学 计算机科学与技术学院,江苏 南京 210016)
基于Minimum Cuts的蛋白质交互识别
张 景,吴红梅,牛 耘
(南京航空航天大学 计算机科学与技术学院,江苏 南京 210016)
蛋白质交互信息对生物、医药研究有着重要意义,是生物医学领域一项重要的研究内容。对基于大规模语料库的蛋白质交互识别,直接利用已有的PPI数据库,能显著降低人工标注的代价。为此,在大规模语料库的基础上,提出了基于Minimum Cuts的蛋白质交互识别方法。在关系相似性框架下,Minimum Cuts分类器不仅采用SVM算法对单个蛋白质对进行初步分类预测,还利用蛋白质对之间的相似性约束判断结果,使分类结果更加准确。实验结果表明,利用Minimum Cuts分类器进行PPI的识别结果优于SVM分类器的识别结果。当训练数据为20%时,Minimum Cuts分类器的识别结果优于训练数据为80%时的SVM分类器的识别结果。
关系相似性;Minimum Cuts;支持向量机;蛋白质交互
0 引 言
蛋白质是组成细胞最重要的成分,通过交互作用执行着细胞内多数重要的分子过程,如DNA复制。活体细胞内几乎所有的生化反应都与蛋白质交互(Protein-Protein Interaction,PPI)作用有关。所以,蛋白质交互作用已成为生物学研究的重要内容,是解决大量医学难题的关键信息[1-3]。随着生物医学的发展,越来越多的蛋白质交互关系被发现,大量PPI信息隐藏在医学文献文本数据库PubMed中[4]。手工从文献提取PPI信息的方式已难以满足生物医学研究的需求。为了帮助生物医学领域的专家从文献中获取有效的信息,基于自然语言处理的蛋白质交互识别已经成为一项重要的研究内容。
目前,常用的文本PPI识别的技术主要包括:基于共现的方法[5]、基于规则的方法[6]和基于机器学习的方法[7-8]。基于共现的方法[9]根据两个蛋白质共现次数来判断这两个蛋白质之间是否存在交互关系。基于规则的方法[10-11]通过构建一些能够刻画蛋白质交互关系的规则,通过文本匹配的方式来寻找对应的交互关系。
机器学习的方法主要包括两大类:基于特征的方法和基于核函数的方法。基于特征的方法主要采用支持向量机(SVM),从标注有交互关系的蛋白质对的句子中抽取单词、短语和依赖关系等作为特征建立模型,进而判断蛋白质对是否存在交互关系[12-13]。基于核函数的方法深入研究句子结构,通过设计核函数衡量不同蛋白质对间的相似度,然后采用支持核函数的分类器进行PPI关系识别。Haussler[14]针对离散结构提出了卷积核;Lodhi等[15]将特征空间特定长度词语子序列的内积作为核函数的计算方式,提出了字符串核;Bunescu等[16]提出了最短依赖路径核,将句子以树的形式表示,用两个实体之间的最短路径表示实体之间的关系。目前的机器学习方法主要以单个句子为研究对象。这些方法能较好地从句子层面对PPI关系进行描述及判断,但是对于语法复杂和交互关系描述间接的句子,仅依赖单个句子中的信息往往难以进行准确判断。针对这些问题,文献[17]直接以现有的PPI数据库为训练数据,提出了一种新的基于文本相似性来比较识别蛋白质交互的方法。
然而,这些监督的学习方法的有效性依赖于足够大且高质量地标注了蛋白质交互信息的文本集合,构造这样的训练集仍需要耗费大量的人力资源。为此,提出了一种新的基于关系相似性的识别方法,充分利用大规模语料库资源和已有的标注信息进行PPI识别。与上述方法不同,在关系相似性框架下,构建蛋白质对的相似性网络,利用Minimum Cuts算法进行蛋白质交互识别。与SVM相比,利用更少的训练数据取得了更高的精度。
1 基于关系相似性的PPI识别
1.1 关系相似性
在PPI识别工作中,蛋白质之间的交互就是两个蛋白质之间的关系。例如,两个蛋白质protein1和protein2交互,它们的关系可表示为“R(protein1,protein2)”,R表示交互关系。
蛋白质对交互关系的文本表达中存在相似性,表现在描述交互关系的句子在表达上是相似的,例如:
(1)SPB association of Plo1 is the earliest fission yeast mitotic event recorded to date.
(2)All proteins of this family have Cdk-binding and anion-binding sites, but only mammalian Cks1 binds to Skp2 and promotes the association of Skp2 with p27 phosphorylated on Thr-187.
(3)A low concentration of GerE activated cotB transcription by final sigma(K) RNA polymerase.
(4)The SpoIIE phosphatase indirectly activates sigmaF by dephosphorylating a protein (SpoIIAA-P) in the pathway that controls the activity of the transcription factor.
句中黑体的单词表示蛋白质名称。在第1、2句,存在交互关系的蛋白质对(SPB,Plo1)和(Skp2,p27)的关系都为“association of”。第3、4句中,有交互关系的蛋白质对(GerE,cotB)和(SpoIIE, sigmaF)的关系为“activated(GerE,cotB)”和“activates(SpoIIE,sigmaF)”,而activated、activates它们的词根都为activate。因此,可以通过比较目标蛋白质对关系与已知关系的蛋白质对的相似性来判断两个蛋白质之间的关系。
1.2 基于关系相似性的PPI识别描述
基于关系相似性的PPI识别主要分成三个模块:收集关系描述、关系表示、关系相似性计算。
1.2.1 关系描述
对于目标蛋白质对,首先利用PubMed数据库提供的接口收集同时包含目标蛋白质对中两个蛋白质的所有句子,将这个句子集合作为目标蛋白质对的签名档。每个签名档都较完整地描述了目标蛋白质对中两个蛋白质之间的关系。
1.2.2 关系表示
以一元词作为特征,采用向量空间模型表示蛋白质对(protein1,protein2)的关系,向量的每一维对应一个特征单词。将签名档中所有的句子去除停止词、单字符单词和数字,选择至少在25篇签名档中出现的单词作为特征。最终得到了4 867个特征,用这些特征单词表示蛋白质对。采用二值权重(0/1)表示特征权重,即特征单词出现时权值为1,不出现权值为0。
1.2.3 关系相似性计算
(1)
根据目标蛋白质对与已知交互关系蛋白质对的相似度,选择合适的分类算法进行PPI识别。
2 基于Minimum Cuts的PPI识别
目前只存在少量可用于监督学习的标注数据集,因而如何有效利用已有的标注数据至关重要。鉴于此,直接以现有PPI数据库为训练数据,提出了基于Minimum Cuts的PPI识别方法。基于Minimum Cuts的分类方法可综合两方面对PPI关系进行判断:对单个目标蛋白质对关系进行初步判断;利用蛋白质对之间的相似性约束判断结果。
2.1 Minimum Cuts算法描述
首先,对目标分类问题建立无向图G=(V,E),源点s和汇点t表示C1和C2两个目标类。图中的节点v1,v2,…,vn表示训练集和测试集中所有的实例x1,x2,…,xn。图G中的边可分E=E(vi,s)∪E(vi,t)∪E(vi,vj),其中E(vi,s)与E(vi,t)表示任意实例点vi与目标类C1、C2之间的关系,边E(vi,s)的权重表示实例xi为C1类的概率大小,E(vi,t)的权重表示实例xi为C2类的概率大小。E(vi,vj)表示实例xi与xt之间是相似的,其权重表示两实例之间的相似度,相似度越大权重越大,相似度越小权重越小。例如,图1是一个Minimum Cuts分类器示意图,s和t分别表示C1和C2类,Y表示训练数据中属于C1的实例,N表示训练集中属于C2的实例,M表示待标记的实例。虚线经过的边就是图1的最小割,并将M与Y分为C1。
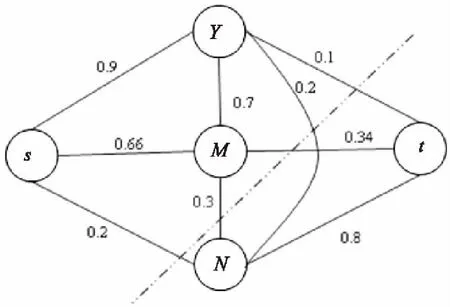
图1 Minimum Cuts分类器
基于Minimum Cuts的分类算法,通过求解无向图的割Cut(S,T)将权值小的边移除,实现实例数据的分类。其中Cut(S,T)将V划分为S和T=V-S两部分,源点s∈S,汇点t∈T。weight(u,v)表示割边的权重,所有割边的权重和为W(S,T)。图G的最小割,就是使得W(S,T)取最小值时的Cut(S,T)。
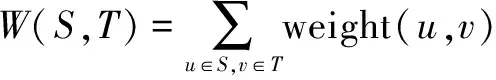
(2)
2.2 基于关系相似性的PPI识别与Minimum Cuts的联系
基于Minimum Cuts的分类器可以通过无向图中边的权值表示实例间的相似度大小。在求无向图的最小割时,将权值较小的边移除,即将相似度小的实例分开,完成了实例分类。
基于关系相似性的PPI识别通过比较目标蛋白质对与已知关系的蛋白质对的相似性来判断目标蛋白质对的交互作用,将相似度大的关系分为一类。这和Minimum Cuts分类算法的核心思想一致。并且PPI识别的目标类为两个(有交互关系和无交互关系),符合Minimum Cuts模型。
因此,在关系相似性框架下,构建Minimum Cuts分类模型,实现PPI识别。
2.3 构建Minimum Cuts分类器
为了建立Minimum Cuts分类器进行PPI识别,建立了无向图表示蛋白质对与目标类(有交互关系、无交互关系)以及蛋白质对与蛋白质对之间的关系。最终通过求解图的最小割,将蛋白质对进行分类,实现PPI识别。
2.3.1 实例点与s、t之间边的权重计算
在交互关系识别问题中,以图1为例,其中s和t分别表示两个目标类,即存在交互关系和不存在交互关系。Y表示训练集中属于有交互关系的一个蛋白质对,N表示训练集中不存在交互关系的一个蛋白质对,M表示待标记的蛋白质对。Y、M和N与s、t之间的边,表示实例与目标类(有交互关系、无交互关系)之间的关系。边的权值表示一个实例为有交互关系、无交互关系的概率值大小。采用SVM算法预测蛋白质对是有交互关系和无交互关系,得到概率值作为相应边的权重。
将训练集中有交互关系的点与s之间边的权值设为100,与t之间边的权值设为0.01。无交互关系的点与s之间的边的权值设为0.01,与t之间的边权值设为100。
2.3.2 实例点之间边的权重计算
实例点之间的边表示两个蛋白质对关系之间是相似的,两个蛋白质对关系相似度值越大,边权重越大。根据表示蛋白质对关系的特征向量,采用余弦距离计算任意两个蛋白质对之间的相似度,余弦值越大权重越大,两个蛋白质对越相似,相应的实例点之间边的权重也越大。
3 实 验
3.1 实验数据
实验中,将有交互关系的蛋白质对作为正类,无交互关系的蛋白质对作为负类。正类蛋白质对来自生物医学专家手工收集信息建立的PPI数据库HPRD,共1 420对。而对于负类,根据HPRD中包含的蛋白质采用随机组合的方法产生负类蛋白质对(删除HPRD已包含的蛋白质对),最后只保留被PubMed数据库中文献记载的蛋白质对作为无交互蛋白质对的训练集,共有1 353对。因此,实验数据集中共包含2 773对蛋白质对。
3.2 实验设置
SVM已被证实为一种非常有效的分类算法,是基于机器学习的蛋白质交互关系识别所采用的重要分类模型。实验部分共包括两个部分:基于SVM的PPI识别和基于Minimum Cuts的PPI识别。
在基于Minimum Cuts的PPI识别中,首先计算实例点对与s、t之间边的权重,即蛋白质对为有交互关系或无交互关系的概率值大小,然后计算实例点之间边的权重。采用SVM算法获得无向图中未标记蛋白质对的分类概率(LIBSVM[18])。在采用余弦距离计算相似度时,当两个蛋白质对间的相似度值小于0.2时,认为两个蛋白质对不相似,在构建无向图时两个点之间不设边。
实验中,采用五折交叉验证的方法进行验证,将全部数据分为五份,每次以其中一份作为测试数据。对同一份测试数据,分别选择了总数的5%、10%、20%、40%、60%和80%的蛋白质对作为训练数据进行实验。然后,将五组测试数据的识别结果的平均值作为一次识别的结果。

3.3 实验结果及分析
表1为基于监督学习的Minimum Cuts的PPI识别结果。表2为SVM分类器的PPI识别结果。表中的第一列序号1,2,…,6依次对应训练数据为5%,10%,20%,40%,60%和80%的分组。图2、图3是对比采用Minimum Cuts分类器与SVM分类器,正类蛋白质对和负类蛋白质对识别结果的F值的变化。

表1 基于监督学习Minimum Cuts的识别结果 %
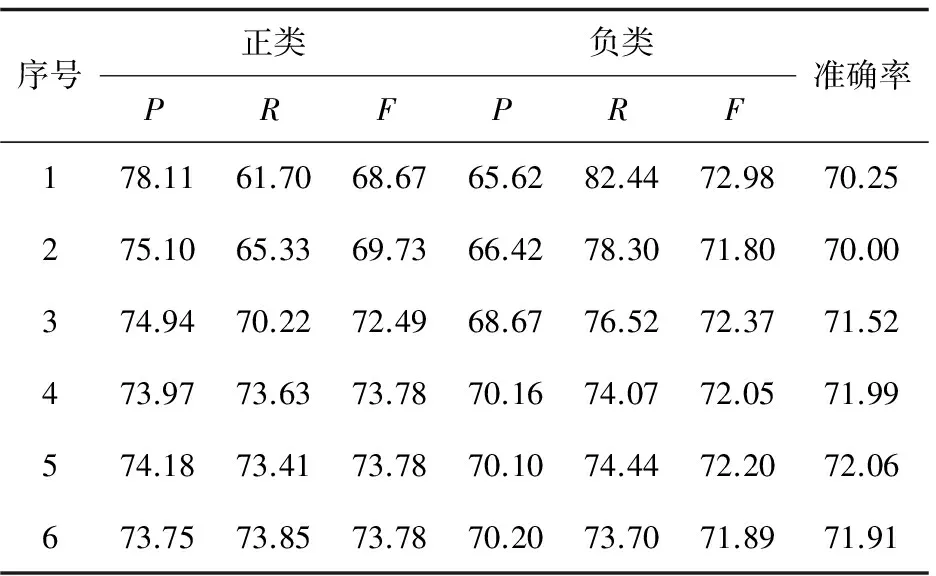
表2 基于SVM分类器的识别结果 %
表1和表2的实验结果说明,对于两种分类器,当训练数据较少时,正类蛋白质对识别结果的召回率较低。随着训练数据的增多,正类蛋白质对的召回率逐渐提高。对SVM分类器,准确率随训练数据增加变化不大,而Minimum Cuts分类器的正、负类的F值及准确率随训练数据增加都有明显提高。说明Minimum Cuts有效利用了蛋白质对签名档相似性进行分类。
负类蛋白质对精确度也逐渐提高,整体的识别准确率也明显提升。实验结果表明,Minimum Cuts分类器的整体识别结果比SVM分类器要好。当训练数据达到80%时,Minimum Cuts分类器识别的正类F值为76.90%,负类F值为76.13%,整体准确率为75.56%,相对SVM分类器识别的正类F值提高了3.12%,负类F值提高了4.24%,整体准确率提高了3.65%。

图2 有交互关系的蛋白质识别结果的F值变化趋势对比
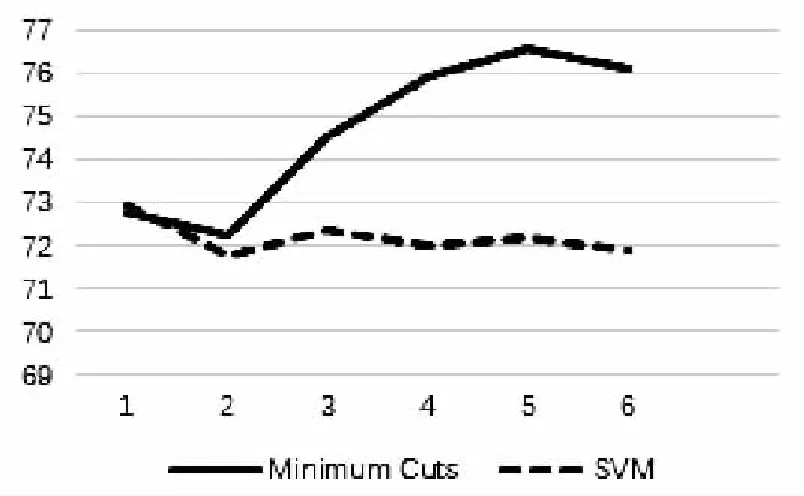
图3 无交互关系的蛋白质识别结果的F值变化趋势对比
从图2、图3中可以看出,采用不同比例的训练数据,基于Minimum Cuts分类器的PPI识别,它的正、负类蛋白质对识别结果的F值都优于SVM分类器。当训练数据为20%时,Minimum Cuts分类器的识别结果优于训练数据为80%时的SVM分类器的识别结果。实验结果表明,Minimum Cuts分类器能更有效地利用标注数据进行PPI识别。
4 结束语
为充分利用已有的PPI数据库实现蛋白质交互识别,降低人工标注的代价,在大规模语料库的基础上,提出了关系相似性框架下基于Minimum Cuts的PPI识别方法。通过构建无向图,不仅对目标蛋白对是否存在交互关系进行了初步判断,还表示了蛋白质对与蛋白质对之间的相似性。通过蛋白质对之间的相似性约束判断结果,用较少的训练数据取得了优于SVM的精度,有效缓解了PPI识别问题中标注数据缺乏带来的困扰。
[1] Prasad T S K,Goel R,Kandasamy K,et al.Human protein reference database-2009 update[J].Nucleic Acids Research,2009,37:767-772.
[2] Kerrien S, Alam-Faruque Y, Aranda B,et al.IntAct-open source resource for molecular interaction data[J].Nucleic Acids Research,2007,35:561-565.
[3] Ceol A,Aryamontri A C,Licata L,et al.MINT,the molecular interaction database:2009 update[J].Nucleic Acids Research,2010,38:532-539.
[4] U.S.National Library of Medicine.PubMed[EB/OL].2000.http://www.ncbi.nlm.nih.gov/pubmed/.
[5] Bunescu R,Mooney R,Ramani A,et al.Integrating co-occurrence statistics with information extraction for robust retrieval of protein interactions from Medline[C]//Proceedings of the workshop on linking natural language processing and biology:towards deeper biological literature analysis.[s.l.]:Association for Computational Linguistics,2006:49-56.
[6] Koike A,Kobayashi Y,Takagi T.Kinase pathway database:an integrated protein-kinase and NLP-based protein-interaction resource[J].Genome Research,2003,13(6a):1231-1243.
[7] 杨志豪,洪 莉,林鸿飞,等.基于支持向量机的生物医学文献蛋白质关系抽取[J].智能系统学报,2008,3(4):361-369.
[8] 崔宝今,林鸿飞,张 霄.基于半监督学习的蛋白质关系抽取研究[J].山东大学学报:工学版,2009,39(3):16-21.
[9] Grimes G R,Wen T Q,Mewissen M,et al.PDQ Wizard:automated prioritization and characterization of gene and protein lists using biomedical literature[J].Bioinformatics,2006,22(16):2055-2057.
[10] Fundel K,Küffner R,Zimmer R.RelEx-relation extraction using dependency parse trees[J].Bioinformatics,2007,23(3):365-371.
[11] Temkin J M,Gilder M R.Extraction of protein interaction information from unstructured text using a context-free grammar[J].Bioinformatics,2003,19(16):2046-2053.
[12] Qian W,Fu C,Cheng H.Semi-supervised method for extraction of protein-protein interactions using hybrid model[C]//Proceedings of the 2013 third international conference on intelligent system design and engineering applications.[s.l.]:IEEE Computer Society,2013:1268-1271.
[13] Niu Y,Otasek D,Jurisica I.Evaluation of linguistic features useful in extraction of interactions from PubMed;application to annotating known,high-throughput and predicted interactions in I2D[J].Bioinformatics,2010,26(1):111-119.
[14] Haussler D.Convolution kernels on discrete structures[R].Santa Cruz:University of California at Santa Cruz,1999.
[15] Lodhi H,Saunders C,Shawe-Taylor J,et al.Text classification using string kernels[J].Journal of Machine Learning Research,2002,2(3):419-444.
[16] Bunescu R C,Mooney R J.A shortest path dependency kernel for relation extraction[C]//Proceedings of the conference on human language technology and empirical methods in natural language processing.[s.l.]:Association for Computational Linguistics,2005:724-731.
[17] Niu Yun,Wang Yuwei.Protein-protein interaction identification using a hybrid model[J].Artificial Intelligence in Medicine,2015,64(3):185-193.
[18] Chang C C,Lin C J.LIBSVM:a library for support vector machines[J].ACM Transactions on Intelligent Systems and Technology,2011,2(3):1-27.
Identification of Protein-protein Interaction with Minimum Cuts
ZHANG Jing,WU Hong-mei,NIU Yun
(School of Computer Science and Technology,Nanjing University of Aeronautics and Astronautics,Nanjing 210016,China)
Protein-Protein Interaction (PPI) information is significant for biological and medical research,and is an important content in biomedicine field.The recognition of PPI with large-scale corpus can significantly reduce the cost of manual annotation by directly using the existing PPI database.Therefore,a method for PPI with Minimum Cuts based on the large-scale corpus has been proposed.Based on the framework of relational similarity,Minimum Cuts classifier not only uses SVM to predict the classification initially of a single protein,but also makes use of the similarity between the protein pairs to determine the results which are more accurate.The experimental results show that the Minimum Cuts classifier is better than the SVM classifier for the recognition of PPI.When the training data is 20%,the recognition results of the Minimum Cuts classifier gets better performance than that of an SVM classifier trained with 80%.
relational similarity;Minimum Cuts;SVM;protein-protein interaction
2016-07-20
2016-10-26 网络出版时间:2017-04-28
国家自然科学基金资助项目(61202132)
张 景(1991-),女,硕士研究生,研究方向为自然语言处理;牛 耘,博士,副教授,研究方向为自然语言处理。
http://kns.cnki.net/kcms/detail/61.1450.TP.20170428.1703.074.html
TP391
A
1673-629X(2017)06-0017-05
10.3969/j.issn.1673-629X.2017.06.004

