野生大麻居群遗传多样性ISSR分析的取样策略
张庆滢 郭蓉 陈璇 许艳萍 郭孟璧 郭鸿彦 杨明


摘要:【目的】分析野生大麻居群的合理取樣样本量,为全面了解我国大麻资源的遗传多样性及制定野生大麻保护策略提供参考依据。【方法】随机抽取96个辽宁科尔沁沙地野生大麻自然居群个体,采用计算机模拟方法从中随机抽取不同样本量(分别为5、10、15、20、25、30、35、40、50、60、70、80、90和96个个体)的抽样群体各20次,利用ISSR分子标记对其遗传多样性进行分析,确定合理的取样样本量。【结果】利用从60条ISSR引物中筛选出的9条引物对随机抽取的96个野生大麻个体进行PCR扩增,共获得76个位点,其中多态位点48个,多态率达63.16%,观测等位基因数(Na)为1.632,有效等位基因数(Ne)为1.250,Neis基因多样性指数(H)为0.156,Shannons信息指数(I)为0.245。由各遗传参数拟合曲线变化趋势分析结果可知,初始的4种抽样群体(分别为5、10、15和20个个体)遗传多样性水平呈急速增加趋势,之后随着样本量的加大,遗传多样性水平增加趋势明显变缓。当抽样群体样本量超过25个个体时,Na、Ne、H和I均包含了野生大麻居群90%以上的遗传变异。【结论】采用ISSR分子标记评估野生大麻群体遗传多样性时,群体取样样本量不宜小于25个个体才能较好地反映该居群总体的遗传多样性水平。
关键词: 野生大麻;ISSR;遗传多样性;取样策略
中图分类号: S563.3 文献标志码:A 文章编号:2095-1191(2017)06-0973-06
Sampling strategy for genetic diversity ISSR analysis of wild
Cannabis sativa L. population
Abstract:【Objective】Reasonable sampling size for wild Cannabis sativa L. was analyzed and determined to provide reference for comprehensive understanding of genetic diversity of domestic C. sativa L. resources and formulating protection strategies for it. 【Method】Ninety-six representative individuals of wild C. sativa L. population from Horqin sandy land were randomly selected. Using computer simulation method, each sampling group with different individuals(5,10, 15,20,25,30,35,40,50,60,70,80,90,96) were randomly sampled 20 times. ISSR molecular marker method was carried out to estimate the genetic diversity and determine the proper sampling size. 【Result】PCR amplification was conducted on 96 sampled C. sativa L. individuals by 9 primers screened out of 60 ISSR primers. The results showed that 76 loci with 48 polymorphic loci were identified, and polymorphic rate was 63.16%. Observed average alleles(Na) number was 1.632, effective alleles(Ne) number was 1.250, Neis gene diversity index(H) was 0.156,and Shannons information index(I) was 0.245. Fitted curves of different genetic parameters indicated that the genetic diversity increased sharply for the first four sampling groups(5,10,15 and 20 individuals respectively). As sampling size enlarged, the increasing trend of genetic diversity was slowed. When the sampling size was over 25 individuals, Na,Ne,H and I contained over 90% of genetic diversity of wild C. sativa L population. 【Conclusion】The sampling size should be no less than 25 individuals when using ISSR molecular marker to evaluate genetic diversity of C. sativa L.,as it can reflect the genetic diversity of the population.
Key words: wild Cannabis sativa L.; ISSR; genetic diversity; sampling strategy
0 引言
【研究意义】大麻(Cannabis sativa L.)是世界上最古老、分布最广泛的栽培作物之一,为一年生草本植物,通常为雌雄异株。大麻在我国种植历史悠久,是我国传统的经济作物,也因其含有具致幻成瘾作用的四氢大麻酚(THC)而被联合国禁毒公约列为毒品源植物之一。近年来,由于工业大麻在造纸、纺织、食用及药用等方面具有突出优势,在高附加值原料产品大麻二酚(CBD)的开发利用和生态保护方面也具有多种优势(如抗病虫草害性能强、对重金属的高吸附作用、可用作环保新材料和农业碳汇等)(张庆滢等,2011;Amaducci et al.,2015),在国内外已成为倍受关注的热点经济作物。野生大麻资源作为大麻研究与利用的天然基因库和宝贵资源,保存着栽培大麻不具备或已消失的遗传基因,是培育低THC含量、优质、高产、抗病(虫)及抗逆大麻新品种的重要物质基础。我国是大麻的重要起源地之一(Gilmore et al.,2007),拥有丰富的大麻资源,但由于人口压力、生境恶化及禁毒铲除等因素影响,野生大麻面临资源严重散失的窘境。因此,全面了解我国大麻资源的遗传多样性,对制定野生大麻保护策略具有重要意义。【前人研究进展】近年来,RAPD、ISSR、AFLP、SRAP和SSR等分子标记技术在种质资源遗传多样性、遗传图谱构建、群体遗传分析等方面已得到广泛应用(倪先林等,2015;肖政等,2015;周娜等,2015)。Hakki等(2007)、Kayis等(2010)在利用RAPD、ISSR分子标记对毒品大麻和工业大麻进行鉴别、毒品来源地追溯等方面均获得了理想效果。汤志成等(2013)利用RAPD分子标记对我国12份野生大麻资源进行了遗传多样性评价,但未涉及对群体内部遗传变异和结构等内容。Gao等(2014)利用SSR分子标记成功将115份大麻栽培品种分成四大类群。宗成堃等(2015)研究认为,联合利用SSR、SRAP和ISSR 3种分子标记技术可有效地构建高密度、高饱和性丹参分子遗传图谱。张影波等(2016)研究认为,SRAP和AFLP标记技术均可用于艾纳香遗传多样性分析。【本研究切入点】进行群体遗传多样性分析时需首先解决代表某一群体总体遗传多样性水平的样本采集问题,但目前针对野生大麻自然居群或大麻群体研究样本量选择的研究未见报道。【拟解决的关键问题】采用ISSR分子标记方法分析辽宁科尔沁沙地野生大麻自然居群的遗传多样性,通过随机抽取不同样本量和进行计算机模拟,确定可代表居群总体遗传多样性水平的样本量,为开展野生大麻群体遗传和保护利用研究提供参考依据。
1 材料与方法
1. 1 试验材料
以辽宁科尔沁沙地的野生大麻自然居群為研究对象,在居群约4 km2范围内随机选取96个野生大麻单株,采集样本时选取干净、无病虫害的正常叶片,及时用硅胶进行干燥,进行总DNA提取并保存于-80 ℃冰箱中备用。取样时取样植株样本间的最小直线距离不低于10 m,96个样本尽量覆盖4 km2的取样范围。
1. 2 试验方法
1. 2. 1 DNA提取 试验采用CTAB法(Doyle,1991)提取大麻基因组DNA。由于大麻叶片含有大量酚类物质,为获得较高纯度的基因组DNA,对该方法进行改良,增加Buffer洗涤(200 mmol/L Tris-Cl,250 mmol/L NaCl,50 mmol/L EDTA)步骤以去除酚类等杂质(陈璇等,2015)。对96 份单株材料进行总DNA提取,经0.8%琼脂糖凝胶电泳及分光光度计检测后保存备用。
1. 2. 2 引物筛选及PCR反应条件 以试验居群的8个野生大麻植株单株基因组DNA为模板,对60条ISSR引物进行筛选,其中18条来自Hakki等(2007)的研究,4条来自Kojoma等(2002)的研究,38条随机选自英国哥伦比亚大学ISSR引物试剂盒。同时对扩增体系进行优化,对各引物进行退火温度梯度(47、50、53、56、58和60 ℃)PCR扩增试验,以确定每条引物的相应退火温度。优化后的PCR反应体系25.0 μL,包含1.5 μL引物(母液浓度10 μmol/L),2.0 μL dNTP(母液浓度10 mmol/L),2.0 μL DNA模板(25~30 ng/μL),0.3 μL Taq DNA聚合酶(5 U/μL),2.5 μL 10×Buffer(10 mmol/L Tris-HCl,pH 8.3,50 mmol/L KCl),以ddH2O补足至25.0 μL。扩增程序:94 ℃预变性4 min;95 ℃ 45 s,47~60 ℃退火(根据不同引物有所不同)50 s,72 ℃ 90 s,进行38个循环;72 ℃延伸10 min。PCR产物在0.5倍TBE缓冲液中用1.8%琼脂糖凝胶电泳,电压4 V/cm。每条引物2次重复扩增。
1. 3 数据处理
以Trans2K PlusⅡ DNA Marker(100~8000 bp)为相对分子量标准,按照扩增DNA片段的有(1)或无(0)构建ISSR数据矩阵。采用计算机随机抽样,从科尔沁沙地野生大麻居群96个单株总体样本中形成由5、10、15、20、25、30、35、40、50、60、70、80、90和96个个体组成的14个抽样群体,每个抽样群体各20次重复,将获得的[0,1]数据矩阵在POPGENE 1.32中进行分析,计算用于评价居群遗传多样性的相关指标,包括观测等位基因数(Na)、有效等位基因数(Ne)、Neis基因多样性指数(H)、Shannons信息指数(I)和多态位点百分率(P)。
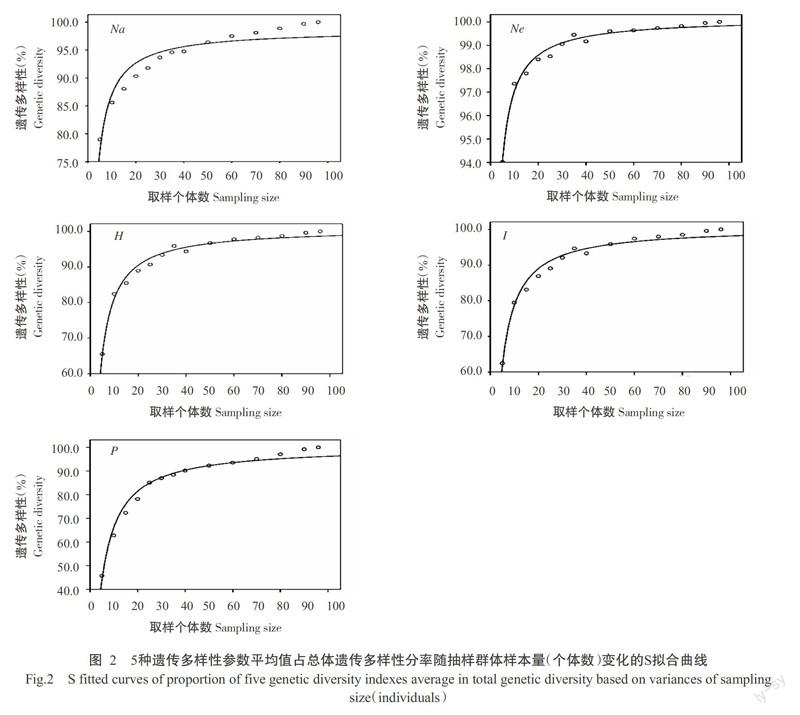
计算不同抽样群体20次重复的遗传多样性参数平均值,并将各平均值转换为占总体遗传多样性参数值的百分比,用SPSS 17.0中的S曲线模型对抽样群体的遗传多样性水平随样本量增加而增加的趋势进行拟合分析,拟合公式为lnY=b0+(b1/t),其中b0和b1为拟合参数,t为样本量,Y为各抽样群体的遗传参数百分比。
将5种遗传参数值占居群总体遗传多样性的百分比与样本量的5条拟合曲线用SPSS 17.0进行协方差分析(ANCOVA),检验截距和斜率的差异,分析Na、Ne、H、I和P 的拟合曲线差异显著性。
2 结果与分析
2. 1 引物筛选结果
根据PCR扩增结果,基于扩增条带数目适中(6~20条条带)、清晰度高及重复性好等原则,最终优选出9条适合野生大麻群体遗传多样性分析的ISSR引物,并确定其最佳退火温度(表1)。其中,1~6号引物来自Hakki等(2007)的研究,7~9号引物筛选自英国哥伦比亚大学ISSR引物试剂盒。图1为9条ISSR引物对科尔沁野生大麻居群第1~10号植株个体样本的扩增结果。
2. 2 群体遗传多样性分析结果
使用筛选出的9条ISSR引物对科尔沁沙地野生大麻居群96个个体进行扩增,在DNA片段长度250~ 2500 bp间共检测出76个清晰、可重复的有效位点,其中48个位点为多态位点,占63.16%。由表2可知,14个抽样群体遗传参数的变化范围:Na为1.289~1.632,Ne为1.176~1.250,H为0.102~0.156,I为0.153~0.245,P为28.881%~63.160%。依据对96个个体的计算机抽样及计算结果(表2),各抽样群体的遗传多样性参数随着样本量的增加而增加,而标准差随着样本量的增加而减少。
2. 3 抽样样本量与遗传多样性参数的关系
对14个抽样群体样本量与其遗传多样性参数(Na、Ne、H、I和P)占总体遗传多样性参数的百分比进行S曲线拟合分析,结果显示,其R2分别为0.911、0.986、0.988、0.984和0.980,拟合度较好。当抽样群体样本量为20个个体时,Na达90.3%;当抽样群体样本量为25个个体时,Na达91.7%,Ne达98.6%,P达85.0%,H和I分别达90.4%和89.8%。从各遗传参数拟合曲线(图2)可看出,初始的4种抽样群体(5、10、15和20个个体)遗传多样性水平呈急速增加趋势,之后随着样本量的加大,遗传多样性水平的增加趋势明显变缓。当样本量达25个个体时,Na、Ne、H和I均超过总体样本遗传多样性的90.0%,P也达到总体样本遗传多样性的85.0%。
对5条拟合曲线用SPSS 17.0进行协方差分析(ANCOVA),通过检验截距与斜率的差异,发现3种遗传多样性参数(Na,He和I)的擬合曲线间差异不显著 (α=0.05水平)。
3 讨论
取样策略是生物多样性有效保护和利用研究面临的最基本问题,关系到植物总体和各居群遗传多样性评价的正确性和估算的准确性,对制定有效的保护及利用策略意义重大(赵茹等,2006)。在生物多样性研究中,H和I是两个最常用的遗传多样性评价参数(李钰莹和董宽虎,2014;李绍臣等,2016;王梦亮等,2016)。本研究采用ISSR分子标记对抽样群体进行分析,所获得的H和I均在取样样本量为25个个体时达到总体样本遗传多样性的90.0%,在样本量超过25个个体后,H和I两个遗传多样性参数随样本量的增加呈微幅上升,而在样本量达到25个个体前,5个遗传多样性参数均呈快速上升趋势。由此判断,ISSR分子标记在样本量不小于25个个体时才能代表总体90%以上的遗传变异。
野生大麻在我国云南、西藏、内蒙古、新疆和东北等地区均有分布,这些地区的野生大麻是我国大麻种质资源的重要组成部分。合理取样是野生大麻群体遗传学研究的前提,在很大程度上受大麻植株自身生物学特性、分布范围、生态环境及取样目的等因素的影响。通常认为植物居群水平上遗传多样性受繁育系统因素的影响最大,风媒传粉异交>动物传粉异交>混交>自交,居群内的杂合程度越高,等位基因在个体间的分布越均匀,较少的个体即可代表整个居群的遗传变异(金燕和卢宝荣,2003)。陈坚等(2015)对异花授粉、种子繁殖植物紫云英进行SSR分子标记取样量研究,认为取样30个个体时遗传多样性指数达最佳值。谭龙涛等(2012)对中苎1号(苎麻为雌雄同株异花植物,兼有无性繁殖和种子繁殖)的研究结果表明,SSR分子标记在中苎1号样本量达50个以上个体时遗传参数值变化不明显,而SRAP分子标记在中苎1号样本量达60个以上个体时结果才较稳定。从上述研究结果可看出,不同的植物由于其生物学特性不一样,在进行群体遗传学研究时,要求抽取的样本量通常不同。就大麻而言,由于具有雌雄异株、异花授粉、风媒等特性,其群体内个体间差异较明显,基因杂合程度高,与自花授粉植物或无性繁殖植物相比,在进行遗传多样性评估时其取样量可相对较小。
大麻通常为雌雄异株(在人工选择压力下可形成雌雄同株品种)植物,具异花授粉、风媒、种子繁殖和一年生等生物学特性。雌雄异株、异花授粉植物群体内遗传多样性丰富,个体间差异较明显,基因杂合程度高,与自花授粉植物或无性繁殖植物相比,在对遗传多样性评估时其取样量相对较小。
4 结论
采用ISSR分子标记评估野生大麻群体的遗传多样性时,群体取样样本量不宜小于25个个体才能较好地反映该居群总体的遗传多样性水平。
参考文献:
陈坚,林新坚,钟少杰. 2015. 取样策略对SSR标记鉴别紫云英品种能力的影响[J]. 植物遗传资源学报,16(6):1249- 1256. [Chen J,Lin X J,Zhong S J. 2015. Effect of sampling strategy on identification of cultivars of Astragalus sinicus L. by SSR marker[J]. Journal of Plant Genetic Resources,16(6):1249-1256.]
陈璇,郭蓉,万睿璇,许艳萍,张庆滢,郭孟璧,郭鸿彦,杨明. 2015. 云南省5个雌雄异株工业大麻群体遗传结构评价[J]. 分子植物育种,13(9):2069-2075. [Chen X,Guo R,Wan R X,Xu Y P,Zhang Q Y,Guo M B,Guo H Y,Yang M. 2015. Genetic structure of five dioecious industrial hemp varieties in Yunnan[J]. Molecular Plant Breeding,13(9):2069-2075.]
金燕,卢宝荣. 2003. 遗传多样性的取样策略[J]. 生物多样性,11(2):155-161. [Jin Y,Lu B R. 2003. Sampling stra-
tegy for genetic diversity[J]. Biodiversity Science,11(2):155-161.]
李紹臣,李凤明,张立民,任军,林玉梅. 2016. 吉林省天然黄檗种群遗传多样性ISSR分析[J]. 生态学报,36(13):4006-4012. [Li S C,Li F M,Zhang L M,Ren J,Lin Y M. 2016. Analysis of genetic diversity in wild populations of Phellodendron amurense Rupr. in Jilin Province using inter simple sequence repeat[J]. Acta Ecologica Sinica,36(13):4006-4012.]
李钰莹,董宽虎. 2014. 山西白羊草种质资源遗传多样性的ISSR分析[J]. 草业学报,23(1):217-222. [Li Y Y,Dong K H. 2014. ISSR analysis of genetic diversity of germplasm resources of Bothriochloa ischaemum in Shanxi[J]. Acta Prataculturae Sinica,23(1):217-222.]
倪先林,赵甘霖,刘天朋,胡炯凌,李元,陈国民,汪小楷,丁国祥. 2015. SSR分子标记在糯高粱种质资源遗传多样性分析中的应用[J]. 江苏农业学报,31(1):16-22. [Ni X L,Zhao G L,Liu T P,Hu J L,Li Y,Chen G M,Wang X K,Ding G X. 2015. Genetic diversity analysis of glutinous sorghum germplasm by simple sequence repeat[J]. Jiangsu Journal of Agricultural Sciences,31(1):16-22.]
谭龙涛,喻春明,陈平,王延周,陈继康,温岚,熊和平. 2012. 样本量对中苎1号群体遗传多样性参数的影响[J]. 中国麻业科学,34(4):179-183. [Tan L T,Yu C M,Chen P,Wang Y Z,Chen J K,Wen L,Xiong H P. 2012. Effects of sample size on genetic diversity parameters for “Zhong Zhu No.1” group[J]. Plant Fiber Sciences in China,34(4):179-183.]
汤志成,陈璇,张庆滢,郭鸿彦,杨明. 2013. 野生大麻种质资源表型及其RAPD遗传多样性分析[J]. 西部林业科学,42(3):62-66. [Tang Z C,Chen X,Zhang Q Y,Guo H Y,Yang M. 2013. Genetic diversity analysis of wild Cannabis in China based on morphological characters and RAPD markers[J]. Journal of West China Forestry Science,42(3):62-66.]
王梦亮,任晓琳,崔晋龙,王俊宏. 2016. 野生红景天的RAPD和ISSR遗传多样性分析[J]. 中草药,47(3):469-473. [Wang M L,Ren X L,Cui J L,Wang J H. 2016. Genetic diversity of wild plants in Rhodiola L. with two molecular marker methods of RAPD and ISSR[J]. Chinese Traditional and Herbal Drugs,47(3):469-473.]
肖政,苏家乐,刘晓青,李畅,何丽斯,陈尚平. 2015. 基于ISSR标记的杜鹃花种质资源遗传多样性分析[J]. 江西农业学报,27(1):6-10. [Xiao Z,Su J L,Liu X Q,Li C,He L S,Chen S P. 2015. Analysis of genetic diversity of Rhododendron germplasm resources based on ISSR markers[J]. Acta Agriculturae Jiangxi,27(1):6-10.]
张庆滢,郭鸿彦,杨明. 2011. 加拿大工业大麻生产贸易概况及科研进展[J]. 中国麻业科学,33(6):302-306. [Zhang Q Y,Guo H Y,Yang M. 2011. Production,trade and research progress on hemp in Canada[J]. Plant Fiber Sciences in China,33(6):302-306.]
张影波,袁媛,庞玉新,王丹,胡璇. 2016. 艾纳香遗传多样性的SRAP和AFLP对比分析[J]. 南方农业学报,47(8):1261-1267. [Zhang Y B,Yuan Y,Pang Y X,Wang D,Hu X. 2016. Comparative analysis of SRAP and AFLP marker for genetic diversity of Blumea balsamifera D C.[J]. Journal of Southern Agriculture,47(8):1261-1267.]
赵茹,程舟,陆伟峰,卢宝荣. 2006. 基于分子标记的野生大豆居群遗传多样性估算与取样策略[J]. 科学通报,51(9):1042-1048. [Zhao R,Cheng Z,Lu W F,Lu B R. 2006. Genetic diversity analysis and sampling strategy of Glycine soja population on molecular marker[J]. Chinese Science Bulletin,51(9):1042-1048.]
周娜,李丹丹,陶伟林,沈虹,罗云米,汝学娟,柳李旺. 2015. 萝卜种质遗传多样性的ISSR,RAPD与RAMP分析[J]. 西南农业学报,28(2):704-712. [Zhou N,Li D D,Tao W L,Shen H,Luo Y M,Ru X J,Liu L W. 2015. Genetic diversity analysis in radish(Raphanus sativus) germplasm with ISSR,RAPD and RAMP[J]. Southwest China Journal of Agricultural Sciences,28(2):704-712.]
宗成堃,宋振巧,陈海梅,刘昶,王建华,郭林林,刘甜,潘玉玲. 2015. 利用SSR、SRAP和ISSR分子标记构建首张丹参遗传连锁图谱[J]. 药学学报,50(3):360-366. [Zong C K,Song Z Q,Chen H M,Liu C,Wang J H,Guo L L,Liu T,Pan Y L. 2015. Construction of the first genetic linkage map of Salvia miltiorrhiza Bge. using SSR,SRAP and ISSR markers[J]. Acta Pharmaceutica Sinica,50(3):360-366.]
Amaducci S,Scordia D,Liu F H,Zhang Q Y,Guo H Y,Testa G,Cosentino S L. 2015. Key cultivation techniques for hemp in Europe and China[J]. Industrial Crops and Pro-
ducts,68(1):2-16.
Doyle J J. 1991. DNA Protocols for Plants-CATB Isolation[M]. Berlin:Springer-Verlage.
Gao C H,Xin P F,Cheng C H,Tang Q,Chen P,Wang C B,Zang G G,Zhao L N. 2014. Diversity analysis in Cannabis sativa based on large-scale development of expressed sequence tag-derived simple sequence repeat markers[J]. PLoS One,9(10):e110638.
Gilmore S,Peakall R,Robertson J. 2007. Organelle DNA haplotypes reflect crop-use characteristics and geographic origins of Cannabis sativa[J]. Forensic Science International,172(2-3):179-190.
Hakki E E,Kayis S A,Pinarkara E,Sag A. 2007. Inter simple sequence repeats separate efficiently hemp from marijuana (Cannabis sativa L.)[J]. Electronic Journal of Biotechno-
logy,10(4):570-581.
Kayis S A,Hakki E E,Pinarkara E. 2010. Comparison of effectiveness of ISSR and RAPD markers in genetic characterization of seized marijuana(Cannabis sativa L.) in Turkey[J]. African Journal of Agricultural Research,5(21):2925-2933.
Kojoma M,Iida O,Makino Y,Sekita S,Satake M. 2002. DNA fingerprinting of Cannabis sativa using inter-simple sequence repeat(ISSR) amplification[J]. Planta Medica,68(1):60- 63.
(責任编辑 思利华)

