南海冷泉区贻贝(Bathymodiolus platifrons)附生菌的分离培养与多样性分析
郭文捷, 赵 瑾, 齐宏涛, 姜 鹏, 李富超, 李荣贵
南海冷泉区贻贝()附生菌的分离培养与多样性分析
郭文捷1, 2, 3, 赵 瑾2, 3, 齐宏涛1, 姜 鹏2, 3, 李富超2, 3, 李荣贵1
(1. 青岛大学生命科学学院分子生物学实验室, 山东青岛 266071; 2. 中国科学院海洋研究所海洋生物学重点实验室, 山东青岛 266071; 3. 青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室, 山东青岛 266071)
以采集后适应培养0~24 h 以及添加甲烷和硫化钠培养24~240 h的南海冷泉区深海贻贝()为材料, 取其鳃部, 分析附生菌的多样性与适应性变化状况。共分离鉴定出贻贝附生菌270 株, 对菌株的16S rDNA进行聚类分析, 结果表明所分离菌株主要分布在4个门, 21个属, 其中变形菌的数量最多且多样性高。分析发现原位新采集的贻贝鳃部附生菌的多样性较高, 6 h后附生菌的多样性明显降低。分别添加甲烷和硫化钠对深海贻贝进行培养, 甲烷组与碳代谢有关的假单胞菌的数量逐渐增多, 且在甲烷组240 h中发现2株食烷菌属菌株。硫化钠组的芽孢杆菌属所占比例升高。此外还发现4株潜在的新种。本研究实现了深海冷泉区贻贝的实验室培养, 并对贻贝鳃部附生菌群结构进行了分析, 丰富了海洋极端环境微生物资源库, 并为深入解析贻贝与其附生菌之间的相互作用关系奠定了基础。
南海; 冷泉区; 贻贝; 附生菌; 多样性
冷泉(cold seep)是由于海底天然气渗漏, 富含水、碳氢化合物(甲烷和石油)、硫化氢等化合物的流体在大陆边缘海底喷溢形成[1], 海底冷泉的温度与周边海水温度相近。对深海冷泉生态系统的研究是继20世纪末对热液生态系统研究热潮以来的又一个重要领域[2]。冷泉区繁衍着管状蠕虫、蛤类、贻贝类、海星、海胆、海虾、珊瑚等生物[3]。这些生物的体内和体表存在大量的微生物, 微生物与宿主之间具有高度相互依赖的共附生关系[4]。共附生微生物参与宿主的物质吸收和能量代谢过程, 可为宿主提供营养物质或清除宿主体内的有害代谢产物[5], 具有多种特殊的功能基因和酶, 是一种很有潜力的生物资源。
贻贝是深海冷泉区广泛分布的海洋无脊椎动物, 属于软体动物门, 双壳纲。目前已有人对贻贝内共生菌进行了研究。Duperron等[6]利用16S rRNA基因测序和系统发育分析以及FISH的方法对墨西哥湾冷泉区贻贝内共生菌进行多样性分析, 结果表明贻贝内共生菌主要包括硫氧化菌和甲烷氧化菌。这些共生菌可以将环境中的硫化物和甲烷转化为贻贝所需要的营养物质[7-9]。但目前对贻贝附生菌的研究尚为空白, 对于共生菌群落结构与化学环境之间的关系以及响应机制也缺乏针对性的研究。
本研究首先实现了深海冷泉贻贝的实验室培养, 并对其鳃部附生菌进行了分离纯化, 通过16S rDNA 序列分析, 对分离得到的附生菌进行了多样性分析。进一步将所采集的贻贝置于不同的化学刺激条件下, 分析附生菌群落结构的变化规律。为深入解析贻贝与其附生菌之间的相互作用关系, 以及对深海新基因、新化合物的开发利用奠定了基础[5]。
1 材料与方法
1.1 样品采集和处理
样品于2014年4月“科学”号冷泉调查航次获得, 由“发现”号ROV采集。采样点位于台湾西南的南海冷泉区(22°06′57.144″N, 119°17′6.580″E), 水深1 113 m(图1[10])。贻贝取回甲板后, 放入过滤海水中, 适应0、6、12、24 h后分别取贻贝的鳃。适应24 h 后开展现场培养实验。现场培养实验分为甲烷组和硫化钠组。其中甲烷实验组每天充甲烷0.5 h, 用过滤海水培养; 硫化钠实验组母液质量浓度为25 g/L,蠕动泵按50 μL/min添加。对照组直接用过滤海水培养。现场培养24、72和240 h分别取贻贝的鳃。本研究中深海冷泉区贻贝为深海偏顶蛤()[10]。
1.2 菌株的分离与培养
在厌氧培养箱中将冻存管中贻贝的鳃用灭菌海水200mL 洗涤5次, 然后分别取50mL 均匀涂布在2216E、HM、142-A、M1四种海水培养基上, 培养基配方见表1。然后置于28℃恒温培养箱培养1~7 d。挑取不同形态单菌落至对应的固体平板, 划线纯化、保种。
1.3 DNA提取及16S rDNA序列聚类分析
DNA提取采用的是CTAB法[11]进行总DNA提取。16S rDNA的扩增参照Li等[12]的方法, 所用引物为27F(5′-AGAGTTTGATCCTGGCTCA-3′)和1492R (5′-GGTTACCTTGTTACGACTT-3′)。PCR 反应体系: Dream Taq Green PCR Master Mix(2x)(Thermo 公司), 20mL; 引物27F(10mmol/L)、1 492R(10mmol/L), 各2mL; 模板, 2mL; dd H2O补足40mL体系。PCR 反应程序: 预变性 94℃ 10 min; 之后为30个循环: 变性 94℃ 50 s, 退火55℃ 50 s, 延伸 72℃ 1 min 30 s,最后72℃, 5 min。
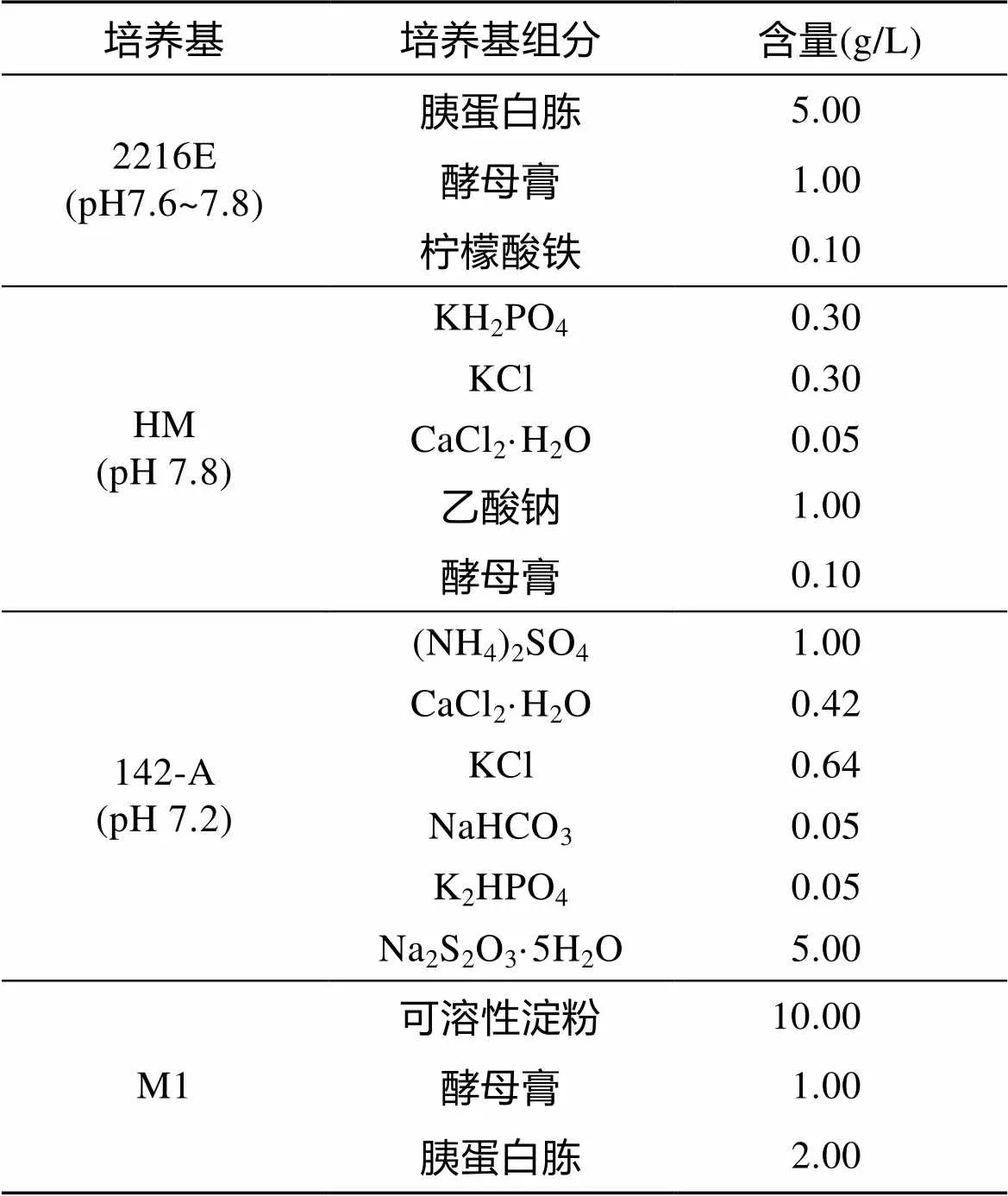
表 1 培养基的组成
PCR产物经琼脂糖凝胶电泳检测后送至上海桑尼进行16S rDNA序列测定。将得到的序列在NCBI的GenBank数据库进行Blast比对。将GenBank中获得的海洋来源的海水、沉积物以及鱼、珊瑚等动物的高同源性序列作为参考序列。然后利用Clustal X软件[13]进行比对分析, 截齐后应用MEGA 4.0[14]软件, 采用邻位相接法[15](Neighbor-Joining)构建系统进化树, 进行菌株的聚类及系统发育分析。
2 结果与分析
2.1 贻贝附生菌多样性的分析
本研究共分离得到270 株纯培养的深海贻贝附生细菌(表2)。经16S rDNA 序列聚类分析, 如图2所示, 所分离的菌株分属于变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)4个门, 21个属。
在门水平上, 变形菌门的菌株在分离菌株中占大部分(243株, 90%)。其次是厚壁菌门(21株, 8%), 放线菌门(3株, 1%), 拟杆菌门(3株, 1%)。Lampert等[16]对珊瑚可培养的共附生菌多样性的研究中, 共分离得到24株细菌, 其中γ变形菌占35%, 是珊瑚共附生菌的优势类群。Webster等[17]对海绵的34株相关共附生菌的研究中, 发现γ变形菌占41%。陈忠等[3]对海胆可培养的共附生微生物多样性的研究中, 共分离到31个菌株, 其中γ变形菌占分离菌株的大部分(20株, 65%)。本研究结果也表明γ变形菌是贻贝附生菌的优势类群, 这与其他海洋无脊椎动物的共附生细菌具有相似之处。

表 2 深海贻贝附生菌的信息表
注: 菌种编号后的数字代表同一处理组同一培养基分离的菌株数目, 只分离到一株的, “1”省略
Note: The figure behind the strain ID indicates the number of bacteria strains isolated from the same sample by using the same medium. When there is only one strain isolated, “1” is omitted
在属的水平上, 假交替单胞菌属占比例最高。假交替单胞菌属能够生存在贫营养的海洋环境中, 在多数情况下它们附生于海鞘、甲壳动物、珊瑚、无脊椎动物幼虫等表面。这种附生关系有助于我们研究微生物与宿主之间相互作用的机制[18]。
在所分离的菌株中, 发现有4个菌株(CS083、CS395、CS578、CS650)的16S rDNA序列与GeneBank中已有序列比对的相似性低于97%, 可能是新种(图2)。在这4个潜在新种中, CS083分离自适应性培养0 h组, 属于厚壁菌门, 与分离自海洋细菌的sp. B20Ydz-xm的16S rDNA 部分序列相似度最高, 相似性为96%, 可能是芽孢杆菌属的一个潜在新种。CS395、CS578和CS650均属于变形菌门。其中CS395是分离自适应性培养12 h 组, 与来自珊瑚中sp.PmeaMuc1的16S rDNA 部分序列相似度为96%; CS578是分离自现场培养实验中硫化钠24 h组, 与从海水中分离出的sp.anaA.si.2 的16S rDNA 部分序列相似度为93%; CS650菌株是分离自现场培养实验中甲烷72 h组, 它与分离自海洋细菌中的sp. ERGCD 的16S rDNA 部分序列相似度为97%。后续, 我们将参照Colwell提出的多相分类学[19]方法对这4株潜在新种进行物种鉴定, 并分析其生态功能及与贻贝之间的相互作用关系。
2.2 适应性培养过程中贻贝附生菌群落结构的变化
将深海冷泉区采集的贻贝在实验室可控条件下进行培养。原位新鲜采集的贻贝的鳃部附生菌多样性较高, 所占比例最多的门为变形菌门, 所占比例最多的属为假交替单胞菌属。适应性培养6 h后, 贻贝的鳃部附生菌的多样性明显降低(图3)。
现场培养实验的甲烷组中, 随着甲烷处理时间的延长, 假交替单胞菌属所占比例逐渐减少, 而与碳分解过程相关的假单胞菌属的比例逐渐增多[20-21]。在甲烷组240 h 中发现2株食烷菌属的菌株, 该属能够以直链和支链的烷烃为唯一的碳源和能源, 该属是海洋专性烷烃降解菌, 可以用于治理海洋中的石油污染[22]。在硫化钠组24~240 h, 海科贝特氏菌属()和芽孢杆菌属的数量逐渐增多, 而且芽孢杆菌属在硫化钠240 h组达到最多, 芽孢杆菌通过自养亚硝化和自养反硝化的脱氮作用, 将含氮有机物转化成贻贝吸收利用的小分子物质, 从而促进贻贝的生长与繁殖[23](图4)。
3 结论
本研究对南海冷泉区贻贝鳃中附生菌进行分离培养与鉴定, 初步的实验结果显示贻贝附生菌包括4个门21个属, 附生菌多样性高。其中变形菌门在所有门类中所占比例最高。在适应培养过程中, 原位新鲜采集的贻贝的鳃部附生菌多样性较高, 培养6 h后, 贻贝的鳃部附生菌的多样性明显降低。现场培养过程中, 随着甲烷处理时间的延长, 与碳分解过程相关的假单胞菌属的比例增多。在硫化钠处理组24~240 h, 海科贝特氏菌属和芽孢杆菌属的数量逐渐增多, 而且芽孢杆菌属在硫化钠240 h 组达到最多。另外, 本研究纯化得到了2株可开发用于治理海洋石油污染的食烷菌属菌株, 并发现了4株潜在的深海微生物新种, 为深海极端环境微生物新资源的发掘和利用奠了基础。
[1] 周琦, 陈建华, 张命桥, 等. 冷泉碳酸盐岩研究进展及成矿意义[J]. 贵州科学, 2007(S1): 103-110. Zhou Qi, Chen Jianhua , Zhang Mingqiao, et al. The advances in study and metallogenic significance of cold seep carbonates[J]. GuiZhou Science, 2007(S1): 103-110.
[2] Zhang C, Li P , Jiang C, et al. Geomicrobiology and biogeo-chemistry of gas hydrates and cold seeps[J]. Chemical Geology, 2004, 205(3-4): 187-194.
[3] 陈忠, 杨华平, 黄奇瑜, 等. 海底甲烷冷泉特征与冷泉生态系统的群落结构[J]. 热带海洋学报, 2007, 26(6): 73-82. Chen Zhong, Yang Huaping, Huang Qiyu, et alCharacteristics of cold seeps and structures of chemoautosynthesis-based communities in seep sediments[J]. Journal of Tropical Oceanography, 2007, 26(6): 73-82.
[4] 苗婷婷, 邢翔, 杜宗军, 等. 柄海鞘共附生细菌的分离培养与系统发育多样性研究[J]. 海洋科学进展, 2012, 30(1): 111-118. Miao Tingting, Xing Xiang, Du Zongjun, et al. Isolation and phylogenetic analysis of associated bacteria from the. [J]. Advances in Marine Science, 2012, 30(1): 111-118.
[5] Hentschel U, Schmid M. Isolation and phylogenetic analysis of bacteria with antimicrobial activities from the Mediterranean spongesand[J]. FEMS Microbiology Ecology, 2001, 35(3): 305-312.
[6] Duperron S, Sibuet M, Gregor M, et al. Diversity, relative abundance and metabolic potential of bacterial endosymbionts in threemussel species from cold seeps in the Gulf of Mexico[J]. Environmental Microbiology, 2007, 9(6): 1423-1438.
[7] Detree C, Chabenat A, Lallier F H, et al. Multiple I-type lysozymes in the hydrothermal vent musseland their role in symbiotic Plasticity[J]. PLoS ONE, 2016, 11(2): e0148988.
[8] Nelson D C, Hagen K D, Edwards D B, et al. The gill symbiont of the hydrothermal vent musselis a psychrophilic, chemoautotrophic, sulfur bacterium[J]. Mar Biol, 1995, 121(3): 487-495.
[9] Distel D L, Cavanaugh C M.Independent phylogenetic origins of methanotrophic and chemoautotrophic bacterial endosymbiosis in marine bivalves[J]. Journal of Bacteriology, 1994, 176: 1932-1938.
[10] 郑平, 王敏晓, 李超伦, 等. 热液和冷泉无脊椎动物基因组大小测定及比较[J]. 海洋学报, 2016, 38(6): 41-50. Zheng Ping, Wang Minxiao, Li Chaolun, et al. Survey of genome size in 10 invertebrates from hydrothermal vent and cold seep[J]. Chinese Journal of Oceanography, 2016, 38(6): 41-50.
[11] Doyle J J, Doyle J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochem Bull, 1987, 19: 11-15.
[12] Li WJ, Xu P, Schumann P, et al.sp. nov., a novel actinobacterium isolated from forest soil in Yunnan (China), and emended description of the genus[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(7): 1424- 1428.
[13] Thompson J D, Gibson T J, Plewniak F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4876- 4882.
[14] Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24: 1596-1599.
[15] Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425.
[16] Lampert Y, Kelman D, Dubinsky Z, et al. Diversity of culturable bacteria in the mucus of the Red Sea coral[J]. FEMS Microbiology Ecology, 2006, 58: 99-108
[17] Webster N S, Wilson K J, BlackallL L, et al. Phylogenetic diversity of bacteria associated with the marine sponge[J]. Applied and Environmental Microbiology, 2001, 67(1): 434–444.
[18] 席宇, 朱大恒, 刘红涛, 等. 假交替单胞菌及其胞外生物活性物质研究进展[J]. 微生物学通报, 2005, 32(3): 108-112. Xi Yu, Zhu Daheng, Liu Hongtao, et al. Advances onspecies and their extracellular bioactive compounds[J]. Microbiology, 2005, 32(3): 108-112.
[19] Colwell R R. Polyphasic Taxonomy of the genus: numerical taxonomy ofcholerae,, and relatedspecies[J]. J Bacteriol, 1970, 104: 410 -433.
[20] Head I M, Jones D M, Roling W F. Marine microorganisms make a meal of oil[J]. Nature Reviews Microbiology, 2006, 4(3): 173-182.
[21] 谭周进, 肖罗, 谢丙炎, 等. 假单胞菌的微生态调节作用[J]. 核农学报, 2004, 18(1): 72-76.Tan Zhoujin, Xiao Luo, Xie Bingyan, et al. Microecological functions of Pseudomonad[J]. Acta Agric Nucleat Sinica, 2004, 18(1): 72-76.
[22] 吴业辉, 邵宗泽. 海洋烷烃降解菌sp. A-11-3 的分离鉴定及其降解酶基因研究[J]. 台湾海峡, 2008, 27(4): 427-434. Wu Yehui, Shao Zongze. Isolation and identification of a novel alkane-degrading bacterial speciessp. A-11-3 and alkane hydroxylase gene detection[J]. Journal of Oceanography In Taiwan Strait, 2008, 27(4): 427-434.
[23] 王文娟, 孙冬岩, 孙笑非, 等. 芽孢杆菌对养殖水体调节作用的研究[J]. 饲料研究, 2011(8): 73-74. Wang Wenjuan, Sun Dongyan, Sun Xiaofei, et al. The study of the regulation effect ofon aquaculture water[J]. Feed Research, 2011(8): 73-74.
Isolation and phylogenetic analysis of epiphytic bacteria from mussels collected from the cold seep
GUO Wen-jie1, 2, 3, ZHAO Jin2, 3, QI Hong-tao1, JIANG Peng2, 3, LI Fu-chao2, 3, LI Rong-gui1
(1. Laboratory of Molecular Biology, College of Life Sciences, Qingdao University, Qingdao 266071, China; 2. Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 3. Laboratory of Marine Biology and Biotechnology, National Laboratory of Marine Science and Technology, Qingdao 266071, China)
Epiphytic bacteria play an important role in substance absorption and metabolism of mussels. Gills of mussels in different experimental groups were collected at different times, and the epiphytic bacteria were cultured and separated. A total of 270 strains were obtained. Four strains, which might be new species, were discovered. The diversity of epiphytic bacteria decreased sharply in 6 h sterile-seawater cultivation. In the methane treatment group, the number ofincreased gradually. In the group treated with Na2S,associated with sulfur circulation increased gradually. It is important to study the interaction between mussels and epiphytic bacteria and to explore the biological resources in extreme deep sea environments.
South China Sea; cold seep; mussel; epiphytic bacteria; diversity
Q938
A
1000-3096(2017)02-0089-07
10.11759/hykx20160930003
2016-09-30;
2017-01-06
中国科学院战略性先导科技专项(XDA11030404); 国家自然科学基金(31300365)
郭文捷(1991-), 女, 山东聊城人, 硕士, 主要从事深海极端环境微生物群落多样性研究, 电话: 17854299753, E-mail: 17854299753@ 163.com。通信作者: 李富超, 053282898500, lifuchao@qdio.ac.cn
Sep.30, 2016
[Special Fund for Strategic Leading Science and Technology Chinese Academy of Sciences, No.XDA11030404; National Natural Science Foundation of China, No.31300365]
(本文编辑: 张培新)

