基于转录组数据的凡纳滨对虾微卫星标记开发
杨 铭, 于 洋, 张晓军, 王全超, 3, 刘敬文, 3, 李富花, 相建海
基于转录组数据的凡纳滨对虾微卫星标记开发
杨 铭1, 2, 于 洋1, 张晓军1, 王全超1, 3, 刘敬文1, 3, 李富花1, 相建海1
(1. 中国科学院海洋研究所, 实验海洋生物学重点实验室, 山东青岛266071; 2. 福州市海洋与渔业技术中心, 福建福州350005; 3. 中国科学院大学, 北京100049)
对虾遗传多样性和育种研究需要大量的分子标记。作者利用凡纳滨对虾()转录组测序数据, 采用MISA软件进行微卫星序列挖掘, 共获得14 767条微卫星序列。统计发现在凡纳滨对虾中微卫星出现的频率为16.76%; 在所有的微卫星序列中, 2碱基微卫星最多, 占59.53%; 其次是3碱基微卫星, 占35.78%。随机选取其中74条序列设计引物, 通过DNA混池扩增和分型的方法进行微卫星标记的开发, PCR扩增的显示有54对(72.97%)能扩增出清晰的目的条带, 进一步分型的结果显示27条(36.49%)微卫星显示出多态性, 从这些多态性的微卫星位点中选择11个位点在“科海1号”种质库材料中进行个体分型, 结果显示在该群体中微卫星标记的等位基因数目为2~11个不等, 期望杂合度值为0.256~0.858, 观察杂合度为0.213~0.875, 多态性信息含量为0.221~0.830, 在11个位点中有4个位点显著偏离HWE (<0.05)。本研究结果为后续使用微卫星标记进行对虾遗传学研究, 促进对虾的遗传选育和种质资源保护提供了重要基础。
凡纳滨对虾(); 微卫星; 转录组; 遗传多样性
凡纳滨对虾()又称南美白对虾, 因其具有生长速度快、抗病能力强、饵料蛋白需求低等突出的养殖特性, 成为中国和世界上最主要的养殖对虾种类, 占虾类总产量的85%。然而, 病害频发和种质退化给对虾养殖造成了巨大经济损失, 通过遗传改良提高对虾的种质, 培育生长快速、抗性能力强的优良品种对于对虾养殖业的稳定发展显得尤为迫切。
在传统的遗传选育基础上, 利用分子标记研究选育种群的遗传多样性、辅助亲本选择能够有效地提高选育效率, 尤其是对于如抗病性状等难以进行表型测定的性状。微卫星(又称简单重复序列, Simple Sequence Repeats, SSR)多态性高、稳定性好, 且为共显性标记, 是一种理想的分子标记。目前, 微卫星标记技术已被广泛用于遗传图谱构建、家系鉴定、品种纯度检测以及目标性状基因定位等研究领域[1-4]。凡纳滨对虾微卫星标记在1996年即有报道[5], 最早批量开发微卫星标记的是Meehan等, 他们通过构建基因组DNA文库, 使用探针筛选的方法共得到含有573个微卫星的173条序列, 据此设计了136对微卫星引物并用于后续的连锁图谱构建[6-7]、家系鉴定[8]等多方面工作。随着测序技术的发展, NCBI上凡纳滨对虾的测序数据显著增多, 利用已有的测序数据, 特别是大量的EST数据, 采用生物信息学方法进行微卫星发掘成为研究热点, 很多的EST-SSR位点被发现和验证[3,9-10]。目前已发表的凡纳滨对虾微卫星标记约有600多个, 然而对于具有如此重要经济价值的物种, 这样少的微卫星标记显然是不相称的。可用遗传标记的缺乏是影响对虾选育的重要原因之一[11]。
近年来, 通过高通量测序, 很多物种获得了海量的序列数据, 从这些序列中发掘微卫星标记成为一种更高效的手段[12-15]。本研究利用已公布的凡纳滨对虾高通量转录组测序数据库, 通过组装的方法构建出凡纳滨对虾转录组参考序列, 运用生物信息学方法分析凡纳滨对虾转录组unigenes中的SSR标记, 并验证了开发出的SSR标记的多态性, 这些微卫星位点和开发出的分子标记为后续对虾遗传研究提供了很好的基础。
1 材料与方法
1.1 数据来源
转录组数据来源为包括5个不同胚胎和幼体发育时期的凡纳滨对虾转录组测序数据(NCBI数据库: SRR1460493, SRR1460494, SRR1460495, SRR1460504, SRR1460505)[16], 测序平台为HiSeq 2000 (Illumina, USA), 共获得263 300 374条经过过滤处理的序列, 总数据量为29.2 Gb。使用Trinity软件(http: //trinityrnaseq. sourceforge.net/)对这些经过过滤处理的序列进行混合拼接形成参考序列。
1.2 微卫星位点的批量发掘和引物设计
微卫星序列的定位和识别使用MISA软件(http: // pgrc.ipk-gatersleben.de/misa/)进行, 由于转录组中存在polyA和polyT, 因此使用转录组进行SSR发掘时不考虑单碱基重复, MISA参数设置如下: 2-6、3-5、4-5、5-5、6-5, 复合微卫星标记的最大长度为100 bp。MISA网站同时提供了用于批量转换微卫星两侧序列的Perl脚本p3_in.pl和p3_out.pl, 利用这两个脚本将MISA结果转换成Primer 3程序可以识别的格式, 在本地运行Primer 3批量设计引物。
1.3 混池方法筛选多态性微卫星位点
从凡纳滨对虾“科海1号”核心种质库中取32尾, 体长13~14 cm, 体质量30~35 g。采用海洋生物基因组提起试剂盒(Tiangen)提取DNA, 提取的DNA使用Nanodrop 2000(Thermo Scientific, USA)测量DNA浓度后稀释至100 ng/μL, 每8个个体的DNA等量混合, 构建4个DNA混池。
在批量设计的引物中随机选取74条, 采用TP-M13方法[17]对4个混池的对虾基因组DNA进行微卫星分型, 为了避免非特异性扩增, 扩增并分型在单个个体中同时进行。PCR扩增上述样品混池和单个个体DNA, PCR反应在20 μL体系中进行, 体系如下: 模板DNA(100 ng/μL)2μL, 10× Buffer 2 μL, dNTP 0.4 μL, MgCl21.2 μL, 正向引物0.1 μL, 反向引物 0.4 μL, M13 引物0.2 μL, Taq(2.5 U/μL)0.25 μL, 超纯水13.35 μL。PCR反应程序如下: 94℃ 4 min, 然后运行94℃ 30 s, 50~60℃30 s, 72℃ 30 s 38个循环; 接着运行94℃ 45 s, 45℃ 45s, 72℃ 45 s 12个循环; 最后72 ℃ 5 min, 4 ℃终止, PCR扩增产物避光保存。
将上述PCR产物使用超纯水稀释20倍, 之后配置上机样品缓冲液, 缓冲液由HiDi甲酰胺和ROX 500标准品(Applied Biosystems, USA)组成, 两者比例为9.85︰0.15。取稀释后的PCR产物1 μL与9 μL的上机缓冲液混合, 95℃变性2 min, 立刻置于冰上以阻止DNA的复性。然后将加入上机缓冲液后的样品在3130xl(Applied Biosystems, USA)进行测序仪分型。分型结果使用GENEMARKER v1.91(SoftGenetics, USA)读取分型数据, 根据分型结果获得每个微卫星位点的多态性。
1.4 个体微卫星分型研究微卫星等位基因频率
在凡纳滨对虾“科海1号”种质库的32个个体中研究微卫星标记的多态性分布, 同时验证混池筛选的微卫星的准确性, 所采用的分型方法同上。获得微卫星分型数据后, 使用PopGen32软件计算群体的等位基因数、观测杂合度和期望杂合度, 利用Cervus 3.0软件计算位点的多态信息含量()利用GENEPOP 4.2软件进行Hardy-Weinberg平衡分析 (http: //genepop. curtin.edu.au/)。
2 结果与分析
2.1 转录组SSR位点开发
凡纳滨对虾的5个转录组组装共获得66 815条非冗余基因, 利用该数据共发掘SSR位点14 767个, 这些SSR位点分布于11 195条基因片段中, 其中以复合微卫星形式存在的有1 844个, 微卫星的发生频率(含有SSR的非冗余基因数量与总非冗余基因数量的比值)为16.76%(表1)。在所发掘的微卫星中, 2碱基微卫星最多, 占59.53%, 其次是3碱基微卫星, 占35.78%(图1)。

表1 凡纳滨对虾转录组SSR发掘位点统计
2.2 微卫星标记验证
从转录组选择的74对微卫星引物, 在“科海1号”DNA混池中进行PCR扩增, 结果显示能够成功扩增的引物有54对, 占设计所选序列的72.97%。经过微卫星分型后有27对(36.49%)显示出多态性(表2), 多态性位点的等位基因数从2~8不等, 以2和3等位基因的居多, 少量微卫星标记含有7~8个等位基因。将这些发掘的标记与已公布的微卫星标记进行BLAST比对, 未发现有共同的部分, 因此这些标记为新发现的凡纳滨对虾微卫星标记。
2.3 多态性微卫星的种群分析
选择11对具有多态性的转录组微卫星引物, 在“科海1号”种质库中进行分型。结果显示, 这11个微卫星引物共包含65个等位基因, 每个位点检测到的等位基因2~11个不等(表3)。遗传多态性分析显示, 这11个标记在“科海1号”种群中的期望杂合度值从0.256到 0.858不等, 观察杂合度从0.213到0.875不等, 多态性信息含量在0.221~0.830, 在11个位点中7个位点符合Hardy-Weinberg平衡, 有4个位点(LvU15501、LvC905、LvC2235、LvU27836)显著偏离HWE (<0.05)。

表2 从凡纳滨对虾转录组中挖掘的具有多态性的微卫星位点
续表
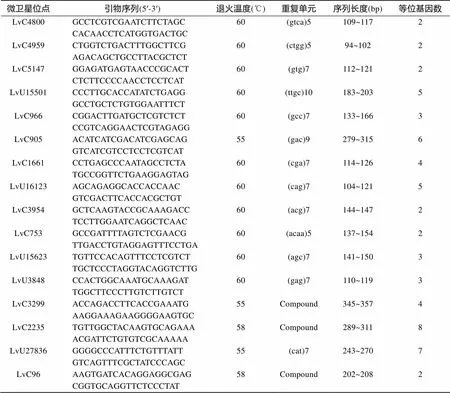
微卫星位点引物序列(5′-3′)退火温度(℃)重复单元序列长度(bp)等位基因数 LvC4800GCCTCGTCGAATCTTCTAGC60(gtca)5109~1172 CACAACCTCATGGTGACTGC LvC4959CTGGTCTGACTTTGGCTTCG60(ctgg)594~1022 AGACAGCTGCCTTACGCTCT LvC5147GGAGATGAGTAACCCGCACT60(gtg)7112~1212 CTCTTCCCCAACCTCCTCAT LvU15501CCCTTGCACCATATCTGAGG60(ttgc)10183~2035 GCCTGCTCTGTGGAATTTCT LvC966CGGACTTGATGCTCGTCTCT60(gcc)7133~1663 CCGTCAGGAACTCGTAGAGG LvC905ACATCATCGACATCGAGCAG55(gac)9279~3156 GTCATCGTCCTCCTCGTCAT LvC1661CCTGAGCCCAATAGCCTCTA60(cga)7114~1264 TGCCGGTTCTGAAGGAGTAG LvU16123AGCAGAGGCACCACCAAC60(cag)7104~1215 GTCGACTTCACCACGCTGT LvC3954GCTCAAGTACCGCAAAGACC60(acg)7144~1472 TCCTTGGAATCAGGCTCAAC LvC753GCCGATTTTAGTCTCGAACG60(acaa)5137~1542 TTGACCTGTAGGAGTTTCCTGA LvU15623TGTTCCACAGTTTCCTCGTCT60(agc)7141~1503 TGCTCCCTAGGTACAGGTCTTG LvU3848CCACTGGCAAATGCAAAGAT60(gag)7110~1193 TGGCTTCCCTTGTCTTGTCT LvC3299ACCAGACCTTCACCGAAATG55Compound345~3574 AAGGAAAGAAGGGGAAGTGC LvC2235TGTTGGCTACAAGTGCAGAAA58Compound289~3118 ACGATTCTGTGTCGCAAAAA LvU27836GGGGCCCATTTCTGTTTATT55(cat)7243~2707 GTCAGTTTCGCTATCCCAGC LvC96AAGTGATCACAGGAGGCGAG58Compound202~2082 CGGTGCAGGTTCTCCCTAT
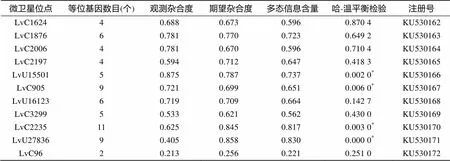
表3 从转录组中发掘的11个多态性位点的遗传多样性信息
注:“*”.显著偏离哈-温平衡(<0.05)
3 讨论
微卫星标记由于其较高的遗传多样性, 一直是研究人员开发的重点。虽然新一代分子标记SNP、Indel等的出现使其关注度有所降低, 但是微卫星标记具有多态性高, 相对于SNP标记多为2态性的特征, 微卫星标记在进行种群遗传关系分析方面具有更大用处。研究人员分析了微卫星标记和SNP标记在进行亲缘关系鉴定方面的功效, 发现SNP标记具有很高的品种与群体特异性, 同一品种不同的群体之间也存在差异, 而中等密度的微卫星标记在种群关系上的作用比SNP效果更好[18], 因此微卫星标记仍然有很大的应用空间。
具有多态性的SSR位点才可能开发为微卫星标记。本研究利用已经获得的高通量测序转录组序列, 采用生物信息学方法从测序拼接的数据中发掘SSR位点, 并进行批量的引物设计, 通过混池法对微卫星标记的多态性进行分析, 最终获得27个新的多态性微卫星标记, 并在一个育种群体中分析了11个微卫星标记的等位基因数、等位基因频率和多态性信息含量等。从11个微卫星标记在育种群体的分型结果看, 91%(10/11)的位点平均多态信息含量分别为都在0.5以上, 根据Botstein等[19]提出的衡量标准, 当>0.5时, 意味着该位点为高度多态位点, 说明本研究开发的标记的多态性较高, 同时也说明了育种群体虽然经过多代的选育, 仍然具有较为丰富的遗传多样性。基因杂合度是衡量群体遗传变异水平的理想参数, 除了位点LvC96外, 其他10个位点的观测杂合度和期望杂合度均较高, 也从一个侧面说明了对虾作为一种人工驯化程度较低的物种, 个体的杂合度仍然较高。
在进行个体分型的11个位点中, 有4个(36%)位点显著偏离哈-温平衡(<0.05)。在已报道的水产动物微卫星挖掘的文章中, 偏离哈-温平衡的标记出现较为普遍, 在菲律宾蛤仔()和拟穴青蟹()的微卫星发掘中, 发现有84%和75%的标记显著偏离哈-温平衡[20-21], 该现象的原因可能是由于海洋动物的遗传变异度较高, 因此在微卫星标记的引物位置在不同个体中可能会存在SNP、Indel等变异, 使其中的等位基因无法扩增出来, 产生无效等位基因, 导致分型结果显著偏离哈-温平衡。在本研究中, 所用的实验材料取自育种群体, 由于在育种过程中是按照育种目的进行的家系构建, 亲本之间的交配并不是随机的, 这也可能是导致有些标记偏离哈-温平衡的原因之一。
凡纳滨对虾基因组复杂, 重复序列所占比例很高, 其中串联重复序列至少占基因组序列的18.59%左右[22]。在本文的研究中, 生物信息学方法预测的微卫星数目明显高于其他物种, 可见无论在转录组中还是基因组中, 对虾微卫星的发生频率均较高。相对于基因组序列, 从转录组中发掘的SSR数量较少, 这与转录组序列为功能基因有关, 在编码区较少有重复序列的出现, 转录组中重复序列多出现在UTR区域。转录组中开发微卫星的效率较低, 这与扩增产物可能跨较大内含子或者引物位于内含子外显子结合处造成扩增失败所致, 这也决定了在扩增转录组中的微卫星序列时一般会选择扩增片段较小的引物。然而由于转录组开发的微卫星来源于基因的调控区或编码区, 可以直接、准确地标记功能基因, 所以更真实地反映该物种的遗传多样性。
本研究表明从高通量测序数据中筛选对虾微卫星是一种高效可行的方法, 研究结果大大增加了凡纳滨对虾可用的微卫星标记, 对于凡纳滨对虾的遗传学研究、分子标记选育以及种质资源保护都具有重要意义。
[1] 王鸿霞, 吴长功, 张留所, 等. 微卫星标记应用于凡纳滨对虾家系鉴别的研究[J]. 遗传, 2006, 28(2): 179- 183. Wang Hongxia, Wu Changgong, Zhang Liusuo, et al. The application of microsatellite markers for parentage determination in selective breeding of Pacific white shrimp ()[J]. Hereditas (Beijing), 2006, 28(2): 179-183.
[2] 程鹏, 杨爱国, 吴彪, 等. 微卫星标记在不同壳色虾夷扇贝家系亲权鉴定的适用性[J]. 水生生物学报, 2011, 35(5): 768-775. Cheng Peng, Yang Aiguo, Wu Biao, et al. The applicability analysis on microsatellite markers for parentage determination of different shell color lines of Japanese scallop[J]. Acta Hydrobiologica Sinica, 2011, 35(5): 768-775.
[3] 王艳红, 胡超群, 张吕平, 等. 凡纳滨对虾EST微卫星标记初步筛选[J]. 水产学报, 2011, 35(7): 969-976. Wang Yanhong, Hu Chaoqun, Zhang Lüping, et al. A preliminary study on microsatellite markers screening from EST sequences of[J]. Journal of Fisheries of China, 2011, 35(7): 969-976.
[4] Norris A T, Bradley D G, Cunningham E P. Parentage and relatedness determination in farmed Atlantic salmon () using microsatellite markers[J]. Aquaculture, 2000, 182(1-2): 73-83.
[5] Garcia D K, Dhar A K, Alcivar-Warren A. Molecular analysis of a RAPD marker (B20) reveals two microsatellites and differential mRNA expression in[J]. Molecular Marine Biology and Biotechnology, 1996, 5(1): 71-83.
[6] Meehan D, Xu Z K, Zuniga G, et al. High frequency and large number of polymorphic microsatellites in cultured shrimp,()[Crustacea : Decapoda][J]. Marine Biotechnology, 2003, 5(4): 311-330.
[7] Alcivar-Warren A, Meehan-Meola D, Park S W, et al. ShrimpMap: A low-density, microsatellite-based linkage map of the pacific whiteleg shrimp,: Identification of sex-linked markers in linkage group 4[J]. Journal of Shellfish Research, 2007, 26(4): 1259- 1277.
[8] 王鸿霞, 吴长功, 张留所, 等. 微卫星标记应用于凡纳滨对虾()家系鉴别的研究[C]//第四届世界华人虾类养殖研讨会, 青岛: 中国科学院海洋研究所, 2004. Wang Hongxia, Wu Changgong, Zhang Liusuo, et al. The application of microsatellite markers for family identification of Pacific white shrimp ()[C]//The 4thWorld Chinese Symposium on Shrimp Aquaculture, Qingdao: Institute of Oceanology, Chinese Academy of Sciences, 2004.
[9] Perez F, Ortiz J, Zhinaula M, et al. Development of EST-SSR markers by data mining in three species of shrimp:,and[J]. Marine Biotechnology, 2005, 7(5): 554-569.
[10] Santos C A, Rossini B C, Marques C G, et al. Characterization and genomic annotation of polymorphic EST-SSR loci ini shrimp[J]. Aquaculture Research, 2011, 43(10): 1567-1570.
[11] Cruz P, Mejia-Ruiz C H, Perez-Enriquez R, et al. Isolation and characterization of microsatellites in Pacific white shrimp()[J]. Molecular Ecology Notes, 2002, 2(3): 239-241.
[12] Zheng X F, Pan C, Diao Y, et al. Development of microsatellite markers by transcriptome sequencing in two species of(Araceae)[J]. BMC Genomics, 2013, 14: 490.
[13] Ma H Y, Jiang W, Liu P, et al. Identification of transcriptome-derived microsatellite markers and their association with the growth performance of the mud crab ()[J]. Plos One, 2014, 9(2): e89134.
[14] Ahn Y K, Tripathi S, Cho Y I, et al.transcriptome assembly and novel microsatellite marker information invarieties Saengryeg 211 and Saengryeg 213[J]. Botanical Studies, 2013, 54: 58.
[15] Huang D N, Zhang Y Q, Jin M D, et al. Characterization and high cross-species transferability of microsatellite markers from the floral transcriptome of(Asparagaceae)[J]. Molecular Ecology Resources, 2014, 14(3): 569-577.
[16] Wei J K, Zhang X J, Yu Y, et al. RNA-Seq reveals the dynamic and diverse features of digestive enzymes during early development of Pacific white shrimp[J]. Comparative Biochemistry and Physiology D-Genomics & Proteomics, 2014, 11(5): 37-44.
[17] Oetting W S, Lee H K, Flanders D J, et al. Linkage analysis with multiplexed short tandem repeat polymorphisms using infrared fluorescence and M13 tailed primers[J]. Genomics, 1995, 30(3): 450-458.
[18] Yang X, Xu Y, Shah T, et al. Comparison of SSRs and SNPs in assessment of genetic relatedness in maize[J]. Genetica, 2011, 139(8): 1045-1054.
[19] Botstein D, White R L, Skolnick M, et al. Construction of a genetic-linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331.
[20] 宋忠魁, 聂振平, 王芳宇. 拟穴青蟹(CA)n微卫星DNA多态性引物筛选[J]. 海洋科学, 2013, 37(10): 98-103.Song Zhongkui, Nie Zhenping, Wang Fangyu. Screening for polymorphic primer pairs of (CA)nmicrosatellite from(mud crab) genome[J]. Marine Sciences, 2013, 37(10): 98-103.
[21] 闫路路, 秦艳杰, 闫喜武, 等. 基于转录组平台的蛤仔微卫星标记筛选[J]. 生态学报, 2015, 35(5): 1573- 1580. Yan Lulu, Qin Yanjie, Yan Xiwu, et al. Development of microsatellite markers inusing next-generation sequencing[J]. Acta Ecologica Sinica, 2015, 35(5): 1573-1580.
[22] Zhao C, Zhang X J, Liu C Z, et al. BAC end sequencing of Pacific white shrimp: a glimpse into the genome of Penaeid shrimp[J]. Chinese Journal of Oceanology and Limnology, 2012, 30(3): 456-470.
Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp ()
YANG Ming1, 2, YU Yang1, ZHANG Xiao-jun1, WANG Quan-chao1, 3, LIU Jing-wen1, 3, LI Fu-hua1, XIANG Jian-hai1
(1. Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Oceanic & Fishery Technology Center of Fuzhou, Fuzhou 350005, China; 3. University of Chinese Academy of Sciences, Beijing 100049, China)
The Pacific white shrimp,, is one of the most economically important marine aquaculture species in China, as well as the world. A large number of molecular markers are required for the studies of genetic diversity and breeding in shrimp. In the present investigation, 14 767 SSRs were detected inunigenes derived from transcriptome sequencing. Among these, dinucleotide SSRs were the most abundant motif (59.53%), followed by trinucleotide (35.78%) SSRs. A total of 74 primer pairs were designed and validated using DNA pools. 54 pairs (72.97%) were amplified with clear band, and 27 pairs (36.49%) were found to be polymorphic. The genetic diversity was analyzed using 11 highly polymorphic SSR markers in the breeding population of “Kehai No.1, ” a selected new variety of. Consequently, the allele number of these microsatellites ranged from 2 to 11. Expected heterozygosity and observed heterozygosity ranged from 0.256 to 0.858 and 0.213 to 0.875, respectively. The polymorphic information varied from 0.221 to 0.830. Four loci deviated significantly from Hardy-Weinberg equilibrium (HWE) (< 0.05). The large number of genetic markers developed in the present study would be highly beneficial to the researches to understand genetic diversity, selective breeding, and germplasm conservation in shrimp.
; microsatellite; transcriptome; polymorphism
S917.4
A
1000-3096(2017)02-0096-07
10.11759//hykx20160322002
2016-03-22;
2016-06-28
国家自然科学基金资助项目(31502161, 31672632); 国家“863”高技术研究发展计划资助项目(2012AA10A404)
杨铭(1971-), 男, 福建福州人, 工程师, 主要从事水产养殖技术研究工作, E-mail: ymfj88@163.com; 张晓军, 通信作者, E-mail: xjzhang@qdio.ac.cn
Mar. 22, 2016
[National Natural Science Foundation of China, No.31502161, 31672632; The National High Technology Research and Development Program of China (863 Program), No.2012AA10A404]
(本文编辑: 谭雪静)

