内参基因在昆虫qRT—PCR中的研究进展
史彩华+胡静荣+李传仁+王文凯
摘要:实时荧光定量PCR(qRT-PCR)具有灵敏度高、特异性强等优点,是昆虫目标基因表达和转录分析最常用的技术手段之一。然而,为了消除样本在RNA产量、质量及逆转录效率上存在的差异,在进行qRT-PCR分析目标基因表达量时必须选择合适的“管家基因”作为内参基因。近年来,大量研究表明不同昆虫和不同试验条件选择的内参基因也不尽相同。因此,选择合适的内参基因是正确判断qRT-PCR结果分析的关键。从内参基因的选择、昆虫内参基因的研究和内参基因稳定性评价等几个方面进行综述,以期为研究者在将来的昆虫试验中选择合适的内参基因提供理论参考依据。
关键词:内参基因;昆虫;qRT-PCR;进展
中圖分类号: S436.33文献标志码: A文章编号:1002-1302(2017)07-0001-07
实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)技术1996年由美国Applied Biosystems公司发明,随后经过进一步技术升华,实现了基因表达检测从定性到定量的飞跃[1]。该技术具有高效快速、重复性好、定量准确、灵敏度高和高通量等优点,尤其可对样本量少、无法用Northern-blot等手段检测的样本进行精确检测[2]。因此该技术是昆虫目标基因表达和转录分析最常用的技术手段之一。
qRT-PCR分为绝对定量和相对定量2种。绝对定量具有高度特异性、重复性好,可进行多重定量,每次试验时必须做标准曲线来分析未知样品中目标核酸序列的绝对拷贝数[3]。由于标记成本太高,通常应用在临床诊断上[4]。针对基因的差异表达分析,科研工作者们最常用的检测手段是采用相对定量法,其优点在于成本低、操作方便[5]。相对定量检测时,需要不同的样本在起始细胞数目、RNA提取效率、质量及逆转录水平等均相同,否则会影响目的基因的真实表达水平。然而,这些条件不可能同时满足,因此,应用qRT-PCR检测目的基因表达水平时,需要选择稳定表达的内参基因进行校正和标准化[6]。
理想的内参基因应该在不同试验条件下均能稳定表达[7]。但是大量的研究表明,随着试验条件的变化,没有哪一种内参基因在不同细胞类型和不同生理状态下的表达是始终恒定不变的[8]。倘若盲目地选择内参基因,可能难以发现某些目的基因表达的微小差异,或者出现错误甚至相反的结论[9],导致目的基因表达水平出现100倍的偏差[10]。因此,选择合适的内参基因是正确判断qRT-PCR结果分析的关键[11]。
近年来,随着qRT-PCR和基因芯片的广泛应用,内参基因的研究报道日益增多[12]。本研究从内参基因的选择、昆虫内参基因的研究和内参基因稳定性评价等几个方面进行综述,以期为研究者在将来的昆虫试验中选择合适的内参基因提供理论参考依据。
1昆虫常用内参基因的选择及具备的条件
内参基因通常选择受环境因素影响较小、表达水平相对稳定的“管家基因”[13]。在细胞中,内参基因的表达量或在基因组中的拷贝数几乎恒定。内参基因在不同类型的细胞、组织及不同试验条件下表达水平存在差异,因此,不同试验需要选择不同的内参基因[14]。理想的内参基因不存在假基因,避免基因组DNA的扩增[15];同时,理想的内参基因具有高度或中度表达特性,Ct值应介于15~30之间[16],能够在昆虫不同类型细胞和组织中稳定表达,不受生理周期的影响,不受任何内外因素的干扰[17];另外,理想的内参基因还需要具有与目的基因相似的表达量[18]。但是,这样的内参基因并不存在,没有任何一种基因在不同的试验条件下始终恒定表达。因此,选择合适的内参基因是qRT-PCR进行基因表达分析的重要前提。
由于内参基因在不同生理条件下的表达并非恒定不变,qRT-PCR试验时使用1个内参基因往往不能得到准确的定量结果。因此,同时使用2个或多个内参基因有助于调整系统偏差,更有利于得到准确的基因表达定量结果,尤其针对表达量只有细微差异的基因非常重要[19]。
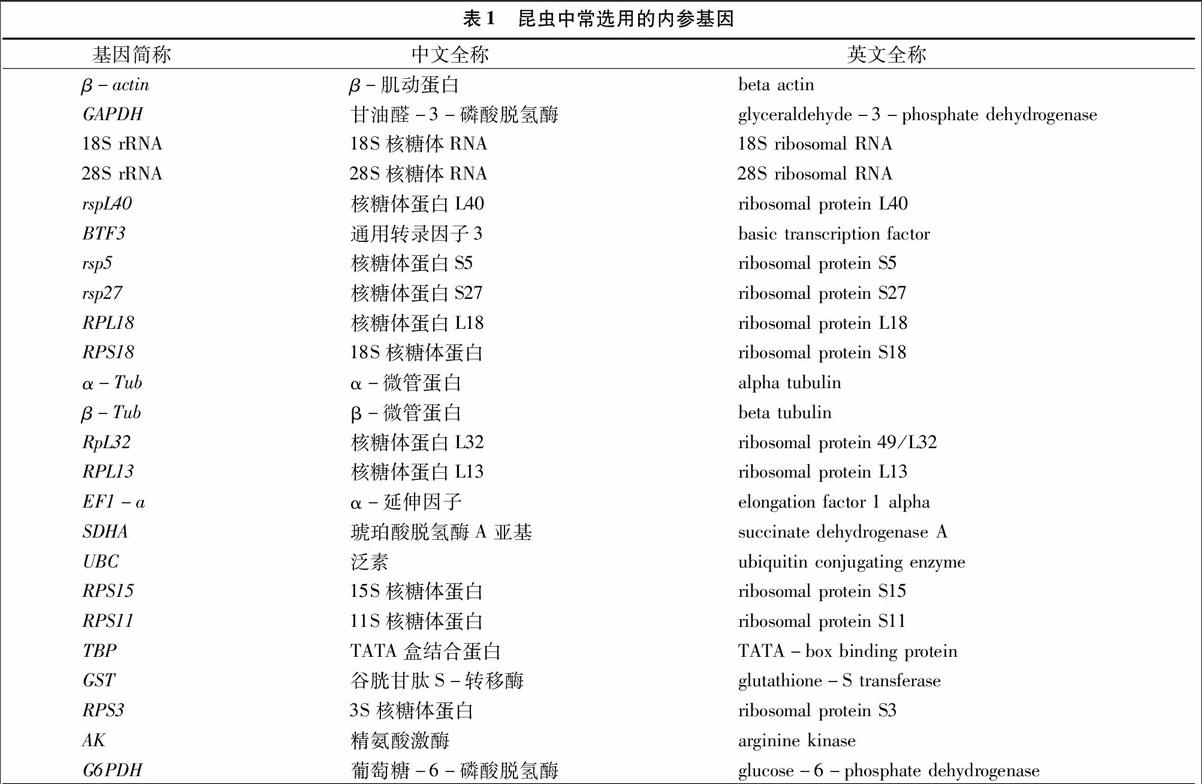
目前,昆虫研究中常见的内参基因包括β-肌动蛋白基因(beta actin,β-actin)、延伸因子1(elongation factor-1 alpha,EF1-a)、甘油醛-3-磷酸-脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、18S核糖体RNA(18S ribosomal RNA,18S rRNA)和琥珀酸脱氢酶复合体A亚基(succinate dehydrogenase complex subunit A,SDHA)基因等[20](表1),这些基因存在于细胞的正常生命代谢中,维持生命活动所需的细胞器骨架的基本组合,参与生命体的基本生化代谢过程。
2特定试验条件下昆虫内参基因的研究
虽然上述常用内参基因一直被作为目的基因的标准化基因,但是,最近大量研究表明,任何一种“管家基因”的所谓恒定表达只是在一定类型的细胞或试验因素作用下“有范围”的恒定[21]。在一种试验条件下稳定表达的基因并非适合另一种试验条件,同种试验条件下不同的昆虫种类选择的内参基因也不尽相同。因此,针对特定试验条件选择合适的内参基因是准确分析目的基因表达的基础[22]。
2.1不同虫态或发育时期内参基因的选择研究
陈立华等研究表明,β-actin基因在蓖麻蚕幼虫、蛹、成虫、卵各发育时期的表达稳定,适合作为不同虫态的内参基因[23]。袁淼研究表明,RPS15、RPS11、α-TUB和EF1-α基因适合作为褐飞虱不同发育历期的内参基因[24]。Sun等研究表明,RPS18和α-TUB基因适合作为朱砂叶螨卵、幼螨、若螨和成螨4个不同历期的内参基因[25]。王佳等研究表明,UBQ、GAPDH和GST基因适合作为柑橘大实蝇不同虫态的内参基因[26];Lü等研究表明,RPL32基因适合作为柑橘大实蝇不同发育历期的内参基因[27];王刘豪研究表明,α-TUB、RPL32和EF1-α基因适合作为柑橘大实蝇不同发育历期的内参基因[28]。
宋旺等研究表明,RPS、GAPDH和α-TUB基因可作为不同存活时间花绒寄甲的内参基因[29]。Bansal等研究表明,TBP基因可作为大豆蚜不同发育历期的内参基因[30]。郭长宁研究表明,β-TUB和β-actin基因可作为金纹细蛾不同发育阶段各虫态间的内参基因[31]。徐素平等研究表明,PPI、RPII和DIMT基因可作为不同发育阶段棉蚜的内参基因[32]。陈芳等研究表明,α-TUB基因可作为棉花粉蚧2龄若虫和3龄若虫的内参基因[33]。杨丽红研究表明,RPII基因适合作为柑橘全爪螨不同发育历期的内参基因[34]。Lu等研究表明,GAPDH和UCCR基因适合作为斜纹夜蛾不同发育历期内参基因[35]。van Hiel等研究表明,RP49、EF1α和ACT基因適合作为5龄沙漠飞蝗幼虫的内参基因[36]。Toutges等研究表明,rps6、rpL13、rps3和rps18基因适合作为赤拟谷盗不同发育时期的内参基因[37]。Bagnall等研究表明,18S rRNA、28S rRNA、GST1、β-TUB和RPLPO基因适合作为丝光绿蝇不同发育历期的内参基因[38]。Mamidala等研究表明,RPL18基因适合作为温带臭虫不同发育历期的内参基因[39]。Zhang等研究表明,28S rRNA和RPS15基因适合作为棉铃虫不同发育历期的内参基因[40]。Shakeel等研究表明,RPL28和RPS15基因适合作为棉铃虫不同发育历期的内参基因[41]。Cardoso等研究表明,ACTIN、GAPDH和RP49基因适合作为丽蝇不同发育历期的内参基因[42]。
综上所述,不同学者对不同昆虫各虫态和发育历期内参基因进行研究,结果表明它们之间并不存在完全的共性,即不同昆虫各虫态和发育历期之间选用的内参基因不尽相同,说明不同昆虫种类同源内参基因之间并不存在绝对的通用性。
2.2不同昆虫组织内参基因的选择研究
李柯等研究表明,黏虫β-actin基因在不同组织间的表达无显著差异,适合作为组织研究的内参基因[43]。吴家红等研究表明,RSPL40和BTF3基因适合作为白纹伊蚊不同组织研究的内参基因[44]。彭然研究表明,ACT3、GAPDH和α-TUB基因在家蚕中肠组织中表达稳定;α-TUB、UBC和TBP在家蚕脂肪体中表达稳定;UBC、α-TUB和ACT3基因在家蚕马氏管中表达稳定,可分别作为不同组织的内参基因[45]。陈立华等研究表明,β-actin基因适合作为蓖麻蚕血液、脂肪体、中肠和丝腺等组织研究的内参基因[23]。王佳等研究表明柑橘大实蝇成虫的不同组织适合采用TUB、GAPDH和GST基因作为内参基因[26]。袁淼研究表明,RPS11、TUB和RPS15基因适合作为褐飞虱组织研究的内参基因[24]。Shen等研究表明,ACTB和α-TUB基因适合作为东方实蝇组织研究的内参基因[46]。吴玉等研究表明,α-TUB和28S rRNA基因在中部丝腺中表达稳定,GAPDH和28S rRNA基因在后部丝腺中表达稳定,α-TUB和UBC基因在脂肪体中表达稳定,适合作为不同组织研究的内参基因[47]。
郭长宁研究表明,β-actin和18S rRNA基因适合作为金纹细蛾成虫不同组织研究的内参基因[31]。徐素平研究表明,PPI和DIMT基因是棉蚜不同组织最优内参基因[32]。Lu等研究表明,RPL10、AK和EF-1α基因适合作为斜纹夜蛾不同组织研究的内参基因[35]。Paim等研究表明,18S rRNA、GAPDH和α-TUB基因适合作为长红锥蝽唾液腺和体腔内研究的内参基因[48]。申光茂等研究表明,α-TUB和ACT5基因组合适合作为橘小实蝇雌虫不同组织的内参基因;α-TUB和ACT3基因组合适合作为橘小实蝇雄虫不同组织的内参基因;α-TUB和ACT2基因组合最适合作为橘小实蝇雄虫和雌虫中肠和马氏管的内参基因;α-TUB和ACT1基因组合适合作为橘小实蝇脂肪体的内参基因[49]。Majerowicz等研究表明,18S rRNA和EF-1α基因适合作为长红锥蝽不同组织器官研究的内参基因[50]。Hornkova等研究表明,AK和PLA2基因适合作为黄蜂唇腺和脂肪体研究的内参基因;EF-1α和PLA2基因适合作为熊蜂唇腺和脂肪体研究的内参基因[51]。Mamidala等研究表明,RPL18基因适合作为温带臭虫不同组织研究的内参基因[39]。Rajarapu等研究表明,EF-1α基因适合作为白蜡窄吉丁虫组织的内参基因[52]。Zhang等研究表明,RPS15和RPL13基因适合作为棉铃虫幼虫组织的内参基因;EF-1α和RPL27基因适合作为棉铃虫成虫组织的内参基因[40]。
根据上述不同学者对不同昆虫组织的内参基因进行研究,结果表明不同昆虫组织之间合适的内参基因不同;另外,同种昆虫不同组织之间选用的合适内参基因也不尽相同。因此,进一步说明特定试验条件下需要选择特定的内参基因,不能盲目借鉴。
2.3不同温度处理后内参基因的选择研究
孟闪闪等研究表明,光滑鳖甲β-actin基因在不同季节、不同温度处理后表达无显著差异,可作为温度胁迫研究的内参基因;而小胸鳖甲随季节和温度的变化,β-actin基因表达呈现显著差异,不适合作为温度胁迫研究的内参基因[53]。
唐婷等研究表明,β-actin基因适合作为荒漠昆虫谢氏宽漠王温度胁迫研究的内参基因[54]。袁淼研究表明,RPS15、TUB和EF-1α基因适合作为褐飞虱温度胁迫研究的内参基因[24]。杨丽红研究表明,α-TUB和RPII基因适合作为柑橘全爪螨温度胁迫研究的内参基因[34]。Lu等研究表明,GAPDH 和EF-1α基因适合作为斜纹夜蛾温度胁迫研究的内参基因[35]。Ponton等研究表明,Actin、Mnf和TUB基因适合作为黑腹果蝇温度胁迫研究的内参基因[55]。Zhang等研究表明,RPS15和RPL27基因适合作为棉铃虫温度胁迫研究的内参基因[40];Shakeel等研究表明,RPL28和RPS15基因适合作为棉铃虫温度胁迫研究的内参基因[41]。
综合上述研究结果表明,温度胁迫对不同昆虫体内同种“管家基因”表达水平的影响不尽相同。同一种“管家基因”在不同昆虫体内反应并非一致,有的经温度胁迫后表达稳定,有的高丰度表达。因此,再次说明不同物种在相同试验条件下也需要筛选特有的内参基因,不能盲目借用。
2.4菌或病毒胁迫后内参基因的选择研究
Scharlaken等研究表明,意大利蜂经细菌胁迫前后ACT、RPS18和GAPDH基因表达稳定,适合作为内参基因[56]。Maroniche等研究表明,UBI、18S rRNA和ACT基因适合作为飞虱感染病毒后的内参基因[57]。Xue等研究表明,28S rRNA基因适合作为昆虫细胞感染病毒后的内参基因[58]。Lord等研究表明,RPS3、RPS18和RPL13基因适合作为赤拟谷盗感染真菌后的内参基因[59]。Niu等研究表明,PPAI、RPL23和UBI基因适合作为欧洲熊蜂感染IAPV病毒后的内参基因[60]。Zhang等研究表明,GAPDH、RPL27和TUB基因适合作为棉铃虫感染核型多角体病毒后的内参基因[40]。
上述研究结果表明,菌或病毒感染昆虫也是一种胁迫,能够影响昆虫体内不同“管家基因”的表达水平。因此,研究菌或病毒感染昆虫后目标基因的表达,需要根据不同的昆虫或不同的病毒进行特定内参基因筛选,不能直接照搬或盲目借鉴其他昆虫的内参基因。
2.5昆虫不同品系或地理种群内参基因的选择研究
袁淼对褐飞虱内参基因筛选研究表明,TUB、RPS11、EF-1α基因在不同地理种群中表达稳定[24]。Lu等对斜纹夜蛾内参基因稳定性分析,结果表明不同种群中RPL10和 EF-1α基因适合作为不同品系或地理种群的内参基因[35]。由此说明,同种昆虫特定基因表达分析时,也需要考虑其地理种群或品系之间的差异,合理选择内参基因。
2.6昆虫不同药剂处理后内参基因的选择研究
高新菊等研究表明,α-TUB基因适合作为二斑叶螨抗甲氰菊酯品系的内参基因研究[61]。Niu等研究表明,EF-1α和GAPDH基因在不同药剂选择压力下的柑橘全爪螨品系中表达最稳定,适合作为理想的内参基因[62]。Sun等研究表明,在朱砂叶螨(Tetranychus cinnabarinus)敏感和抗甲氰菊酯品系中最理想的内参基因是RPS18和5.8S rRNA基因组合[25]。袁淼对褐飞虱内参基因筛选研究表明,RPS11、EF-1α和TUB基因在不同药剂处理下表达稳定,适合作为药剂研究的内参基因[24]。郭长宁研究表明,β-actin和β-TUB基因适合作为金纹细蛾药剂研究中的内参基因[31]。申光茂等研究表明,橘小实蝇经药剂处理后,幼虫中EF-1α基因表达稳定,脂肪中RPL13基因表达稳定[49]。Jiang等研究表明,18S rRNA基因适合作为书虱经杀虫剂诱导后的内参基因[63]。Zhang等研究表明,RPS15和RPL32基因适合作为棉铃虫经杀虫剂处理后的内参基因[40]。
综上研究表明,药剂处理也会导致昆虫体内部分“管家基因”发生表达差异,有些“管家基因”表达恒定,有些“管家基因”发生明显的上下调。因此,在研究药剂处理后昆虫目标基因的表达差异时,也需要根据昆虫种类和药剂类型来提前筛选合适的内参基因。
2.7其他
袁淼对褐飞虱内参基因筛选研究表明,RPS15、TUB、RPS11和EF-1α在不同食物饲喂时表达稳定;RPS11、TUB、RPS15在饥饿处理下表达稳定[24]。Ponton等研究表明,RPL32和α-TUB适合作为黑腹果蝇食物胁迫下的内参基因[64]。Spanier等研究表明,Xbp1、Tbp、CAPON和Stx16适合作为水生蚤类被捕食诱导研究的内参基因[65]。Chapuis等研究表明,Arm和EF-1α适合作为澳大利亚蝗虫高密度饲养条件下的内参基因[66]。Wang等研究表明,UBE3和RPL22适合作为芫菁雌虫内参基因;UBE3、TAF5和RPL22适合作为芫菁雄虫内参基因[67]。Shakeel等研究表明,棉铃虫在饥饿状态下,RPL28和RPS15适合作为内参基因;经过不同光周期胁迫幼虫,HSP90和TUBB表达最稳定;TUBB和GAPDH在机械损伤中表达最稳定[41]。
总之,综合国内外学者对不同昆虫、不同试验条件下内参基因的筛选研究,结果表明同一物种同一内参基因在不同生理条件下通常并不稳定。而不同物种中同源内参基因也没有绝对的通用性,它们的稳定性不尽相同。因此,在所有条件下都稳定表达的理想内参基因并不存在,在研究目标基因表达水平时,应根据具体条件选择合适的内参基因。
3内参基因稳定性评价及数据分析方法
内参基因进行表达稳定性评价之前,首先需要通过 qRT-PCR分析内参基因引物的扩增效率。只有扩增效率在一定范围内(90%~105%);熔解曲线以单峰最佳,且Tm值(扩增产物解链温度)至少要大于75 ℃,才符合进行基因表达稳定性评价的要求[68]。
目前,评价内参基因稳定性的分析方法有多种,主要包括GeNorm[69]、NormFinder[70]和BestKeeper[71]等程序。Xie等将这3种分析方法整合成一个网络分析工具——RefFinder,能够综合评价和筛选合适的内参基因[72]。
GeNorm是Jo Vandesompele于2002年编写的一款软件,专门用于qRT-PCR方法分析内参基因的稳定性。其原理是不管在何种试验下,2个理想内参基因表达水平的比值均保持一致,不受基因表达丰度差异的影响,表达水平比值变异度的增加意味着二者中其一或全部基因表达稳定性的降低[73]。该程序将某一“管家基因”与其他“管家基因”表达水平进行两两比值,转换为对数后将其平均标准差作为基因表达稳定性的平均值M。对所有候选“管家基因”的表达稳定度排序,M值越大,稳定性越低;M值越小,稳定性越高。GeNorm程序可以筛选不同试验条件下最适内参基因的数量,选择出2个以上最优组合的内参基因,并非传统的单一内参基因,有利于減少系统偏差,得到更加可靠的相对定量结果[74]。另外,GeNorm程序可以对标准化因子的配对变异V值进行分析,以确定所需内参基因的最适数目。将Vn/Vn+1比值与默认的V值(0.15)进行比较,如果Vn/Vn+1大于0.15,则有必要引入第n+1个基因;反之,则不必引入新的内参基因[75]。GeNorm软件下载地址:http://medgen.ugent.be/~jvdesomp/genorm/index.php。
NormFinde是Claus于2004年編写的一款软件,建立在一个固定的统计学框架上,通过测定每个基因的稳定性来评估不同条件下候选内参基因和样本之间的变化[76],筛选出合适的内参基因。其运行原理与GeNorm程序类似,通过测定每个基因的稳定值,再根据稳定值的大小排序,值越大稳定性越差;反之,稳定性越好,最终将表达稳定值最小的基因作为内参基因。不足之处是只能选择1个合适的内参基因作标准[77]。NormFinder软件下载地址:http://www.mdl.dk/publicationsnormfinder.htm。
BestKeeper是Pfaffl等于2004年编写的一款分析软件,用于分析内参基因的表达稳定性和目标基因的表达水平,其优点在于可以同时比较100个样品中10个内参基因和10个目标基因的表达水平[71,78]。其运行原理是先比较每个基因之间Ct值产生配对的相关系数(r)、变异系数(CV)和标准偏差(s),再根据标准偏差和变异系数的大小排序,其中标准偏差和变异系数越小,稳定性越好;反之,稳定性越差[79]。BestKeeper不仅可以分析内参基因表达的稳定性,也可以比较目标基因的表达水平。BestKeeper软件下载地址:http://www.gene-quantif ication.de/best-keeper.html。
GeNorm、NormFinder和Best-Keeper均是通过不同的统计学分析方法筛选合适的内参基因,但由于这些软件具有不同的算法,可能导致彼此之间得出的最佳内参基因不尽相同[80]。因此,可以用RefFinder整合上述3种分析方法,并对每个基因单独用3种分析方法评价排名求几何平均值,得到一个综合排名指数,若指数越小,说明该内参基因越稳定[81]。
4小结
目前还没有一种理想的内参基因可以完全用于同种昆虫的不同细胞和组织,更没有一种理想的内参基因可以完全用于不同种昆虫的细胞和组织,即不同昆虫同一生物因素或非生物因素条件下,选择的内参基因不同;同种昆虫不同生物因素或非生物因素条件下,选择的内参基因也不尽相同。另外,不同学者在同种昆虫相同试验条件下,筛选出并非完全一致的内参基因,这是由不同学者选择不同候选“管家基因”所致。
为了获得真实可靠的试验结果,在进行基因相对定量研究时,由于不同的样本存在RNA的产量、质量和反转录效果上的差异,需要选择合适的内参基因进行校正[82-83]。从目前已有的报道来看,不同物种在不同条件下的最佳内参基因往往存在较大差异[84-86]。因此,不能盲目地选择校正目的基因的内参基因。任何一种“管家基因”均只在一定类型的细胞或试验因素下表达恒定,在其他类型的细胞或试验因素下可能发生较大变化,有时可以是十几倍、几十倍甚至上百倍的差异[87-88]。倘若盲目地选择内参基因,可能导致本应微量表达的基因难以发现,或者产生错误甚至相反的结论。如研究烟粉虱目标基因表达时往往采用常见的ACT作为内参基因[89-90],而Su等发现烟粉虱ACT在许多条件下的表达极不稳定[7]。因此,研究目标基因的表达时,需要根据样本的不同,选择合适而稳定的内参基因进行校正和标准化。
另外,在基因组测序和基因芯片分析结果的推动下,新内参基因的筛选有了新的途径[91]。常用内参基因由于表达稳定性差可能逐渐被新的内参基因取代。目前,基于基因芯片的表达数据和EST数据库筛选出不同试验条件下均能稳定表达的基因,这些基因可作为候选内参基因,并通过qRT-PCR试验验证其表达稳定性,再与传统内参基因的表达稳定性进行比较,有助于准确无误地揭示昆虫基因表达的内在规律。然而,近年来使用基因芯片数据和EST数据库筛选新内参基因的研究较少,尤其在昆虫研究中更是未见报道,许多研究者仍然沿用常用基因作为内参。因此,在挖掘新内参基因的道路上,还需要寻找更加精确有效的方法。
参考文献:
[1]Li R M,Xie W,Wang S L,et al. Reference gene selection for qRT-PCR analysis in the sweetpotato whitefly,Bemisia tabaci(Hemiptera:Aleyrodidae)[J]. PLoS One,2013,8(1):e53006.
[2]Nolan T,Hands R E,Bustin S A. Quantification of mRNA using real-time RT-PCR[J]. Nature Protocols,2006,1(3):1559-1582.
[3]Peters I R,Helps C R,Hall E J,et al. Real-time RT-PCR:considerations for efficient and sensitive assay design[J]. Journal of Immunological Methods,2004,286(1/2):203-217.
[4]Wang H,Hanash S. Mass spectrometry based proteomics for absolute quantification of proteins from tumor cells[J]. Methods,2015,81(15):34-40.
[5]Li Z Y,Zhu Y B,Wang N,et al. Selection of optimal qRT-PCR reference genes for gene expression studies in foxtail millet[J]. Indian Journal of Genetics and Plant Breeding,2015,75(2):225-231.
[6]Suzuki T,Higgins P J,Crawford D R. Control selection for RNA quantitation[J]. BioTechniques,2000,29(2):332-337.
[7]Su Y,He W B,Wang J,et al. Selection of endogenous reference genes for gene expression analysis in the Mediterranean species of the Bemisia tabaci(Hemiptera:Aleyrodidae) complex[J]. Journal of Economic Entomology,2013,106(3):1446-1455.
[8]Yuan M,Lu Y,Zhu X,et al. Selection and evaluation of potential reference genes for gene expression analysis in the brown planthopper,Nilaparvata lugens(Hemiptera:Delphacidae) using reverse-transcription quantitative PCR[J]. PLoS One,2014,9(1):e86503.
[9]Huggett J,Dheda K,Bustin S,et al. Real-time RT-PCR normalisation;strategies and considerations[J]. Genes and Immunity,2005,6(4):279-284.
[10]Gutierrez L,Mauriat M,Guénin S,et al. The lack of a systematic validation of reference genes:a serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR) analysis in plants[J]. Plant Biotechnology Journal,2008,6(6):609-618.
[11]Liang P,Guo Y J,Zhou X G,et al. Expression profiling in Bemisia tabaci under insecticide treatment:indicating the necessity for custom reference gene selection[J]. PLoS One,2014,9(1):e87514.
[12]Bonaventure A O,Jose M L,Ivana H R O,et al. Evaluation of reference genes for insect olfaction studies[J]. Parasites & Vectors,2015,8(1):1-15.
[13]Wanchin Y,Jiamayne L,Yickching W,et al. Evaluation of suitable reference genes for qRT-PCR gene expression normalization in reproductvie,vegetative tissues and during fruit development in oil palm[J]. PCTOC,2014,116(1):55-66.
[14]Teng X L,Zhang Z,He G L,et al. Validation of reference genes for quantitative expression analysis by real-time RT-PCR in four lepidopteran insects[J]. Journal of Insect Science,2012,12(60):1-17.
[15]Cao S,Zhang X,Ye N,et al. Evaluation of putative internal reference genes for gene expression normalization in Nannochloropsis sp. by quantitative real-time RT-PCR[J]. Biochemical and Biophysical Research Communications,2012,424(1):118-123.
[16]Wan H,Zhao Z,Qian C,et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry,2010,399(2):257-261.
[17]Lilly S T,Drummond R S,Pearson M N,et al. Identification and validation of reference genes for normalization of transcripts from virus-infected Arabidopsis thaliana[J]. Molecular Plant-Microbe Interactions,2011,24(3):294-304.
[18]Ferdous A S,Islam M T,Alam S S,et al. Identification of stable reference genes for quantitative PCR in jute under different experimental conditions:an essential assessment for gene expression analysis[J]. Australian Journal of Crop Science,2015,9(7):646-655.
[19]Khanlou K M,van Bockstaele E. A critique of widely used normalization software tools and an alternative method to identify reliable reference genes in red clover(Trifolium pratense L.)[J]. Planta,2012,236(5):1381-1393.
[20]Sekalska B,Ciechanowicz A,Dolegowska B,et al. Optimized RT-PCR method for assaying expression of monocyte chemotactic protein type 1 (MCP-1) in rabbit aorta[J]. Biochemical Genetics,2006,44(3/4):133-143.
[21]Provenzano M,Moceilin S. Complementary techniques:validation of gene expression data by quantitative real time PCR[J]. Adv Exp Med Biol,2007,593:66-73.
[22]Radonic A,Thulke S,Mackay I M,et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochem Biophys Res Commun,2004,313(4):856-862.
[23]陳立华,张月华,何庆玲,等. 蓖麻蚕基因转录表达分析的内参基因筛选[J]. 河北农业大学学报,2014,37(6):78-84.
[24]袁淼. 褐飞虱内参基因的筛选及精氨酸激酶基因的分子特性研究[D]. 武汉:华中农业大学,2014:50-54.
[25]Sun W,Jin Y,He L,et al. Suitable reference gene selection for different strains and developmental stages of the carmine spider mite,Tetranychus cinnabarinus,using quantitative real-time PCR[J]. Journal of Insect Science,2010,10(208):1-12.
[26]王佳,赵静,刘映红. 柑橘大实蝇内参基因的评估[J]. 昆虫学报,2014,57(12):1375-1380.
[27]Lü Z C,Wang L H,Dai R L,et al. Evaluation of endogenous reference genes of Bactrocera(Tetradacus) minax by gene expression profiling under various experimental conditions[J]. Florida Entomologist,2014,97(2):597-604.
[28]王刘豪. 柑橘大实蝇转录组测序和滞育相关基因克隆研究[D]. 新乡:河南科技学院,2013:53-59
[29]宋旺,王小纪,郭瑞坚,等. 花绒寄甲荧光定量PCR分析中内参基因的选择[J]. 西北农业学报,2015,24(2):156-161.
[30]Bansal R,Mamidala P,Mian M A,et al. Validation of reference genes for gene expression studies in Aphis glycines (Hemiptera:Aphididae)[J]. Entomological Society of America,2012,105(4):1432-1438.
[31]郭长宁. 金纹细蛾内参基因克隆分析及稳定性评价[D]. 杨凌:西北农林科技大学,2014:32-49.
[32]徐素平. 棉蚜气味结合蛋白基因克隆和表达分析[D]. 武汉:华中农业大学,2014:35-47.
[33]陈芳,陆永跃. 热胁迫下棉花粉蚧内参基因的筛选[J]. 昆虫学报,2014,57(10):1146-1154.
[34]杨丽红. 柑橘全爪螨Panonychus citri(McGregor)对热胁迫的响应机制研究[D]. 重庆:西南大学,2011:99-106.
[35]Lu Y,Yuan M,Gao X,et al. Identification and validation of reference genes for gene expression analysis using quantitative PCR in Spodoptera litura(Lepidoptera:Noctuidae)[J]. PLoS One,2013,8(7):e68059.
[36]van Hiel M B,van Wielendaele P,Temmerman L,et al. Identification and validation of housekeeping genes in brains of the desert locust Schistocerca gregaria under different developmental conditions[J]. BMC Molecular Biology,2009,10(1):56.
[37]Toutges M J,Hartzer K,Lord J,et al. Evaluation of reference genes for quantitative polymerase chain reaction across life cycle stages and tissue types of Tribolium castaneum[J]. Journal of Agricultural and Food Chemistry,2010,58(16):8948-8951.
[38]Bagnall N H,Kotze A C. Evaluation of reference genes for real-time PCR quantification of gene expression in the Australian sheep blowfly,Lucilia cuprina[J]. Medical and Veterinary Entomology,2010,24(2):176-181.
[39]Mamidala P,Rajarapu S P,Jones S C,et al. Identification and validation of reference genes for quantitative real-time polymerase chain reaction in Cimex lectularius[J]. Journal of Medical Entomology,2011,48(4):947-951.
[40]Zhang S,An S,Li Z,et al. Identification and validation of reference genes for normalization of gene expression analysis using qRT-PCR in Helicoverpa armigera(Lepidoptera:Noctuidae)[J]. Gene,2015,555(2):393-402.
[41]Shakeel M,Zhu X,Kang T H,et al. Selection and evaluation of reference genes for quantitative gene expression studies in cotton bollworm,Helicoverpa armigera(Lepidoptera:Noctuidae)[J]. Journal of Asia-Pacific Entomology,2015,18(2):123-130.
[42]Cardoso G A,Matiolli C C,Azeredoespin A M L D,et al. Selection and validation of reference genes for functional studies in the Calliphoridae family[J]. Journal of Insect Science,2014,14(2):2.
[43]李柯,陰环,奚耕思,等. 黏虫β-actin基因cDNA的克隆、序列分析及表达量检测[J]. 昆虫知识,2010,47(6):1089-1094.
[44]吴家红,程金芝,孙宇,等. 白纹伊蚊基因表达定量PCR内参基因的选择[J]. 中国人兽共患病学报,2011,27(5):432-435.
[45]彭然. 家蚕常用内参基因的稳定性分析及两种实时荧光定量PCR方法比较[D]. 苏州:苏州大学,2012:19-33.
[46]Shen G M,Jiang H B,Wang X N,et al. Evaluation of endogenous references for gene expression profiling in different tussues of the oriental fruit fly Bactrocera dorsalis(Diptera:Tephritidae)[J]. BMC Molecular Biology,2011,11(1):76.
[47]吴玉,翟渊粉,黄明霞,等. 家蚕常用内参基因稳定性分析及丝蛋白相关基因表达调控研究[J]. 中国细胞生物学学报,2013,35(4):423-431.
[48]Paim R M,Pereira M H,Ponzio R D,et al. Validation of reference genes for expression analysis in the salivary gland and the intestine of Rhodnius prolixus(Hemiptera:Reduviidae) under different experimental conditions by quantitative real-time PCR[J]. BMC Research Notes,2012,5(1):128.
[49]Shen G M,Jiang H B,Wang X N,et al. Evaluation of endogenous references for gene expression profiling in different tissues of the oriental fruit fly Bactrocera dorsalis(Diptera:Tephritidae)[J]. BMC Molecular Biology,2010,11(1):76.
[50]Majerowicz D,Alves-Bezerra M,Logullo R,et al. Looking for reference genes for real-time quantitative PCR experiments in Rhodnius prolixus(Hemiptera:Reduviidae)[J]. Insect Molecular Biology,2011,20(6):713-722.
[51]Hornkova D,Matoukova P,Kindl J,et al. Selection of reference genes for real-time polymerase chain reaction analysis in tissues from Bombus terrestris and Bombus lucorum of different ages[J]. Analytical Biochemistry,2010,397(1):118-120.
[52]Rajarapu S P,Mamidala P,Mittapalli O. Validation of reference genes for gene expression studies in the emerald ash borer (Agrilus planipennis)[J]. Insect Science,2012,19(1):41-46.
[53]孟闪闪,马文静,刘小宁,等. β-actin基因在荒漠昆虫光滑鳖甲和小胸鳖甲中表达的稳定性研究[J]. 天津农业科学,2014,20(2):1-6.
[54]唐婷,柳峰松,任国栋. 谢氏宽漠王β-actin基因cDNA克隆、序列分析及表达量检测[J]. 昆虫学报,2008,51(11):1210-1215.
[55]Ponton F,Chapuis M P,Pernice M,et al. Evaluation of potential reference genes for reverse transcription-qPCR studies of physiological responses in Drosophila melanogaster[J]. Journal of Insect Physiology,2011,57(6):840-850.
[56]Scharlaken B,de Graaf D C,Goossens K,et al. Reference gene selection for insect expression studies using quantitative real-time PCR:The head of the honeybee,apis mellifera,after a bacterial challenge[J]. Journal of Insect science,2008,8(33):1-10.
[57]Maroniche G A,Sagadín M,Mongelli V C,et al. Reference gene selection for gene expression studies using RT-qPCR in virus-infected planthoppers[J]. Virology Journal,2011,8(1):1-8.
[58]Xue J L,Salem T Z,Turney C M,et al. Strategy of the use of 28S rRNA as a housekeeping gene in real-time quantitative PCR analysis of gene transcription in insect cells infected by viruses[J]. Journal of Virological Methods,2010,163(2):210-215.
[59]Lord J C,Hartzer K,Toutges M,et al. Evaluation of quantitative PCR reference genes for gene expression studies in Tribolium castaneum after fungal challenge[J]. Journal of Microbiological Methods,2010,80(2):219-221.
[60]Niu J Z,Cappelle K,de Miranda J R,et al. Analysis of reference gene stability after Israeli acute paralysis virus infection in bumblebees Bombus terrestris[J]. Journal of Invertebrate Pathology,2014,115(1):76-79.
[61]高新菊,沈慧敏. 二斑葉螨对甲氰菊酯的抗性选育及解毒酶活力变化[J]. 昆虫学报,2011,54(1):64-69.
[62]Niu J Z,Dou W,Ding T B,et al. Evaluation of suitable reference genes for quantitative RT-PCR during development and abiotic stress in Panonychus citri(McGregor) (Acari:Tetranychidae)[J]. Molecular Biology Reports,2012,39(5):5841-5849.
[63]Jiang H B,Liu Y H,Tang P A,et al. Validation of endogenous reference genes for insecticide-induced and developmental expression profiling of Liposcelis bostsrychophila(Psocoptera:Liposcelididae)[J]. Molecular Biology Reports,2010,37(2):1019-1029.
[64]Ponton F,Chapuis M P,Pernice M,et al. Evaluation of potential reference genes for reverse transcription qPCR studies of physiological responses in Drosophila melanogaster[J]. Journal of Insect Physiology,2011,57(6):840-850.
[65]Spanier K I,Leese F,Mayer C,et al. Predator-induced defences in Daphnia pulex:selection and evaluation of internal reference genes for gene expression studies with real-time PCR[J]. BMC Molecular Biology,2010,11(1):50.
[66]Chapuis M P,Tohidi-Esfahani D,Dodgson T,et al. Assessment and validation of a suite of reverse transcription quantitative PCR reference genes for analyses of density-dependent behavioural plasticity in the Australian plague locust[J]. BMC Molecular Biology,2011,12(1):7.
[67]Wang Y,Wang Z K,Huang Y,et al. Identification of suitable reference genes for gene expression studies by qRT-PCR in the blister beetle Mylabris cichorii[J]. Journal of Insect Science,2014,14:94.
[68]Tellinghuisen J,Spiess A N. Comparing real-time quantitative polymerase chain reaction analysis methods for precision,linearity,and accuracy of estimating amplification efficiency[J]. Analytical Biochemistry,2014,449(15):76-82.
[69]Vandesompele J,de Preter K,Pattyn F,et al. Accurate normalization of real-time quantitativeRT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol,2002,3(7):34.
[70]Andersen C,Ledet-Jensen J,Orntoft T. Normalization of real-time quantitative RT-PCR data:a model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J]. Cancer Res,2004,64(15):5245-5250.
[71]Pfaffl M W,Tichopad A,Prgomet C,et al. Determination of stable housekeeping genes,differentially regulated target genes and sample integrity:BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters,2004,26(6):509-515.
[72]Xie F,Sun G,Stiller J W,et al. Genome-wide functional analysis of the cotton transcriptome by creating an integrated EST database[J]. PLoS One,2011,6(11):e26980.
[73]胡金川. 如何利用geNorm軟件筛选基因表达测定的内参基因[J]. 中华检验医学杂志,2008,31(8):918.
[74]张艳君,朱志峰,陆融,等. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展,2007,34(5):546-550.
[75]Rivera-Vega L,Mamidala P,Koch J L,et al. Evaluation of reference genes for expression studies in ash (Fraxinus spp.)[J]. Plant Molecular Biology Reporter,2012,30(1):242-245.
[76]Ferreira E,Cronjé M J. Selection of suitable reference genes for quantitative real-time PCR in apoptosis-induced MCF-7 breast cancer cells[J]. Molecular Biotechnology,2012,50(2):121-128.
[77]Gao Z H,Wei J H,Yang Y,et al. Selection and validation of reference genes for studying stress-related agarwood formation of Aquilaria sinensis[J]. Plant Cell Reports,2012,31(9):1759-1768.
[78]Kumar V,Sharma R,Trivedi P C,et al. Traditional and novel references towards systematic normalization of qRT-PCR data in plants[J]. Australian Journal of Crop Science,2011,5(11):1455-1468.
[79]Zhang L,Cao Y L,Liu X,et al. In-depth analysis of the endogenous reference genes used in the quantitative PCR detection systems for rice[J]. European Food Research and Technology,2012,234(6):981-993.
[80]Wang Q,Ishikawa T,Michiue T,et al. Stability of endogenous reference genes in postmortem human brains for normalization of quantitative real-time PCR data:comprehensive evaluation using geNorm,NormFinder,and BestKeeper[J]. International Journal of Legal Medicine,2012,126(6):943-952.
[81]Huang L,Yan H,Jiang X,et al. Reference gene selection for quantitative real-time reverse-transcriptase PCR in orchardgrass subjected to various abiotic stresses[J]. Gene,2014,553(2):158-165.
[82]Bionaz M,Loor J J. Identification of reference genes for quantitative real-time PCR in the bovine mammary gland during the lactation cycle[J]. Physiological Genomics,2007,29(3):312-319.
[83]Borges A F,Fonseca C,Ferreira R B,et al. Reference gene validation for quantitative RT-PCR during biotic and abiotic stress in Vitis vinifera[J]. PLoS One,2014,9(10):e111399.
[84]Rodrigues T B,Khajuria C,Wang H,et al. Validation of reference housekeeping genes for gene expression studies in western corn rootworm (Diabrotica virgifera virgefera)[J]. PLoS One,2013,9(10):e109825.
[85]Han G Y,Li X M,Zhang T,et al. Cloning and tissue-specific expression of a chitin deacetylase gene from Helicoverpa armigera(Lepidoptera:Noctuidae)and its response to Bacillus thuringiensis[J]. Journal of Insect Science,2015,15(1):95.
[86]Zheng Y T,Li H B,Lu M X,et al. Evaluation and validation of reference genes for qRT-PCR normalization in Frankliniella occidentalis(Thysanoptera:Thripidae )[J]. PLoS One,2014,9(10):e111369.
[87]Iskandar H M,Simpson R S,Casu R E,et al. Comparison of reference genes for quantitative real-time polymerase chain reaction analysis of gene expression in sugarcane[J]. Plant Molecular Biology reporter,2004,22(4):325-337.
[88]Liu D W,Chen S T,Liu H P. Choice of endogenous control for gene expression in nonsmall cell lung cancer[J]. The European Respiratory Journal,2005,26(6):1002-1008.
[89]Li J M,Su Y L,Gao X L,et al. Molecular characterization and oxidative stress response of an intracellular Cu/Zn superoxide dismutase (CuZnSOD) of the whitefly,Bemisia tabaci[J]. Archives of Insect Biochemistry and Physiology,2011,77(3):118-133.
[90]Luan J B,Li J M,Varela N,et al. Global analysis of the transcriptional response of whitefly to tomato yellow leaf curl China virus reveals the relationship of coevolved adaptations[J]. Journal of Virology,2011,85(7):3330-3340.
[91]Chang C W,Cheng W C,Chen C R,et al. Identification of human housekeeping genes and tissue-selective genes by microarray meta-analysis[J]. PLoS One,2011,6(7):e22859.

