油桃花芽自然休眠诱导期FAR1/FHY3基因的表达分析
郇 蕾,王旭旭,刘文海,李 玲,2,高东升,2*
1.山东农业大学园艺科学与工程学院,山东 泰安 271018
2.作物生物学国家重点实验室,山东 泰安 271018
3.潍坊工程职业学院,山东 潍坊 261000
油桃花芽自然休眠诱导期FAR1/FHY3基因的表达分析
郇 蕾1,王旭旭1,刘文海3,李 玲1,2,高东升1,2*
1.山东农业大学园艺科学与工程学院,山东 泰安 271018
2.作物生物学国家重点实验室,山东 泰安 271018
3.潍坊工程职业学院,山东 潍坊 261000
FAR-RED ELONGATED HYPOCOTYL 3(FHY3)和它的同源基因FAR-RED-IMPAIREDRESPONSE 1(FAR1)参与远红光、脱落酸(Abscisic acid,ABA)对拟南芥种子休眠的调节,然而FHY3/FAR1在木本植物花芽休眠中的表达尚不清楚。本研究以八年生‘中油四号’油桃花芽为材料,利用NCBI找出桃树上的FAR1/FHY3同源基因,测定了同源性最高的5个基因在休眠诱导期的表达,并在休眠诱导期分别用脱落酸及远红光处理桃树,荧光定量检测了基因的表达量变化。结果表明,桃中同源性最高的Prupe.3G186100、Prupe.3G186200、Prupe.4G115100、Prupe.3G186000 4个基因在休眠诱导期均有上调,且受ABA诱导表达上调,受远红光诱导表达下调,而Prupe.2G327900在休眠诱导期表达下调,受ABA诱导下调,受远红光诱导上调。我们推测,PpFRSs基因可能通过不同的模式参与油桃花芽休眠诱导的调控。
ABA;花芽休眠诱导;表达分析;远红光;FHY3/FAR1
光是影响植物生长发育的重要环境信号。高等植物在不断进化的过程中,形成了一个光感受器的网络来感受光环境的变化,适应环境,影响自身生长发育[1]。在这些光感受器中,光敏色素能够
调控种子的休眠过程。光敏色素A(phytochrome A,phyA)是高等植物中调控远红光诱导反应的主要光受体。phyA介导的远红光能促进种子休眠,这可能由phyA介导的高辐照度反应(High irradiance reaction,HIR)引起的[2]。
FAR-RED ELONGATED HYPOCOTYL 3(FHY3)和它的同源基因FAR-RED-IMPAIREDRESPO NSE 1(FAR1)编码一类起源于转座子酶的植物所特有的转录因子[3-5],被认为是phyA信号传导途径中重要的调控因子[6,7]。FHY3和FAR1能够直接结合到FAR-REDELONGATED HYPOCOTYL 1(FHY1)和FHY1-like(FHL)的启动子上,激活它们的表达,而FHY1和FHL的蛋白产物,在phyA的进核途径中起着重要的作用[5,8]。对fhy3和far1突变体进行基因芯片分析,发现一系列由远红光诱导的基因在转录水平上受到影响,进一步说明该同源基因参与phyA信号传导[3]。FAR1,FHY3作为转录因子能够转录调节不同的靶基因来控制多种细胞过程。
目前对FAR1,FHY3的研究主要涉及UV-B信号[9]、生物钟[10-12]、开花、叶绿体的生物起源[12]、叶绿素的生物合成[13]、细胞程序性死亡及活性氧的平衡[14]、ABA信号[15]、光信号与淀粉合成[16]。在拟南芥中,atfar1和atfhy3突变体植株的种子萌发率都比野生型的低,而双突变体的更低,说明了FAR1、FHY3正调节拟南芥种子的萌发。
木本植物芽休眠是植物生长发育过程中的一种暂停现象,是植物经过长期演化而获得的一种对环境及季节性变化的生物学适应性[17]。休眠的诱导是植物开始休眠的前提,植物一旦被诱导进入休眠就必须满足需冷量以解除休眠,方可正常生长发育[18]。休眠的发生受温度、光照等多种自然因子诱导[19],其中,光照被认为是主要的环境信号因子,脱落酸(Abscisic acid,ABA)作为一种重要的植物激素,调控种子休眠、芽休眠[20]。研究花芽诱导的分子机制,找到诱导芽休眠的主要调控途径及关键基因,对调控设施果树的栽培模式,改变设施鲜果的上市时间具有重要意义。本研究中以油桃‘中油四号’(Prunus persica var.nectariana cv.Zhongyousihao)为研究材料,利用实时荧光定量PCR技术检测了桃树花芽FAR1,FHY3在休眠诱导期间及脱落酸、远红光处理条件下的表达变化,旨在为进一步揭示FAR1,FHY3在转录水平上与芽休眠诱导的关系奠定基础。
1 材料与方法
1.1 材 料
试验于2015~2016年在山东农业大学设施果树实验室及山东农业大学园艺试验站温室大棚内进行,选取中油四号油桃(Prunus persica var.nectariana cv.Zhongyousihao)为试材。
1.2 方 法
1.2.1 ‘中油四号’油桃花芽休眠诱导的界定 据新梢停长时间初步界定休眠诱导。测量新梢生长长度,并观察叶片。新梢生长长度不再增加且枝条上只有老叶片,完全没有新叶片分生出来时,判定为新梢停长。休眠诱导发生在新梢停长后。
采用清水插枝法进一步测定休眠状态。每隔7 d采集树冠外围带有花芽和叶芽的10根一年生枝条,除去叶片,剪去长的顶稍,将枝条基部剪齐,插入盛水2~3 cm深的塑料小桶中,在植物光照培养箱中进行培养。培养条件为:气温25°C,光照强度40 μmol·m-2·s-1,昼/夜为14 h/10 h,相对空气湿度80%~90%,每天换水1次,每次剪掉基部2~3 mm,露出新茬。记录第一颗叶芽萌发的时间(d),依此界定休眠状态。当10 d≤第1颗芽萌发所需时间≤6周时,表明芽体已进入自然休眠诱导期。若连续6周未萌发,确定桃芽休眠诱导期结束,转入深休眠期。随着休眠的进行,需冷量不断满足,萌芽所需的时间变短,当再次出现10 d≤第1颗芽萌发所需时间≤6周时,表明芽体进入休眠解除期。
1.2.2 自然条件下自然休眠诱导期取材 每隔7 d取一年生枝上的花芽,立刻用液氮速冻,-80℃保存备用。
1.2.3 外源ABA处理 随机采集一年生枝条200根,插入清水中预培养两天(温度25℃,光照强度为40 μmol·m-2·s-1,昼/夜:12 h/12 h,相对空气湿度80%),分为两组,分别为①对照(CK),清水+0.5%Trtion 100②脱落酸处理(ABA),100 μM ABA+0.5%Trtion 100,分时间段取样,用液氮冷冻备用。
1.2.4 远红光处理 自8月份选择生长一致、长势健壮的桃树进行远红光补光处理,并以不补光为对照。光源为18 W植物生长LED灯管,主峰波长730 nm。桃树成直线排列,与补光灯管垂直,且在灯管的投影范围之内。每天20:00~4:00进行人工补光8 h,补光期间,进行常规管理,9月初取一年生枝上的花芽、树皮和叶片,立刻用液氮速冻,-80℃保存备用。
1.2.5 目的基因的查找及生物信息学分析 根据拟南芥FAR1、FHY3登陆的基因,在拟南芥基因组数据库(https://www.arabidopsis.org/)中查得FAR1、FHY3的蛋白序列,与已建立的桃全基因组氨基酸序列(https://www.rosaceae.org/species/prunus/prunus_persica)进行Blast序列比对,初步筛选出同源氨基酸序列的候选基因,将这些基因通过Pfam(http://pfam.xfam.org/)进行分析,去除无FAR1/FHY3保守结构域的基因序列;再将候选的FAR1/FHY3相关基因序列通过Clustal W 工具进行多序列比对,去除重复序列。用MEGA 5.0软件进行系统发育树的构建,采用邻接法(Neighbor-joining method)构建进化树,自举检测(Bootstrap)次数为1000。
从桃数据库获得每一个FAR1相关基因的基因组序列和编码区序列,借助在线工具GSDS(http:// gsds.cbi.pku.edu.cn)绘制其内含子和外显子结构图。
1.2.6 总RNA的提取和qRT-PCR采用RNeasy Plus Mini Kit(Qiagen,Valencia,CA,USA)试剂盒提取样品RNA,通过大连TaKaRa公司PrimeScript™RT reagent Kit with gDNA Eraser(Perfect Real Time,Takara)试剂盒反转录获得cDNA。根据Prupe.3G186100、Prupe.3G186200、Prupe.4G115100、Prupe.3G186000、Prupe.2G327900的基因序列设计荧光定量特异性引物。采用Bio-Rad iCycler iQ荧光定量PCR仪,使用SYBR Premix Ex Taq试剂盒(TaKaRa,中国大连)进行荧光定量PCR反应。反应体系为SYBR Premix Ex Taq(TaKaRa,中国大连)12.5 μL,上游、下游引物各1 μL,cDNA 1 μL,ddH2O 9.5 μL,总反应体系25 μL。PCR反应条件:95℃预变性30 s,95℃变性5 s,60℃退火30 s,40次循环,反应结束后分析荧光值变化曲线和融解曲线。每个样品重复3次,取平均值,采用Comparative CT(2−ΔΔCt)的方法计算实验结果,采用GraphPad Prism 6.0软件作图。

表1 引物序列Table 1 Primer sequences of 5 FRS genes used for qRT-PCR
2 结果与分析
2.1 休眠进程界定

图1 中油四号桃树休眠诱导的界定Fig.1 Definition for dormancy induction process in‘Zhong You Tao 4’peach
由图1可知,自然条件下,9月11日中油四号桃树新梢平均长度达到52.63 cm后不再增加,此时第一芽萌发所需时间为12 d,初步确定休眠诱导期的开始。2015年8月7日至10月22日采集的‘中油四号’油桃新梢进行清水插枝法培养,统计第一颗芽萌发时间结果如图1所示。8月28至9月4日第1颗芽的萌发时间均小于10 d,认为其仍处于自然生长期;而在9月11日,新梢第1颗芽的萌发时间为12 d,9月11日到10月2日,第1颗芽的萌发时间大于10 d小于6周,表明芽体处于休眠诱导期。10月9日,第一颗芽萌发时间为51 d,大于6周,已经进入深休眠期。
2.2 桃FAR1基因的系统进化分析
经BLASTp同源搜索和Pfam在线工具分析结构域,共获得71条具典型FAR1结构域的序列。将其与拟南芥FAR1/FHY3基因一起进行进化树分析。根据系统发育进化树(图2),得到5个与拟南芥 FAR1/FHY3同源性较高基因,Prupe.3G186100、Prupe.3G186200、Prupe.4G115100、Prupe.3G186000、Prupe.2G327900,我们将其统称为FHY3/FAR1-related genes(FRSs)。由图三可知,拟南芥FAR1(AT4G15090)、FHY3(AT3G22170)基因均含内含子6个,外显子7个,总长度分别为4079 bp和3867 bp。根据桃基因组公布的基因序列及基因结构图分析表明,Prupe.3G186100基因组序列为5216 bp,含有长度为2520 bp的CDS序列,编码839个氨基酸,位于基因组第三条染色体上,由7个外显子和6个内含子构成;Prupe.3G186200基因组序列为6247 bp,含有长度为2592 bp的CDS序列,编码863个氨基酸,位于基因组第三条染色体上,由7个外显子和6个内含子构成;Prupe.4G115100基因组序列为5004 bp,含有长度为2673 bp的CDS序列,编码890个氨基酸,位于基因组第四条染色体上,由7个外显子和6个内含子构成;Prupe.3G186000基因组序列为4269 bp,含有长度为2574 bp的CDS序列,编码857个氨基酸,位于基因组第三条染色体上,由8个外显子和7个内含子构成;Prupe.2G327900基因组序列为4348 bp,含有长度为2352 bp的CDS序列,编码783个氨基酸,位于基因组第二条染色体上,由8个外显子和7个内含子构成。
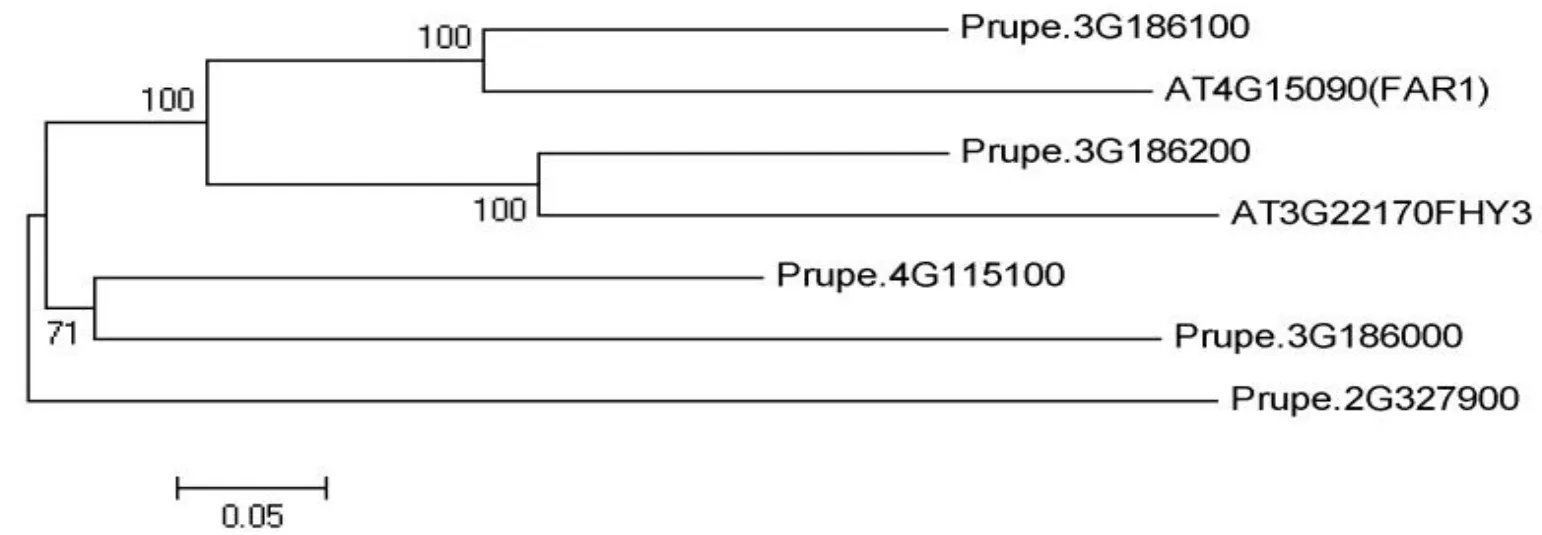
图2 桃与拟南芥中FAR1、FHY3基因系统进化树分析Fig.2 Phylogenetic tree of FAR1、FHY3 from nectarine and Arabidopsis

图3 桃与拟南芥中FAR1、FHY3基因的内含子分布图Fig.3 The distribution of introns in nectarine and Arabidopsis FAR1、FHY3 genes
2.3 自然条件下PpFHY3/PpFAR1在花芽休眠诱导期的表达分析
用RT-PCR分析FAR1s基因在‘中油四号’油桃花芽休眠诱导过程中的表达模式,结果如图4所示。Prupe.3G186100在整个自然休眠诱导期表达量呈上升趋势,至10月9日进入深休眠期,表达量持续上升,在9月25日差异显著。Prupe.3G186200和Prupe.4G115100从9月11日到9月25日,表达持续显著上升,在9月25日表达水平均达到最高值,Prupe.4G115100的表达量变化达到10倍以上,然后呈现降低的趋势。Prupe.3G186000在整个自然休眠诱导期表达量呈逐渐上升趋势,10月9日进入深休眠期,表达量下降。而Prupe.2G327900在整个自然休眠诱导期直至10月9日进入深休眠,表达量呈逐渐下降趋势。
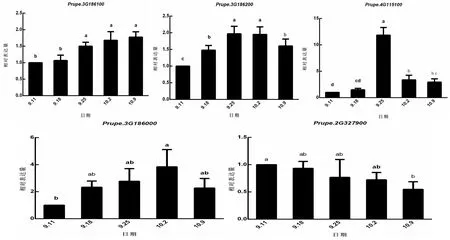
图4 PpFRSs基因在自然休眠诱导过程中的表达变化Fig.4 Relative gene expression of peach FRSs genes during dormancy induction process
2.4 ABA处理对桃花芽中PpFRSs基因的表达调控
对‘中油四号’油桃枝条进行100μM ABA处理后,分时段取花芽,提取cDNA为模板,用PpFRSs成员的特异引物进行了qRT-PCR分析。
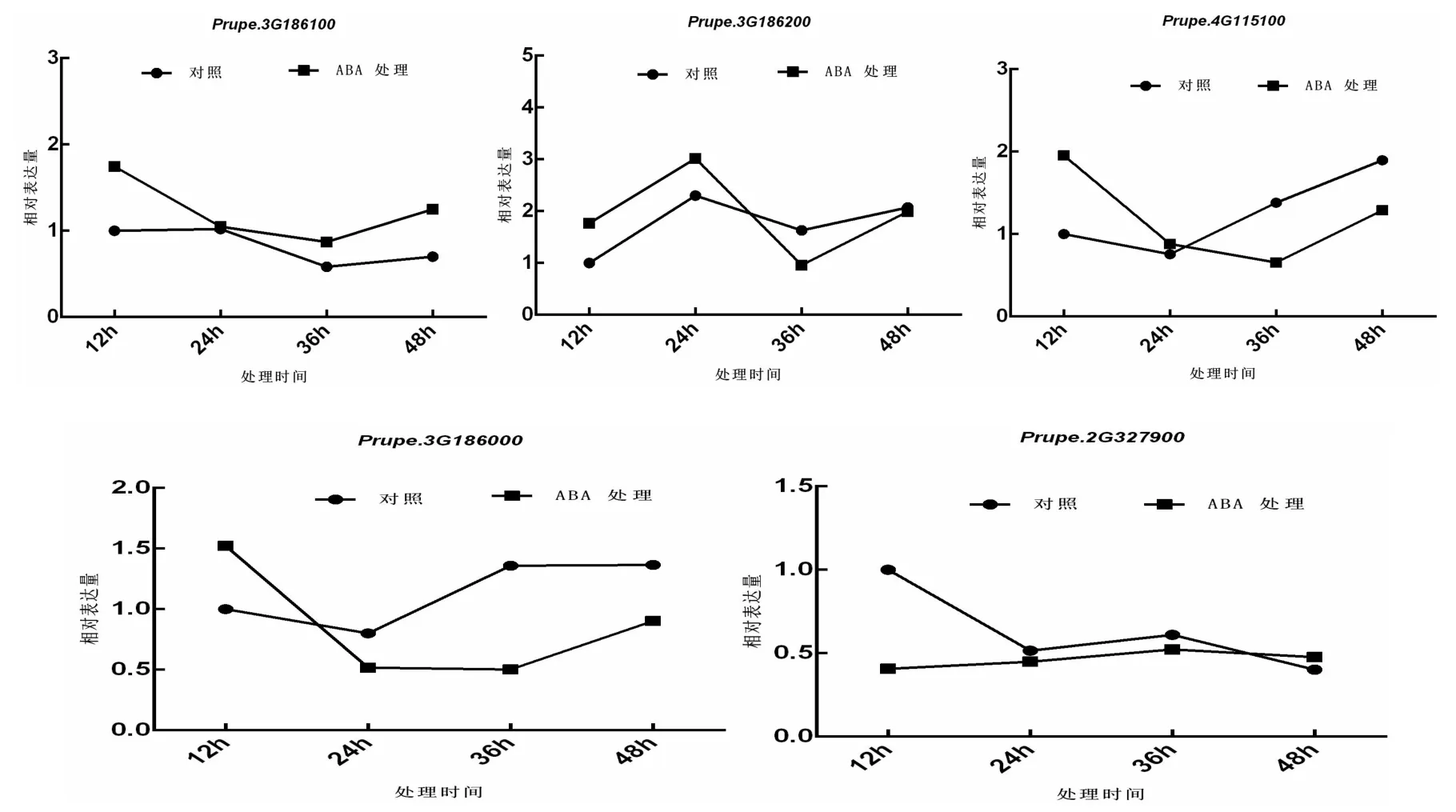
图5 PpFRSs在100 μM脱落酸处理下的表达变化Fig.5 Relative gene expression of PpFRSs treated by 100 μM ABA
Prupe.3G186100受ABA诱导表达上调,在处理12 h时差异最显著。Prupe.3G186200和Prupe.4G115100趋势一致,在处理0~24 h内,表达量上调,36~48 h表达量下调。Prupe.3G186000在处理12 h时表达量高于对照,24~48 h处理组表达下调。Prupe.2G327900在处理12~36 h表达量低于,48 h表达量高于对照。处理 12 h,除Prupe.2G327900表达下调,Prupe.3G186100、Prupe.3G186200、Prupe.4G115100、Prupe.3G186000的表达均上调。
2.5 远红光处理对桃花芽中FHY3/FAR1基因家族成员的表达调控

图6 PpFRSs在远红光处理下的表达变化Fig.6 Relative gene expression of PpFRSs treated by far-red
为了研究远红光对PpFRSs的诱导作用及在花芽中是否具有特异性,我们以一年生树皮和叶片作为参照,对PpFRSs的表达进行了分析。荧光定量显示(图6),远红光处理后,在花芽中,Prupe.3G186100、Prupe.3G186200、Prupe.4G115100、Prupe.3G186000的表达受诱导下调,Prupe.2G327900表达上调。在一年生韧皮部中,Prupe.3G186100、Prupe.2G327900的表达下调,Prupe.3G186200、Prupe.4G115100、Prupe.3G186000表达上调。叶片中,Prupe.3G186100、Prupe.3G186200、Prupe.2G327900受远红光诱导下调,Prupe.4G115100、Prupe.3G186000上调。Prupe.3G186100、Prupe.3G186200在不同组织中的变化趋势基本一致,均受远红光诱导下调,而Prupe.4G115100、Prupe.3G186100在花芽中特异性下调,一年生韧皮部和叶片中都诱导上调。Prupe.2G327900受远红光诱导在花芽中特异性上调,一年生韧皮部和叶片中表达下调。
3 讨论
FAR1、FHY3是一对同源蛋白,在phyA控制的远红光反应中起着重要的作用。在拟南芥基因组中,除FAR1、FHY3外,还有12个FHY3/FAR1相关的基因,命名为FRS(FHY3/FAR1-related genes)家族基因[4]。在以往的研究中指出,FHY3和FARl是由古老的MURA类转座酶进化而来,获得调控基因表达的能力[21]。本试验将拟南芥中的FAR1、FHY3蛋白序列比对桃树基因组,找到了71条类似序列。通过生物信息学分析,发现PpFRSs都含有FRS家族保守的核心转座酶结构域。序列比对及系统发育进化树表明,Prupe.3G186100与拟南芥FAR1(AT4G15090),Prupe.3G186200与拟南芥FHY3(AT3G22170)同源性最高,其次分别为Prupe.4G115100、Prupe.3G186000、Prupe.2G327900。基因结构图显示,Prupe.3G186100、Prupe.3G186200、Prupe.4G115100与拟南芥FAR1、FHY3外显子数目一致。
落叶果树的休眠是长期自然选择的结果,是其度过冬季不良环境的一种生物学适应性[22]。休眠的诱导是植物开始休眠的前提,植物一旦被诱导进入休眠就必须满足需冷量以解除休眠,方可正常生长发育,休眠的开始受环境因素诱导,而光照在休眠诱导中起主要作用[18]。phyA作为远红光的主要光受体,通过细胞质到细胞核的转移来诱导一系列远红光响应基因的表达,进而调控种子的萌发、幼苗的去黄化等过程。在模式植物拟南芥中,远红光能够诱导FAR1、FHY3表达下调[5],而FAR1/FHY3作为远红光途径中重要的调控因子,还能够结合脱落酸相关的转录因子ABI5,连接光信号途径和激素信号途径,共同影响种子休眠,且FAR1、FHY3的表达可以被ABA诱导[15]。Leida等[23]发现桃花芽休眠和种子休眠具有一定的相似性。远红光能够诱导种子休眠,因此,PpFRSs也可能参与了桃树花芽的休眠诱导。本研究发现,Prupe.3G186100、Prupe.3G186000在整个休眠诱导期表达量持续升高,Prupe.3G186200、Prupe.4G115100在休眠诱导期呈先升高后降低的趋势,但表达水平一直高于自然生长期,可能参与了桃花芽的休眠诱导过程。用ABA处理桃花芽,PpFRSs也响应了ABA信号。远红光处理桃树,以一年生树皮和叶片作对照,发现4个同源性最高的PpFRSs基因在花芽中表达下调,与拟南芥中的研究一致。Prupe.2G327900在整个休眠诱导过程中及受ABA、远红光处理后的表达量趋势均与其它4个基因趋势相反,可能通过不同的模式参与休眠诱导的调控。
参考文献
[1]陈春峰,陈 由,王亚琴.植物光敏色素入核机理的研究进展[J].热带亚热带植物学报,2012,20(5):533-538
[2]Franklin KA,Davis SJ,Stoddart WM,et al.Mutant analyses define multiple roles for phytochrome C in Arabidopsis photomorphogenesis[J].Plant Cell,2013,15(9):1981-1989
[3]Hudson ME,Lisch DR,Quail PH.The FHY3 and FAR1 genes encode transposase-related proteins involved in regulation of gene expression by the phytochrome A-signaling pathway[J].Plant J,2003,34(4):453-471
[4]Lin RC,Wang HY.Arabidopsis FHY3/FAR1 gene family and distinct roles of its members in light control of Arabidopsis development[J].Plant Physiol,2004,136(4):4010-4022
[5]Lin RC,Ding L,Casola C,et al.Transposase-derived transcription factors regulate light signaling in Arabidopsis[J]. Science,2007,318(5854):1302-1305
[6]Hudson M,Ringli C,Boylan MT,et al.The FAR1 locus encodes a novel nuclear protein specific to phytochrome A signaling[J].Genes Dev,1999,13(15):2017-2027
[7]Wang HY,Deng XW.Arabidopsis FHY3 defines a key phytochrome A signaling component directly interacting with its homologous partner FAR1[J].Embo J,2002,21(6):1339-1349
[8]Hiltbrunner A,Tscheuschler A,Viczián A,et al.FHY1 and FHL act together to mediate nuclear accumulation of the phytochromeAphotoreceptor[J].Plant&Cell Physiol,2006,47(8):1023-1034
[9]Huang X,Ouyang X,Yang PY,et al.Arabidopsis FHY3 and HY5 positively mediate induction of COP1 transcription in response to photomorphogenic UV-B light[J].Plant Cell,2012,24(11):4590-4606
[10]Allen T,Koustenis A,Theodorou G,et al.Arabidopsis FHY3 specifically gates phytochrome signaling to the circadian clock[J].Plant Cell,2007,146(4):2506-2516
[11]Li G,Siddiqui H,Teng YB,et al.Coordinated transcriptional regulation underlying the circadian clock in Arabidopsis[J].Nat Cell Biol,2011,13(5):616-622
[12]Ouyang XH,Li JG,Li G,et al.Genome-wide binding site analysis of FAR-RED ELONGATED HYPOCOTYL3 reveals its novel function inArabidopsis development[J].Plant Cell,2011,23(7):2514-2535
[13]Tang W J,Wang W Q,Chen D Q,et al.Transposase-derived proteins FHY3/FAR1 interact with PHYTOCHROME-INTERACTING FACTOR 1 to regulate chlorophyll biosynthesis by modulating HEMB 1 during deetiolation in Arabidopsis[C].北京:从植物科学到农业发展”2012全国植物生物学大会,2012:1984-2000
[14]Stirnberg P,Zhao S,Williamson L,et al.FHY3 promotes shoot branching and stress tolerance in Arabidopsis in an AXR1-dependent manner[J].Plant J,2012,71(6):907-920
[15]Tang WJ,Ji Q,Huang Y,et al.FAR-RED ELONGATED HYPOCOTYL3 and FAR-RED IMPAIRED RESPONSE1 transcription factors integrate light and abscisic acid signaling inArabidopsis[J].Plant Physiol,2013,163(2):857-866
[16]Ma L,Xue N,Fu XY,et al.Arabidopsis thaliana FAR-RED ELONGATED HYPOCOTYLS3(FHY3)and FAR-RED-IMPAIRED RESPONSE1(FAR1)modulate starch synthesis in response to light and sugar[J].New Phytol, 2016,213(4):1682-1696
[17]高东升,束怀瑞,李宪利.几种适宜设施栽培果树需冷量的研究[J].园艺学报,2001,28(4):283–289
[18]王海波,王孝娣,程存刚,等.桃芽休眠的自然诱导因子及钙在休眠诱导中的作用[J].应用生态学报,2008,19(11):2333-2338
[19]Olsen JE.Mechanisms of dormancy regulation[J].Acta Horic Sin,2006,727(727):157-165
[20]段成国,李宪利,高东升,等.内源ABA和GA3对欧洲甜樱桃花芽自然休眠的调控[J].园艺学报,2004,31(2):149–154
[21]Babu MM,Mlyer L,Balaji S,et al.The natural history of the WRKY–GCM1 zincfingers and the relationship between transcription factors and transposons[J].NucleicAcids Res,2006,34(22):6505-6520
[22]庄维兵,高志红,章 镇,等.木本植物季节性休眠的分子机制[J].西北植物学报,2010,30(9):1919-1928
[23]Leida C,Terol J,Martí G,et al.Identification of genes associated with bud dormancy release in Prunus persica by suppression subtractive hybridization[J].Tree Physiol,2010,30(5):655–666
Expression Analysis of FAR1/FHY3-related Genes Involved in Nectarine Floral Buds during Dormancy Induction
HUAN Lei1,WANG Xu-xu1,LIU Wen-hai3,LI Ling1,2,GAO Dong-sheng1,2*
1.College of Horticulture Science and Engineering/Shandong Agricultural University,Tai’an 271018,China
2.State Key Laboratory of Crop Biology/Shandong Agricultural University,Tai’an 271018,China
3.Weifang Engineering Vocational College,Weifang 261000,China
FAR-RED ELONGATED HYPOCOTYL3(FHY3)and its homolog FAR-RED IMPAIRED RESPONSE1(FAR1),are involved in the regulation of seed germination induced by far red and abscisic acid,but the transcriptional changes of FHY3/FAR1 in dormancy induction process of flower bud is still to be explored.The homologous genes of Arabidopsis thaliana in peach were firstly identified by NCBI blast toolkit.At the same time the expression pattern of the five highest homology genes was characterized during the dormancy induction course,far-red light conditions and ABA-treated conditions, respectively, in floral buds of seven-year-old‘Zhongyousihao’nectarine(Prunus persica var.nectariana cv.‘Zhongyousihao’).Quantitative real-time PCR analysis demonstrated that the expression of Prupe.3G186100,Prupe. 3G186200,Prupe.4G115100,and Prupe.3G186000 have a up-regulated tendency during the dormancy induction course, also,their transcripts are up-regulated by ABA,down-regulated by far-red light.Conversely,Prupe.2G327900 transcript is down-regulated during both the dormancy induction course and ABA-treated conditions,but up-regulated by far-red light. According to the aforementioned results,we could speculate that PpFRSs may be involved in dormancy induction of flower bud through different methods.
ABA;bud dormancy induction;expression analysis;far-red;FHY3/FAR1
S662.1
:A
:1000-2324(2017)02-0229-07
10.3969/j.issn.1000-2324.2017.02.014
2016-11-23
:2016-12-10
山东省自然科学基金(NO.ZR2014CM015)
郇 蕾(1991-),女,硕士研究生,研究方向:果树生理与分子.E-mail:huanleisheshi@163.com
*通讯作者:Author for correspondence.E-mail:dsgao@sdau.edu.cn
——记嘉荫县红光乡燎原村党支部

