哺乳动物合成生物学在生物医学领域的研究进展
杨林凤,尹剑丽,王美艳,叶海峰
哺乳动物合成生物学在生物医学领域的研究进展
杨林凤,尹剑丽,王美艳,叶海峰
华东师范大学生命科学学院上海市调控生物学重点实验室,上海 200241

杨林凤, 尹剑丽, 王美艳, 等. 哺乳动物合成生物学在生物医学领域的研究进展. 生物工程学报, 2017, 33(3): 436–455.Yang LF, Yin JL, Wang MY, et al. Research progress of mammalian synthetic biology in biomedical field. Chin J Biotech, 2017, 33(3): 436–455.
虽然合成生物学还处于早期研究阶段,但最近十年,该领域取得了非常显著的研究进展。合成生物学是以工程学思想为基础,通过人工设计、改造基因线路,从而赋予细胞或生物体新的功能,现已广泛应用于各个领域。随着人们对基因线路设计的深入研究,使得合成生物学研究走向临床应用成为可能。本文将围绕哺乳动物合成生物学在疾病治疗方面的研究进展,介绍基因线路的设计思路和方法、不同诱导因子调控的开环式基因线路以及用于疾病诊疗的闭环式基因环路在生物医学领域的应用。最后对合成生物学走向临床治疗的应用前景和挑战进行展望。
合成生物学,基因线路,基因治疗,细胞治疗
合成生物学是一门综合性的学科,致力于合成一些功能元件、模块、系统,从而对生命体进行有目的的设计、改造,使细胞或生物体具有特定的新功能[1-2]。随着基因组学和蛋白质组学的发展,大量生物分子的功能被挖掘出来,包括DNA、RNA调控序列,配体响应蛋白,相互作用蛋白等。这为合成生物学家设计、合成各种基因线路提供良好的素材[3-4]。近几年,随着基因、细胞治疗理念的深化,科学家们开始将人工合成的基因网络导入哺乳动物细胞内用于各种疾病的治疗。因此,为了能更好地概述哺乳动物合成生物学在生物医学领域的研究进展,本文首先介绍哺乳动物合成基因线路设计的思路和方法,之后再介绍各种开环式和闭环式基因线路用于各种疾病治疗的案例,最后探讨哺乳动物合成生物学走向临床应用的前景和挑战。
1 构建哺乳动物合成生物学基因线路的设计思路和方法
生物遗传信息的传递需要转录和翻译的过程,因此在不同的生物体中存在具有特定生物功能的大分子和基因线路。而合成生物学的目的就在于设计和改造这些功能元件,使生物体具备某种新的特定功能。因此,为了构建具有医学治疗价值的复杂生物系统,合成生物学家通过理性设计和组装各种生物功能元件来达到目的。本节主要介绍合成生物学在转录水平、翻译水平以及翻译后水平对信号通路设计、合成的思路和方法以及生物信息学在基因线路设计方面的应用。
1.1 转录水平调控环路
目前,大多数基因治疗和自体细胞疗法都利用病毒启动子或组织特异性和内源性的启动子来进行转基因表达,然而病毒性启动子可能会存在基因毒性[5],在体内也容易引起基因沉默[6]。而组织特异性和生理学的启动子可以在特定细胞系中以相似的表达模式表达适量蛋白,以减少基因毒性和细胞毒性[7-8]。但是由于内源性的启动子和调控元件在不同细胞系中的表达量和含量会有所差异[9],会出现表达量不足以致难以达到治疗效果的情况[10],因此在构建基因线路中不易于设计、改造[11]。相比较而言,合成的最小启动子更加适于应用在ON和OFF基因开关的设计上,这些开关可以响应内源或外源的诱导剂,从而对基因表达进行精确的时空调控 (图1)。
早期研究报道表明,细菌中的转录调控蛋白可与特定的DNA操纵序列结合,将该DNA结合域与转录调控功能域在哺乳动物细胞内融合表达,可用于调控下游基因表达。具体调控方式如下:将该蛋白与转录激活蛋白 (如Viral protein 16,VP16等) 融合表达,该融合蛋白将结合至下游的DNA操纵序列上,转录激活子将招募转录激活因子,启动最小启动子表达下游基因[12-14];或者将该蛋白与转录抑制蛋白 (如Kruppel-associated box,KRAB等) 融合表达,则可以通过与强启动子下游的DNA操纵序列结合并阻遏下游基因的表达[15](图1A)。
为了能够在恶劣的环境中生存,细菌进化出大量的刺激物调控转录响应系统[16]。类似于大肠杆菌中的四环素调控的TetR系统,大多数的原核生物基因调控蛋白都是转录抑制子,当诱导物分子不存在时,调控蛋白结合至相应的DNA操纵序列,该结合具有很高特异性和亲和力;当有刺激物分子存在时,该分子会与转录调控蛋白结合,使其空间构象改变,从操纵序列上解离下来[17]。这一系列诱导物调控的基因线路系统可以用于激活或抑制基因的表达,可作为基因编辑调控的工具。将这些小分子调控系统用于哺乳动物细胞,可以构建一系列有价值的模式系统,包括基因开关[18–19]、筛选器[20]、时间延迟基因线路[21–22]和振荡器[23–25]等。迄今为止,已有多篇文章发表通过化学物质调控基因表达的转录水平调控装置,其在临床医学治疗方面的潜力也逐渐被发掘出来,随后本文就其在疾病治疗中的应用展开讨论。
近几年,原核生物转录因子作为DNA结合域的应用已经逐渐被锌指蛋白(Zinc-fingerproteins,ZF)、转录激活因子样效应物核酸酶 (Transcription activator-like effectors,TALEs) 和CRISPR (clustered regular interspaced short palindromic repeats)/Cas9 (CRISPR-associated protein) 所代替,这种转录因子不受DNA操纵序列的限制,原则上可以靶向任何DNA序列。前两个可编程的转录系统都是基于共价键结合的形式,锌指蛋白可以通过Cys2-His2锌指结构域与特定核苷酸序列结合,TALEs是通过33−35个保守氨基酸序列去识别DNA序列[26-27]。如果将这两种蛋白与转录调控域融合表达,这些系统则可以调控任何内源或外源基因的转录表达[28-34]。CRISPR/Cas9技术则是基于Watson-Crick碱基配对原则,核酸内切酶Cas9可以在一条20个碱基的非编码向导RNA (sgRNA) 的作用下结合至与sgRNA序列互补的核苷酸序列处,并对靶基因进行切割,引起DNA双链断裂 (double-stranded breaks,DSBs)。断裂的双链DNA在生物体内的修复方式主要包括非同源末端连接 (non-homologous end-joining,NHEJ) 和同源指导修复 (homologous directed repair,HDR)。随后,通过突变得到的dCas9 (dead Cas9) 保留了其DNA结合的功能而失去了切割活性,利用这一特点,科学家们将dCas9与不同类型的转录调控因子进行组合,从而形成各种转录激活、抑制装置。基于CRISPR/Cas9系统的激活装置可分为以下几种类型:1) dCas9直接与各种转录激活因子融合表达,其中激活因子可包括:多个拷贝的单纯疱疹病毒蛋白 (VP16)n[35-36]、VP16的四聚衍生物VP64[37-38]、NF-κB 的转录激活域p65[39]、VPR (VP64-P65-Rta)[40]、SunTag[41]、组蛋白乙酰转移酶p300[42]和DNA去甲基化酶Tet1[43-45]等;2) 改造sgRNA使其含有一个茎环结构可与噬菌体外壳蛋白MS2结合,从而可通过表达MS2-VP64[46]、MS2-p65-HSF1[47]等形式的激活装置间接实现基因的激活表达。目前CRISPR/Cas9系统的抑制装置也可通过dCas9融合转录抑制子KRAB[48-49]、甲基化转移酶DNMT3a[50-51]、组蛋白去甲基化酶LSD1[52]等形式实现基因的抑制。此外,改变sgRNA的长度也可实现基因的激活和抑制[53]。目前,一些调控的CRISPR/Cas9系统不断被开发出来,如雷帕霉素调控Cas9系统[54]、蓝光调控Cas9系统[39]。相比较于ZNF和TALEN技术,CRISPR因设计简单、效率高、作用范围广而被快速地应用于生物医学领域。令人更为激动的是,四川大学华西医院肿瘤学家卢铀领衔的团队首次将CRISPR/Cas9应用于临床中,即通过CRISPR/ Cas9系统敲除T细胞中抑制免疫功能的PD-1基因,并在体外进行细胞扩增。该类细胞扩增至一定量后再植入患者体内并发挥杀伤肿瘤的功能[55](图1A)。
1.2 翻译水平调控环路
随着功能性RNA的研究日益深入,合成生物学家们也开始寻求从功能性RNA方面去设计、合成基因线路。RNA干扰是真核细胞中非常重要的转录抑制系统,干扰RNA和microRNAs (miRNA) 可以通过调控内源的蛋白复合体并结合至与它们的碱基序列互补配对的信使RNA,阻碍其翻译过程从而调控基因表达[56]。这种miRNA调控在维持组织特异性和发育阶段特异性方面具有重要作用[57-58]。因此,科学家们通过利用miRNA可特异性结合至信使RNA的3'端非翻译区 (untranslated regions,UTRs) 的特点,将其用于基因治疗和细胞治疗中以降低脱靶效应。在基因治疗中,利用病毒载体传输,可以在特定的细胞系中表达miRNA抑制特定基因的表达[59],比如通过抑制相关基因在抗原递呈细胞中的表达可以增加基因治疗的免疫耐受力[60-61]。在利用工程化的离体造血干细胞 (hematopoietic stem cells,HSCs) 进行细胞治疗的过程中,为了降低在HSC分化过程中的细胞毒性[62-63]并保证治疗基因在HSC来源的细胞系中特异性表达[64],研究者们通过设计、合成不同发育阶段、不同细胞系中特异性表达miRNA的基因线路来实现这一目标。
RNA干扰介导的沉默机制作为一个非常有价值的工具已经广泛运用于基因治疗和细胞治疗方面,miRNA介导的基因沉默在合成生物学中的应用也十分广泛,目前,RNA干扰装置可用于构建紧密式的基因开关[65]调控特定基因表达的合成基因线路[66]、可调控基因表达的逻辑门基因线路[67-68]和可识别肿瘤细胞的分选装置[69](图1B)。
另外,存在于mRNA中的核糖体开关 (riboswitch) 于2002年被首次发现[70],关于riboswitch的研究日益深入,随着生物信息学的快速发展和大量DNA数据库的建立,越来越多的核糖体开关被发现,同时也发表了许多人工合成的RNA适配器[71]。核糖体开关可以特异性地识别其靶向配体并改变mRNA的构象,在转录或翻译水平上调节基因表达[72]。合成生物学家们利用其可与小分子或蛋白特异性结合的特性,构建了一系列可在转录或翻译水平上调控基因表达的合成基因线路[73-76]。这些RNA调控装置作为一个高度合成的装置[77-78],可以作为基本框架合成一系列用于调控细胞命运的配体调控转录装置。此外,引入非天然氨基酸 (nonstandard amino acids,nsAAs) 也是在翻译水平上调控的常见方式[79],相较于传统的nsAAs引入方法如化学合成直接掺入法和定点突变法,体外无细胞表达系统可以实现最大比例的nsAAs掺入,且不伤害细胞活性,展示出广阔的应用前景[80-82]。
1.3 信号通路水平和翻译后水平调控环路
哺乳动物细胞在处理、传递和响应信息并执行信息所传递的信号通路方面有极大的优势,然而基于转录因子的感受器的激活剂大部分局限于扩散性的小分子,哺乳动物细胞在处理信号的过程中会涉及到大量的细胞受体,这些受体可以感受到过量内源或外源分子和刺激物。利用这些特征,基于合成生物学和生物工程学的技术和方法,科学家们通过利用细胞自身的或者工程化改造后的细胞受体、信号转导蛋白和下游响应元件,从而构建组装具有新功能的虚拟基因网络系统,让细胞执行新功能。
细胞信号通路激活的信号分子有很多,从光等物理信号到气味、信息素等复杂大分子,从蛋白、多肽到小分子物质,这些胞外信号分子可通过G蛋白偶联受体 (G-protein-coupled receptors,GPCRs) 传递至胞内并引起不同细胞中的信号通路的激活,执行不同的生理功能。基于GPCRs结构和功能上的特点,科学家们通过对其进行工程化的改造使其可以响应合成的生物配体,这种工程化的受体被称为合成配体特异性激活受体 (receptors activated solely by synthetic ligands,RASSL),即通过对内源性的GPCR进行合理的工程化改造,使它的结合域对天然配体的亲和力下降,而与合成的配体结 合[83]。然而内源的GPCRs通常会对合成配体有较小的亲和力,因此这种工程化的受体会缺少特异性[84],但是药物特异性激活受体 (designer receptor exclusively activated by designer drugs,DREADD) 技术成功地解决这个问题,该技术通过将分子进化与活细胞实验结合在一起,设计出了诱导物与内源GPCRs完全没有亲和力的工程化GPCRs[85]。自从首次报道一种改造后的人M型乙酰胆碱受体可特异性识别一种抗精神失常药氯氮平后[86],DREADDs迅速成为研究生物进程的重要工具[87],也逐渐将其应用于构建各种人工合成基因线路[88-89]。合成生物学家们通过将可感知信号的GPCRs与相关的转录调控装置结合,将这种工具发展成为可响应几乎所有刺激物的远程调控细胞装置。
翻译后水平调控装置的设计主要通过利用蛋白与蛋白之间的相互作用以及蛋白的泛素化降解作用,该装置相对于转录水平和翻译水平的调控具有反应速度快的优势,可直接通过刺激物诱导蛋白结构改变,调控细胞通路,解决后两者的调控延迟效应。比如,在肿瘤免疫治疗过程中会发生细胞因子风暴等副作用,这时候就需要终止治疗过程。现已有研究报道通过FK506结合蛋白 (FK506 binding protein,FKBP12) 与促凋亡蛋白融合表达,在加入合成药物AP1903后,FKBP12蛋白二聚化,即凋亡蛋白二聚化,激活下游凋亡信号通路,引起T细胞凋亡,该系统可以精确调控工程化的T细胞迅速凋亡,可作为防止免疫排斥反应的安全开关[90–93]。另一篇研究报道表明,调控基因工程化嵌合型抗原受体 (chimeric antigen receptor,CAR) 在进行肿瘤免疫治疗过程中,利用雷帕霉素诱导并结合的FKBP (FK506 binding protein)和FRB (FKBP-rapamycin binding domain)两种蛋白去调控CAR 的表达,从而实现对CAR-T细胞的精确调控[94](图1C)。此外,基于蛋白相互作用的翻译后水平调控装置除了雷帕霉素诱导FKBP和FRB结合的系统外,还有内含肽 (intein) 系统[95]、蓝光诱导的Cry2/CIB1系统[39,96]、紫外光诱导的UVR8/UVR8系统[97]以及光诱导的依赖于藻青素的PIF6/PhyB系统[98]等,这些系统可与各种效应蛋白如转录调控蛋白、翻译调控蛋白、离子通道调控蛋白等相结合,有效地调控转基因表达,应用已十分广泛。

图1 用于构建哺乳动物合成基因线路的工具
1.4 生物信息学与基因线路
生物信息学是一门对生物学数据进行采集、处理和模拟的学科,随着各种基因组测序计划的开展和生物技术的发展以及计算机的普及,大量的生物学数据库已经成功构建,这对于合成生物学的发展有极大的便利[99]。通过对生物数据库进行筛选,可快速获得用于构建基因线 路的目的基因或蛋白,例如,Yang等基于DNA 可通过不可逆的重组酶记录信号的记忆能力,通过生物信息学的方法进行分析、对比,筛选出34种噬菌体整合酶并构建了11种完全正交性的记忆开关,将这些开关在大肠杆菌中构建、表达,已被证明可以用于永久记录转录过程中的瞬时信息,储存容量可达1 375个字节[100]。另外,通过对实验数据的分析、计算,结合数学逻辑方法构建实验模型,可作为基因线路设计的参考,例如,William Bacchus等发表的细胞间交流的系统,通过对两个系统中细胞的密度、mRNA和蛋白的表达动力学以及蛋白与DNA的亲和度等进行常微分方程 (ordinary differential equations,ODEs) 解析,从而构建一个细胞交流的动力学模型,再结合模型参数进行实验验证,大大便利了基因线路的设计和构建[101]。
2 用于疾病治疗的基因线路
在过去的一段时间里,合成生物学仅仅停留于概念式的网络设计,然而近几年,更多的合成生物学家致力于研究设计应用式的基因线路。在早期的研究中,合成生物学便产生了一系列简单的基因表达系统,如拨动开关[102]、基因振荡器[103-105]、计数器[106]、计时器[107]、定时器[108]、模式探测器[109]、过滤器[110]和细胞通讯系统[111-112]等。然而,这些概念式的表达系统并不能满足当今社会对医药的需求,因此需要继续发掘合成生物学在医学领域的潜力,近年来构建的一系列基因线路已逐渐应用于药物合成[113-114]、药物筛选[115]等方面,同时一些与疾病治疗相关的基因线路陆续被设计出来,它们可以通过感应外界的刺激或机体内的代谢信号,重新编程细胞行为,从而实现临床治疗的目的。这些生物感应的基因线路一般可分为开环式和闭环式控制线路,前者属于线性的刺激-响应系统,后者属于环状的刺激-响应-反馈系统[116]。
2.1 开环式基因线路在生物医学领域的应用
开环式的基因线路是属于线性式的刺激-响应系统,因此,按不同的刺激因子可将其分为:1) 化学物质调控的开环式基因线路;2) 光调控的开环式基因线路;3) 微波调控的开环式基因线路;4) 磁调控的开环式基因线路 (图2)。
2.1.1 化学物质调控的开环式基因线路
四环素[12,117-118]、大环内脂类抗生素[119]、链阳菌素[117,120]响应的基因线路是早期经典的化学物质调控系统。但由于这些基因线路是由一些会对人体产生副作用的抗生素物质诱导调控的,极大地限制了其应用范围。最近,一些新的健康安全的小分子物质调控的基因线路被陆续开发出来,如维生素[121]、氨基酸[122]、根皮素[123]、食品添加剂香草酸[124]、苯甲酸[125]和沐浴乳成分对羟基苯甲酸酯[126]和中药成分齐墩果酸[127]等诱导的基因线路。这些安全的基因线路有效地促进了哺乳动物合成生物学在疾病治疗中的应用。例如利用降血压药物胍那苯治疗代谢综合征[128]。在该环路中,胍那苯通过刺激嵌合型痕胺受体 (cTAAR1),活化G蛋白,进一步激活膜上的腺苷环化酶,将ATP转变为cAMP;胞内的cAMP进一步激活蛋白激酶A,使活化的蛋白激酶A入核,cAMP反应结合蛋白1 (CREB1) 磷酸化,进而激活人工合成响应cAMP的启动子 (PCRE),启动表达胰高血糖素样肽-1和瘦素 (Leptin) 的融合蛋白,实现在治疗高血压的同时治疗高血糖和肥胖,达到“三合一”的治疗效果 (图2A)。
2.1.2 光调控的开环式基因线路
相比于化学物质调控系统,光控的基因线路具有高度的时空特异性、便捷性等优势,因此越来越受到人们的关注。据研究报道,目前合成生物学家已开发出由蓝光、红光、紫外光以及远红光诱导的基因线路,包括光受体视黑素系统[129]、LOV系统[130-131]、CRY2/CIB1系 统[132]、植物光敏素系统[133-134]、隐花色素[135-136]和细菌光敏色素系统[137]等。这些光控的基因线路在疾病治疗上具有极大的应用前景:1) 视黑素蓝光系统治疗Ⅱ型糖尿病[129];2) LOV蓝光系统治疗Ⅰ型糖尿病[138];3) Opto-CRAC近红外光系统治疗肿瘤[139]等。此外,光控的基因线路可实现远程控制內源基因的激活、删除和插 入[140-142],这为将来实现基因、细胞水平治疗提供理论和实践基础 (图2B)。
2.1.3 微波调控的开环式基因线路
相比于光,微波具有更强的穿透性,这使微波调控的基因线路在临床治疗上更具优势。现有研究报道一种微波调控的基因线路可用于糖尿病治疗[143]。该基因线路中的微波感受装置是一种与生物磁珠相结合的温度敏感型的离子通道蛋白TRPV1,该蛋白在外界温度升高 (不超过42 ℃) 时可被激活,引起钙离子内流,激活下游信号通路。因此,该基因线路在微波辐照下,TRPV1上的生物纳米磁珠产生磁热效应并释放热量,导致细胞内局部温度升高,从而激活TRPV1,引起钙离子内流,打开NFAT信号通路,从而启动胰岛素的表达,调节体内葡萄糖稳态。此外,该微波调控的基因线路中由于存在加入纳米磁珠材料的安全问题,因此科学家对其环路作了进一步的优化:1) 强表达铁蛋白 (Ferritin),铁蛋白不仅是机体贮存铁的一种可溶性蛋白,而且当24个铁蛋白亚基聚合成一个12 nm的聚合物时,该聚合物同样可在低频微波或磁场作用下产热,从而实现了无侵入、安全的远程基因调控方式;2) 通过将铁蛋白与GFP融合表达,并结合到被抗GFP抗体修饰的TRPV1上,从而使铁蛋白聚合物定位至细胞膜上,使得该系统的灵敏度增强,更加有效地远程调控小鼠体内的葡萄糖稳态[144-145](图2C)。
2.1.4 磁调控的开环式基因线路
光遗传学和化学遗传学作为研究工具已经被广泛应用于神经行为研究领域,然而这两种研究方法的刺激方式都是植入式的,会对实验动物产生巨大伤害,同时也会降低神经反应速度,因此,亟需进行磁遗传学的研究。2015年Wheeler等发现磁场远程可以调控神经系统将可感受力的瞬时感受器电位通道V型成员4受体 (transient receptor potential cation channel subfamily V member 4,TRPV4) 与Ferritin融合表达,该融合蛋白可使强表达的Ferritin与其结合,从而使磁蛋白复合体定位至细胞膜上,当磁场存在时,磁蛋白可感受磁场变化,产生拉力,激活TRPV4,打开钙离子通道,引起膜电位的变化,从而实现磁场调控神经系统的目的。将磁控系统用于神经学研究具有无侵入性、远程调控、更加安全的优势,相信未来该系统在其他疾病治疗方面也会展现其优越性[146](图2D)。
2.2 自我诊断及治疗的闭环式基因线路在医学领域的应用
不同于开环式基因线路,闭环式的基因环路是通过识别机体内的代谢物质,重新编程细胞内部的代谢活动,同时还具备反馈调节机制,从而实现监测-治疗的双重功效。因此,越来越多的闭环式基因环路被用于疾病的诊断和治疗,如尿酸感受器用于治疗痛风[147]、脂肪酸感受器用于治疗肥胖[148]、甲状腺激素感受器用于治疗甲 亢[150]、细胞因子交换器用于治疗银屑病[152]、以及胰岛素感受器用于治疗Ⅱ型糖尿病[153](图3)。
2.2.1 治疗痛风的闭环式基因环路
尿酸是哺乳动物嘌呤代谢的终端物质,若体内尿酸水平过高,则会引发痛风症。现有一种人工合成的尿酸传感器,它能将体内尿酸水平维持在正常范围内,从而缓解痛风症状[147]。该尿酸传感器的感受装置是由KRAB和含有尿酸配体结合域的转录因子HucR融合表达形成,其应答装置则是由尿酸诱导型的启动子和尿酸氧化酶 (urate oxidase) 组成。尿酸诱导型的启动子是由SV40强启动子3'端和HucR结合的操纵子位点(hucO)组成。当尿酸浓度处于极低状态,KRAB-HucR结合至操纵子位点 (hucO) 上,阻遏下游基因的表达;而当尿酸水平处于过高状态,尿酸分子主动与HucR结合,使转录抑制子从操纵子上解离下来,启动表达尿酸氧化酶,清除尿酸并使其恢复至正常浓度范围内。这种闭环式的基因环路利用尿酸反馈机制,不仅能实时监测尿酸浓度的变化,并且能及时对其作出反应,可以将机体内的尿酸水平维持在正常的生理范围内,进而改善痛风的病症 (图3A)。

图2 开环式调控的基因线路
2.2.2 治疗肥胖的闭环式基因环路
人体能量过剩,脂肪囤积则会引发肥胖。体内脂肪酸的含量是衡量过食性肥胖的重要指标之一。现有一种人工合成的脂肪酸传感器,它能通过监测血液中脂肪酸的含量,定量表达能抑制食欲的短肽——普兰林肽 (pramlintide),从而达到治疗肥胖的目的[148]。该脂肪酸传感器中的脂质感应受体 (lipid-sensing receptor,LSR) 是由转录抑制子TtgR与过氧化物酶增生物激活受体 (peroxisome proliferator-activated receptorα,PPARα) 融合表达形成。这种闭环式的反馈机制主要是利用脂质感应受体中的PPARα,根据脂肪酸的含量分别招募共阻遏因子或共激活因子,进而关闭或启动目的基因的表达。此外,在系统中加入根皮素,可使转录抑制子TtgR从其结合位点上解离下来,从而关闭整个环路的运行,确保环路的安全性 (图3B)。
2.2.3 治疗甲亢的闭环式基因环路
甲亢是一种由体内甲状腺激素过量引起的甲状腺机能亢进的自身免疫性疾病。自身抗体通过刺激甲状腺中的促甲状腺激素受体使机体分泌甲状腺激素,从而使控制其分泌的下丘脑-脑垂体-甲状腺反馈机制发生紊乱[149]。现有人工合成的甲状腺激素传感器,它能监测体内甲状腺激素的含量,同时通过表达促甲状腺激素受体拮抗剂来维持体内甲状腺激素的稳态[150]。在甲状腺激素不存在时,甲状腺传感器中的甲状腺激素受体招募转录共阻遏子,关闭目的基因的表达,但当甲状腺激素存在时,甲状腺激素受体招募转录共激活子,启动表达促甲状腺激素受体拮抗剂,阻碍自身抗体与促甲状腺激素受体的结合,进而改善甲亢的病症 (图3C)。
2.2.4 治疗银屑病的闭环式基因环路
银屑病是一种慢性炎症皮肤病,并具有病程长、易复发的特点,传统的药物治疗往往不能达到根除的效果[151]。银屑病发生时,肿瘤坏死因子和白介素22的表达量处于“上调”状态。因此,这两种细胞因子可作为银屑病发生时的特异性生物标记物。现有一种人工合成的细胞因子交换器,它能将上述两种促炎症细胞因子转换输出白介素4、白介素10的抗炎症因子,从而缓解病症[152]。该转换器是通过一个“AND”逻辑门实现细胞因子之间的转换。当TNF作用于人工合成的肿瘤坏死因子受体 (TNFR),激活NF-κB信号通路,启动表达白介素22受体α (hIL22RA)。同时,hIL22RA与细胞內源表达的白介素10受体β (hIL10RB) 异二聚化组装成白介素22受体,能识别IL22,激活JAK-STAT信号通路,启动IL4和IL10的表达。因此,在该闭环式传感器中,只有TNF和IL22同时存在时,系统才能输出双因子信号,进而提高系统的特异性 (图3D)。
3 哺乳动物合成生物学走向临床研究的方法手段
随着哺乳动物合成生物学的发展,更多人工合成的治疗系统被陆续开发出来,然而这些系统在临床应用中仍面临以下4个挑战:1) 运输效率;2) 系统在病人体内的持久性;3) 临床疗效;4) 免疫学特性。最新研究报道自体干细胞疗法虽可部分解决上述临床治疗问题,但由于自体干细胞的不易获取性很大程度上限制了它的应用范围。因此,开发一种安全、有效的治疗输送系统是目前哺乳动物合成生物学走向临床应用亟待解决的问题。本节将主要介绍一些新型的输送方式以及各自的优缺点。
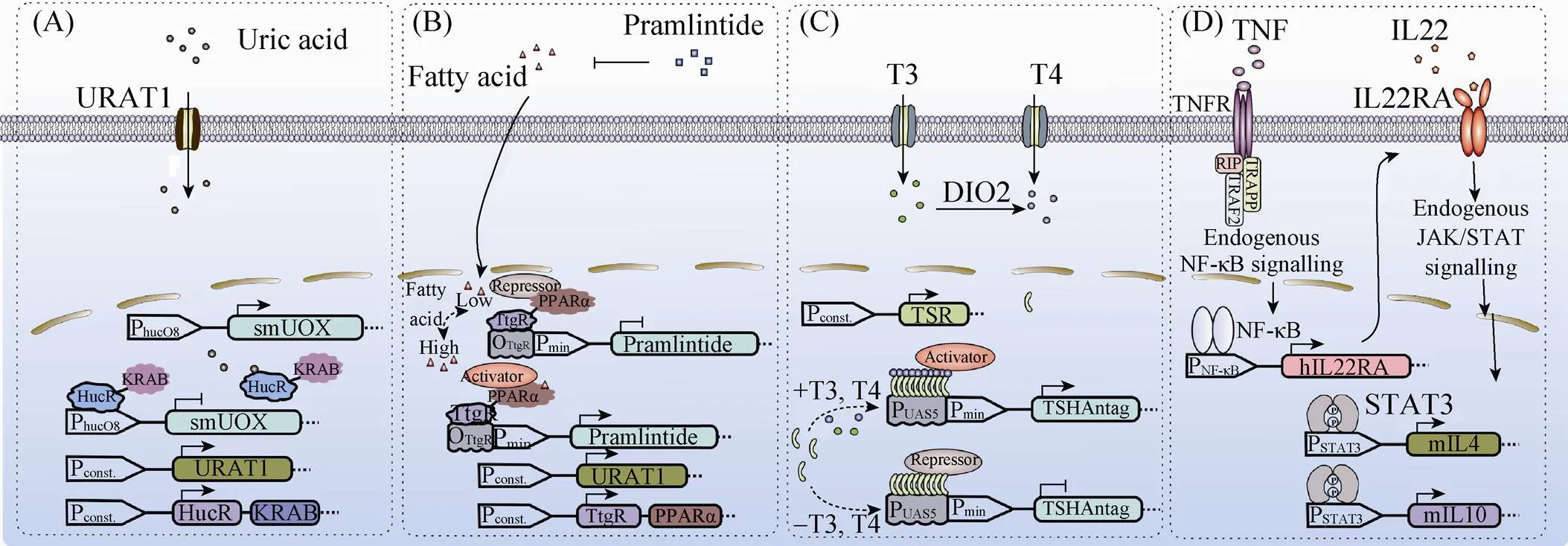
图3 闭环式调控的基因环路
3.1 细胞胶囊化
若将外源细胞直接注入机体内会引起体内的免疫反应并对其进行清除。因此,如何使植入的异源细胞避开体内免疫系统的攻击是目前细胞治疗的难题之一[154]。近年来,合成生物学家成功找到一种新的细胞输送方式——细胞胶囊化。这类包裹细胞的胶囊是由海藻酸-聚- L-赖氨酸-海藻酸组成的半透膜,它不仅可以阻止免疫球蛋白分子和免疫活性细胞的进入,而且还能允许机体内的养分、氧气等自由交换[155-156]。这种半封闭式的胶囊系统不仅可以使植入的治疗细胞免受免疫系统的清除,而且还可通过保证治疗细胞与机体之间的交流来提高细胞的活力。但是,一些海藻酸钠聚合物的拉伸强度低,植入细胞的寿命等问题都会影响胶囊化细胞的长期治疗效果[154,157-158]。尽管细胞胶囊化的输送方式有待改善,但目前仍是一个经济有效的手段。
3.2 病毒输送载体
病毒输送载体需要将治疗系统整合至宿主基因组内,从而实现长期有效的治疗目的。理想的病毒输送载体需要满足以下几个条件:1) 具备有效的基因转移能力;2) 无免疫副作用;3) 病毒基因无法复制、以保证生物安全。目前使用最广泛的病毒载体包括逆转录病毒、腺病毒和腺相关病毒。
逆转录病毒是一类正链RNA病毒,是可将外源基因整合至基因组内并使其得以长期稳定的表达。目前最常见的逆转录病毒载体是人类免疫缺陷病毒-1 (HIV-1) 来源的病毒载体——慢病毒载体[159]。携带治疗系统的慢病毒载体在体外包装成有感染力的病毒颗粒,通过感染机体组织将系统整合至机体内,从而实现系统长期稳定的运作。虽然慢病毒载体具有基因表达持久、稳定,感染效率高等优点,但是载体容量小,与宿主基因组随机整合方式可引起基因突变等问题仍有待解决。
腺病毒是一类DNA病毒,其基因组不整合至宿主细胞的基因组中,始终游离在染色体外。最初的腺病毒载体Ad2和Ad5因基因转移效率高、宿主范围广而备受瞩目。这两种经典的腺病毒载体通过一系列改造后产生了4代不同的载体,第2、3代的腺病毒载体在去除E1、E3、E4编码序列后,相比于第1代,具有更大的载体容量和更低的免疫原性[160]。第4代病毒载体仅保留反向末端重复序列ITR和包装信号序列,载体容量可达37 kb,并进一步降低了免疫原性。尽管腺病毒载体具有基因转移效率高、宿主范围广、易制备、较安全等优势,但仍存在靶向性差、转基因表达时间短的问题[161]。
腺相关病毒是一类目前结构最为简单的单链DNA缺陷病毒,只有在辅助病毒 (腺病毒、单纯疱疹病毒、痘苗病毒) 的参与下才能进行复制。腺相关病毒的基因组包括两个末端的反向重复序列 (ITR) 和编码蛋白cap、rep的基因序列。鉴于腺相关病毒的感染效率高、安全性好、自身免疫原性低及具有定点整合功能[162],现已被用于抗肿瘤免疫基因治疗、肿瘤血管新生治疗等。然而,由于腺相关病毒的结构较为简单,其上载容量小,上限是5 kb左右,限制了其进一步的临床应用。
3.3 其他输送载体
上述常见的病毒输送载体可实现治疗基因的转移,但由于存在潜在免疫原、插入突变、特异性和上载容量等问题限制其应用范围。近年来,科学家陆续找到一些新的基因转移载体,包括1-型单纯疱疹病毒载体、杆状病毒载体、阳离子聚合物和脂质载体等。其中,1-型单纯疱疹病毒和杆状病毒载体具有更大的上载容量,容量可达到150 kb左右。此外,阳离子聚合物和脂质的材料载体能将RNA和环状DNA分子包裹成纳米颗粒进行运送,但同时存在输送效率、免疫原性和不能长期表达等局限性[163]。
4 总结与展望
随着合成生物学的发展,哺乳动物细胞内调控系统的构建已完成以下几大转变:1) 从概念式的系统设计转变为合成更具医用价值的环路;2) 从化学分子调控转变为无踪迹的光、微波和磁场调控;3) 从时间上的调控转变为时间和空间的调控;4) 从线性化的开环式调控转变为回馈式的闭环式调控等。这些复杂化、综合化、多元化的调控系统为合成生物学在医学领域中的应用提供了基础。
近年来,哺乳动物合成生物学在临床上的应用已经有了很大的发展,也为以前无法治愈的疾病提供了新颖的治疗策略,但同时也存在许多局限性和问题,如合成生物学的伦理性、安全性和有效性问题。然而随着科技的进步与发展,这些问题也逐步被解决,例如,第1代的基因与细胞治疗在临床上遭受了很大的挫折,然而随着病毒载体和合成生物学调控装置的深入研究,新一代的基因与细胞治疗的安全性和有效性有了很大的提高,在临床上的应用也有很大的进展;现有的细胞治疗需培养易从体内分离得到的细胞再将其植回患者体内,这种线性式治疗有很大的局限性,而新兴的干细胞疗法和基因重编辑技术的应用则可以解决这一问题。
随着合成调控装置的优化改进及其实际应用性的提高,未来将会有更多的合成生物学设计理念被用于基因与细胞治疗,如通过调控内源的控制元件来长期控制、调节相关基因的表达,或者通过植入闭环式的调控装置,使其能够感知患者自身的相关信号从而调控基因表达,释放合理的治疗药物以满足患者的需求。我们坚信,随着科技的进步及人类社会的发展,合成生物学将会展示其更大的价值和意义。
[1] Pai MP. Drug dosing based on weight and body surface area: mathematical assumptions and limitations in obese adults. Pharmac Therapy, 2012, 32(9): 856–868.
[2] Lienert F, Lohmueller JJ, Garg A, et al. Synthetic biology in mammalian cells: next generation research tools and therapeutics. Nat Rev Mol Cell Biol, 2014,15(2): 95–107.
[3] Lu TK, Khalil AS, Collins JJ. Next-generation synthetic gene networks. Nat Biotechnol, 2009, 27(12): 1139–1150.
[4] Ausländer S, Fussenegger M. From gene switches to mammalian designer cells: present and future prospects. Trends Biotechnol, 2013, 31(3): 155–168.
[5] Montini E, Cesana D, Schmidt M, et al. The genotoxic potential of retroviral vectors is strongly modulated by vector design and integration site selection in a mouse model of HSC gene therapy. J Clin Invest, 2009, 119(4): 964–975.
[6] Ellis J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Hum Gene Ther, 2005, 16(11): 1241–1246.
[7] Zychlinski D, Schambach A, Modlich U, et al. Physiological promoters reduce the genotoxic risk of integrating gene vectors. Mol Ther, 2008, 16(4): 718–725.
[8] Toscano MG, Romero Z, Muñoz P, et al. Physiological and tissue-specific vectors for treatment of inherited diseases. Gene Ther, 2011, 18(2): 117–127.
[9] Frecha C, Toscano MG, Costa C, et al. Improved lentiviral vectors for wiskott-aldrich syndrome gene therapy mimic endogenous expression profiles throughout haematopoiesis. Gene Ther, 2008, 15(12): 930–941.
[10] Manno CS, Pierce GF, Arruda VR, et al. Successful transduction of liver in hemophilia by AAV-Factor IX and limitations imposed by the host immune response. Nat Med, 2006, 12(3): 342–347.
[11] Lisowski L, Sadelain M. Current status of globin gene therapy for the treatment of β-thalassaemia. Br J Haematol, 2008, 141(3): 335–345.
[12] Gossen M, Bujard H. Tight control of gene expression in mammalian cells by tetracycline-responsive promoters. Proc Natl Acad Sci USA, 1992, 89(12): 5547–5551.
[13] Labow MA, Baim SB, Shenk T, et al. Conversion of the lac repressor into an allosterically regulated transcriptional activator for mammalian cells. Mol Cell Biol, 1990, 10(7): 3343–3356.
[14] Seipel K, Georgiev O, Schaffner W. Different activation domains stimulate transcription from remote (‘enhancer’) and proximal (‘promoter’) positions. EMBO J, 1992, 11(13): 4961–4968.
[15] Deuschle U, Meyer WK, Thiesen HJ. Tetracycline-reversible silencing of eukaryotic promoters. Mol Cell Biol, 1995, 15(4): 1907–1914.
[16] Stanton BC, Siciliano V, Ghodasara A, et al. Systematic transfer of prokaryotic sensors and circuits to mammalian cells. ACS Synth Biol, 2014, 3(12): 880–891.
[17] RamosJ L, Martínez-Bueno M, Molina-Henares AJ, et al. The TetR family of transcriptional repressors. Microbiol Mol Biol Rev, 2005, 69(2): 326–356.
[18] Kramer BP, Weber W, Fussenegger M. Artificial regulatory networks and cascades for discrete multilevel transgene control in mammalian cells. Biotechnol Bioeng, 2003, 83(7): 810–820.
[19] Kramer BP, Viretta AU, Daoud-El-Baba M, et al. An engineered epigenetic transgene switch in mammalian cells. Nat Biotechnol, 2004, 22(7): 867–870.
[20] Greber D, Fussenegger M. An engineered mammalian band-pass network. Nucleic Acids Res, 2010, 38(18): e174.
[21] Weber W, Stelling J, Rimann M, et al. A synthetic time-delay circuit in mammalian cells and mice. Proc Natl Acad Sci USA, 2007, 104(8): 2643–2648.
[22] Weber W, Kramer BP, Fussenegger M. A genetic time-delay circuitry in mammalian cells. Biotechnol Bioeng, 2007, 98(4): 894–902.
[23] Swinburne IA, Miguez DG, Landgraf D, et al. Intron length increases oscillatory periods of gene expression in animal cells. Genes Dev, 2008, 22(17): 2342–2346.
[24] Tigges M, Marquez-Lago TT, Stelling J, et al. A tunable synthetic mammalian oscillator. Nature, 2009, 457(7227): 309–312.
[25] Tigges M, Dénervaud N, Greber D, et al. A synthetic low-frequency mammalian oscillator. Nucleic Acids Res, 2010, 38(8): 2702–2711.
[26] Bogdanove AJ, Voytas DF. TAL effectors: customizable proteins for DNA targeting. Science, 2011, 333(6051): 1843–1846.
[27] Beerli RR, Barbas CF III. Engineering polydactyl zinc-finger transcription factors. Nat Biotechnol, 2002, 20(2): 135–141.
[28] Li YQ, Jiang Y, Chen H, et al. Modular construction of mammalian gene circuits using TALE transcriptional repressors. Nat Chem Biol, 2015, 11(3): 207–213.
[29] Lohmueller JJ, Armel TZ, Silver PA. A tunable zinc finger-based framework for Boolean logic computation in mammalian cells. Nucleic Acids Res, 2012, 40(11): 5180–5187.
[30] Khalil AS, Lu TK, Bashor CJ, et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell, 2012, 150(3): 647–658.
[31] Rebar EJ, Huang Y, Hickey R, et al. Induction of angiogenesis in a mouse model using engineered transcription factors. Nat Med, 2002, 8(12): 1427–1432.
[32] Liu PQ, Morton MF, Reik A, et al. Cell lines for drug discovery: elevating target-protein levels using engineered transcription factors. J Biomol Screen, 2004, 9(1): 44–51.
[33] Gao XF, Yang J, Tsang JCH, et al. Reprogramming to pluripotency using designer TALE transcription factors targeting enhancers. Stem Cell Reports, 2013, 1(2): 183–197.
[34] Zhang ZH,Wu E, Qian ZJ, et al. A multicolor panel of TALE-KRAB based transcriptional repressor vectors enabling knockdown of multiple gene targets. Sci Rep, 2014, 4: 7338.
[35] Cheng AW, Wang HY, Yang H, et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res, 2013, 23(10): 1163–1171.
[36] Chakraborty S, Ji H, Kabadi AM, et al. A CRISPR/Cas9-based system for reprogramming cell lineage specification. Stem Cell Reports, 2014, 3(6): 940–947.
[37] Maeder ML, Linder SJ, Cascio VM, et al. CRISPR RNA-guided activation of endogenous human genes. Nat Methods, 2013, 10(10): 977–979.
[38] Perez-Pinera P, Kocak DD, Vockley CM, et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods, 2013, 10(10): 973–976.
[39] Polstein LR,Gersbach CA. A light-inducible CRISPR-Cas9 system for control of endogenous gene activation. Nat Chem Biol, 2015, 11(3): 198–200.
[40] Chavez A, Scheiman J, Vora S, et al. Highly efficient Cas9-mediated transcriptional programming. Nat Methods, 2015, 12(4): 326–328.
[41] Tanenbaum ME, Gilbert LA, Qi LS, et al. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 2014, 159(3): 635–646.
[42] Hilton IB, D'Ippolito AM, Vockley CM, et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol, 2015, 33(5): 510–517.
[43] Xu XX,Tao YH, Gao XB, et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discov, 2016, 2: 16009.
[44] Morita S, Noguchi H, Horii T, et al. Targeted DNA demethylationusing dCas9-peptide repeat and scFv-TET1 catalytic domain fusions. Nat Biotechnol, 2016, 34(10): 1060–1065.
[45] Liu XS, Wu H, Ji X, et al. Editing DNA methylation in the mammalian genome. Cell, 2016, 167(1): 233–247. e17.
[46] Zalatan JG, Lee ME, Almeida R, et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell, 2015, 160(1/2): 339–350.
[47] Konermann S, Brigham MD, Trevino AE, et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2015, 517(7536): 583–588.
[48] Lawhorn IEB, Ferreira JP, Wang CL. Evaluation of sgRNA target sites for CRISPR-mediated repression of TP53. PLoS ONE, 2014, 9(11): e113232.
[49] Thakore PI, D'Ippolito AM, Song LY, et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods, 2015, 12(12): 1143–1149.
[50] McDonald JI, Celik H, Rois LE, et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biol Open, 2016, 5(6): 866–874.
[51] Vojta A, Dobrinić P, Tadić V, et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Res, 2016, 44(12): 5615–5628.
[52] Kearns NA, Pham H, Tabak B, et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods, 2015, 12(5): 401–403.
[53] Kiani S, Chavez A, Tuttle M, et al. Cas9 gRNA engineering for genome editing, activation and repression. Nat Methods, 2015, 12(11): 1051–1054.
[54] Zetsche B, Volz SE, Zhang F. A split-Cas9 architecture for inducible genome editing and transcription modulation. Nat Biotechnol, 2015, 33(2): 139–142.
[55] Cyranoski D. CRISPR gene-editing tested in a person for the first time. Nature, 2016, 539(7630): 479.
[56] Carthew RW, Sontheimer EJ. Origins and mechanisms of miRNAs and siRNAs. Cell, 2009, 136(4): 642–655.
[57] Ambros V. The functions of animal microRNAs. Nature, 2004, 431(7006): 350–355.
[58] Guo ZY, Maki MD, Ding RF, et al. Genome-wide survey of tissue-specific microRNA and transcription factor regulatory networks in 12 tissues. Sci Rep, 2014, 4: 5150.
[59] Colin A, Faideau M, Dufour N, et al. Engineered lentiviral vector targeting astrocytes. Glia, 2009, 57(6): 667–679.
[60] Brown BD, Cantore A, Annoni A, et al. A microRNA-regulated lentiviral vector mediates stable correction of hemophilia B mice. Blood, 2007, 110(13): 4144–4152.
[61] Annoni A, Brown BD, Cantore A, et al.delivery of a microRNA-regulated transgene induces antigen-specific regulatory T cells and promotes immunologic tolerance. Blood, 2009, 114(25): 5152–5161.
[62] Gentner B, Visigalli I, HiramatsuH, et al. Identification of hematopoietic stem cell-specific miRNAs enables gene therapy of globoid cell leukodystrophy. Sci Transl Med, 2010, 2(58): 58ra84.
[63] Cavazzana-Calvo M, Payen E, Negre O, et al. Transfusion independence andactivation after gene therapy of human β-thalassaemia. Nature, 2010, 467(7313): 318–322.
[64] Brown BD,Venneri MA, Zingale A, et al. Endogenous microRNA regulation suppresses transgene expression in hematopoietic lineages and enables stable gene transfer. Nat Med, 2006, 12(5): 585–591.
[65] Deans TL, Cantor CR, Collins JJ. A tunable genetic switch based on RNAi and repressor proteins for regulating gene expression in mammalian cells. Cell, 2007, 130(2): 363–372.
[66] Lapique N, Benenson Y. Digital switching in a biosensor circuitprogrammable timing of gene availability. Nat Chem Biol, 2014, 10(12): 1020–1027.
[67] Rinaudo K, Bleris L, Maddamsetti R, et al. A universal RNAi-based logic evaluator that operates in mammalian cells. Nat Biotechnol, 2007, 25(7): 795–801.
[68] Leisner M, Bleris L, Lohmueller J, et al. Rationally designed logic integration of regulatory signals in mammalian cells. Nat Nanotechnol, 2010, 5(9): 666–670.
[69] Xie Z, Wroblewska L, Prochazka L, et al. Multi-input RNAi-based logic circuit for identification of specific cancer cells. Science, 2011, 333(6047): 1307–1311.
[70] Winkler W, Nahvi A, Breaker RR. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression. Nature, 2002, 419(6910): 952–956.
[71] Gallivan JP. Toward reprogramming bacteria with small molecules and RNA. Curr Opin Chem Biol, 2007, 11(6): 612–619.
[72] Coppins RL, Hall KB, Groisman EA. The intricate world of riboswitches. Curr Opin Microbiol, 2007, 10(2): 176–181.
[73] Wieland M, Ausländer D, Fussenegger M. Engineering of ribozyme-based riboswitches for mammalian cells. Methods, 2012, 56(3): 351–357.
[74] Ausländer S, Stücheli P, RehmC, et al. A general design strategy for protein-responsive riboswitches in mammalian cells. Nat Methods, 2014, 11(11): 1154–1160.
[75] Kennedy AB, Vowles JV, d'Espaux L, et al. Protein-responsive ribozyme switches in eukaryotic cells. Nucleic Acids Res, 2014, 42(19): 12306–12321.
[76] Win MN, Smolke CD. A modular and extensible RNA-based gene-regulatory platform for engineering cellular function. Proc Natl Acad Sci USA, 2007, 104(36): 14283–14288.
[77] Beisel CL, Bayer TS, Hoff KG, et al. Model-guided design of ligand-regulated RNAi for programmable control of gene expression. Mol Syst Biol, 2008, 4: 224.
[78] Win MN, Smolke CD. Higher-order cellular information processing with synthetic RNA devices. Science, 2008, 322(5900): 456–460.
[79] Des Soye BJ, Patel JR, Isaacs FJ, et al. Repurposing the translation apparatus for synthetic biology. Curr Opin Chem Biol, 2015, 28: 83–90.
[80] Jewett MC, Calhoun KA, VoloshinA, et al. An integrated cell-free metabolic platform for protein production and synthetic biology. Mol Syst Biol, 2008, 4: 220.
[81] Carlson ED, Gan R, Hodgman CE, et al. Cell-free protein synthesis: applications come of age. Biotechnol Adv, 2012, 30(5): 1185–1194.
[82] Bundy BC, Swartz JR. Site-specific incorporation of-propargyloxyphenylalanine in a cell-free environment for direct protein-protein click conjugation. Bioconjug Chem, 2010, 21(2): 255–263.
[83] Conklin BR, Hsiao EC, ClaeysenS, et al. Engineering GPCR signaling pathways with RASSLs. Nat Methods, 2008, 5(8): 673–678.
[84] Nichols CD, Roth BL. Engineered G-protein coupled receptors are powerful tools to investigate biological processes and behaviors. Front Mol Neurosci, 2009, 2: 16.
[85] Dong SY, Rogan SC, Roth BL. Directed molecular evolution of DREADDs: a generic approach to creating next-generation RASSLs. Nat Protoc, 2010, 5(3): 561–573.
[86] Armbruster BN, Li X, Pausch MH, et al. Evolving the lock to fit the key to create a family of G protein-coupled receptors potently activated by an inert ligand. Proc Natl Acad Sci USA, 2007, 104(12): 5163–5168.
[87] Urban DJ, Roth BL. DREADDs (designer receptors exclusively activated by designer drugs): chemogenetic tools with therapeutic utility. Annu Rev Pharmacol Toxicol, 2015, 55(1): 399–417.
[88] Barnea G, Strapps W, Herrada G, et al. The genetic design of signaling cascades to record receptor activation. Proc Natl Acad Sci USA, 2008, 105(1): 64–69.
[89] Airan RD, Thompson KR, Fenno LE, et al. Temporally precisecontrol of intracellular signalling. Nature, 2009, 458(7241): 1025–1029.
[90] Clackson T, Yang W, Rozamus LW, et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proc Natl Acad Sci USA, 1998, 95(18): 10437–10442.
[91] Thomis DC, Marktel S, Bonini C, et al. A fas-based suicide switch in human T cells for the treatment of graft-versus-host disease. Blood, 2001, 97(5): 1249–1257.
[92] Junker K, Koehl U, Zimmerman S, et al. Kinetics of cell death in T lymphocytes genetically modified with two novel suicide fusion genes. Gene Ther, 2003, 10(14): 1189–1197.
[93] Straathof KC, Pulè MA, Yotnda P, et al. An inducible caspase 9 safety switch for T-cell therapy. Blood, 2005, 105(11): 4247–4254.
[94] Wu CY, Roybal KT, Puchner EM, et al. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science, 2015, 350(6258): aab4077.
[95] Berrade L, Kwon Y, Camarero JA. Photomodulation of protein trans-splicing through backbone photocaging of the DnaE split intein. Chembiochem, 2010, 11(10): 1368–1372.
[96] Hughes RM, Freeman DJ, Lamb KN, et al. Optogenetic apoptosis: light-triggered cell death. Angew Chem Int Ed, 2015, 54(41): 12064–12068.
[97] Tucker CL, Vrana JD, Kennedy MJ. Tools for controlling protein interactions usinglight. Curr Protoc Cell Biol, 2014, 64: 17.16.1–17.16.20.
[98] Müller K, Zurbriggen MD, Weber W. Control of gene expression using a red- and far-red light—responsive bi-stable toggle switch. Nat Protoc, 2014, 9(3): 622–632.
[99] Hogeweg P. The roots of bioinformatics in theoretical biology. PLoS Comput Biol, 2011, 7(3): e1002021.
[100] Yang L, Nielsen AAK, Fernandez-RodriguezJ, et al. Permanent genetic memory with>1-byte capacity. Nat Methods, 2014, 11(12): 1261–1266.
[101] Bacchus W, Lang M, El-Baba MD, et al. Synthetic two-way communication between mammalian cells. Nat Biotechnol, 2012, 30(10): 991–996.
[102] Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in. Nature, 2000, 403(6767): 339–342.
[103] Fung E, Wong WW, Suen JK, et al. A synthetic gene-metabolic oscillator. Nature, 2005, 435(7038): 118–122.
[104] Stricker J, Cookson S, Bennett MR, et al. A fast, robust and tunable synthetic gene oscillator. Nature, 2008, 456(7221): 516–519.
[105] Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335–338.
[106] Friedland AE, Lu TK, Wang X, et al. Synthetic gene networks that count. Science, 2009, 324(5931): 1199–1202.
[107] Danino T, Mondragón-Palomino O, Tsimring L, et al. A synchronized quorum of genetic clocks. Nature, 2010, 463(7279): 326–330.
[108] Ellis T, Wang X, Collins JJ. Diversity-based, model-guided construction of synthetic gene networks with predicted functions. Nat Biotechnol, 2009, 27(5): 465–471.
[109] Tabor JJ, Salis HM, Simpson ZB, et al. A synthetic genetic edge detection program. Cell, 2009, 137(7): 1272–1281.
[110] Basu S, Gerchman Y, Collins CH, et al. A synthetic multicellular system for programmed pattern formation. Nature, 2005, 434(7037): 1130–1134.
[111] Bulter T, Lee SG, Wong WW, et al. Design of artificial cell-cell communication using gene and metabolic networks. Proc Natl Acad Sci USA, 2004, 101(8): 2299–2304.
[112] You LC, Cox RSIII, Weiss R, et al. Programmed population control by cell-cell communication and regulated killing. Nature, 2004, 428(6985): 868–871.
[113] Martin VJJ, Pitera DJ, Withers ST, et al. Engineering a mevalonate pathway infor production of terpenoids. Nat Biotechnol, 2003, 21(7): 796–802.
[114] Ajikumar PK, Xiao WH, Tyo KEJ, et al. Isoprenoid pathway optimization for taxol precursor overproduction in. Science, 2010, 330(6000): 70–74.
[115] Weber W, Schoenmakers R, Keller B, et al. A synthetic mammalian gene circuit reveals antituberculosis compounds. Proc Natl Acad Sci USA, 2008, 105(29): 9994–9998.
[116] Jakobus K, Wend S, Weber W. Synthetic mammalian gene networks as a blueprint for the design of interactive biohybrid materials. Chem Soc Rev, 2012, 41(3): 1000–1018.
[117] Fussenegger M, Morris RP, Fux C, et al. Streptogramin-based gene regulation systems for mammalian cells. Nat Biotechnol, 2000, 18(11): 1203–1208.
[118] Urlinger S, Baron U, Thellmann M, et al. Exploring the sequence space for tetracycline-dependent transcriptional activators: novel mutations yield expanded range and sensitivity. Proc Natl Acad Sci U S A, 2000, 97(14): 7963–7968.
[119] Weber W, Fux C, Daoud-El Baba M, et al. Macrolide-based transgene control in mammalian cells and mice. Nat Biotechnol, 2002, 20(9): 901–907.
[120] Weber W, Schoenmakers R, Spielmann M, et al.-derived quorum-sensing systems engineered for adjustable transgene expression in mammalian cells and mice. Nucleic Acids Res, 2003, 31(14): e71.
[121] Weber W, Link N, Fussenegger M. A genetic redox sensor for mammalian cells. Metab Eng, 2006, 8(3): 273–280.
[122] Hartenbach S, Daoud-El Baba M, Weber W, et al. An engineered L-arginine sensor ofenables arginine-adjustable transcription control in mammalian cells and mice. Nucleic Acids Res, 2007, 35(20): e136.
[123] Gitzinger M, Kemmer C, El-Baba MD, et al. Controlling transgene expression in subcutaneous implants using a skin lotion containing the apple metabolite phloretin. Proc Natl Acad Sci USA, 2009, 106(26): 10638–10643.
[124] Gitzinger M, Kemmer C, Fluri DA, et al. The food additive vanillic acid controls transgene expression in mammalian cells and mice. Nucleic Acids Res, 2012, 40(5): e37.
[125] Xie MQ, Ye HF, Charpin-El Hamri G, et al. Antagonistic control of a dual-input mammalian gene switch by food additives. Nucleic Acids Res, 2014, 42(14): e116.
[126] Wang H, Ye HF, Xie MQ, et al. Cosmetics-triggered percutaneous remote control of transgene expression in mice. Nucleic Acids Res, 2015, 43(14): e91.
[127] Xue S, Yin JL, Shao JW, et al. A synthetic-biology-inspired therapeutic strategy for targeting and treating hepatogenous diabetes. Mol Ther, 2017, 25(2): 443–445.
[128] Ye HF, Charpin-El Hamri G, Zwicky K, et al. Pharmaceutically controlled designer circuit for the treatment of the metabolic syndrome. Proc Natl Acad Sci USA, 2013, 110(1): 141–146.
[129] Ye HF, Daoud-El Baba M, Peng RW, et al. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice. Science, 2011, 332(6037): 1565–1568.
[130] G N, Tan A, Farhatnia Y, et al. Channelrhodopsins: visual regeneration and neural activation by a light switch. New Biotechnol, 2013, 30(5): 461–474.
[131] Kawano F, Suzuki H, Furuya A, et al. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun, 2015, 6: 6256.
[132] Kennedy MJ, Hughes RM, Peteya LA, et al. Rapid blue-light–mediated induction of protein interactions in living cells. Nat Methods, 2010, 7(12): 973–975.
[133] Gomez EJ, Gerhardt K, Judd J, et al. Light-activated nuclear translocation of adeno-associated virus nanoparticles using phytochrome B for enhanced, tunable, and spatially programmable gene delivery. ACS Nano, 2016, 10(1): 225–237.
[134] Levskaya A, Weiner OD, Lim WA, et al. Spatiotemporal control of cell signalling using a light-switchable protein interaction. Nature, 2009, 461(7266): 997–1001.
[135] Lee S, Park H, Kyung T, et al. Reversible protein inactivation by optogenetic trapping in cells. Nat Methods, 2014, 11(6): 633–636.
[136] Taslimi A, Vrana JD, Chen D, et al. An optimized optogenetic clustering tool for probing protein interaction and function. Nat Commun, 2014, 5: 4925.
[137] Kaberniuk AA, Shemetov AA, Verkhusha VV. A bacterial phytochrome-based optogenetic system controllable with near-infrared light. Nat Methods, 2016, 13(7): 591–597.
[138] Wang X,Chen XJ, Yang Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods, 2012, 9(3): 266–269.
[139] Xu YX, Hyun YM, Lim K, et al. Optogenetic control of chemokine receptor signal and T-cell migration. Proc Natl Acad Sci USA, 2014, 111(17): 6371–6376.
[140] Nihongaki Y, Kawano F, Nakajima T, et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing. Nat Biotechnol, 2015, 33(7): 755–760.
[141] Taslimi A, Zoltowski B, Miranda JG, et al. Optimized second-generation CRY2–CIB dimerizers and photoactivatable Cre recombinase. Nat Chem Biol, 2016, 12(6): 425–430.
[142] Jain PK, Ramanan V, Schepers AG, et al. Development of light-activated CRISPR using guide RNAs with photocleavable protectors. Angew Chem Int Ed, 2016, 55(40): 12440–12444.
[143] Stanley SA, Gagner JE, Damanpour S, et al. Radio-wave heating of iron oxide nanoparticles can regulate plasma glucose in mice. Science, 2012, 336(6081): 604–608.
[144] Stanley SA, Sauer J, Kane RS, et al. Remote regulation of glucose homeostasis in mice using genetically encoded nanoparticles. Nat Med, 2015, 21(1): 92–98.
[145] Leibiger IB, Berggren PO. Regulation of glucose homeostasis using radiogenetics and magnetogenetics in mice. Nat Med, 2015, 21(1): 14–16.
[146] Wheeler MA, Smith CJ, Ottolini M, et al. Genetically targeted magnetic control of the nervous system. Nat Neurosci, 2016, 19(5): 756–761.
[147] Kemmer C, Gitzinger M, Daoud-El Baba M, et al. Self-sufficient control of urate homeostasis in mice by a synthetic circuit. Nat Biotechnol, 2010, 28(4): 355–360.
[148] Rössger K, Charpin-El-Hamr iG, Fussenegger M. A closed-loop synthetic gene circuit for the treatment of diet-induced obesity in mice. Nat Commun, 2013, 4: 2825.
[149] Zoeller RT, Tan SW, Tyl RW. General background on the hypothalamic-pituitary-thyroid (HPT) axis. Crit Rev Toxicol, 2007, 37(1/2): 11–53.
[150] Saxena P, Charpin-El Hamri G, Folcher M, et al. Synthetic gene network restoring endogenous pituitary-thyroid feedback control in experimental Graves’ disease. Proc Natl Acad Sci USA, 2016, 113(5): 1244–1249.
[151] Carey W, Glazer S, Gottlieb AB, et al. Relapse, rebound, and psoriasis adverse events: an advisory group report. J Am Acad Dermatol, 2006, 54(4): S171–S181.
[152] Schukur L, Geering B, Charpin-El Hamri G, et al. Implantable synthetic cytokine converter cells with AND-gate logic treat experimental psoriasis. Sci Transl Med, 2015, 7(318): 318ra201.
[153] Ye HF, Xie M, Xue S, et al. Self adjusting synthetic gene circut for correcting insulin resistance. Nat Biomed Engin, 2016, 1(1): 0005.
[154] Lathuiliere A, Cosson S, Lutolf MP, et al. A high-capacity cell macroencapsulation system supporting the long-term survival of genetically engineered allogeneic cells. Biomaterials, 2014, 35(2): 779–791.
[155] Yang HK, Yoon KH. Current status of encapsulated islet transplantation. J Diabetes Complications, 2015, 29(5): 737–743.
[156] Catena R, Santos E, Orive G, et al. Improvement of the monitoring and biosafety of encapsulated cells using the SFGNESTGL triple reporter system. J Control Release, 2010, 146(1): 93–98.
[157] De Castro M, Orive G, HernándezRM, et al. Comparative study of microcapsules elaborated with three polycations (PLL, PDL, PLO) for cell immobilization. J Microencapsul, 2005, 22(3): 303–315.
[158] Wilson JT, Cui WX, Chaikof EL. Layer-by-layer assembly of a conformal nanothin PEG coating for intraportal islet transplantation. Nano Lett, 2008, 8(7): 1940–1948.
[159] Bukrinsky MI, Haggerty S, Dempsey MP, et al. A nuclear localization signal within HIV-1 matrix protein that governs infection of non-dividing cells. Nature, 1993, 365(6447): 666–669.
[160] Gao GP, Yang Y, Wilson JM. Biology of adenovirus vectors with E1 and E4 deletions for liver-directed gene therapy. J Virol, 1996, 70(12): 8934–8943.
[161] Alba R, Bosch A, Chillon M. Gutless adenovirus: last-generation adenovirus for gene therapy. Gene Ther, 2005, 12 Suppl 1: S18–S27.
[162] Xiao X, Li J, Samulski RJ. Production of high-titer recombinant adeno-associated virus vectors in the absence of helper adenovirus. J Virol, 1998, 72(3): 2224–2232.
[163] Yin H, Kanasty RL, Eltoukhy AA, et al. Non-viral vectors for gene-based therapy. Nat Rev Genet, 2014, 15(8): 541–555.
(本文责编 陈宏宇)
叶海峰 华东师范大学生命科学学院,研究员,博士生导师。主要从事合成生物学与生物医学工程领域研究。相关研究成果发表在、、、、、等杂志。2013年获瑞士苏黎世联邦理工大学荣誉奖章“ETH Silver Medal”。2014年入选国家中央组织部第11批“青年千人计划”。2015年获得国家自然科学基金“优秀青年基金”资助。目前参与国家科技部重大专项子课题1项、主持国家自然科学基金面上项目2项、上海市科委基础研究重点项目1项、上海市科技启明星项目1项。申请发明专利6项。先后担任、、等学术期刊审稿人。

Research progress of mammalian synthetic biology in biomedical field
Linfeng Yang, Jianli Yin, Meiyan Wang, and Haifeng Ye
Shanghai Key Laboratory of Regulatory Biology, School of Life Sciences, East China Normal University, Shanghai 200241, China
Although still in its infant stage, synthetic biology has achieved remarkable development and progress during the past decade. Synthetic biology applies engineering principles to design and construct gene circuits uploaded into living cells or organisms to perform novel or improved functions, and it has been widely used in many fields. In this review, we describe the recent advances of mammalian synthetic biology for the treatment of diseases. We introduce common tools and design principles of synthetic gene circuits, and then we demonstrate open-loop gene circuits induced by different trigger molecules used in disease diagnosis and close-loop gene circuits used for biomedical applications. Finally, we discuss the perspectives and potential challenges of synthetic biology for clinical applications.
synthetic biology, synthetic gene circuits, gene-based therapy, cell-based therapy
November 7, 2016; Accepted:January 6, 2017
Haifeng Ye. Tel: +86-21-54341058; E-mail: hfye@bio.ecnu.edu.cn
Supported by:National Key Research and Development Program of China, Stem Cell and Translational Research (No. 2016YFA0100300), National Natural Science Foundation of China (Nos. 31470834, 31522017, 31670869), Science and Technology Commission of Shanghai Municipality (Nos. 15QA1401500, 14JC1401700), Thousand Youth Talents Plan.
国家重大研究计划 (No. 2016YFA0100300),国家自然科学基金 (Nos. 31470834, 31522017, 31670869),上海市科委 (Nos. 15QA1401500, 14JC1401700),青年千人计划资助。
2017-01-17
http://www.cnki.net/kcms/detail/11.1998.Q.20170117.0853.002.html

