滇牡丹ω-3脂肪酸脱氢酶基因克隆与功能分析
朱金鑫,孙金金,原晓龙,王 娟,杨宇明,王 毅
( 1.西南林业大学林学院,昆明 650224; 2.国家林业局重点开放性实验室云南珍稀濒特森林植物保护和繁育实验室,云南省森林植物培育与开发利用重点实验室,昆明 650204; 3.云南省林业科学院,昆明 650204)
生物工程
滇牡丹ω-3脂肪酸脱氢酶基因克隆与功能分析
朱金鑫1,2,孙金金1,2,原晓龙2,3,王 娟2,3,杨宇明2,3,王 毅2,3
( 1.西南林业大学林学院,昆明 650224; 2.国家林业局重点开放性实验室云南珍稀濒特森林植物保护和繁育实验室,云南省森林植物培育与开发利用重点实验室,昆明 650204; 3.云南省林业科学院,昆明 650204)
ω-3脂肪酸脱氢酶是多不饱和脂肪酸亚麻酸生物合成途径的关键酶。依据转录组数据设计特异性引物,使用RT-PCR方法从滇牡丹克隆得到1个FAD3基因的cDNA全长序列,命名为PdFAD3(GeneBank登录号为KX289610)。生物信息学分析结果表明PdFAD3基因片段序列全长2 134 bp,包含完整的cDNA开放阅读框,编码含有450个氨基酸残基的蛋白质;Blast比对结果显示该蛋白质属于FAD蛋白质家族;系统进化树分析结果显示与芍药属芍药组植物芍药亲缘关系较近;该蛋白质属于跨膜蛋白质,亲水性较低;RT-PCR结果显示其在种子中的表达随着种子成熟出现双峰型的表达量变化,与目前所报道的其他物种的FAD3表达情况相似。为进一步研究其调控机理提供理论依据及获得含高不饱和脂肪酸转基因滇牡丹品种奠定理论基础。
滇牡丹;ω-3脂肪酸脱氢酶;基因克隆;基因功能分析
牡丹是我国传统名花,具有很高的观赏和药用价值,牡丹籽油作为中药丹皮(牡丹根皮)的副产品,其食用药用价值逐渐被人们发现并不断加强重视。研究发现牡丹籽油中含有丰富的不饱和脂肪酸[1],具有重要的研究价值和应用潜力,自2011年中华人民共和国卫生部将牡丹籽油列入新资源食品后,油用牡丹迅速成为研究热点。滇牡丹(Paeoniadelavayi)主要分布在我国云南中部至西北部、西藏东南部和四川西南部海拔在1 900~3 600 m的部分区域,是我国西南地区特有珍稀种质资源,滇牡丹虽然自然分布狭窄,但其生态适应性强,栽培范围广[2],因此滇牡丹具有极大的引种驯化和育种空间,应用价值高。
多不饱和脂肪酸(PUFAs)是指含有两个或两个以上双键的长链脂肪酸[3],在生物体内具有重要作用[4-5]。自20世纪90年代后高血压、肥胖症及心血管疾病等对人类健康的威胁越来越大,美国、日本等国将研究重点转向功能食品,其中主要研究对象为磷脂与多不饱和脂肪酸[6]。但由于高等动物体内缺乏一系列脂肪酸脱氢酶而不能自身合成α-亚麻酸、亚油酸等多不饱和脂肪酸,因此研究人员希望通过研究多不饱和脂肪酸的合成途径,获得高含量不饱和脂肪酸的植物品种。
牡丹籽油中α-亚麻酸属于ω-3 PUFAs,ω-3脂肪酸脱氢酶催化饱和脂肪酸脱氢反应生成不饱和脂肪酸,是α-亚麻酸合成的关键酶,ω-3脂肪酸脱氢酶基因已经在拟南芥[7]、大豆[8]、小麦[9]等多种植物中得到克隆。研究人员将ω-3脂肪酸脱氢酶基因导入烟草后发现其不饱和脂肪酸含量显著增加[10],转FAD3基因番茄叶片和果实中不饱和脂肪酸含量均有增加[11],而对ω-3脂肪酸脱氢酶基因进行基因沉默处理后不饱和脂肪酸含量降低[12],由此可见ω-3脂肪酸脱氢酶在植物体内扮演调控多不饱和脂肪酸含量的角色。从滇牡丹中克隆得到ω-3 脂肪酸脱氢酶基因并进行生物信息学分析,为进一步研究其调控机理及获得含高不饱和脂肪酸转基因品种奠定理论基础。
1 材料与方法
1.1 实验材料
滇牡丹种子于2014年6— 9月采集于云南省林业科学院树木园苗圃,样本经液氮速冻后于-80℃ 冰箱保存待用。总RNA小量提取试剂盒购自美国Qiagen, pESY-T3克隆试剂盒与感受态细胞购自北京全式金生物技术有限公司,LA-Taq酶、Reverse Transcriptase M-MLV购自大连宝生物工程有限公司。
1.2 实验方法
1.2.1 PdFAD3基因克隆
实验方法参照李苏雨等[13]的方法。具体操作如下:通过转录组分析获得1个只含有部分滇牡丹ω-3脂肪酸脱氢酶(PdFAD3)基因的序列片段,根据该基因片段序列设计特异引物5′-RACE引物(本实验所用引物见表1)对5′-基因片段进行克隆。按照总RNA小量提取试剂盒操作步骤提取总RNA, 检测总RNA浓度及完整性后反转录合成cDNA第一链。按照试剂盒操作步骤扩增获得PdFAD3 基因5′端基因序列片段,利用ATG序列拼接软件将5′-RACE获得基因片段和转录组数据分析获得的基因片段进行拼接后获得PdFAD3全长基因序列。以PdFAD3基因全长序列为模板,设计含有起始密码子和终止密码子的特异引物PdFAD3F0和PdFAD3R0,以滇牡丹种子cDNA为模板,PdFAD3F0和PdFAD3R0为引物扩增滇牡丹FAD3基因cDNA开放阅读框全长序列。PCR反应体系参考PrimeScriptTM Kit. 扩增程序为: 94℃ 5 min; 94℃ 30 s; 58℃ 45 s; 72℃ 90 s; 30个循环后72℃延伸7 min。将PCR产物转入pESY-T3载体中送测序公司检测并验证。

表1 引物序列
1.2.2 生物信息学分析
应用NCBI(http://www.ncbi.nlm.nih.gov/) Blast和DNAMAN生物学软件对PdFAD3的氨基酸序列进行多序列比对分析;利用软件MEGA 5.0以邻位连接法(Neighbor-Joining)构建PdFAD3的氨基酸序列的系统进化树;使用ProtScale(http://web.expasy.org/protscale/)在线工具进行蛋白质亲疏水性预测分析;使用ProtScale(http://web.expasy.org/protparam)在线工具分析蛋白质理化性质;使用Tmpred(http://www.ch.embnet.org/software/TMPRED_form.html)在线工具分析PdFAD3蛋白质序列跨膜结构域。
1.2.3 荧光定量PCR检测PdFAD3组织表达分析
以获得的PdFAD3基因序列为模板设计特异性引物PdFAD3Ft和PdFAD3Rt(见表1);分别采集滇牡丹授粉后,30、50、70 d及90 d的种子,采集后立即放入液氮中。用RNA小量提取试剂盒提取滇牡丹不同发育时期的种子总RNA,电泳检测后-80℃保存;参照Reverse Transcriptase M-MLV试剂盒合成cDNA,保存于-20℃; Real-time RT-PCR反应用荧光染料SYBR Green Ⅰ, GAPDH为内参基因。PCR反应体系20 μL, 反应程序为:94℃ 4 min;95℃ 15 s; 57℃ 15 s; 72℃ 25 s; 35个循环,72℃单点检测信号。反应结束后,60℃连续检测信号产生溶解曲线。用Opticon monitor 3.1软件进行数据的记录和分析。每个样品3次重复,灭菌水对空白对照。
2 结果与分析
2.1 PdFAD3基因克隆与序列分析
通过分析滇牡丹种子转录组获得1 452 bp的ω-3脂肪酸脱氢酶基因片段,依据获得基因片段设计5′-RACE的基因扩增特异引物,以cDNA为模板进行PCR扩增, 5′-RACE获得812 bp基因片段 (见图1),利用ATGC序列拼接软件,最终获得PdFAD3全长cDNA序列2 134 bp。利用NCBI ORF Finder软件对拼接数据结果进行序列分析,结果显示PdFAD3基因片段包含完整的cDNA开放阅读框(ORF),序列全长1 353 bp,5′端非编码区 302 bp,3′端非编码区 448 bp;编码450个氨基酸残基的蛋白质,起始密码子为ATG,终止密码子TGA。并根据获得的PdFAD3基因全长设计含有起始密码子和终止密码子的特异引物,以cDNA为模板扩增获得PdFAD3 基因开放阅读框(见图1),并将扩增产物连接到克隆载体中,进行永久保存。
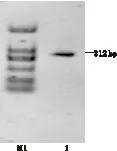
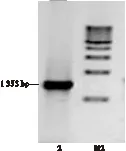
注:M1. Trans 2k DNA marker; M2. Takara 1k DNA marker;1. 5′-RACE;2.滇牡丹FAD3开放阅读框PCR产物。
图1 滇牡丹FAD3基因克隆
2.2 PdFAD3氨基酸序列比对分析
将克隆出来的PdFAD3基因编码的蛋白质氨基酸序列与已报道的FAD3的氨基酸序列进行多重序列比对。
结果显示该蛋白质与已报道的FAD3蛋白质超家族的保守结构域具有高度一致性,氨基酸序列中有3个保守组氨酸中心位点[14],表明该蛋白质属于FAD3家族蛋白质。PdFAD3的氨基酸序列与大豆(Glycinemax)氨基酸序列(NP_001237361.1)相似性达74.18%, 与胡桃(Juglansregia)氨基酸序列(AHJ79158.1)相似性达75.93%, 与芍药(Paeonialactiflora)氨基酸序列(NP_001046132.1)相似性达94.67%, 与蓖麻(Ricinuscommunis)氨基酸序列(CAA13176.1)相似性达71.52%(见图2)。
蛋白质理化性质分析结果显示PdFAD3分子式为C2364H3558N646O632S14, 相对分子质量为51 589.0, 理论等电点为9.11, 亲水性平均系数为-0.291, 在线软件ProtScale进一步分析结果显示蛋白质亲水性较低(见图3)。在线软件Tmpred软件分析蛋白质跨膜结构结果显示存在5个跨膜区,表明PdFAD3为跨膜蛋白质(见表2)。
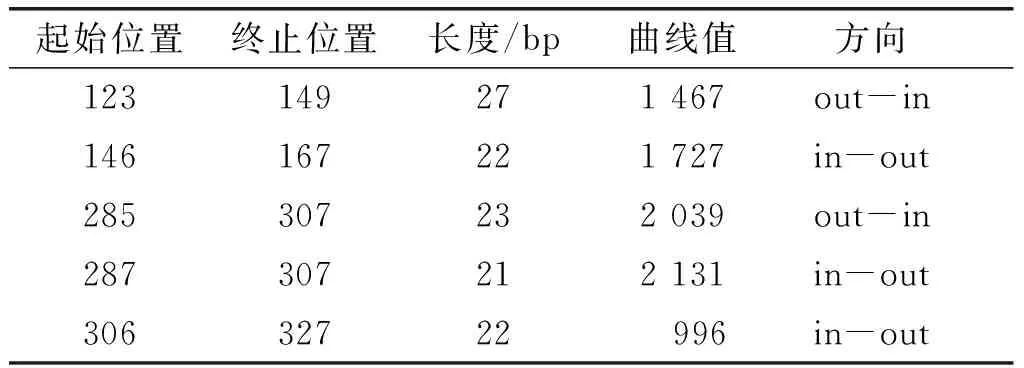
表2 滇牡丹FAD3蛋白质跨膜结构预测
2.3 PdFAD3基因系统进化树分析
利用MEGA 5.0软件构建基PdFAD3氨基酸的系统进化树(见图4)。结果表明,PdFAD3与芍药属植物芍药PlFAD3聚为一类,亲缘关系最近,与双子叶植物亲缘关系次之,与单子叶植物聚为不同分支,亲缘关系最远。由基因系统进化树可知,FAD3基因的聚类与植物分类大体一致。
2.4 荧光定量PCR检测PdFAD3
利用RT-PCR方法和PdFAD3的基因特异引物检测其在授粉后30、50、70、90 d的种子中的表达情况(见图5)。结果显示PdFAD3的表达情况为双峰型,在50 d时PdFAD3的表达量达到第1个高峰,此后表达水平降低,在70 d后表达水平再次升高。结果显示在授粉后50 d内因合成生物膜需求PdFAD3 表达水平较高,而在种子发育后期,即授粉50~70 d内,PdFAD3表达水平降低,α-亚麻酸等不饱和脂肪酸合成量降低,以积累饱和脂肪酸等物质以备萌发所需,PdFAD3表达状况与目前报道的其他物种的FAD3的表达状况相似[15]。

注:PdFAD3为滇牡丹FAD3;GmFAD3为大豆FAD3;JrFAD3为胡桃FAD3;PlFAD3为芍药FAD3;
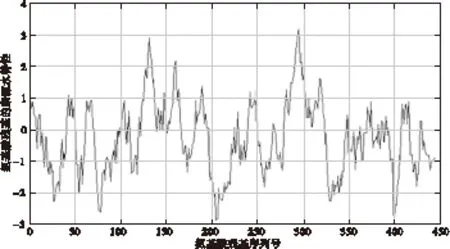
图3 滇牡丹FAD3蛋白质亲水性分析
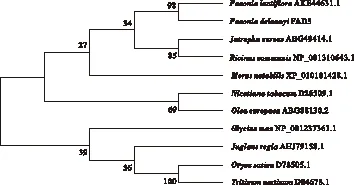
图4 PdFAD3与其他植物FAD3系统进化树
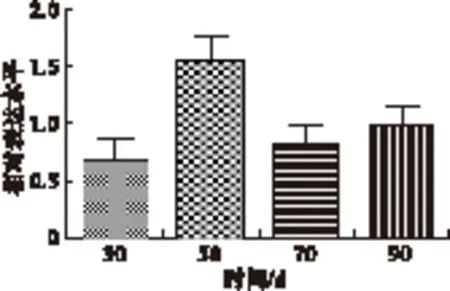
图5 滇牡丹种子不同时期FAD3表达水平
3 结 论
ω-3脂肪酸脱氢酶是多不饱和脂肪酸亚麻酸生物合成途径中的关键酶。本研究对转录组数据分析后克隆得到滇牡丹PdFAD3基因序列,GeneBank登录号为KX289610, 包含完整的cDNA开放阅读框,开放阅读框全长1 353 bp, 编码450个氨基酸,生物信息学分析显示PdFAD3与芍药FAD3蛋白质氨基酸序列相似性高达94.67%, 亲缘关系较近,属于FAD3蛋白质家族,该蛋白质属于跨膜蛋白质,亲水性较低。分析结果显示ω-3脂肪酸脱氢酶基因比较保守,可以作为未来滇牡丹不饱和脂肪酸高产型新品种培育的调控研究重点。
[1] 周海梅, 马锦琦, 苗春雨,等. 牡丹籽油的理化指标和脂肪酸成分分析[J]. 中国油脂, 2009, 34(7):72-74.
[2] 李奎. 滇牡丹保护生物学与遗传多样性研究[D]. 北京:中国林业科学研究院, 2013.
[3] 杭晓敏, 唐涌濂. 多不饱和脂肪酸的研究进展[J]. 中国生物工程杂志, 2001, 21(4):18-21.
[4] MARTINS L P, SILVA S D M, ALVES R E. Chilling injury physiology in red mombin fruit (SpondiaspurpureaL.)[J]. Rev Bras Frutic, 2003, 25(1):23-26.
[5] 毛峰, 秦振华. 长链多不饱和脂肪酸与人体健康[J]. 医药产业资讯, 2006, 3(3):40-41.
[6] 刘红梅. 油脂中功能性成分的研究进展[J]. 轻工科技, 2015(8):24-25.
[7] GIBSON S,ARONDEL V, IBA K, et al. Cloning of a temperature-regulated gene encoding a chloroplastomega-3 desaturase fromArabidopsisthaliana[J]. Plant Physiol, 1995, 106(4):1615-1621.
[8] 刘文浩, 李伟, 陈相艳,等.ω-3脂肪酸脱氢酶在高、低油大豆品种中的克隆与序列分析[J]. 大豆科学, 2008, 27(4):569-571.
[9] 李敏, 高翔, 陈其皎,等. 小麦品种陕253ω-醇溶蛋白基因的克隆及序列分析[J]. 农业生物技术学报, 2010, 18(5):853-860.
[10] SHIMADA T,WAKITA Y, OTANI M, et al. Modification of fatty acid composition in rice plants by transformation with a tobacco microsomal.Omega-3 fatty acid desaturase gene (NtFAD3)[J]. Plant Biotechnol, 2000, 17(1):43-48.
[12] MURAKAMI Y, TSUYAMA M, KOBAYASHI Y, et al. Trienoic fatty acids and plant tolerance of high temperature[J]. Science, 2000, 287(5452):476-479.
[13] 李苏雨, 王毅, 原晓龙,等. 滇牡丹ω-6脂肪酸脱氢酶基因的克隆与功能分析[J]. 西部林业科学, 2016,45(2):22-28.
[14] 官玲亮, 侯凯, 陈郡雯,等.ω-6和ω-3脂肪酸脱氢酶家族系统进化与功能分化[J]. 遗传, 2013, 35(5):643-654.
[15] LI S S, WANG L S, SHU Q Y, et al. Fatty acid composition of developing tree peony (PaeoniasectionMoutanDC.) seeds and transcriptome analysis during seed development[J]. BMC Genomics, 2015, 16(1):1-14.
Cloning and functional analysis ofomega-3 fatty acid desaturase gene fromPaeoniadelavayi
ZHUJinxin1,2,SUNJinjin1,2,YUANXiaolong2,3,WANGJuan2,3,YANGYuming2,3,WANGYi2,3
(1.ForestryCollege,SouthwestForestryUniversity,Kunming650224,China; 2.YunnanProvincialKeyLaboratoryofCultivationandExploitationofForestPlants,YunnanLaboratoryforConservation>ofRare,Endangered&EndemicForestPlants,PublicKeyLaboratoryoftheStateForestryAdministration,Kunming650204,China;3.YunnanAcademyofForestry,Kunming650204,China)
Omega-3 fatty acid desaturase is the key enzyme in the biosynthesis pathway of linolenic belonging to polyunsaturated fatty acids. A full-length cDNA sequences of a new FAD3 gene was successfully cloned fromPaeoniadelavayiby RT-PCR method with the specific primers designed based on transcriptome datas, named PdFAD3(GeneBank accession number: KX289610).Bioinformatics analysis results showed that full-length sequence of PdFAD3 gene was 2 134 bp, containing the complete cDNA open reading frame (ORF), encoded a protein with 450 amino acids. Blast comparison results showed that the protein belonged to the FAD family. Analysis results of phylogenetic tree showed thatPaeoniadelavayihad close genetic relationship withPaeonialactiflorabelonging toPaeoniaSect.Paeonia. The protein belonged to the transmembrane protein and was less hydrophilic. RT-PCR results showed that the expression in the seed as maturation occured bimodal change, similar to the FAD3 reported in other species. The PdFAD3 gene cloned fromPaeoniadelavayiprovided a scientific basis for further study on its regulation mechanism and obtainingPaeoniadelavayitransgenic variety with highly unsaturated fatty acids.
Paeoniadelavayi;omega-3 fatty acid desaturase; gene clone; gene functional analysis
2016-06-14;
2016-12-11
云南省林业科学院重点实验室自主创新项目(22CX2016-05);云南省科技计划项目(2015A005);云南省应用基础研究重点项目(2013FA054)
朱金鑫(1993),男,硕士研究生,研究方向为植物分子生物学(E-mail)1639807959@qq.com。
王 毅,助理研究员(E-mail)22825818@qq.com。
TS222;S502
A
1003-7969(2017)02-0102-05

