基于微球和微柱阵列芯片的乳滴数字PCR定量方法*
程祖乐, 王 琨, 毛红菊, 夏文薇, 金庆辉, 赵建龙
(1.中国科学院 上海微系统与信息技术研究所 传感技术联合国家重点实验室,上海 200050;2.中国科学院 研究生院,北京 100039;3.上海交通大学 医学院 附属第九人民医院,上海 200011)
基于微球和微柱阵列芯片的乳滴数字PCR定量方法*
程祖乐1,2, 王 琨1,2, 毛红菊1, 夏文薇3, 金庆辉1, 赵建龙1
(1.中国科学院 上海微系统与信息技术研究所 传感技术联合国家重点实验室,上海 200050;2.中国科学院 研究生院,北京 100039;3.上海交通大学 医学院 附属第九人民医院,上海 200011)
BEAMing是一种基于磁珠表面核酸扩增的乳滴数字聚合酶链反应(PCR)技术,具有很高的灵敏度, 然而后续检测目标磁珠比较困难。通过修饰链霉亲和素的聚苯乙烯微球捕获BEAMing实验中生物素修饰的目标磁珠并利用微柱阵列芯片拦截微球,可以达到统计磁珠的目的。微柱阵列芯片采用基于尺寸差异的拦截原理。该芯片组装简单成本低,降低了BEAMing技术的磁珠统计难度。利用该方法对不同浓度的特定DNA序列做了检测,验证了该方法的实用性。
微柱阵列芯片; 乳滴数字聚合酶链反应; 微球; 生物传感器
0 引 言
BEAMing(beads,emulsion,amplification,and magnetics)是一种基于磁珠固相扩增的乳滴数字聚合酶链反应(PCR)技术[1],由于扩增磁珠与靶标分子个数存在对应关系,通过检测扩增磁珠即可得到靶标分子的拷贝数[2]。传统的BEAMing实验磁珠计数困难且成本高[1~3]。
本文在先前实验室微球过滤方法[4]的基础上提出了一种基于微球和微柱阵列芯片传感器[5,6]的BEAMing核酸检测方法,该方法在保证了BEAMing检测灵敏度的同时提高了检测效率,降低了检测难度。
1 实验方法
1.1 BEAMing实验
实验引物序列如表1所示。将10 μL SA修饰磁珠Dynabeads M—270(美国Invitrogen公司)与1 μL 100 mmol/L 引物1在结合缓冲液(5 mmol/L pH 7.5三羟甲基氨基甲烷—氯化氢(Tris—HCl),0.5 mmol/L 乙二胺四乙酸(EDTA),1 mol/L NaCl)中室温孵育15 min。利用磁场富集磁珠并用重悬液(20 mmol/L pH 8.4 Tris—HCl,50 mmol/L KCl)清洗3次,用10 μL重悬液重悬。油乳混合液由质量浓度7 %ABIL WE09(德国Evonik Degussa公司),体积分数20 %的矿物油(美国Sigma公司)和体积分数73 %的Tegosoft DEC(德国Evonik Degussa公司)组成。反应预混液成分如表2所示。在一个1.5 mL离心管中加入一颗5 mm钢珠,200 μL油乳混合液和40 μL反应预混液,放入全自动多样品组织研磨仪(Tissuelyser—24,上海净信科技公司)中33Hz频率振荡20s形成油包水乳液。转移乳液至500 μL PCR反应管中,在标准PCR仪(日本Takara公司)中扩增,反应条件包括:94 ℃/2 min热启动激活,98 ℃/15 s变性,60 ℃/45 s退火,72 ℃/75 s延伸,共45个从变性到延伸循环。扩增完成之后,收集乳液至1.5 mL离心管中,加入600 μL破乳缓冲液(10 mmol/L Tris—HCl (pH 7.5),1 % 氚核Triton—X 100,1 % SDS,100 mmol/L NaCl,1 mmol/L EDTA),振荡后放入离心机(德国Eppendof公司)中,3 200gn下,离心3 min,吸去上层油相之后,重复以上步骤1次。富集磁珠,移除上清后加入40 μL结合缓冲液重悬。
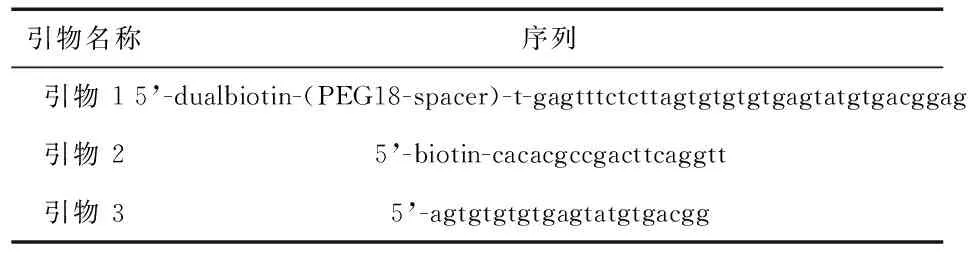
表1 引物序列
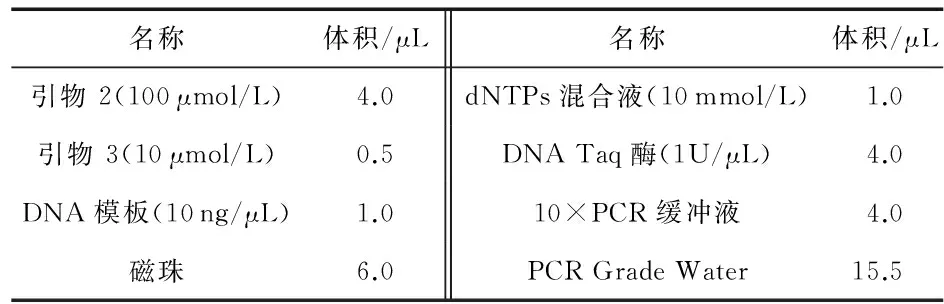
表2 预混液成分
1.2 微柱阵列芯片的制作
如图1所示,将硅片置于煮沸的清洗液(H2SO4∶H2O2=10∶1)中浸泡10 min。取出硅片,放入去离子水清洗腔中喷淋甩干。将LC100A正胶旋涂在清洗后的硅片上,旋涂厚度为3 μm。涂完后前烘,利用曝光显影工艺,将掩膜版上的图案转移到光刻胶上,暴露出硅片上被刻蚀的部分。利用深反应离子刻蚀(DRIE)在硅片上刻蚀出一系列微井阵列,微井大小为130 μm×30 μm,微井间距随着行数减小,变化范围为50~6 μm,微井阵列为0.8 μm×0.8 cm,刻蚀深度为30 μm,用等离子去胶工艺去除残胶。对制作好硅片模具硅烷化,然后取聚二甲基硅氧烷(PDMS)预聚体:固化剂为10∶1配制PDMS。将配制好的PDMS浇筑在硅片模具上,静置1~2 h,90 ℃加热1 h。待PDMS完全固化后剥离,微井阵列转化为微柱阵列,然后用蓝膜包覆,手动切割打孔。最后将载玻片与芯片通过等离子体键合。至此芯片组装完成[7]。

图1 硅片模具的制作流程
1.3 检测流程
取1 μL SA聚苯乙烯微球(约5 000个),用结合缓冲液稀释至10 μL,每次1 μL分5次逐步加入到制备好的40 μL磁珠悬浊液中充分吹打,室温孵育30 min,随后 6 000 r/min,离心5 min,吸去上清液。加入40 μL PBST溶液(pH 7.4 聚丁二酸丁二醇酯(PBS)溶液,0.01 %失水山梨醇聚氧乙烯醚酯(Tween 20)重悬,吹打混匀。富集磁珠,吸去上清液。重复3次清洗步骤后,用40 μL PBST溶液重悬。
微柱阵列芯片采取负压进样,用进样仪将200 μL PBST溶液通入到装置中,以10 mL/h的速率冲洗。取20 μL微球和磁珠重悬液,用PBST溶液将其重悬至100 μL,以5 mL/h的速率通入到装置中。进样完成后,用200 μL PBST冲洗2次。
2 结果讨论
2.1 检测原理
BEAMing实验中加入磁珠数远大于靶标分子数,满足单个磁珠与靶标分子共存在一个液腔中的泊松分布条件。引物2参与靶标分子的扩增使目标磁珠带上生物素,引物3提高了磁珠表面扩增效率[2]。SA修饰微球捕获目标磁珠形成复合物并在磁场下富集,游离微球则随着上清液一起吸除。微球磁珠复合物会因尺寸大而被截留在微柱阵列通道内,游离的磁珠则流出芯片。每个微球饱和结合磁珠数量约为20个,通过统计截留的微球,即可知道目标核酸大致个数。
2.2 乳液的热稳定性与试剂配比
为了验证乳滴热稳定性,分别在热循环前后吸取0.5 μL乳液在倒置显微镜(日本OLYMPUS公司)400倍下观察,乳滴状态没有发生变化。为了保证大于80 %的液滴最多只能包含一个磁珠,根据泊松分布,反应中加入的磁珠数最多应为乳滴的80 %。按照乳滴直径为10~15 μm的标准,40 μL的预混液可以打出3×106~1×107个乳滴,每毫升磁珠悬浊液含有(6~7)×108个磁珠,据此计算,应加入的磁珠量为5~8 μL。本文选取了6 μL作为加入量,结果如图2所示。磁珠较为均匀地分散在乳滴中,且大部分乳滴只含有一个磁珠。

图2 微乳滴与磁珠分布示意图
2.3 定量结果分析
实验采用分步加入微球的方法保证SA微球能与目标磁珠充分结合,每一步加入的微球数量约在500个左右,其具体加入的数量亦可调整。孵育之后,利用磁铁聚集磁珠,吸取上清液放置在显微镜下观察,上清液中的微球如图3(a)所示,图3(b)显示的是磁珠微球复合物的形貌。
为了验证该方法可行性,选取一段长度为66 bp的cDNA片段作为检测标的。将含有该序列的cDNA溶液按2倍梯度稀释之后分别对这3个溶液样本进行了检测,图4为芯片中截取部分微球拦截结果。3张芯片分别截取到287,148,85个微球,转化为核酸拷贝数约为5 740,2 960,1 700个拷贝,而空白对照试验无微球截获。该结果与稀释倍数相对照呈现了较好的线性关系,表明本方法具有可行性。

图3 上清液和沉淀中微球形态
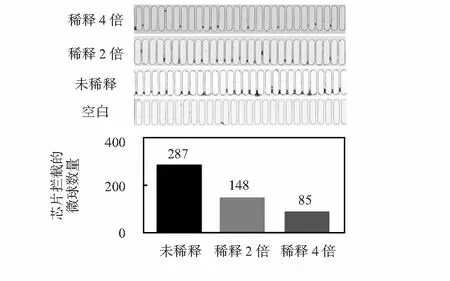
图4 不同模板浓度溶液样本的芯片拦截结果
3 结束语
本文根据BEAMing技术和微流控芯片技术设计了一种核酸检测方法,其特点包括:1)计数简单,只要统计微球
数便可知道整个溶液中磁珠数量的大致范围;2)BEAMing具有乳滴数字PCR的特点,可以很容易达到几十万级甚至百万级的反应腔数,同时又不增加实验操作难度;3)根据检测要求和通量的不同,下一步还可以增加微柱数和芯片面积,改变微球直径大小参数等,并且可以对微球进行不同基团修饰,做到多重检测。综上,本文提出的核酸检测方法,检测成本低,制备操作简单,具有广泛的应用前景。
[1] Dressman D,Yan H,Traverso G,et.al.Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations[C]∥Proceedings of the National Academy of Sciences,2003:8817-8822.
[2] Diehl F,Li M,He Y,et al.BEAMing:Single-molecule PCR on microparticles in water-in-oil emulsions[J].Nature Methods,2006,3:551-559.
[3] Boulanger J,Muresan L,Tiemann-Boege I.Massively parallel haplotyping on microscopic beads for the high-throughput phase ana-lysis of single molecules[J].Summaries of Technical Papers of Annual Meeting Architectural Institute of Japan,D:Environmental Engineering,2012,7(4):292-294.
[4] 范晓云,贾春平,李 刚,等.一种用于循环肿瘤细胞检测的生物传感器芯片[J].传感器与微系统,2015,34(11):119-121.
[5] Mohamed H,Murray M,Turner J N,et al.Isolation of tumor cells using size and deformation[J].Journal of Chromatography A,2009,1216(47):8289-8295.
[6] Koh Y,Lee B R,Yoon H J,et al.Bead affinity chromatography in a temperature-controllable microsystem for biomarker detec-tion[J].Analytical & Bioanalytical Chemistry,2012,404(8):2267-2275.
[7] 王 丽,李 刚,金庆辉,等.玻璃-PDMS薄膜-玻璃夹心微流控芯片制作[J].传感器与微系统,2016,35(1):101-103.
程祖乐(1990 -),男,硕士研究生,主要研究方向为微流控技术在核酸检测中的应用。
毛红菊,通讯作者,E—mail:hjmao@mail.sim.ac.cn。
Emulsion digital PCR quantitative method based on microbeads and micropillar array chip*
CHENG Zu-le1,2, WANG Kun1,2, MAO Hong-ju1, XIA Wen-wei3, JIN Qing-hui1, ZHAO Jian-long1
(1.State Key Laboratory of Transducer Technology,Shanghai Institute of Microsystem and Information Technology,Chinese Academy of Sciences,Shanghai 200050,China;2.Graduate School of Chinese Academy of Sciences,Beijing 100039,China;3.Shanghai Ninth People’s Hospital, School of Medicine,Shanghai Jiao Tong University,Shanghai 200011,China )
BEAMing is an emulsion digital polymerase chain reaction(PCR)technology with high sensitivity.However,the available beads detecting method is complex,which limits its' applications.Through counting the streptavidin modified polystyrene latex microspheres which have fully captured the target magnetic beads,the number of target magnetic beads can be calculated easily.The microsphere trapping mechanism is based on the difference of microsphere diameter and micropillars intervals.The fabrication of this chip is simple and low-cost.This chip-and-beads-based method reduces difficulty of magnetic beads counting in BEAMing.Feasibility of this method is demonstrated by detecting a DNA sequence in different concentrations.
micropillar array chip; emulsion digital polymerase chain reaction(PCR); microspheres; biosensor
10.13873/J.1000—9787(2017)05—0016—03
2016—05—18
国家重大科学研究计划资助项目( 2012CB933303);国家自然科学基金资助项目( 61571429,61271161);上海市科委资助项目(15140902402)
TP 212.3; O 482.54
A
1000—9787(2017)05—0016—03

