凭祥睑虎线粒体基因组序列测定与分析
刘健翔++秦新民
摘要:采用PCR扩增方法测定了凭祥睑虎(Goniurosaurus luii)线粒体基因组全序列。经测序线粒体基因组全长16 519 bp,含有13个蛋白质编码基因、2个rRNA基因、22个tRNA基因和1个控制区。凭祥睑虎线粒体基因组碱基组成分别为A(34.11%)、T(27.43%)、C(26.01%)、G(12.45%)。除tRNA-Ser(AGY)外,其余21个tRNA基因的二级结构均为典型的三叶草结构。13个蛋白质编码基因中,7个基因(ND1、ND4、ND5、COⅡ、COⅢ、ATP8、ATP6)的起始密码子为ATG,2个基因(ND2、ND3)为ATA,2个基因(ND4L、Cytb)为ACA,剩余2个分别为ATC(COI)和GTG(ND6)。终止密码子一般都是TAA、AGA或者TAA,ND2、ND3、ND4、ND6、ATP6、COⅢ 6个基因由不完全终止密码子终止(TA或T)。控制区全长1 147 bp,含有7个串联重复序列、6个终止相关序列TAS和2个保守序列(CBS-2,CBS-3)。
关键词:凭祥睑虎(Goniurosaurus luii);睑虎科;线粒体基因组;D-环
中图分类号:Q953+.3 文献标识码:A 文章编号:0439-8114(2017)05-0976-05
DOI:10.14088/j.cnki.issn0439-8114.2017.05.048
Sequencing and Analysis of the Complete Mitochondrial Genome of Goniurosaurus luii
LIU Jian-xiang1,2,QIN xin-min1
(1.College of Life Science,Guangxi Normal University,Guilin 541004,Guangxi,China;2.Guilin Medical University,Guilin 541004,Guangxi,China)
Abstract:The complete nucleotide sequence of the mitochondrial genome of Goniurosaurus luii was determined by using PCR amplification and DNA sequencing. The results showed that the entire mitochondrial genome of G. luii is 16 519 bp in length and contained 13 protein-coding genes,2 rRNA genes,22 tRNA genes and 1 control region(D-loop). The average nucleotide composition were 34.11% A,27.43% T,26.01% C and 12.45% G. The 21 tRNA genes can be fold in the typical cloverleaf structure except tRNA-Ser(AGY). Among 13 protein-coding genes of the G. luii mitochondrial genome,seven of the 13 protein coding genes(ND1,ND4,ND5,COⅡ,COⅢ,ATP8 and ATP6) used ATG as the start codon,two genes(ND2 and ND3) used ATA,Cytb and ND4L used ACA,COI and ND6 started with ATC and GTG as the start codon,respectively. The remaining six genes had incomplete stop codons(ND3,ND4 and ATP6 with TA-,ND2,COⅢ and ND6 with T-). In addition,the control area is 1 147 bp, there are 6 tandem repeat sequences,7 terminal associated sequence(TAS) and 2 conserved sequence blocks (CSB-2,CSB-3) in the control region of G. luii.
Key words:Goniurosaurus luii; eublepharidae; mitochondrial genome; D-loop
憑祥睑虎(Goniurosaurus luii)属有鳞目(Squamata)睑虎科(Eublepharidae)爬行动物,主要分布于广西壮族自治区。由于栖息地环境的破坏和人工大量捕猎,凭祥睑虎种群数量急剧减少,该物种已被列入中国国家林业局2000年8月1日发布的《国家保护的有益的或者有重要经济、科学研究价值的陆生野生动物名录》。目前,关于凭祥睑虎的研究较少,仅在睑虎属物种的研究中有所涉及[1-3]。动物线粒体mtDNA具有分子结构简单、进化速度快等特点,被广泛应用于物种起源、进化和亲缘关系的研究,成为研究近缘种间以及种内群体之间遗传分化的有力工具[4-7]。目前,凭祥睑虎线粒体基因组DNA全序列的研究鲜见报道,本研究测定了凭祥睑虎的线粒体基因组全序列,并对其结构进行了分析,以期为凭祥睑虎的系统发育和物种多样性保护提供分子依据。
1 材料与方法
1.1 材料
凭祥睑虎尾部肌肉。
1.2 DNA的提取
总DNA的提取采用改进的SDS/蛋白酶K的方法[8]。
1.3 引物的设计
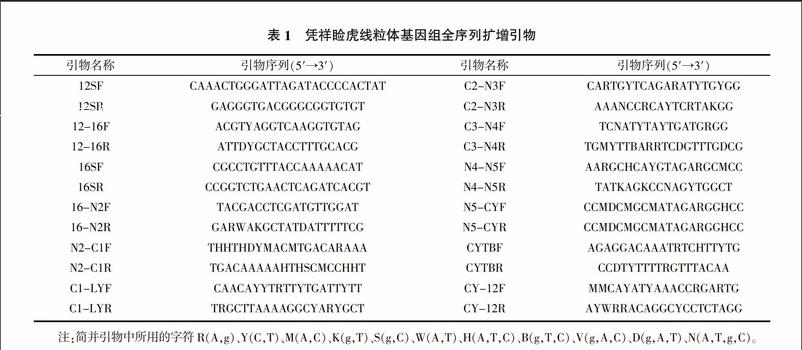
参考爬行动物通用引物扩增12S rRNA、16S rRNA、COI、ND2、ND5和Cytb基因的部分片段,根据上述片段的测序结果,利用Primer 5.0软件设计新引物,用来扩增短片段之间的长片段DNA,所用引物见表1。
1.4 PCR扩增和序列测定、拼接
PCR的反应体系和PCR反应参数参考文献[9]进行。PCR扩增产物由华大基因科技股份有限公司进行双向测序。获得的序列通过Chromas 2.22软件分析,运用Clustal X1.8[10]软件比对校正后,使用DNASTAR和BLASTN软件根据测得片段间的重叠区域拼接成完整的线粒体基因组全长。各蛋白质编码基因和rRNA基因通过与GenBank中近缘物种的线粒体基因进行同源比对定位。tRNA基因的查找、定位及二级结构预测使用在线版的tRNAscan-SE Search Server软件[11]。用MEGA 5.0软件[12]对蛋白质编码基因进行翻译,最后用Sequin 11.0软件对全序列进行注释,并提交至GenBank。
2 结果与分析
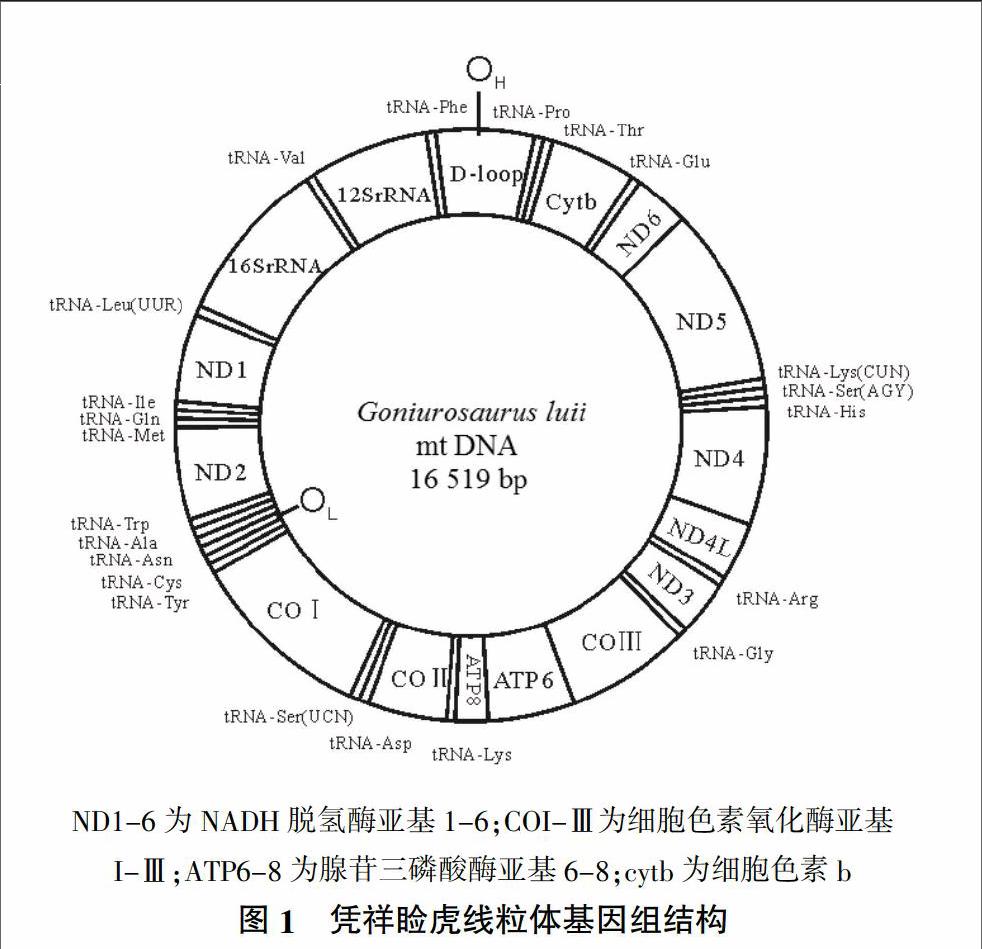
2.1 凭祥睑虎线粒体基因组的结构和碱基组成
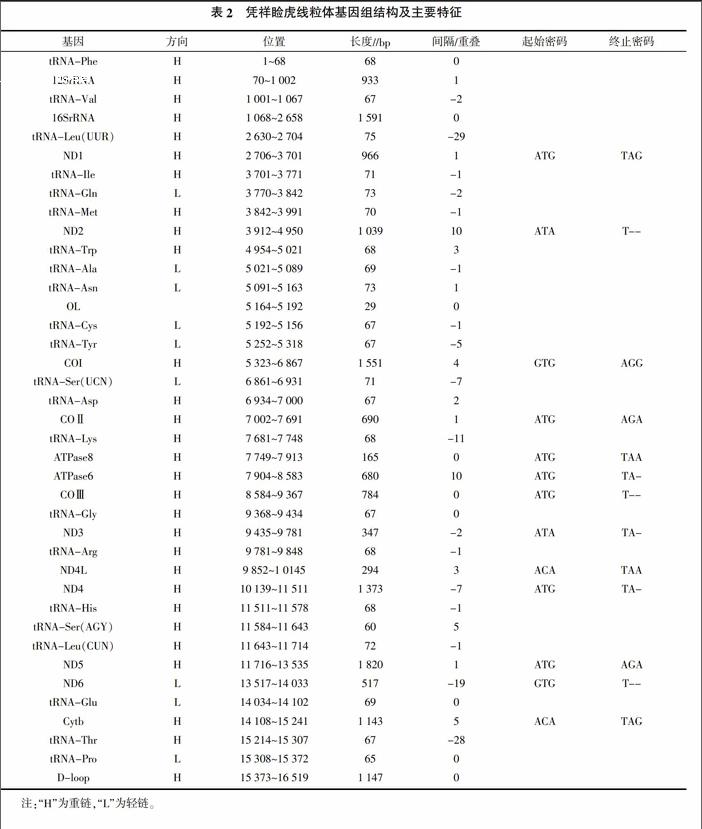
凭祥睑虎线粒体DNA是闭合的环状分子,全长为16 519 bp(GenBank登录号:KM455054),其中包括了13个蛋白质编码基因、22个tRNA基因、2个rRNA基因和1个控制区(D-loop)(图1)。除了tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser(UCN)、tRNA-Glu、tRNA-Pro和ND6 這几个基因在L-链上编码以外,其余基因均在H链编码。凭祥睑虎线粒体基因组碱基含量分别为A(34.11%)、T(27.43%)、C(26.01%)、G(12.45%),总的A+T含量为61.54%,与报道的其他爬行动物相符[13,14]。
2.2 蛋白质编码基因
凭祥睑虎13个蛋白编码基因总长度为11 384 bp,占整个基因组的69.0%。除ND6由L链编码,其余12个基因均由H链编码。最长的蛋白质编码基因是ND5,长度为1 820 bp,最短的是ATPase8基因,长度为165 bp。13蛋白质编码基因中,7个基因(NDI、ND4、ND5、COⅡ、COⅢ、ATP8、ATP6)的起始密码子为ATG,2个基因(ND2、ND3)为ATA,2个基因(ND4L、Cytb)为ACA,剩余2个分别为ATC(COI)和GTG(ND6)。终止密码子一般都是TAA、AGA 或者TAA,ND2、ND3、ND4、ND6、ATP6、COⅢ 6个基因由不完全终止密码子终止(T或TA),不完全密码子(TA或T)可以通过转录后聚腺苷酸化转变为标准的终止密码子(表2)。
2.3 rRNA和tRNA基因
与其他脊椎动物线粒体基因组一样,凭祥睑虎包括1个12S rRNA和1个16S rRNA,它们位于tRNA-Phe和tRNA-Leu(UUR)基因之间,12S rRNA与16S rRNA基因被tRNA-Val基因分隔开。凭祥睑虎线粒体基因组中12S rRNA和16S rRNA基因的长度分别为933、1 591 bp(表2)。12S rRNA基因的碱基组成为36.12% A、24.33% C、17.15% G、22.40% T,16S rRNA基因为36.46% A、23.51% C、15.96% G、24.07% T。
凭祥睑虎线粒体DNA中tRNA基因共有22个,长度在60~75 bp之间,其中14个tRNA由重链编码,8个tRNA由轻链编码。tRNA基因的二级结构见图2。
2.4 D-loop
凭祥睑虎序列控制区(D-loop)是位于tRNA-Phe和tRNA-Pro之间长1 147 bp的序列,它占整个基因的6.94%,其碱基组成为35.92% A、30.08 C、10.37% G、23.63% T。在控制区内存在6个91 bp串联重复单元(AATACCTTAAAGTAAGAGTCTA TTCCTAATCTATTACCTATATACAAATAAGGGGCC- TAAAGGCCCCTTTTTACCCACTACTTATTACAAT),7个DNA复制终止相关序列TAS(ATTCCTAATCTATT),1个CSB-2(CAAACCCCCCTTACCCCC)和1个CSB-3(TCGCCAAACCCCTAAAACGA)。
3 讨论
睑虎属(Goniurosaurus)动物种类较少,目前全世界仅报道了14个种,其中中国已命名了6个种[3]。一些学者分别采用线粒体12S rRNA、16SrRNA、ND2等基因构建了NJ、MP、BI等系统发育树探讨该属物种的系统发育和亲缘关系,但结果并不一致[2,15-17],其原因之一是所采用的单个基因片段包含的遗传信息数量不多。相比之下,整个线粒体基因组序列所含遗传信息量更为丰富,因此以线粒体基因组全序列进行物种的系统发育分析,其结果更为可信。近年来,一些学者利用线粒体基因组全序列信息在解决鳄类、龟类和鸟类的分类地位和系统发生关系等方面取得了重要进展[7,18-21]。
目前,睑虎属物种线粒体基因组全序列鲜见报道。本研究测定了凭祥睑虎线粒体基因组序列,mtDNA全长为16 519 bp,含有13个蛋白质编码基因、2个rRNA基因、22个tRNA基因和1个控制区。碱基含量为A(34.11%)、T(27.43%)、C(26.01%)、G(12.45%)。除tRNA-Ser(AGY)外,其余21个tRNA基因的二级结构均为典型的三叶草结构。13蛋白质编码基因中,7个基因(ND1、ND4、ND5、COⅡ、COⅢ、ATP8、ATP6)的起始密码子为ATG,2个基因(ND2、ND3)为ATA,2个基因(ND4L、Cytb)为ACA,剩余2个分别为ATC(COI)和GTG(ND6)。终止密码子一般都是TAA、AGA或者TAA,ND2、ND3、ND4、ND6、ATP6、COⅢ 6个基因由不完全终止密码子终止(T或TA)。控制区全长1 147 bp,含有7个串联重复序列、6个终止相关序列TAS和2个保守序列(CBS-2、CBS-3)。结果可为凭祥睑虎的分类、进化和系统发育等方面提供分子依据。
参考文献:
[1] GRISMER L L,VIETS B E,BOYLE L J. Two new continental species of Goniurosaurus(Squamata;Eublepharidae)with a phylogeny and evolutionary classification of the genus[J].J Herpetol,1999,33:382-393.
[2] KRATOCHVFL L S, FRYNTA D. Body size, male combat and the evolution of sexual dimorphism in eublepharid geckos (Squamata: Eublepharidae)[J]. Biol J Linn Soc,2002,2:303-314.
[3] 张 洁,庆 宁,易祖盛,等.Goniurosaurus indet.的有效性及中国睑虎属(Squamata:Sauria:Eublepharidae)种间的亲缘关系[J].华南师范大学学报,2014,46(2):92-98.
[4] LOPEZ J V,CEVARIO S O,BRIEN S J.Complete nucleotide sequences of the domesticcat(Felis catus) mitochondrial genome and a transposed mtDNA tandem repeat (Numt) in the nuclear genome[J].Genomics,1996,33:229-246.
[5] IVANOV P L,WADHAMS M J,ROBY R K,et a1.Mitochondrial DNA sequence heteroplasmy in the grand duke of Russia georgij romanov establishes the remains of Tsar Nicholas Ⅱ[J].Nature Genetics,1996,12:417-420.
[6] DELPORT W,FERGUSON J W,BLOOMER P.Characterization and evolution of the mitochondrlal DNA control region in Hornbills (Bucerotifoormes)[J].Mol Evol,2002,54:794-806.
[7] SHEN Y Y,LIANG L,SUN Y B,et al.A mitogenomic perspective on the ancient,rapid radiation in the galliformes with an emphasis on the Phasianidae[J].BMC Evolutionary Biology,2010, 10:132-141.
[8] 汪永慶,王新国,徐来详,等.一种动物基因组DNA提取方法的改进[J].动物学杂志,2001,36(1):27-29.
[9] 覃屏生,陶春荣,佘 莹,等.鳄蜥线粒体基因组全序列及其结构分析[J].湖北农业科学,2014,53(4):925-928.
[10] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The Clustal X windows interface flexible strategies for multiple sequence aligrunent aided by quality analysis tools[J].Nucleic Acid Res,1997,25(24):4876-4882.
[11] SCHATTNER P,BROOKS A N,LOWE T M. The tRNAscan-SE,snoscan and snoGPS web servers for the detection of tRNAs and snoRNAs[J]. Nucleic Acids Res,2005,33;686-689.
[12] TAMURA K,PETERSON D,PETERSON N,et al. MEGA 5: Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28:2731-2739.
[13] BOHME M U,FRITZSCH G,TIPPMANN A,et al. The complete mitochondrial genome of the green lizard Lacerta viridis viridis(Reptilia:Lacertidae) and its phylogenetic position within squamate reptiles[J]. Gene,2007,394(1-2):69-77.
[14] Li H M,ZENG D L,GUAN Q X,et al. Complete mitochondrial genome of Gekko swinhonis(Squamata,Gekkonidae)[J].Mitochondr DNA,2013,24:86-89.
[15] OTA H,HONDA M,KOBAYASHI M,et al. Phylogenetic relationships of eublepharid geckos(Reptilia:Squamata):A molecular approach[J].Zoological Science,1996,16:659-666.
[16] JONNIAUX P,KUMAZAWA Y. Molecular phylogenetic and dating analyses using mitochondrial NDA sequences of eyelid geckos(Squamata:Eublepharidae)[J].Gene,2008,407:105-115.
[17] WANG Y Y,YANG J H,GRISMER L L. A new species of Goniurosaurs(Squamata:Eublepharidae) from Libo,Guizhou Province,China[J].Herpetologica,2013,69:214-226.
[18] JANKE A,ERPENBCEK D,NILSSON M,et al. The mitochondrial genomes of the iguana (Iguana iguana) and the caiman (Caiman crocodylus):Implications for amniote phylogey[J].Proc R Soc Lond B,2001,268:623-631.
[19] PARHAM J F,MACEY J R,PAPENFUSS T J,et al. The phylogeny of mediterranean tortoises and their close relatives based on complete mitochondrial genome sequences from museum specimens[J].Mol Phylogenet Evol,2006,38:56-64.
[20] LI H M,SHI J P,ZENG D L,et al. The complete mitochondrial genome of Chrysolophus pictu(Galliformes:Phasianidae) and phylogenetic analysis with related species[J].Mitochondrial DNA,2011,22:159-161.
[21] 張 颖,聂刘旺,宋娇莲.缅甸陆龟线粒体全基因组的测序及分析[J].动物学报,2007,53(1):151-158.

