DNA条形码技术在仔稚鱼鉴定中的实践
刘守海,秦玉涛,刘材材,季晓,张昊飞
(1.国家海洋局东海环境监测中心 上海 201206;2.海洋赤潮灾害立体监测技术与应用国家海洋局重点实验室 上海 201206)
DNA条形码技术在仔稚鱼鉴定中的实践
刘守海1,2,秦玉涛1,2,刘材材1,2,季晓1,2,张昊飞1,2
(1.国家海洋局东海环境监测中心 上海 201206;2.海洋赤潮灾害立体监测技术与应用国家海洋局重点实验室 上海 201206)
对物种准确鉴定是进行生物多样性研究的前提。为提高物种鉴定的准确性,文章采用DNA条形码技术结合形态学对长江口仔稚鱼样品进行种类鉴定。结果表明,44个样品中,通过线粒体COI基因的分析鉴定有17个种、1个属;部分形态上难以鉴定的种类,如虾虎鱼科的矛尾虾虎鱼、斑尾刺虾虎鱼、睛尾蝌蚪虾虎鱼、褐吻虾虎鱼和斑点竿虾虎鱼,可通过DNA条形码实现有效鉴定;形态较为相似的种类,通过DNA条形码可鉴定到种,淡水鲤科的有青鱼、鲢、鳊,鲻科的有龟鮻和前鳞龟鮻。利用DNA条形码技术结合形态进行仔稚鱼种类的鉴定,能提高仔稚鱼鉴定的精确度。
DNA条形码;生物多样性;COI基因;海洋生物;物种鉴定
准确地鉴别仔鱼种类是进行鱼类浮游生物多样性研究的前提,也是进行海洋生态系统科学研究的基础。根据仔鱼形态特征差异可以对其进行种类鉴别[1],但鱼类早期发育阶段时间较短,其形态变化复杂,形态特征差异不明显或比较相似,有资料可查仔、稚鱼种类不超过300种[2]。因此,仅凭借鱼类早期发育阶段的外部形态特征很难对仔鱼进行精确分类。
随着DNA条形码技术[3]的提出和快速发展,尤其是DNA条形码数据库的成功构建[4],为这一技术在仔鱼种类鉴定中应用奠定基础[5-7]。迄今国内对DNA条形码技术应用于仔鱼鉴定的报道较少[8-9]。本文以2016年5月长江口及其邻近海域的仔稚鱼样品为材料,采用形态学与DNA条形码技术结合的形式进行种类鉴定,讨论种类鉴定的精确度,同时探索DNA条形码技术在海洋生物多样性监测中的应用可行性。
1 实验材料与方法
1.1 样品采集
于2016年5月9—29日在长江口及其邻近海域设置采样站点(图1)。
图1 采样站位
按照《海洋调查规范》(GB/T12763.6—2007)使用大型浮游生物网(网长 280 cm、网口内径80 cm、网口面积0.5 m2、网目0.505 mm)在站点进行水平拖网。采集到的标本现场进行初步处理,用无水乙醇保存,回实验室后对样品进行形态鉴定、拍照和分子鉴定。
1.2 实验方法
取背部肌肉约100 mg,使用海洋动物组织基因组DNA提取试剂盒(北京天根生化科技有限公司)提取总DNA。使用通用引物 F1:TCR ACY AAY CAY AAA GAY ATY GGC AC和R1:TAG ACT TCW GGG TGR CCR AAG AAT CA扩增仔鱼的线粒体DNA的COI部分序列,PCR总反应体系为50 μL,其中包括:10×PCR buffer 5 μL、dNTP 4 μL(2.5 mmol/L)、上下游引物各2 μL(10 mmol/L)、Taq DNA聚合酶0.8 μL(5 U/μL)、模板DNA 1μL,加双蒸水至总体积50 μL。样品在AG-22331型PCR仪(Eppendorf)上进行扩增,94℃预变性5 min;94℃ 30 s、52℃ 45 s、72℃ 1 min,35个循环;72℃延伸10 min;4℃保存。扩增产物使用1.0%的琼脂糖凝胶电泳检测后送至上海杰李生物技术有限公司胶回收双向测序后拼接。
1.3 数据处理
利用DNAStar软件包对测序和GenBank下载序列进行校对,用Clustal X对所有序列进行比对排序并去除两端冗余列。利用MEGA6.0分析序列的核苷酸组成等,基于K2P双参数模型计算种间及种内的遗传距离。
2 实验结果
2.1 测序结果
本研究共获得43条COI基因序列。获得的COI基因序列长度为680 bp。
2.2 遗传距离
对COI基因的K2P遗传距离分析结果表明,种内的遗传距离在0~0.011,平均值为0.004。COI基因序列的属内种间遗传距离在0.075~0.080,平均值为0.077。
2.3 种类鉴定
在44个样品中,分子实验将样品鉴定到17个种、1个属;而形态鉴定结果鉴定到种的仅有6个,鉴定到属的有2个,鉴定到科的有2个,还有2个仔鱼未能进行种类鉴定。相对形态鉴定结果,分子实验结果的精确度更高。
分子技术对虾虎鱼科、鲤科、鯔科等类群的鉴定结果更为精确。分子鉴定结果将所获得的虾虎鱼科样品全部鉴定到种:矛尾虾虎鱼(Chaeturichthys stigmatias)、斑尾刺虾虎鱼(Acanthogobius ommaturus)、睛尾蝌蚪虾虎鱼(Lophiogobius ocellicauda)、褐吻虾虎鱼(Rhinogobius brunneus)、斑点竿虾虎鱼(Luciogobius guttatus)(图2)。形态相似不易鉴定的淡水鲤科仔鱼3种:青鱼(Mylopharyngodon piceus)、鲢(Hypophthalmichthys molitrix)、鳊(Parabramis peknensis)。鲻科中鉴定出2种:龟鮻(Chelon haematocheilus)和前鳞龟鮻(Chelon affinis)。
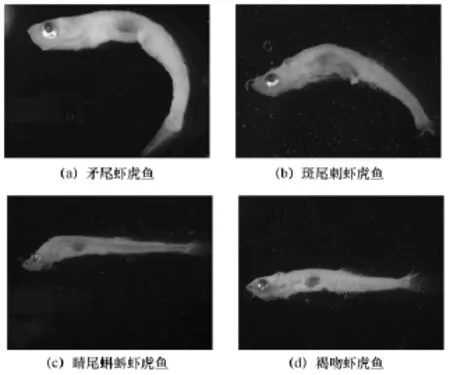
图2 形态无法鉴定的虾虎鱼科仔鱼种类
3 讨论
3.1 长江口仔稚鱼的种类组成
关于长江口仔稚鱼的报道已有不少[10-14]。由于长江径流带来大量营养物质,长江口水域成为多种经济鱼类的产卵、索饵和育幼的重要场所,仔稚鱼的种类多、类群复杂(包括淡水种类、咸淡水种类、近海种类),具有明显的河口特征。
从20世纪80年代开始,每10年对长江口及其邻近水域的鱼卵和仔稚鱼进行调查,发现种类数有下降趋势(表1)。本研究中鱼卵和仔稚鱼的调查结果为10科17种,较2007年调查结果科的数量有所减少、种类数相仿;使用条形码技术更有助于种类鉴定,将之前的虾虎鱼科及鲤科仔稚鱼鉴定到种,更获得鲻科龟鮻属的龟鮻和前鳞龟鮻。由此可见,条形码技术能完善种类组成资料。

表1 不同时期春季长江口及其邻近水域仔稚鱼调查结果对比
3.2 DNA条形码技术鉴定仔稚鱼的精确度
由于仔鱼阶段的形态特征不甚明显,形态学鉴定结果往往会使部分仔鱼样品鉴定到科或属。因此,在形态特征不明显或分类依据有疑问的时候,严谨的做法是只鉴定到科或属。加之不同鉴定者水平不一,也会对鉴定结果有一定影响。基于COI的条形码辅助仔鱼鉴定能够克服形态特征和人为干扰,将形态鉴定特征不明显的样品准确鉴定到种,可提高仔鱼鉴定的精确度。条形码辅助仔鱼鉴定可以区分形态学较难鉴定的科类或相似种类。
温带河口的仔稚鱼一般以河口定居物种(如虾虎鱼科)或季节性进入河口产卵者占优势[15],长江径流也会带来一些淡水种类的仔稚鱼。长江口水域环境复杂多变,分布的虾虎鱼科常见种类有10种左右;淡水鱼类主要以鲤形目、鲈形目和鲶形目为主,其中又以鲤形目的鲤科鱼类最多,常见种类有20多种[16]。本研究通过形态学结合DNA条形码技术将调查所获得的虾虎鱼科和淡水鲤科等仔鱼样品全部鉴定到种,共获得虾虎鱼科的5个种即矛尾虾虎鱼、斑尾刺虾虎鱼、睛尾蝌蚪虾虎鱼、褐吻虾虎鱼、斑点竿虾虎鱼,淡水种类有鲤科的青鱼、鲢和鳊。在以往虾虎鱼科和鲤科仔鱼的研究中存在部分样品鉴定到科级[11-14,17],鉴定到种的则较少[11,18-19],说明基于COI序列的DNA条形码可对形态学较难鉴定的科类或相似种类进行有效的物种鉴定。
3.3 DNA条形码技术在仔稚鱼多样性监测中的应用展望
DNA条形码技术辅助仔稚鱼鉴定,其准确性取决于比对数据库的准确与完备程度。很多国际组织已建立自己的DNA条形码数据库,如BOLD、ABBI、AllLeps、TBI、MBI等。其中BOLD数据库收录的鱼类DNA条形码信息基本来自于COI基因;截至2016年4月,BOLD数据库收录近18.69万条辐鳍鱼纲鱼类的DNA条形码序列信息,隶属于46目1.75万种。BOLD等数据库条形码信息的不断丰富,为DNA条形码技术在鱼卵和仔、稚鱼鉴定领域的应用提供可比对的鱼类条形码数据。
传统的形态学鉴定仍然是目前海洋生物多样性监测的主要手段,但海洋环境监测中鱼卵和仔、稚鱼样品形态鉴定存在样品形态特征不明显、样品鉴定水平参差不齐、形态鉴定工作量大且耗时的问题,寻找一个结果更精确、过程可标准化的方法对样品进行快速准确的鉴定是迫切工作。利用形态结合DNA条形码技术既可以有效提高仔稚鱼种类鉴定的精确度,也有利于对海洋生物多样性进行客观科学的评价。
[2]卞晓东,张秀梅,高天翔,等.沙氏下鱵鱼卵的形态学及遗传学鉴别[J].水产学报,2008,32(3):342-352.
[3]ARNOT D E,ROPER C,BAYOUMI R A L.Digital codes from hypervariable tandemly repeated DNA-sequences in the Plasmodium-Falciparum Circumsporozoite gene can genetically barcode isolates[J].Molecular and Biochemical Parasitology,1993,61(1):15-24.
[4]HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identification through DNA barcodes[J].Proceedings of the Royal Society B:Biological Sciences,2003,270:313-322.
[5]KO H L,WANG Y Z,CHIU T S,et al.Evaluating the accuracy of morphological identification of larval fishes by applying DNA barcoding[J].PLOS ONE,2013,8(1):1-7.
[6]HUBERT N,DELRIEU-TROTTIN E,IRISSON J O,et al.Identifying coral reef fish larvae through DNA barcoding:A test case with the families Acanthuridae and Holocentridae[J].Molecular Phylogenetics and Evolution,2010,55:1195-1203.
[7]HUBERT N,ESPIAU B,MEYER C,et al.Identifying the ichthyoplankton of a coral reef using DNA barcodes[J].Molecular Ecology Resources,2015,15:57-67.
[8]何文平,程飞,黎雨轩,等.刀鲚和凤鲚的分子鉴定及其在仔鱼种类鉴定中的应用[J].水生生物学报,2011,35(4):565-571.
[9]周美玉,陈晓,杨圣云.采用DNA条形码技术对厦门海域鱼卵、仔稚鱼种类的鉴定[J].海洋环境科学,2015,34(1):120-135.
[10]杨东莱,吴光宗,孙继仁.长江口及其邻近海区的浮性鱼卵和仔稚鱼的生态研究[J].海洋与湖沼,1990,21(4):346-355.
[11]朱鑫华,刘栋,沙学绅.长江口春季鱼类浮游生物群落结构与环境因子的关系[J].海洋科学集刊,2002,44:169-178.
[12]蒋玫,沈新强,王云龙,等.长江口及其邻近水域鱼卵、仔鱼的种类组成与分布特征[J].海洋学报,2006,28(2):171-174.
[13]王金辉,孙亚伟,刘材材,等.长江口鱼卵仔鱼资源现状的调查与分析[J].海洋学研究,2007,25(4):40-50.
[14]刘淑德,线薇微.长江口及其邻近水域鱼类浮游生物群落的时空格局[J].生物多样性,2009,17(2):151-159.
[15]STRYDOM N A.Occurrence of larval and early juvenile fishes in the surf zone adjacent to two intermittently open estuaries:South Africa[J].Environmental Biology of Fishes,2003,66:349-359.
[16]庄平,王幼槐,李圣法,等.长江口鱼类[M].上海:上海科学技术出版社,2007:201-222.
[17]闫欣,钟俊生,王明星,等.杭州湾北部水域仔稚鱼种类组成和多样性研究[J].上海海洋大学学报,2014,23(5):765-773.
[18]蒋日进,钟俊生,张冬良,等.长江口沿岸碎波带仔稚鱼的种类组成及其多样性特征[J].动物学研究,2008,29(3): 297-304.
[19]陈渊戈,张宇,钟俊生,等.长江口南支和杭州湾北岸碎波带水域仔稚鱼群聚的比较[J].上海海洋大学学报,2011,20 (5):688-696.
Molecular Identification of Fish Larvae and Juveniles Based on DNA Barcoding
LIU Shouhai1,2,QIN Yutao1,2,LIU Caicai1,2,JI Xiao1,2,ZHANG Haofei1,2
(1.East China Sea Environmental Monitoring Center,SOA,Shanghai 201206,China;2.Key Lab of Integrated Monitoring and Applied Technology for Marine Harmful Algal Blooms,SOA,Shanghai 201206,China)
Accurate identification of species is a prerequisite for biodiversity research.To improve the identification accuracy of fish larvae and juveniles in the Yangtze Estuary,the method of morphology and DNA barcoding were used based on mitochondrial the COI gene.The results showed that among the 44 samples analyzed,17 of which could be identified to the species level using DNA barcoding,while only one to the genus level.Some species without available taxonomic keys for morphological identification,including Chaeturichthys stigmatias,Acanthogobius ommaturus,Lophiogobius ocellicauda,Rhinogobius brunneus,Luciogobius guttatus in Gobiidae,were difficult to identify by morphology but were successfully identified by using DNA barcoding.Some species with similar morphology were hard to be distinguished,such as Mylopharyngodon piceus,Hypophthalmichthys molitrix,Parabramis peknensis in Freshwater Cyprinidae,Chelon haematocheilus,Chelon affinis in Mugilidae.DNA barcoding based on the COI gene could provideeffective identification to the species level.Collectively,DNA barcoding,together with morphological identification,could largely increase the identification accuracy for fish larvae and juveniles.
DNA barcoding,Biodiversity,COI gene,Marine organism,Species identification
P7
A
1005-9857(2017)02-0092-04
2016-10-20;
2016-12-09
海洋公益性行业科研专项(201305027,201505004-6);海洋赤潮灾害立体监测技术与应用国家海洋局重点实验室基金资助课题(MATHAB201402,MATHAB201408);东海分局青年海洋科技基金项目(201501,201608).
刘守海,工程师,硕士,研究方向为海洋鱼类浮游生物学,电子信箱:liush@eastsea.gov.cn
张昊飞,高级工程师,博士,研究方向为海洋环境科学,电子信箱:zhanghf@eastsea.gov.cn

