植物隐花色素及其分子调控机制
王红霞+郝建梅+丁谦+张一卉+李景娟+高建伟


摘要:蓝光受体隐花色素(cryptochrome)对植物生长发育有重要的调节作用,其调节反应主要有去黄化、抑制下胚轴伸长、刺激子叶伸展、促使叶绿体发育、调节光周期、调节生物钟、促进保卫细胞发育、调节气孔开度和调控基因表达等。本文概述了近年来植物蓝光受体隐花色素及其分子调控机制方面的研究进展。
关键词:植物;隐花色素;分子调控;研究进展
中图分类号:S184文献标识号:A文章编号:1001-4942(2017)03-0148-06
AbstractCryptochrome is the blue-light receptor which mediates light regulation in the growth and development of plants, including de-etiolation, inhibition of hypocotyl elongation, stimulation of cotyledon extension, promotion of chloroplast development, photoperiodic control, regulation of circadian clock, stimulation of guard cell development, regulation of stomata opening and gene expression. Here, we summarized the research progresses of plant cryptochromes and their molecular regulation mechanism.
Key wordsPlants; Cryptochrome; Molecular regulation; Research progress
光是影响植物生长发育的一个重要环境因素,它不仅是光合作用的能量来源,同时也是生长发育的信号来源。植物通过光受体介导的光信号转导途径来调控自身的生长发育。到目前为止,植物中发现的光受体有两类:一类是红光/远红光受体光敏色素(phytochrome,PHY),一类是蓝光/近紫外光受体隐花色素 (crytochrome,CRY) 和向光素(phototropin,PHOT)。其中蓝光受体CRY是重要的光受体,参与调控光形态建成、开花时间、生物节律性、气孔开放和气孔发育等重要生理过程[1]。本文将重点综述近年来在隐花色素功能及其与其它光信号转导因子相互作用的分子调控机制方面的研究进展。
1隐花色素的结构
隐花色素包含2个重要结构域(图1):N端PHR(photolyase-homologous region)结构域,包含500个残基;C端CCE(cryptochrome C-terminal extension)結构域,具有不同的长度[2-5]。隐花色素与DNA光裂合酶(photolyase)在氨基酸序列、发色团成分以及各自光依赖型的生物化学活性方面都有相似性,但是隐花色素没有DNA 修复活性[6-9]。
PHR结构域是隐花色素绑定发色团的结构域,它具有与DNA光裂合酶相类似的序列[10]。拟南芥CRY1、CRY2蛋白PHR结构域与FAD(flavin adenine dinucleotide,黄素腺嘌呤二核苷酸)以非共价闭合的方式进行绑定[11-13]。许多与FAD绑定相关的残基无论是在拟南芥还是人类的隐花色素中都很保守。相反,隐花色素中与叶酸或DNA绑定相关的光裂合酶相似结构域较不保守[6,14,15]。拟南芥CRY1蛋白PHR结构域具有与之功能相适应的特点[3,6]:(1)含3个色氨酸残基,它们参与光诱导相关的从蛋白表面到FAD的电子传递活动;(2)连接环Cys-190与α/β结构域的Cys-80形成一个二硫键,参与光诱导的CRY蛋白构象变化;(3)CRY1-PHR结构域与ATP相连接,对拟南芥CRY1蛋白磷酸化有重要作用;(4)CRY1-PHR表面主要含有阴离子,不与DNA结合,因此CRY1蛋白不具有DNA修复的功能。CRY1-PHR表面静电位与蛋白磷酸化、带阴电荷的CCE结构域形成静电排斥,从而引起光诱导下的构象变化。
植物隐花色素CCE结构域具有特异的序列。除衣藻(Chlamydomonas reinhardtii)外,地钱、苔藓、蕨类直至被子植物的隐花色素CCE结构域都含有DAS(DQXVP-Acidic-STAESSS)[10]。
2隐花色素的生物学功能
目前,人们主要利用拟南芥、水稻等模式植物,通过观察其基因缺失突变体、基因过表达植株与野生型之间的差异来研究隐花色素的生物学功能。本文根据近年来发表的相关研究结果,将植物隐花色素的生物学功能进行归纳整理,结果见表1[1, 16-29]。
3隐花色素与其它光信号转导蛋白的互作及其调控机制隐花色素在调控植物生长发育的过程中,与蛋白激酶、泛素连接酶等多种蛋白因子相互作用,共同完成蓝光信号转导的生物学功能。
3.1光敏色素
光敏色素A(phyA)是第一个被发现的与隐花色素相互作用的蛋白[1]。在红光和远红光下,phyA在体内促进隐花色素磷酸化,而CRY蛋白调控幼苗发育的主要生理活动仅在蓝光下发生,所以受phyA影响的CRY1蛋白磷酸化反应不是CRY1调控幼苗发育的主要信号转导机制。CRY2蛋白能与phyB相互作用,这种相互作用影响CRY2蛋白的信号转导,调控植物的开花时间。phyB还能通过影响CRY1-COP1的互作来调节相关基因的表达,从而抑制植物下胚轴生长[30,31]。
3.2ZTL蛋白
拟南芥含有两种LOV结构域类型的蓝光受体[1]:(1)向光素(photo1和photo2),控制各种向光反应;(2)F-box/Kelch重复蛋白、ZTL(ZEITLUPE)等蛋白,调节生物钟组分蛋白的降解。ZTL蛋白能与CRY1和phyB在酵母杂交、体外pull-down试验中相互作用,调节生物钟和开花时间。
3.3COP1蛋白
COP1蛋白是一种E3连接酶,在植物光形态建成反应和光受体信号转导过程中起调控作用。
(1)COP1蛋白与CRY蛋白相互作用,参与蓝光信号转导。将隐花色素CCE结构域或CCE结构域的一部分与GUS(β-glucuronidase)报告基因相融合,导入野生型拟南芥中过量表达,转基因植株与cop1功能缺失突变体相比,表型变化相似,高于80%的基因表达变化也相似[1]。OsCRY1是水稻的蓝光受体,有两个拷贝(OsCRY1a和OsCRY1b)。OsCRY1b的C端可以与OsCOP1互作,同时两者在细胞中定位在同一位置,说明OsCRY1与OsCOP1直接互作从而介导蓝光的信号传导(http://www.ricedata.cn/gene/list/951.htm)。
(2)CRY蛋白与COP1蛋白以直接或间接的方式相互作用来抑制COP1蛋白的E3连接酶活性,调节其它受光调节蛋白的降解,从而影响植物的生长。COP1蛋白能调控多种蛋白的降解,包括HY5/HYH、LAF1、HFR1、CRY2、phyA、CO和GI等蛋白[1]。
(3)SPA1蛋白参与隐花色素对COP1蛋白的调控。SPA1是COP1的正调控因子,隐花色素与SPA1蛋白相互作用,抑制了蓝光下依赖于COP1的转录蛋白的降解。Lian等[31]发现拟南芥隐花色素CRY1、CRY2在蓝光下均能直接与SPA1相互作用,黑暗下不能与SPA1相互作用。CRY1和CRY2与SPA1在拟南芥原生质体和洋葱表皮细胞核内均发生共定位。免疫共沉淀(Co-IP)试验证明,拟南芥CRY1与SPA1只能在蓝光下产生相互作用,而在黑暗处理、红光和远红光下都不能产生相互作用。通过对cry1/spa1/spa2/spa3/spa4五突变体、cry2/spa1/spa2/spa3和cry2/spa1/spa3/spa4四突变体的遗传分析,发现SPA1、SPA2、SPA3和SPA4在遗传上位于CRY1和CRY2的下游调控光形态建成、气孔发育和光周期开花时间。蓝光诱导的CRY1-SPA1互作能抑制COP1与SPA1的结合,促进COP1-SPA1复合体解离,导致COP1活性下降,其底物HY5蛋白积累增多,最终促进光形态建成。Zuo等[32]证实拟南芥CRY2与COP1互作蛋白SPA1发生了依赖于蓝光的直接相互作用。SPA1在蓝光照射条件下可抑制开花时间调控因子CONSTANS(CO)的COP1蛋白质水解,蓝光依赖的CRY2-SPA1互作促进CRY2-COP1互作。
3.4CIB1蛋白
CIB1蛋白(CRY-interacting bHLH1)是一种bHLH蛋白,能促进植物开花,其功能依赖于隐花色素。CRY2蛋白在蓝光下抑制CO蛋白的降解,从而调节植物开花,这种调控作用受CRY2对COP1调节的影响。近年来的研究结果表明,CRY2蛋白与CIB1蛋白相互作用直接调节FT(成花素)的表达;也可以与COP1、CO等蛋白形成復杂的蛋白复合体,共同调控植物开花。CRY2-COP1-CO-FT和CRY2-CIB1-FT的信号转导途径相交汇[33]。
3.5PIF蛋白
转录因子PIF(phytochrome interacting factor)是一种bHLH蛋白。以往研究表明,黑暗中PIF蛋白活性高,能促进细胞伸长相关基因的表达,引起下胚轴伸长;在红光/远红光下,光敏色素与PIF结合,促进PIF蛋白的降解,进而解除了PIF促进细胞伸长的作用。2016年初,美国索克研究所(Salk Institute for Biological Studies)的Joanne Chory实验室、中国科学院上海生命科学研究院的刘宏涛实验室分别在《细胞》、《美国科学院院报》上公布了各自新的研究结果:蓝光也通过PIF来调控植物下胚轴伸长[34-36]。
Casal等[34]发现,低光照度蓝光照射下,植物蓝光受体隐花色素(CRY1/CRY2)能与转录因子PIF直接结合,调控PIF的蛋白活性。借助多种突变体,包括pif4、pif5、pif7、pif1/pif3/pif4/pif5、cry1、cry1/pif4和cry1/pif4/pif5突变体,研究了PIF在低光照度蓝光下的生物学功能,结果表明:在低光照度蓝光照射下,PIF4、PIF5作用于隐花色素下游基因,介导拟南芥下胚轴伸长生长。通过免疫印迹(immunolotting)、免疫共沉淀(co-immunoprecipitation)试验证明了PIF4和PIF5能直接与CRY1、CRY2相互作用。转录组分析结果表明,pif4/pif5突变体差异表达的基因主要与细胞壁修饰、细胞壁发生、细胞生长密切关联。PIF4/PIF5与CRY2处于同一个基因组位点,CRY2能与PIF4/PIF5的转录因子结合来调控低光照度蓝光下拟南芥下胚轴伸长生长。以上结果说明,低光照度蓝光下,隐花色素能直接与PIF相互作用来调控植物的生长发育。
Ma等[35]发现,蓝光抑制温度升高引起的下胚轴伸长,这一过程依赖于CRY1。CRY1对温度响应至关重要,cry1缺失突变体对温度升高敏感,下胚轴伸长显著;CRY1过表达,转基因植株对温度变化不敏感。在蓝光照射下,CRY1与PIF4结合能调控PIF4在高温条件下的转录活性,增强生长素合成基因YUC8的表达,促进生长素合成及下胚轴伸长。CRY1和PIF4能直接结合到YUC8等基因的启动子上,快速调控基因表达。由于PIF4也是光敏色素phyB的受体,所以可以得出结论:PIF4是蓝光、红光/远红光和温度信号转导途径交互作用的关键节点,蓝光、红光/远红光和温度可以使用同一个PIF4实现功能整合,协同调控植物生长发育。
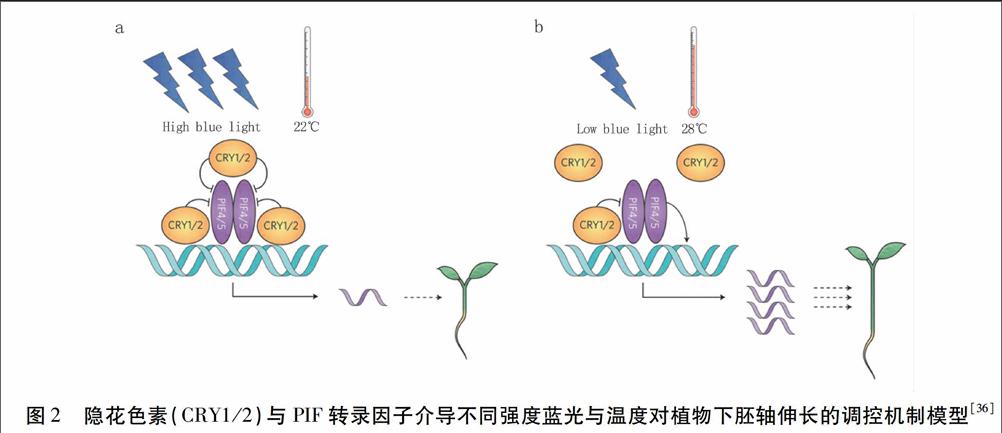
综合上述两实验室的研究结果,得出以下结论:低光照度蓝光下,隐花色素与PIF之间的互作主要调控细胞壁合成相关基因的表达;而在高温下,隐花色素与PIF之间的互作主要调控生长素合成相关基因的表达。因此,隐花色素与PIF之间的互作,在不同环境条件下最后的信号输出方式是有差异的。为此,Zhu等[36]提出了隐花色素与PIF互作模型:高强度蓝光照射或正常温度条件下,隐花色素与PIF之间的蛋白相互作用强,隐花色素抑制PIF的转录因子活性从而抑制下游基因表达,使植物表现为缩短的下胚轴;而在低蓝光照度或高温条件下,隐花色素与PIF之间的蛋白相互作用变弱,从而提高了PIF活性激活下游基因表达,促进下胚轴伸长(见图2)。
4問题与前景
自1993年首次发现隐花色素以来,人们在植物隐花色素结构、生物学功能、光激发及信号转导调控机制研究方面取得长足进展[17,37-38]。但是,仍有很多问题亟待人们去解答,如CRY3的生理功能是什么?如何利用隐花色素晶体研究其光诱导下的构象变化机制?依赖蓝光的隐花色素蛋白磷酸化/脱磷酸化、泛酸化作用机制是什么?伴随着对上述问题研究的不断深入,人们将会从根本上揭示隐花色素对光信号转导的分子调控机制。
参考文献:
[1]Yu X, Liu H, Klejnot J, et al. The cryptochrome blue light receptors [J].The Arabidopsis Book, 2010, 8:e0135.
[2]Lin C. Blue light receptors and signal transduction [J]. The Plant Cell, 2002,14: 207-225.
[3]Yu X, Shalitin D, Liu X, et al. Derepression of the NC80 motif is critical for the photoactivation of Arabidopsis CRY2 [J]. Proc. Natl. Acad. Sci. USA, 2007, 104(17): 7289-7294.
[4]Yang H Q, Wu Y J, Tang R H, et al. The C termini of Arabidopsis cryptochromes mediate a constitutive light response [J]. Cell, 2000, 103(5): 815-827.
[5]Rosenfeldt G, Viana R M, Mootz H D, et al. Chemically induced and light-independent cryptochrome photoreceptor activation [J]. Mol. Plant, 2008, 1(1):4-14.
[6]Brautigam C A, Smith B S, Ma Z, et al. Structure of the photolyase-like domain of cryptochrome 1 from Arabidopsis thaliana [J]. Proc. Natl. Acad. Sci. USA, 2004, 101(33):12142-12147.
[7]Klar T, Pokorny R, Moldt J, et al. Cryptochrome 3 from Arabidopsis thaliana: structural and functional analysis of its complex with a folate light antenna [J]. J. Mol. Biol., 2007, 366(3): 954-964.
[8]Pokorny R, Klar T, Hennecke U, et al. Recognition and repair of UV lesions in loop structures of duplex DNA by DASH-type cryptochrome [J]. Proc. Natl. Acad. Sci. USA, 2008, 105(52): 21023-21027.
[9]Hitomi K, DiTacchio L, Arvai A S, et al. Functional motifs in the (6-4) photolyase crystal structure make a comparative framework for DNA repair photolyases and clock cryptochromes [J]. Proc. Natl. Acad. Sci. USA, 2009, 106(17): 6962-6967.
[10]Lin C, Shalitin D. Cryptochrome structure and signal transduction [J]. Annu. Rev. Plant Biol., 2003, 54(1): 469-496.
[11]Malhotra K, Kim S T, Batschauer A, et al. Putative blue-light photoreceptors from Arabidopsis thaliana and Sinapis alba with a high degree of sequence homology to DNA photolyase contain the two photolyase cofactors but lack DNA repair activity [J]. Biochemistry, 1995, 34(20): 6892-6899.
[12]Banerjee R, Schleicher E, Meier S, et al. The signaling state of Arabidopsis cryptochrome 2 contains flavin semiquinone [J]. J. Biol. Chem., 2007, 282(20): 14916-14922.
[13]Bouly J P, Schleicher E, Dionisio-Sese M, et al. Cryptochrome blue light photoreceptors are activated through interconversion of flavin redox states [J]. J. Biol. Chem., 2007, 282(13): 9383-9391.
[14]Lin C, Robertson D E, Ahmad M, et al. Association of flavin adenine dinucleotide with the Arabidopsis blue light receptor CRY1 [J]. Science, 1995, 269(5226): 968-970.
[15]Berndt A, Kottke T, Breitkreuz H, et al. A novel photoreaction mechanism for the circadian blue light photoreceptor Drosophila cryptochrome [J]. J. Biol. Chem., 2007, 282(17): 13011-13021.
[16]Koornneef M, Rolff E, Spruit C J P. Genetic control of light-inhibited hypocotyl elongation in Arabidopsis thaliana (L.) Heynh [J]. Z. Pflanzenphysiol. Bd.,1980, 100(2): 147-160.
[17]Ahmad M, Cashmore A R. HY4 gene of A. thaliana encodes a protein with characteristics of a blue-light photoreceptor [J]. Nature, 1993, 366(6451): 162-166.
[18]Ahmad M, Lin C, Cashmore A R. Mutations throughout an Arabidopsis blue-light photoreceptor impair blue-light-responsive anthocyanin accumulation and inhibition of hypocotyl elongation [J]. Plant J., 1995, 8(5): 653-658.
[19]Lin C, Ahmad M, Cashmore A R. Arabidopsis cryptochrome 1 is a soluble protein mediating blue light-dependent regulation of plant growth and development [J]. Plant J., 1996, 10(5): 893-902.
[20]Lin C, Ahmad M, Gordon D, et al. Expression of an Arabidopsis cryptochrome gene in transgenic tobacco results in hypersensitivity to blue, UV-A, and green light [J]. Proc. Natl. Acad. Sci. USA, 1995, 92(18): 8423-8427.
[21]Mockler T C, Guo H, Yang H, et al. Antagonistic actions of Arabidopsis cryptochromes and phytochrome B in the regulation of floral induction [J]. Development,1999,126(10):2073-2082.
[22]Ahmad M, Grancher N, Heil M, et al. Action spectrum for cryptochrome-dependent hypocotyl growth inhibition in Arabidopsis [J]. Plant Physiol., 2002, 129(2): 774-785.
[23]Blum D E, Neff M M, Van Volkenburgh E. Light-stimulated cotyledon expansion in blu3 and hy4 mutants of Arabidopsis thaliana [J]. Plant Physiol., 1994, 105(4): 1433-1436.
[24]Lin C, Yang H, Guo H, et al. Enhancement of blue-light sensitivity of Arabidopsis seedlings by a blue light receptor cryptochrome 2 [J]. Proc. Natl. Acad. Sci. USA, 1998, 95(5): 2686-2690.
[25]Ma L, Li J, Qu L, et al. Light control of Arabidopsis development entails coordinated regulation of genome expression and cellular pathways [J]. The Plant Cell,2001,13(12):2589-2607.
[26]Nagashima A, Hanaoka M, Shikanai T, et al. The multiple-stress responsive plastid sigma factor, SIG5, directs activation of the psbD blue light-responsive promoter (BLRP) in Arabidopsis thaliana[J]. Plant Cell Physiol.,2004,45(4):357-368.
[27]Guo H, Yang H, Mockler T C, et al. Regulation of flowering time by Arabidopsis photoreceptors [J]. Science, 1998, 279(5355): 1360-1363.
[28]Mao J, Zhang Y C, Sang Y, et al. A role for Arabidopsis cryptochromes and COP1 in the regulation of stomatal opening [J]. Proc. Natl. Acad. Sci. USA, 2005, 102(34): 12270-12275.
[29]Kang C Y, Lian H L, Wang F F, et al. Cryptochromes, phytochromes, and COP1 regulate light-controlled stomatal development in Arabidopsis[J]. The Plant Cell, 2009, 21(9): 2624-2641.
[30]Strasser B, Sanchez-Lamas M, Yanovsky M J, et al. Arabidopsis thaliana life without phytochromes [J]. Proc. Natl. Acad. Sci. USA, 2010, 107(10): 4776-4781.
[31]Lian H L, He S B, Zhang Y C, et al. Blue-light dependent interaction of cryptochrome 1 with SPA1 defines a dynamic signaling mechanism [J]. Genes Dev., 2011, 25(10): 1023-1028.
[32]Zuo Z Z, Liu H T, Liu B, et al. Blue light-dependent interaction of CRY2 with SPA1 regulates COP1 activity and floral initiation in Arabidopsis [J]. Curr. Biol., 2011, 21(10): 841-847.
[33]Liu H T, Wang Q, Liu Y W, et al. Arabidopsis CRY2 and ZTL mediate blue-light regulation of the transcription factor CIB1 by distinct mechanisms [J]. Proc. Natl. Acad. Sci. USA, 2013, 110(43): 17582-17587.
[34]Casal J J. Photoreceptor signaling networks in plant responses to shade [J]. Annu. Rev. Plant Biol., 2013, 64(1): 403-427.
[35]Ma D B, Li X, Guo Y X, et al. Cryptochrome 1 interacts with PIF4 to regulate high temperature-mediated hypocotyl elongation in response to blue light [J]. Proc. Natl. Acad. Sci. USA, 2016, 113(1): 224-229.
[36]Zhu Z Q, Lin C T. Photomorphogenesis: when blue meets red [J]. Nature Plant, 2016, 2(2):16019.
[37]Li L,Li X,Liu Y W, et al. Flowering responses to light and temperature [J]. Sci. China Life Sci.,2015, 59(4): 403-408.
[38]Liu Y W, Li X, Liu H T, et al. Multiple bHLH proteins form heterodimers to mediate CRY2-dependent regulation of flowering-time in Arabidopsis[J]. PLoS Genet., 2013, 9(10): e1003861.

