抗生素最小抑制浓度法(MICs)评估环境大肠杆菌抗生素抗性
伍娇,饶大伟,严涛,王爽,陈鹏,张永奎,杨坤,*
1. 四川大学化学工程学院,成都 610065 2. 夏威夷马诺大学土木与环境工程系,美国 夏威夷 96822
细菌抗药性(antibiotic resistance,AR)热点不仅仅局限在医疗行业,许多其他环境场所,如市政污水处理系统、制药厂的排污、水产及畜牧养殖厂,因受到人为抗生素压力的作用,也成为抗生素抗性释放进入环境的热点[1]。越来越多的证据表明:环境中的抗生素抗性可以传递给临床致病菌,从而威胁人类健康[2-4],这使得细菌的抗药性成为紧迫的环境问题。而抗生素抗性基因(antibiotic resistance genes,ARGs)也越来越被认为是新兴的环境污染物[5]。它们和重金属一样,具有其他持久化学污染物 “易形成,难消除”的特性[6-7]。在抗生素的选择性压力下,细菌抗药性可以持久地存在于环境中。针对单个菌和细菌群落水平的适应性代价研究表明:在没有抗生素选择性压力的情况下,抗性菌通常也能续存相当长的一段时间[8]。抗性基因与其他被选择基因之间在基因水平上的链接会导致所谓的共选择效应。由此,一个非被选择的抗药性由于其与被选择基因之间存在某种链接而能够续存在生态群落中[9]。有报道证明这种共选择效应不仅存在于不同的细菌抗药性之间[10-12],也存在于抗生素与其他选择性压力如重金属[13-14]、清洁剂和杀虫剂[15]之间,从而使得细菌抗药性在没有抗生素选择性压力下,也能在环境中续存相当长的时间。由此,估计环境中细菌抗药性的影响范围及程度并评估抗药性感染风险而非单纯检测释放入环境中的抗生素,对于维护人类健康显得尤为重要。
但时至今日,在评估环境抗药性方面仍存在技术障碍[1]。到目前为止,直接用于环境样品的抗药性测试方法仍未开发。一种好的方法应该能实现从地理上相隔较远的环境区划采集到的样品间的对比,并能够追踪抗性的动态变化。世界范围内的微生物学家和环境科学家均借鉴临床标准判断菌体是否抗药,需要预设抗药阈值。这一预设的抗药阈值通常具有临床意义,但对于环境研究,其缺乏统计意义,且并不适用于所有的环境区划。此外,也有可能忽略一些潜在的抗药性机制。尽管基于非培养的分子生物学手段(特别是定量PCR)已经成功用于环境抗药性的评估[16-21],而此类方法的普遍运用由于一些技术原因而受到了限制。这类方法主要基于之前的临床研究。但是最近的环境研究表明,环境中的抗药性组成及其规模还远未完全了解[12, 22, 23]。而其他内在的技术因素,如DNA的提取方法,目标抗性的基因型以及所使用的引物集均会影响最终的结果,因此在没有统一规范的前提下,很难实现不同环境区划间的直接比较。
为解决上述环境抗药性评估中存在的问题,在本研究中,使用一种基于培养的表型分析方法来评估环境抗药性。在2个不同的地理区域(中国成都和美国夏威夷),对从不同环境区划(天然水系和市政污水)分离到的大肠杆菌,分别确定其各抗生素的最小抑制浓度(minimum inhibitory concentrations,MIC)。对MIC数据进行系统的统计学分析以评估环境中的抗药性,实现环境抗药性的跨时空对比。不同环境区划的抗药性通过MIC分位数、抗性百分比及多抗药性指数(multiple antibiotic resistance indexes,MARI)进行评价。通过对标准化的MIC数据进行聚类和非度量多维测度(non-metric multidimensional scaling,NMDS)解析两地环境细菌的抗药模式,比较了3个星期分离到的细菌的抗药性,分析中国内陆河流(锦江)中抗药性的时间变化情况。对环境菌株进行广谱β-内酰胺酶(extended-spectrumβ-lactamases,ESBLs)基因的PCR筛查证明抗性基因与抗性表型之间的联系。结合实验数据及两地现有的抗生素使用状况信息,分析两地抗药性水平与当地人类活动及抗生素实践之间的联系。
1 材料与方法(Materials and methods)
1.1 样品采集
分别于2014年3月26日、4月2日和4月9日在沿锦江的5个固定采样点采集水样。采样地点限于东经104°5'18.2''至 104o5'31.6'',北纬30°37'43.9''至 30°38'27.5''范围内。5个固定采样点的位置如图1所示。每个采样日,从5个采样点分别收集500 mL水样。采集的水样迅速送往实验室及时处理(3 h内)。在成都市污水处理系统的主干管上收集一个城市污水水样。夏威夷Manoa河的水样及采集详见文献[24]。沿着Manoa河设置6个采样点,其中3个位于热带丛林中的河流上游,另外3个位于市区的河流中下游,在设定的采样点分别采集水样。夏威夷污水样品采自当地污水处理厂Honouliuli wastewater treatment plant (WWTP)。
1.2 实验材料
夏威夷菌株测试所用抗生素卡那霉素A(kanamycin A)、阿米卡星(amikacin)、庆大霉素(gentamicin)、多粘菌素(colistin)、环丙沙星(ciprofloxacin)购自美国Enzo Life Sciences公司,链霉素(Streptomycin)、头孢氨苄(cefalexin)、头孢曲松钠(ceftriaxone)、氨苄青霉素(ampicillin)购自美国TCI公司,四环素(tetracycline)、萘啶酮酸(nalidixic acid)购自美国Alfa Aesar公司,氯霉素(chloramphenicol)购自美国VWR公司,头孢西丁(cefoxitin)购自加拿大Toronto Research Chemicals公司。成都菌株所用抗生素除了卡那霉素A(kanamycin A)购自美国Amresco公司,阿米卡星、庆大霉素、链霉素、头孢氨苄、环丙沙星、萘啶酮酸、青霉素、四环素、多粘菌素、头孢曲松钠均购自中国上海恒远公司(所有抗生素纯度信息具体见附表S1),蛋白胨大豆琼脂(TSA)、mTEC(membrane-Thermotolerant E. coli)琼脂购自北京陆桥技术公司,0.45 μm S-PAK®滤膜来自美国Merck Millipore公司。
1.3 实验方法
菌体计数及大肠杆菌分离:锦江河水样品分别进行异养菌平板计数(HPC)和大肠杆菌列举。HPC中用灭菌蒸馏水10倍系列稀释后,取适当浓度稀释液400 μL涂布在蛋白胨大豆琼脂板上。在37 ℃培养箱中培养22 h后,记录平板上的菌落数目,并估算原水品的HPC数值。大肠杆菌计数:取1 mL稀释水样(10-1和10-2)通过0.45 μm S-PAK®滤膜(Merck Millipore, Billerica, USA)过滤(微孔)进行过滤。过滤后将截留有细菌的过滤膜转移至mTEC琼脂平板上。大肠杆菌的列举采用EPA标准方法(方法1103)[25]。随机选取mTEC琼脂平板上的大肠杆菌在LB培养基上划线纯化。挑选的大肠杆菌的通过IMViC(indole-methyl red-Voges-Proskauer-citrate)实验进行测试以确定其属性,大肠杆菌验证率大于90%,经过验证的大肠杆菌制成甘油管于-80 ℃下保存并用于随后的分析。从锦江河水样中共收集96株大肠杆菌。依相同的方法从成都污水水样中分离出46株大肠杆菌。在夏威夷大学实验室中采用类似的方法从环境样品中分离了大肠杆菌。从Manoa河的水样和土样中共分离到190株大肠杆菌[24]。从当地的污水样品中分离到94株大肠杆菌。
MICs值测定:运用改良的微量肉汤稀释法[26]确定环境大肠杆菌的抗生素MIC值。首先,将分离得到大肠杆菌接种在含有LB肉汤的96孔板中,置于37oC过夜培养至稳定生长区。然后,将细胞培养物接种到含一定浓度范围抗生素(0.25,0.5,1,2,4,8,16,32,64,和128 μg·mL-1)的LB培养基测试板中。对于2种抑菌效价较高的抗生素,ceftriaxone和 ciprofloxacin,在培养液中的浓度范围设置为0.0125,0.025,0.05,0.1,0.2,0.4,0.8,1.6,3.2和6.4 μg·mL-1。接种采用火焰灭菌的48-针复制器以确保每株菌统一的接种量。在37 ℃培养20 h后,MIC终点值记为没有明显生长的最低抗生素浓度。每个浓度进行一次重复的测试,计算2个重复的平均值为最终的MIC值。不含抗生素的阳性和阴性对照分别用于检验环境大肠杆菌在实验室条件下的生长情况和灭菌的彻底性。整个过程的质量控制采用大肠杆菌标准菌株ATCC 25922,结果显示标准菌株ATCC 25922的抗生素MIC值与文献报道类似[27]。
环境大肠杆菌菌株的ESBL基因筛查:β-内酰胺酶是最常见的抗生素抗药性机制,尤其在革兰氏阴性菌中。其中,广谱β-内酰胺酶(ESBLs)因其多抗药性和世界范围内的散播而受到广泛关注。本研究对环境菌株的TEM 、SHV和OXA型β-内酰胺酶基因[28]进行了多重PCR的筛查以证明这些菌株经由MIC数据确定的对β-内酰胺类抗生素的抗性。利用夏威夷市政污水环境DNA提取物作为模板扩增这3个ESBL基因来作为blaTEM、blaSHV和blaOXA PCR检测的阳性对照,ESBL基因的PCR产物通过测序进行验证。
1.4 数据分析
将得到的MIC数据评价各环境区划的AR。在确定环境分离菌株的抗生素MIC值时,大肠杆菌标准菌株ATCC 25922作为敏感控制菌。该标准菌株对本研究中所使用的所有抗生素均敏感[27]。从不同环境区划分离到的各组大肠杆菌的MIC50和MIC90值分别从获得的MIC数据获得。以标准菌株MIC值的16倍作为截断点,判断各环境菌株是否抗药。即当分离菌株MICs值大于该截断点时,则认为该菌株抗药。环境样品的多抗性通过计算其多抗药性指数(MARIs)来衡量,采用下列公式计算[29]。
c代表从单个的环境样品中分离菌株的数目,ai为菌株i所抗的抗生素数目,b是受测抗生素的数量(成都菌株为11,夏威夷的为13)。在对MIC数据进行统计学分析时,首先将成都和夏威夷环境大肠杆菌的MICs数据对当地所确定的标准菌株ATCC 25922的MIC值分别进行标准化。然后,标准化的MIC数据乘以100后以2为底进行对数转换。转换后的各抗生素或大肠杆菌的MIC数据可以被看作多维向量,分别表示抗生素的抑菌谱或大肠杆菌的抗药谱。利用MATLAB R2012a (MathWorks, Natick, USA)内置的聚类分析软件Clustergram将转换后的数据绘制成聚类热图。各抗生素或大肠杆菌分别依据其MIC向量间的线性相关性和Euclidean距离完成聚类。各菌株MIC向量的Euclidean距离被用于NMDS分析,从而比较从不同环境区划和采样日期获得的各大肠杆菌的抗药谱。NMDS采用100次重复的随机启动配置样本点(大肠杆菌菌株),其准确性通过计算Kruskal应力指数(Kruskal stress)确定[30]。
2 结果(Results)
2.1 HPC和大肠杆菌计数
根据HPC和大肠杆菌计数结果(附表S2),不同采样日锦江水体中细菌的数量彼此不同。4月2号水样中细菌浓度明显高于其他2个采样日,尤其是大肠杆菌数目 (P < 0.01)。4月2日当天有中雨,河流的水位显著增加,这意味着在流域的其他地方有暴雨。地表径流给锦江河水体带来更多的细菌。在各采样日期,沿着河流水中的细菌浓度呈现规律性的变化,即在采样点1的细菌丰度(特别是大肠杆菌的丰度)显著高于其他4个地点(P < 0.05)。在采样点1的上游有一个挡水坝,形成的落差导致河床底部的污泥悬浮,从而搅起额外的细菌。随着河水流向下游部分细菌再次沉降,而细菌的浓度又回落到一个相对稳定的水平。
2.2 MICs数据
成都和夏威夷实验室确定的标准菌株的MIC值大致相等。其中MIC值存在偏差的4种抗生素是streptomycin, tetracycline, cefalexin 和 colistin,这可能是由于2个实验室所使用的这4种抗生素的效价和培养基成分存在差异所造成的。各组大肠杆菌的MIC分位数(MIC50和MIC90)总结在表1中,可以看出成都的环境抗药性总体高于夏威夷,仅有一种抗生素(Colistin)例外。
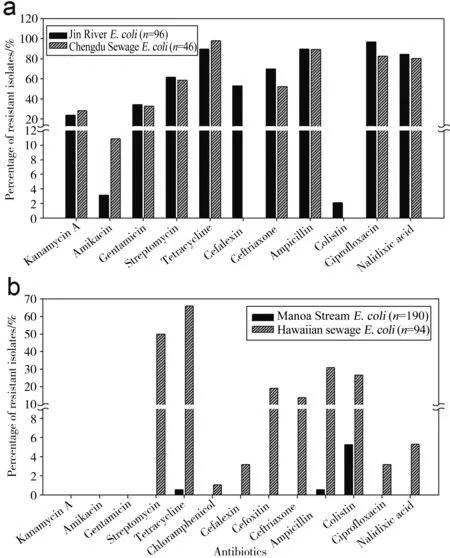
图2 成都(a)和夏威夷(b)大肠杆菌的抗生素抗性Fig. 2 Antibiotic resistance (AR) among the E. coli strains isolated at Chengdu (a) and Hawai'i (b)

AntibioticsMIC50/MIC90/(μg·mL-1)JinRiverEscherichiacoliisolates(n=96)ChengdusewageEscherichiacoliisolates(n=46)ManoaStreamEscherichiacoliisolates(n=190)HawaiiansewageEscherichiacoliisolates(n=94)KanamycinA32/>12816/>12816/168/16Amikacin16/3216/>1288/168/16Gentamicin8/>1288/>1284/88/8Streptomycin>128/>128>128/>12816/16120/>128Tetracycline128/>128128/>1281/232/128ChloramphenicolND*ND*4/48/8Cefalexin>128/>12864/1288/168/32CefoxitinND*ND*4/84/64Ampicillin>128/>128>128/>1284/84/>128Colistin2/44/81/82/32Nalidixicacid>128/>128>128/>1284/44/8Ceftriaxone>6.4/>6.41.6/>6.40.05/0.10.05/>6.4Ciprofloxacin>6.4/>6.40.8/>6.40.025/0.0250.025/0.05
注: ND表示未检测出结果。
Note:*ND denotes not determined.
2.3 抗生素抗性分析
锦江大肠杆菌对多数抗生素均表现出高的抗性百分比(超过50%),除了kanamycin A, amikacin, gentamicin和colistin(图2a)。有趣的是当地污水大肠杆菌也呈现相似的抗药性水平,多抗药性的情况也非常严重(图3和表2)。其中有一株菌对所有11种抗生素均具抗性。锦江的多抗药性指数(MARI,0.553)甚至稍高于当地污水的MARI值(0.484)。而夏威夷污水中大肠杆菌的抗药性和多抗药性均低得多(MARI值为0.169),但仍然明显高于Manoa河大肠杆菌的抗药性(图2b和图3)。夏威夷的大肠杆菌对3种氨基糖苷类抗生素:kanamycin A, amikacin和gentamicin不具抗性。从2个地区分离到的大肠杆菌对tetracycline(TC)均表现出最高的抗药性。
2.4 环境AR的时间-空间对比
针对锦江抗药性的时空特性进行了分析。对单一采样事件而言,从不同采样点获得的大肠杆菌抗药性水平没有表现出显著的差异(表2)。而不同的采样日获得的水样却表现出不同的抗药性。其中3月25日的样品所分离的大肠杆菌显示出最高的抗性水平,而4月2号的样品所分离出大肠杆菌抗药性最低(表2)。同样的结果也反映在图4中。
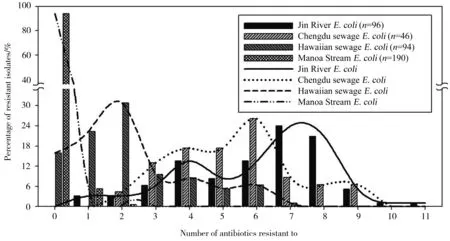
图3 大肠杆菌多抗药性Fig. 3 Multiple drug resistance (MDR) among the E. coli isolates
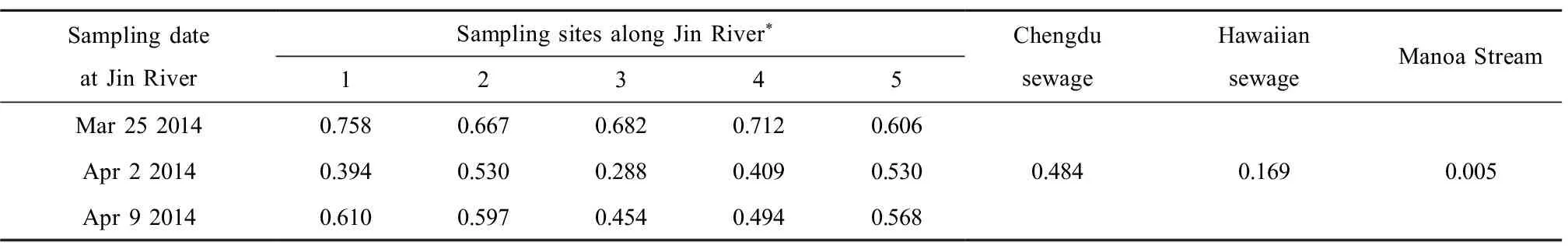
SamplingdateatJinRiverSamplingsitesalongJinRiver*12345ChengdusewageHawaiiansewageManoaStreamMar2520140.7580.6670.6820.7120.606Apr220140.3940.5300.2880.4090.5300.4840.1690.005Apr920140.6100.5970.4540.4940.568
注:在不同的取样日期中,3月25日锦江河大肠杆菌的多抗药性指数远远高于其他2天(P<0.05),并且4月2日的锦江河大肠杆菌多抗药性指数最低(P<0.05),不同的采样点的多抗药性指数无太大差异。
Note:*Among different sampling dates, the Jin River water samples of Mar. 25 showed significantly higher MDR than other two days (P<0.05), while the water samples of Apr. 2 gave the lowest MDR (P<0.05). There is no significant difference in MDR among different sampling sites.

图4 不同采样日锦江水样中大肠杆菌的抗药(a)及多抗药性(b)Fig. 4 AR (a) and MDR (b) among E. coli strains isolated from Jin River water samples of different dates
聚类热图(图5)中抗生素(行)和大肠杆菌(列)依据相应的MIC向量完成Euclidean聚类。由于MIC数据进行了归一化、乘以100及对数转化等处理,并且设定抗药截断点为标准菌株MIC值的16倍。大于log2(1600)=10.64的数据代表抗药,也就是说图中暖色块(不同饱和度的红色)意味着抗药,而冷色块(不同饱和度的蓝色)意味着敏感。大肠杆菌依据其抗药谱的相似性形成聚类,而抗生素依据其抑菌谱的相似性形成聚类。
MIC数据反映大肠杆菌的抗药谱,而菌体的抗药谱反映其抗生素接触史,菌体的抗药谱曾作为细菌的表型“指纹”对细菌进行溯源[29, 31-34]。MIC数据的NMDS分析可以解析2个地区不同环境区划大肠杆菌的抗药谱。大肠杆菌依据其抗药谱标示为二维空间的散点(图6)。散点间的距离表示各菌株之间的抗药谱的差异。对菌体多抗药性数目进行插值连续化处理之后生成多抗药性的等高图衬在了NMDS图下。可以看出,锦江河受到持续的抗药性污染,而成都污水菌株(白色方点)的抗药模式较为多样,表明其来源的多样性。夏威夷污水菌(红色三角)散落在图中多抗药性相对较弱的区域,Manoa河菌(绿色星形)聚集在无多抗药性的区域。成都地区污水及锦江河分离大肠杆菌整体较夏威夷污水中大肠杆菌有更严重的多抗药性。

图5 锦江(a)及夏威夷污水(b)大肠杆菌MIC数据的聚类热图Fig. 5 Clustered heat map generated from antibiotic MIC data of E. coli strains isolated from Jin River water (a) and Hawaiian sewage (b)
2.5 ESBLs结果
对于夏威夷大肠杆菌,我们检测了头孢菌素耐药株中3种常见的ESBL基因(blaTEM、blaSHV和blaOXA)。在污水中分离到15株耐头孢菌素大肠杆菌,其中只有4株检测到blaTEM基因。对于成都大肠杆菌,对所有锦江河和污水分离株进行了全面调查,其中66.9%(142株中的95株)的大肠杆菌检测到TEM基因,SHV 4.2%(n=6)和OXA 2.8%(n=4)。对3种β-内酰胺类抗生素均无抗性的10株大肠杆菌中,其中有9株没有检测到任何一种ESBL基因。图7显示了检测到的ESBL基因和β-内酰胺类抗生素(头孢氨苄、头孢曲松和氨苄青霉素)MIC值及细菌多抗药性的正相关性。
3 讨论(Discussion)
在本研究中,依据环境分离大肠杆菌的抗生素最小抑制浓度评估环境抗生素抗性。引入标准菌株(ATCC 25922)对MIC数据进行标准化,屏蔽了由于培养基成分、不同抗生素供货商提供的抗生素纯度和效价的差异等因素对实验数据造成的影响。环境分离株和标准均属于同个菌种,设置抗性截断点为16倍于敏感标准菌株(ATCC 25922)的MIC值,而环境菌株在明显较高的抗生素浓度下仍然能够生长,环境菌株必然存在某种抗药性机制使其能够在如此高的抗生素浓度下生存。这样的设定赋予了环境样品的抗生素抗性统计学意义。在报道该方法确定的环境抗生素抗性时,明确指出了MIC截断点,而标准菌株可从菌种库获得,其性质的表征也已为人熟知,任何人均可重复上述实验并获得类似的结果。除此之外,该方法另外一个好处是得到的结果比较全面,实现环境抗药性的跨时空对比。从空间角度看,环境大肠杆菌抗生素MIC数据的多极化分布直观地反映出抗性污染的存在以及抗性污染的程度。典型的,从夏威夷污水中分离到的大肠杆菌组,仅有3种抗生素(kanamycin A, amikacin和gentamicin)的MIC值没有表现出多极分布(附录Table S6)。而它们正是污水大肠杆菌不具抗性的3种抗生素(图2b)。而对于Manoa河中分离到的大肠杆菌组,因其很可能没有受到抗生素抗性污染,几乎所有抗生素的MIC均无多极分布,而其中3个例外的抗生素(tetracycline, ampicillin和colistin)呈现出几乎可以忽略的多极化分布(附录Table S5)。对于从锦江和成都污水中分离的的大肠杆菌组,几乎所有的抗生素都表现出多极的MIC分布。其中amikacin对锦江大肠杆菌的MIC数据,cefalexin和colistin对成都污水大肠杆菌的MIC数据例外,而这正是那些相应大肠杆菌组对抗生素不具抗性的情况(附录Table S3,S4)。由此,通过该方法,可以实现抗生素抗性的跨地域对比。从时间角度看,锦江菌体抗药性是随时间变化的(图4)。4月2日有中雨。尽管地表径流给河道带来了更多的细菌,但这些细菌包含来源于非抗性污染地区的抗药性较弱的菌株。因此,降雨可以看作河道抗性菌的稀释事件。根据NMDS分析(图6),锦江河道受到持续的抗药性污染,而且此污染呈现出如下具体的特征:单一来源(NMDS显示出单一的多抗药性模式,即图中红圈圈住的点)及其严重的多抗药性(平均抗药数目在7左右)。4月2日(黑色菱形)大肠杆菌的抗药模式分布更为分散,表明雨水的确为水体带来了不同来源的大肠杆菌。4月9日(紫色圆点)的菌株中亦包含抗性模式不同的菌株,应该是降雨持续影响的结果。而成都污水菌株(白色方点)的抗药模式较为多样,表明其来源的多样性。夏威夷污水菌(红色三角)散落在图中多抗药性相对较弱的区域,Manoa河菌(绿色星形)聚集在无多抗药性的区域。总之,成都地区污水及锦江河分离大肠杆菌整体较夏威夷污水中大肠杆菌有更严重的多抗药性。环境抗生素抗性的跨区域的比较和动态追踪均得以实现。但该方法的缺点是涉及的样品较多,实验中工作量较大,且只适用与能分离到大肠杆菌的情况。
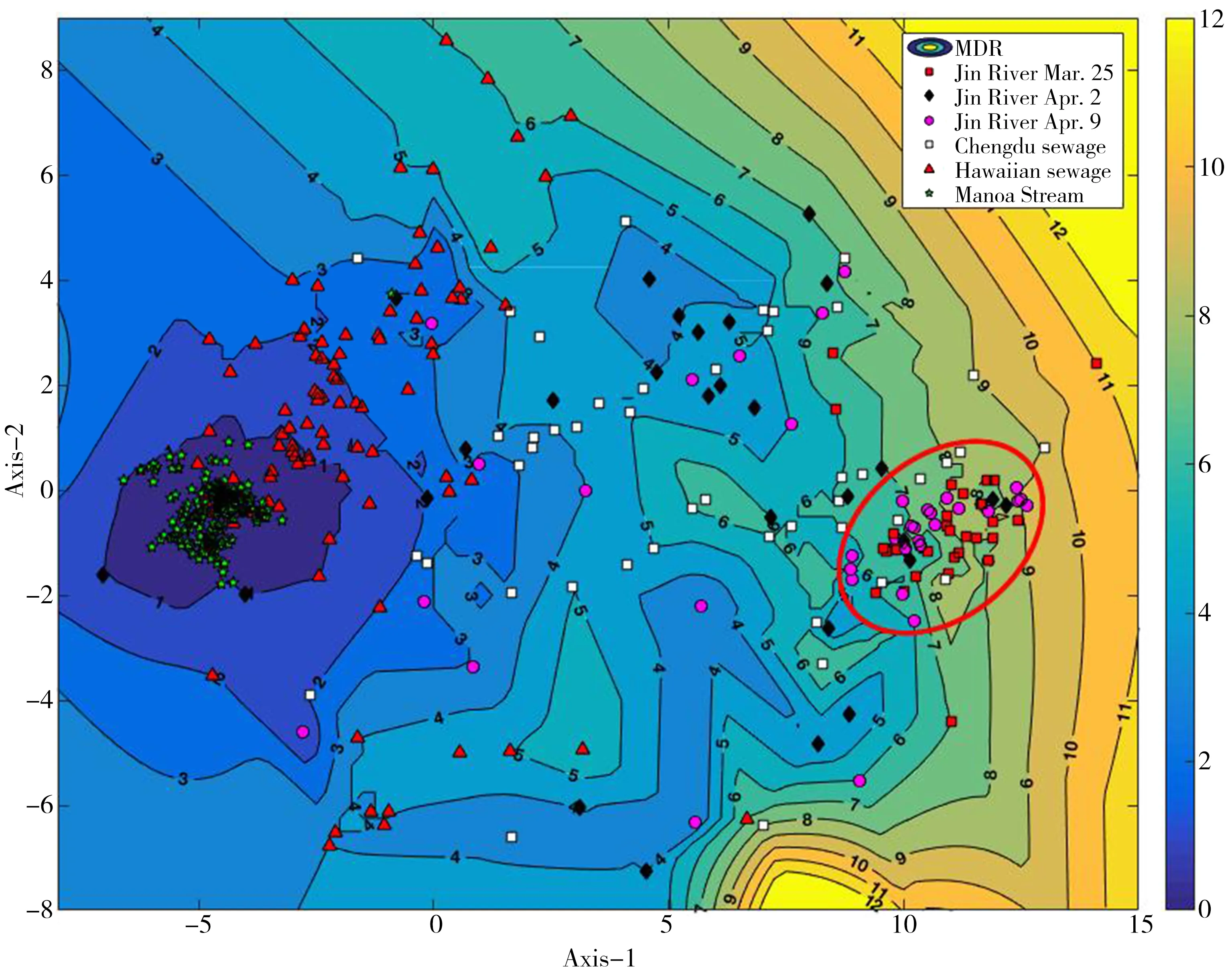
图6 大肠杆菌抗药谱的NMDS分析Fig. 6 NMDS analysis of AR patterns of the E. coli isolatesNote: MDR, Contour map of multi-drug resistance; the numbers on the contour lines are the numbers of antibiotics that one strain is resistant to; the Kruskal stress is 0.0988.

图7 ESBL基因与β-内酰胺类抗生素MICs(a)及多抗药性(b)之间的关系Fig. 7 The correlation of ESBL genes with MICs of β-lactam antibiotics (a) and MDR (b)
地理上相隔甚远的2个地区,其天然水体的细菌抗药性存在巨大的差异,这反映了两地人类活动对水体所施加影响的差异。中国不仅是全球最大的抗生素生产国也是最大的消费国[35]。之前的研究表明,抗生素及细菌抗药性在中国的不同环境区划均有例行检出[21, 36-44]。而这些研究主要集中在东南沿海地区。中国的地理地形决定了其主要河流发源于西部的大山而流向东部海岸。因此,要全面了解整个国家环境抗药性的形势并追寻其形成和发展的内在机制,监控中国中西部内陆水系中的抗药性具有重要意义。作为一条内陆水道,锦江河是2条河流(府河,其上游被称为柏条河,与南河,其上游被称为清水河)的汇流,这2条河都流经成都市区。它们都是岷江(长江上游主要支流之一)的分支,都由都江堰引流而至,都江堰是我国古代最著名的水利灌溉工程之一,至今仍在使用。在流入成都市区之前,它们都流经开阔的农业灌溉区。水样的采集点则正好位于2条河流汇流点的下游位置(图1)。在我国,对畜牧业与水产养殖业的抗生素使用没有严格禁止。数十种抗生素作为饲料添加剂使用。在全球用于食用动物生产的抗生素消费中,中国占据最大的份额(2010年占23%)[45]。畜牧业以及水产养殖业的开放性排放可能对水体带来严重的抗药性污染。流经成都后,锦江重新汇入岷江。由于联合下水排污系统在城市的部分地区仍在使用,锦江水体兼具农业与城市的双重影响。而夏威夷的Manoa河发源于Koolau山脉的热带雨林,流经Manoa山谷居民区,最后经由Ala Wai Canal注入大海。夏威夷拥有自19世纪末便开始构建的完备的污水收集、处理和排放系统。因此Manoa河水很少受到人类活动的影响,而没有观察到明显的抗生素抗性。
两地市政污水中的抗生素抗性也存在差异。由于Honouliuli污水处理厂的污水收集区域中不存在畜牧业,市政污水中的抗生素抗性在一定程度上反映了当地抗生素的临床使用状况。2010年抗生素的销售数据显示,美国当年各抗生素的销售量为头孢菌素类抗生素502 561 kg,喹诺酮类281 557 kg,四环素类129 183 kg,氨苄青霉素42 643 kg及氨基糖苷类6 991 kg[46]。污水中对各抗生素的抗药性(图3-2b)与各抗生素的销售量(使用量)之间似乎是正相关的。最明显的结果是,3种氨基糖苷类抗生素(kanamycin A, amikacin和gentamicin)没有观察到抗性菌株可能源于这3种抗生素较低的使用量(销售量)。2010年间,中国是继印度之后的第二大临床抗生素消费国[47]。这正是为什么在一个如此人口稠密的中国内陆城市(成都)的市政污水中检测到严重细菌抗药性的原因。对抗生素临床使用的监管不力也加剧了这一形势。尽管中国政府近年来正逐步规范抗生素的临床使用,但仍缺乏严格的行政和法律约束。在中国,没有医生处方的情况下一些常规抗生素仍能从普通药店买到。通过比较两地细菌的抗生素抗药性,在成都具有更高的抗药性感染风险。要扭转这一形势,当地的首要任务是进一步规范各类抗生素的使用并强化污水的收集及集中处理。而在夏威夷,尽管完备的污水收集和处理系统有效控制了自然环境中的抗生素抗性并降低了抗性菌感染的风险,保险起见,对污水处理系统出水中的细菌抗药性的检测仍然是必要的,这可能涉及到现有污水处理手段的改进。对于已经存在严重抗药性的抗生素的使用,如在成都tetracycline,streptomycin,ceftriaxone,ampicillin,ciprofloxacin和nalidixic acid的使用,在夏威夷streptomycin和tetracycline的使用都需特别谨慎,对这些抗生素的使用必须严格限制。
不同抗生素的抑菌效价不同,而且菌体对各抗生素的抗药性也各有差异,这使得不同抗生素对同组大肠杆菌的MIC数据经过标准化后呈现数量级上的差异。因此,标准化后的数据随后进行了对数转化,不同抗生素MIC向量间的线性相关性作为其聚类分析的依据,该聚类分析可显示不同抗生素在抑菌谱上的相似性。根据聚类分析的结果(图5),不同抗生素的聚类机制至少有2种。其一,不同的抗生素属于同一种类,其抗药机制类似,即抗性决定子相同亦或处在接近的遗传位点上。其二,同组环境大肠杆菌的抗生素接触史决定了不同抗生素间的聚类。如果几种抗生素并存于同组菌体的抗生素接触史中,那么细菌在多种抗生素选择性压力下将表现出对这些抗生素的多抗药性,而这些抗生素在聚类分析时便会聚类在一起。在我们的研究中,2种情形似乎都遇到了。对于2个地理区域采集到的大肠杆菌,3种头孢菌素cefoxtin (对于成都的分离菌株未检测)、cefalexin和ceftriaxone,2种氨基糖苷类抗生素kanamycin A和amikacin,以及2种喹诺酮ciprofloxacin和nalidixic acid,均分别紧密聚类在一起。另一方面,不同种类抗生素间的聚类可能源于这些抗生素曾协同出现在两地细菌的抗生素接触史中。如对于锦江河的分离株(图5a),ampicillin与喹诺酮类的抗生素聚类在一起,而对于夏威夷污水的分离株(图5b),colistin与头孢菌素类抗生素聚类在一起。
colistin,也被称作多粘菌素E (polymyxin E),是一种环状多肽的混合物,亦是作为治疗多抗药性细菌感染的“最后手段”之一。之前的研究表明,带有NMD-1型金属β-内酰胺酶的多抗药性肠杆菌科对于colistin仍然敏感[48]。colistin曾经因为其肾和神经毒性而被中断使用。但最近它又被用于与其他一种或多种抗生素连用以治疗抗药性感染[49-51]。成都分离株的colistin抗性很少见,这一点是意料之外的。在中国,多粘菌素是使用最为广泛的兽用抗生素之一,通常作为畜牧业饲料添加剂使用。最近在中国已经发现了人和动物体内质粒介导的colistin抗性[52]。colistin作为一种多肽混合物,有报道指出其在消化道中被快速降解[53, 54]。这可能正是为什么在如此大的一个猪生产省份(四川)的环境中观察到低于预期的colistin抗药性的原因。此外,根据IMS Health MIDAS数据库(https://resistancemapcddep.org/AntibioticUse.php)发布的数据,在中国自2007年起,多粘菌素就已经很少作为临床抗生素使用,而在美国其临床使用仍在继续,并且每1 000个居民,其平均使用量为7个标准单位(standard unit per 1000 inhabitants)。成都污水分离株对这种抗生素均无抗性(图2a),而夏威夷污水分离株却表现出较高的抗性(抗性百分比为26%,图2b),并且在进行聚类分析时,colistin与头孢类抗生素聚类在一起(图5b)。两地具有高MIC值(≥128 μg·mL-1)的colistin大肠杆菌(成都1株,夏威夷3株)均表现出严重的多抗药性。成都抗药株的抗药数目高达11,而夏威夷抗药株的抗药数目均超过6。考虑到最近发现的质粒介导的colistin抗性,这些研究事实严重挑战了colistin与其他抗生素的临床联合使用。
ESBLs基因的筛查证明:环境大肠杆菌的抗生素抗性与其抗性决定子(抗性基因)之间存在正相关。然而这种正相关性低于我们的预期,表明基因型与表型间的偏差。表型和基因手段已联合使用于环境抗生素抗性的评估,其结果可彼此互补。而本研究的重点是表型分析手段,旨在实验环境抗生素抗性的时空追踪。而适用于环境研究的基因手段将是我们接下来的工作重点之一。
致谢:感谢国家自然科学基金(21677104)对本研究的支持。
[1] Berendonk T U, Manaia C M, Merlin C, et al. Tackling antibiotic resistance: The environmental framework [J]. Nature Reviews Microbiology, 2015, 13(5): 310-317
[2] Forsberg K J, Reyes A, Wang B, et al. The shared antibiotic resistome of soil bacteria and human pathogens [J]. Science, 2012, 337(6098): 1107-1111
[3] Bonnedahl J, Drobni M, Gauthier-Clerc M, et al. Dissemination of Escherichia coli with CTX-M type ESBL between humans and yellow-legged gulls in the south of France [J]. PLOS One, 2009, 4(6): e5958
[4] Hernandez J, Johansson A, Stedt J, et al. Characterization and comparison of extended-spectrum beta-lactamase (ESBL) resistance genotypes and population structure of Escherichia coli isolated from Franklin's gulls (Leucophaeus pipixcan) and humans in Chile [J]. PLOS One, 2013, 8(9): e76150
[5] Pruden A, Pei R, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: Studies in northern Colorado [J]. Environmental Science & Technology, 2006, 40(23): 7445-7450
[6] Aminov R I, Mackie R I. Evolution and ecology of antibiotic resistance genes [J]. FEMS Microbiology Letters, 2007, 271(2): 147-161
[7] Zhang X X, Zhang T, Fang H H. Antibiotic resistance genes in water environment [J]. Applied Microbiology and Biotechnology, 2009, 82(3): 397-414
[8] Andersson D I. The biological cost of mutational antibiotic resistance: Any practical conclusions? [J]. Current Opinion in Microbiology, 2006, 9(5): 461-465
[9] Enne V I. Enhancement of host fitness by the sul2-coding plasmid p9123 in the absence of selective pressure [J]. Journal of Antimicrobial Chemotherapy, 2004, 53(6): 958-963
[10] Funk J A, Lejeune J T, Wittum T E, et al. The effect of subtherapeutic chlortetracycline on antimicrobial resistance in the fecal flora of swine [J]. Microbial Drug Resistance, 2006, 12(3): 210-218
[11] Marti R, Scott A, Tien Y-C, et al. Impact of manure fertilization on the abundance of antibiotic-resistant bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest [J]. Applied and Environmental Microbiology, 2013, 79(18): 5701-5709
[12] Wright G D. The antibiotic resistome: The nexus of chemical and genetic diversity [J]. Nature Reviews Microbiology, 2007, 5(3): 175-186
[13] Baker-Austin C, Wright M S, Stepanauskas R, et al. Co-selection of antibiotic and metal resistance [J]. Trends in Microbiology, 2006, 14(4): 176-182
[14] Seiler C, Berendonk T U. Heavy metal driven co-selection of antibiotic resistance in soil and water bodies impacted by agriculture and aquaculture [J]. Frontiers in Microbiology, 2012, 3(69): 399
[15] Gaze W H, Zhang L, Abdouslam N A, et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment [J]. ISME Journal, 2011, 5(8): 1253-1261
[16] Volkmann H, Schwartz T, Bischoff P, et al. Detection of clinically relevant antibiotic-resistance genes in municipal wastewater using real-time PCR (TaqMan) [J]. Journal of Microbiological Methods, 2004, 56(2): 277-286
[17] Wang F H, Qiao M, Su J Q, et al. High throughput profiling of antibiotic resistance genes in urban park soils with reclaimed water irrigation [J]. Environmental Science & Technology, 2014, 48(16): 9079-9085
[18] Schwartz T, Kohnen W, Jansen B, et al. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms [J]. FEMS Microbiology Ecology, 2003, 43(3): 325-335
[19] Negreanu Y, Pasternak Z, Jurkevitch E, et al. Impact of treated wastewater irrigation on antibiotic resistance in agricultural soils [J]. Environmental Science and Technology, 2012, 46(9): 4800-4808
[20] Czekalski N, Gascon Diez E, Burgmann H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake [J]. ISME Journal, 2014, 8(7): 1381-1390
[21] Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms [J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440
[22] D'Costa V M, McGrann K M, Hughes D W, et al. Sampling the antibiotic resistome [J]. Science, 2006, 311(5759): 374-377
[23] Davies J, Davies D. Origins and evolution of antibiotic resistance [J]. Microbiology and Molecular Biology Reviews, 2010, 74(3): 417-433
[24] Goto D K, Yan T. Genotypic diversity of Escherichia coli in the water and soil of tropical watersheds in Hawaii [J]. Applied & Environmental Microbiology, 2011, 77(12): 3988-3997
[25] US EPA. Method 1103.1: Escherichia coli (E. coli) in water by membrane filtration using membrane-thermotolerant Escherichia coli Agar (mTEC) [R]. Washington DC: US EPA, 2002
[26] Bell T H, Yergeau E, Juck D F, et al. Alteration of microbial community structure affects diesel biodegradation in an Arctic soil [J]. FEMS Microbiology Ecology, 2013, 85(1): 51-61
[27] Andrews J M. Determination of minimum inhibitory concentrations [J]. Journal of Antimicrobial Chemotherapy, 2001, 48(suppl 1): 5-16
[28] Dallenne C, Da Costa A, Decre D, et al. Development of a set of multiplex PCR assays for the detection of genes encoding important beta-lactamases in Enterobacteriaceae [J]. Journal of Antimicrobial Chemotherapy, 2010, 65(3): 490-495
[29] Krumperman P H. Multiple antibiotic resistance indexing of Escherichia coli to identify high-risk sources of fecal contamination of foods [J]. Applied and Environmental Microbiology, 1983, 46(1): 165-170
[30] Cui H, Yang K, Pagaling E, et al. Spatial and temporal variation in enterococcal abundance and its relationship to the microbial community in Hawaii beach sand and water [J]. Applied and Environmental Microbiology, 2013, 79(12): 3601-3609
[31] Wiggins B, Andrews R, Conway R, et al. Use of antibiotic resistance analysis to identify nonpoint sources of fecal pollution [J]. Applied and Environmental Microbiology, 1999, 65(8): 3483-3486
[32] Parveen S, Murphree R L, Edmiston L, et al. Association of multiple-antibiotic-resistance profiles with point and nonpoint sources of Escherichia coli in Apalachicola Bay [J]. Applied and Environmental Microbiology, 1997, 63(7): 2607-2612
[33] Hagedorn C, Robinson S L, Filtz J R, et al. Determining sources of fecal pollution in a rural Virginia watershed with antibiotic resistance patterns in fecal streptococci [J]. Applied and Environmental Microbiology, 1999, 65(12): 5522-5531
[34] Wiggins B A. Discriminant analysis of antibiotic resistance patterns in fecal streptococci, a method to differentiate human and animal sources of fecal pollution in natural waters [J]. Applied and Environmental Microbiology, 1996, 62(11): 3997-4002
[35] Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance [J]. Environmental Science & Technology, 2015, 49(11): 6772-6782
[36] Xu W, Zhang G, Li X, et al. Occurrence and elimination of antibiotics at four sewage treatment plants in the Pearl River Delta (PRD), South China [J]. Water Research, 2007, 41(19): 4526-4534
[37] Zou S, Xu W, Zhang R, et al. Occurrence and distribution of antibiotics in coastal water of the Bohai Bay, China: Impacts of river discharge and aquaculture activities [J]. Environmental Pollution, 2011, 159(10): 2913-2920
[38] Wei R, Ge F, Huang S, et al. Occurrence of veterinary antibiotics in animal wastewater and surface water around farms in Jiangsu Province, China [J]. Chemosphere, 2011, 82(10): 1408-1414
[39] Luo Y, Mao D, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China [J]. Environmental Science & Technology, 2010, 44(19): 7220-7225
[40] Tao R, Ying G G, Su H C, et al. Detection of antibiotic resistance and tetracycline resistance genes in Enterobacteriaceae isolated from the Pearl rivers in South China [J]. Environmental Pollution, 2010, 158(6): 2101-2109
[41] Su H C, Ying G G, Tao R, et al. Occurrence of antibiotic resistance and characterization of resistance genes and integrons in Enterobacteriaceae isolated from integrated fish farms in South China [J]. Journal of Environmental Monitoring, 2011, 13(11): 3229-3236
[42] Su H C, Ying G G, Tao R, et al. Class 1 and 2 integrons, sul resistance genes and antibiotic resistance in Escherichia coli isolated from Dongjiang River, South China [J]. Environmental Pollution, 2012, 169(15): 42-49
[43] Xiong W, Sun Y, Zhang T, et al. Antibiotics, antibiotic resistance genes, and bacterial community composition in fresh water aquaculture environment in China [J]. Microbial Ecology, 2015, 70(2): 425-432
[44] Yang J F, Ying G G, Zhao J L, et al. Simultaneous determination of four classes of antibiotics in sediments of the Pearl Rivers using RRLC-MS/MS [J]. Science of the Total Environment, 2010, 408(16): 3424-3432
[45] Van Boeckel T P, Brower C, Gilbert M, et al. Global trends in antimicrobial use in food animals [J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(18): 5649-5654
[46] Food and Drug Administration (FDA). Drug Use Review [R]. Department of Health and Human Services, Public Health Service, Food and Drug Administration Center for Drug Evaluation and Research, Office of Surveillance and Epidemiology, 2012
[47] Van Boeckel T P, Gandra S, Ashok A, et al. Global antibiotic consumption 2000 to 2010: An analysis of national pharmaceutical sales data [J]. The Lancet Infectious Diseases, 2014, 14(8): 742-750
[48] Kumarasamy K K, Toleman M A, Walsh T R, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK: A molecular, biological, and epidemiological study [J]. The Lancet Infectious Diseases, 2010, 10(9): 597-602
[49] D'Souza B B, Padmaraj S R, Rekha P D, et al. In vitro synergistic activity of colistin and ceftazidime or ciprofloxacin against multidrug-resistant clinical strains of Pseudomonas aeruginosa [J]. Microbial Drug Resistance, 2014, 20(6): 550-554
[50] Le Minh V, Thi Khanh Nhu N, Vinh Phat V, et al. In vitro activity of colistin in antimicrobial combination against carbapenem-resistant Acinetobacter baumannii isolated from patients with ventilator-associated pneumonia in Vietnam [J]. Journal of Medical Microbiology, 2015, 64(10): 1162-1169
[51] Petrosillo N, Ioannidou E, Falagas M. Colistin monotherapy vs. combination therapy: Evidence from microbiological, animal and clinical studies [J]. Clinical Microbiology and Infection, 2008, 14(9): 816-827
[52] Liu Y-Y, Wang Y, Walsh T R, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study [J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168
[53] Rhouma M, Beaudry F, Theriault W, et al. Gastric stability and oral bioavailability of colistin sulfate in pigs challenged or not with Escherichia coli O149: F4 (K88) [J]. Research in Veterinary Science, 2015, 102(5): 173-181
[54] Guyonnet J, Manco B, Baduel L, et al. Determination of a dosage regimen of colistin by pharmacokinetic/pharmacodynamic integration and modeling for treatment of G.I.T. disease in pigs [J]. Research in Veterinary Science, 2010, 88(2): 307-314

