全基因组预测希金斯炭疽菌中碳水化合物酶类蛋白
韩长志
(西南林业大学林学院/云南省森林灾害预警与控制重点实验室,云南昆明 650224)
全基因组预测希金斯炭疽菌中碳水化合物酶类蛋白
韩长志
(西南林业大学林学院/云南省森林灾害预警与控制重点实验室,云南昆明 650224)
希金斯炭疽菌侵染菜心等十字花科植物引起的炭疽病,给各国农业生产造成了巨大的经济损失。基于前期研究结果,以658个分泌蛋白为基础序列,利用CAZymes Analysis Toolkit预测程序,分析上述蛋白中的碳水化合物酶类(CAZymes)蛋白,明确该菌中含有238个CAZymes,分为主要类别和复合类别2类,前者包括75个糖苷水解酶(GHs)、48个碳水化合物绑定结构(CBMs)、33个辅助酶类家族(AAs)、30个碳水化合物酯酶(CEs)、23个多糖裂解酶(PLs)、4个糖基转移酶(GTs),后者则包括17个GHs/CBMs、4个AAs/CBMs、4个CEs/CBMs等。研究结果可为深入开展该病菌侵染植物作用机制的研究提供一定的理论基础。
希金斯炭疽菌;碳水化合物酶类蛋白;预测程序;全基因组预测
碳水化合物酶类(carbohydrate-active enzymes,简称CAZymes)是植物病原菌侵染过程中突破寄主细胞第一道屏障——细胞壁的关键因素[1]。同时,对不同营养类型的植物病原真菌进行分析,可以明确非活体营养型病原菌基因组较活体营养型编码更多的CAZymes[2]。随着大量植物病原菌基因组测序的完成,近些年关于CAZymes的研究进展较快[3]。根据最新研究成果,以涵盖木质纤维素降解所需要的相关酶类进行分类,主要涉及以下6类:糖苷水解酶(glycoside hydrolases,简称GHs)[4]、糖基转移酶(glycosyl transferases,简称GTs)[5]、多糖裂解酶(polysaccharide lyases,简称PLs)[6]、碳水化合物酯酶(carbohydrate esterases,简称CEs)[6]、辅助酶类家族(auxiliary activities,简称AAs)[7]以及碳水化合物绑定结构(carbohydrate-binding modules,简称CBMs)[8]。
希金斯炭疽菌(ColletotrichumhigginsanumSacc.)作为半活体营养型植物病原菌,可以侵染菜心、白菜、羽衣甘蓝、大白菜、萝卜、结球甘蓝等十字花科蔬菜引起炭疽病[9-10],是农业生产上较为严重的病害之一[11]。国内外学者对其生物学特性、生防菌筛选以及遗传转化[12]、致病基因[13-14]、转化子的突变体库建立[15-16]、防治[17]、丝裂原活化蛋白激酶(MAPK)级联信号通路绘制[18],以及G蛋白信号调控因子(RGS)[19]、磷脂酰肌醇转移蛋白(PITP)[20]、G蛋白偶联受体(GPCR)[21]、腺苷酸环化酶及其相关蛋白[22-23]、14-3-3[24]、胞裂蛋白(septin)[25]、磷酸二酯酶(PDE)[26]等在内的诸多蛋白进行了生物信息学分析。
本研究基于前期所获得的希金斯炭疽菌中658个分泌蛋白,利用CAZymes Analysis Toolkit(CAT)预测工具明确该菌中CAZymes不同亚家族的分布情况,以期为深入开展其功能研究打下坚实的基础。
1 材料与方法
1.1 希金斯炭疽菌分泌蛋白序列来源
希金斯炭疽菌分泌蛋白,来自笔者在前期研究中获得的658个分泌蛋白[27]。
1.2 CAZymes预测
利用在线工具CAZymes Analysis Toolkit[28]进行预测。
2 结果与分析
2.1 希金斯炭疽菌中含有的CAZymes
通过CAT分析,明确希金斯炭疽菌中共含有238个CAZymes,其中GHs含量较高,所占比例为31.51%;其后依次为CBMs、AAs、CEs、PLs、GTs,所占比例分别为20.17%、13.87%、12.61%、9.66%、1.68%(图1)。此外,在上述CAZymes的预测中,不仅具有单一的酶类型,而且有部分蛋白同属于2种酶的类型,涉及GHs/CBMs、AAs/CBMs、CEs/CBMs 等(图1)。由于CAZymes分类系统的理论基础是建立在氨基酸序列的相似性上, 不同蛋白具有不同的结构折叠类型,因此,对于同一蛋白具有不同酶类型的情况,推测病原菌在侵染植物过程中上述酶所发挥的功能具有较多特性。
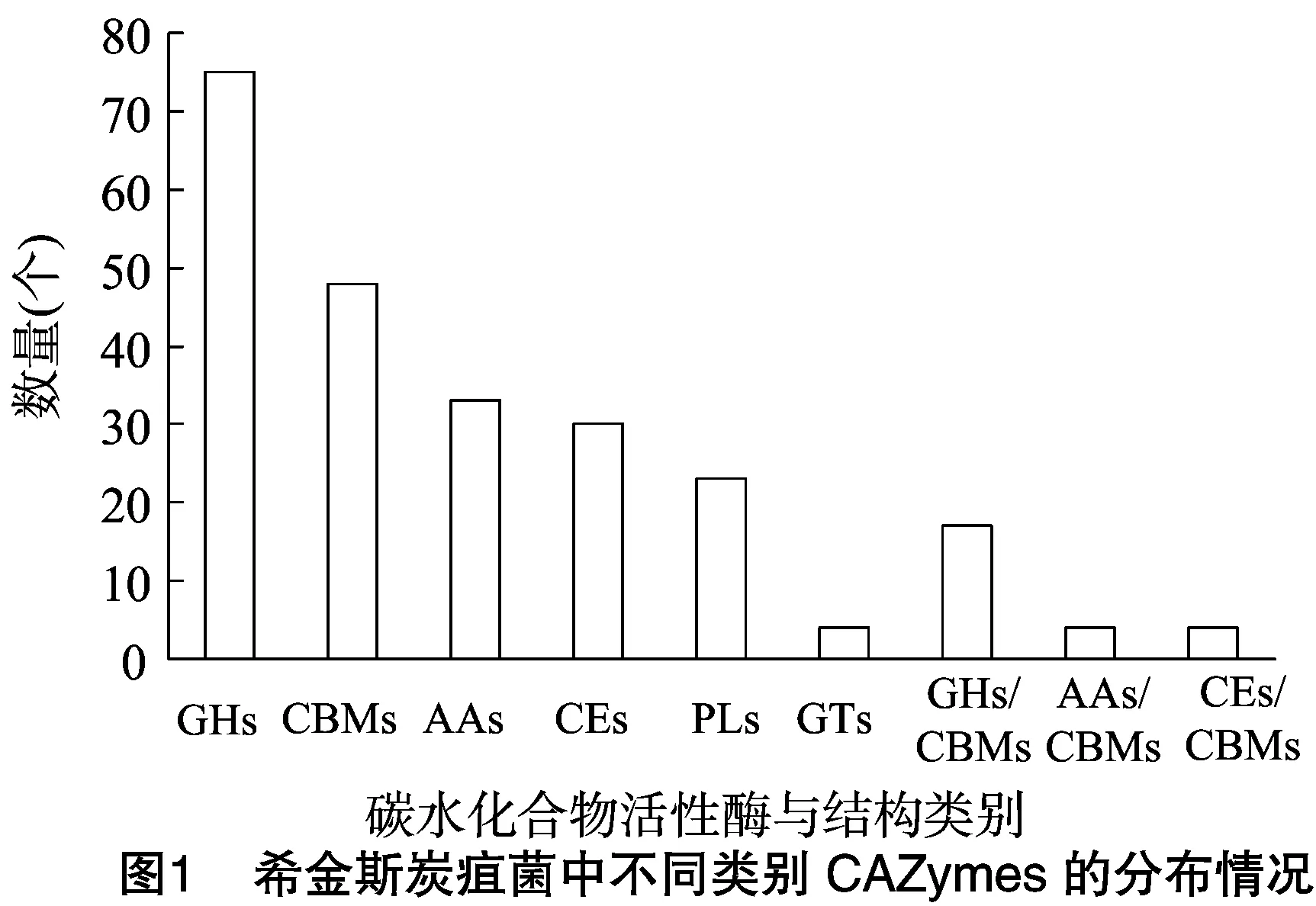
2.2 希金斯炭疽菌中含有的GHs
通过对上述糖基水解酶进行分析,发现GH28中蛋白数量较多,为9个,其次为GH16,数量为7个(表1)。值得关注的是,GH105、GH128、GH132、GH20、GH30等类别的酶均只含有1个蛋白,推测希金斯炭疽菌侵染不同植物所利用的酶并不相同,也从分泌酶角度可以解释其寄主广泛的原因。然而,对于为什么有些酶类所含数量较多,而大多数所含数量较少,有待进一步研究。
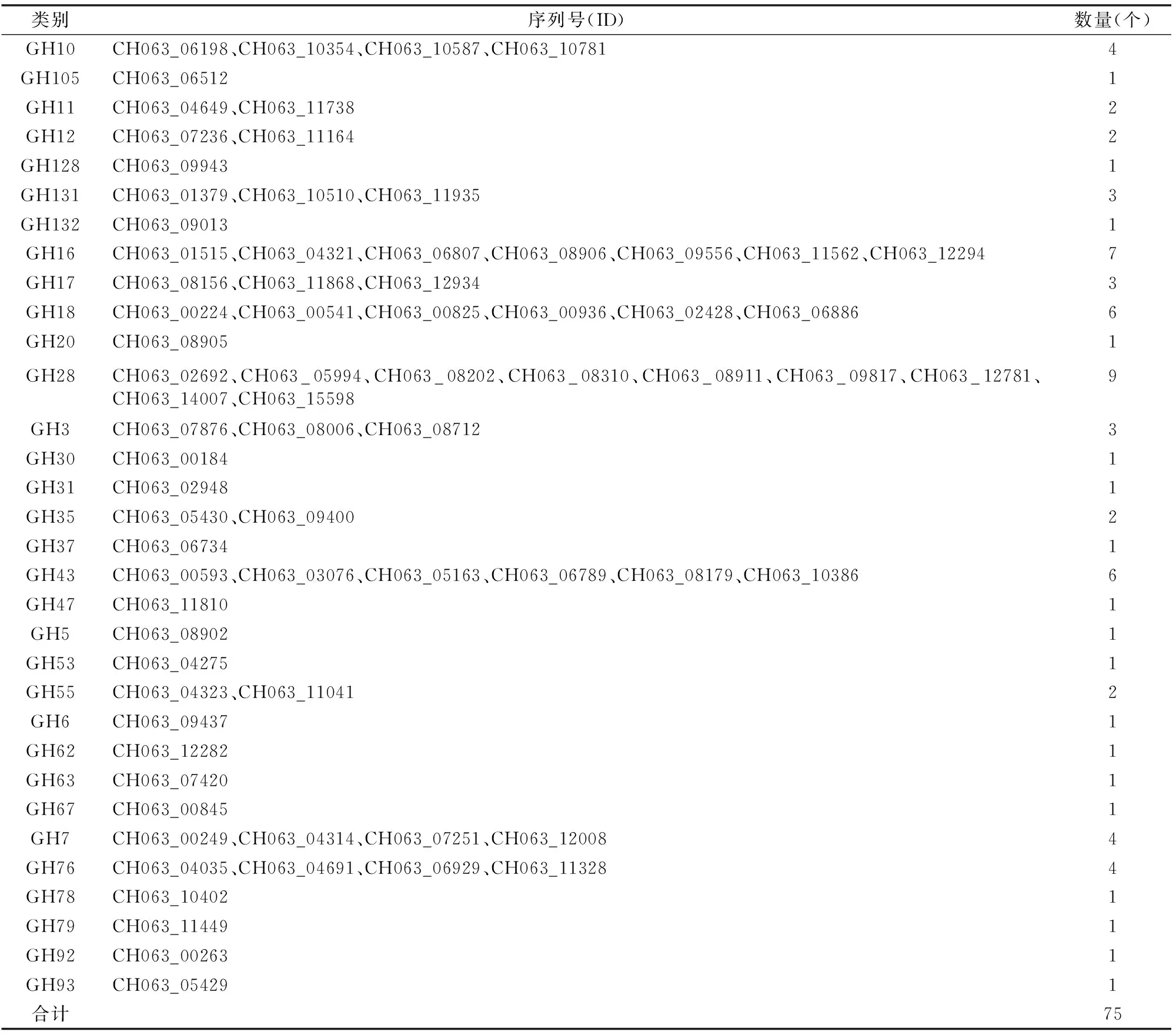
表1 希金斯炭疽菌中糖基水解酶分布情况
注:ID来自Broad数据库。下表同。
2.3 希金斯炭疽菌中含有的CBMs
通过对碳水化合物绑定结构蛋白进行分析,发现CBM50数量最多,为18个,其次为CBM13,数量为10个,上述蛋白所占比例为58.33%;此外,还涉及CBM1、CBM2、CBM3、CBM5、CBM12、CBM32、CBM63、CBM67等类别蛋白(表2)。值得关注的是,CH063_04845属于CBM10/CBM2类别,是CZAymes酶类主要类别中存在的唯一1个CBM蛋白,推测它在发挥CBM功能方面具有特殊性。因此,对于该蛋白有待于开展更为细致的工作,以期解释它们与其他蛋白之间的差别。
2.4 希金斯炭疽菌中含有的AAs
通过对辅助酶家族进行分析,发现AA9数量较多,为12个,其次为AA7,为6个,上述蛋白所占比例为54.55%;此外,还涉及AA1、AA2、AA3、AA5等类别蛋白(表3)。该类蛋白作为近些年新分类蛋白,具有重要的生物学功能,因此,有待于开展后续生物学研究,从而更好地解析该类蛋白的功能。
2.5 希金斯炭疽菌中含有的CEs
通过对碳水化合物酯酶蛋白进行分析,发现CE5、CE10数量较多,均为9个,其次为CE1,数量为3个,上述蛋白所占比例为70.00%;此外,还涉及CE3、CE4、CE6、CE8、CE10等类别蛋白(表4)。
2.6 希金斯炭疽菌中含有的PLs
通过对多糖裂解酶蛋白进行分析,发现PL1数量较多,为10个,其次为PL3,数量为8个,上述蛋白所占比例为 78.26%;此外,还涉及PL4、PL9等类别蛋白,其数量较少(表5)。

表2 希金斯炭疽菌中碳水化合物绑定结构分布情况

表3 希金斯炭疽菌中辅助酶类家族分布情况

表4 希金斯炭疽菌中碳水化合物酯酶的分布情况
2.7 希金斯炭疽菌中含有的GTs
通过对糖基转移酶蛋白进行分析,发现仅包含GT2、GT8、GT22、GT34等4个蛋白,其ID分别为CH063_00779、CH063_11721、CH063_11413、CH063_15057(表6)。与其他酶类进行比较,该酶类较少,推测它们在希金斯炭疽菌危害植物过程中发挥着特有功能。
2.8 希金斯炭疽菌中含有的复合类别蛋白
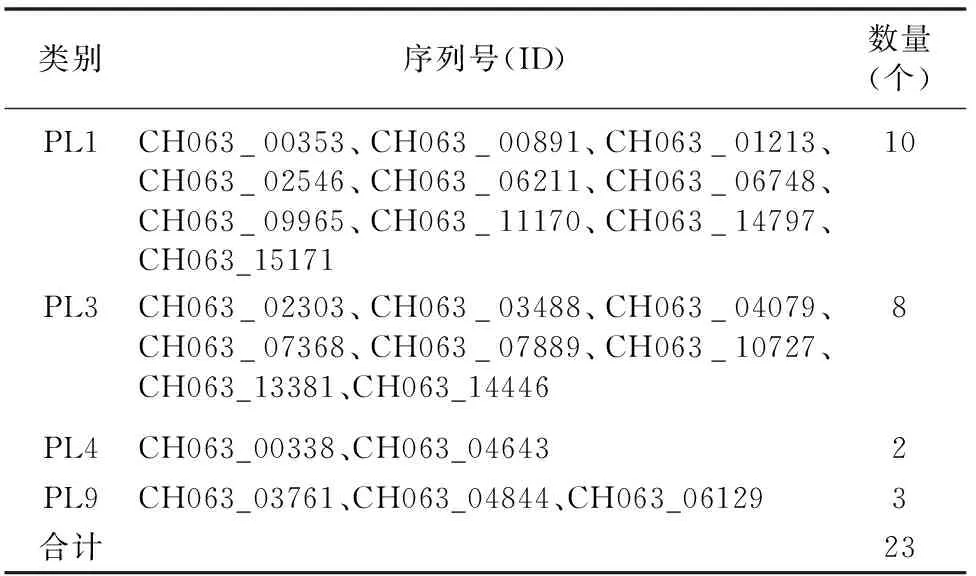
表5 希金斯炭疽菌中多糖裂解酶的分布情况

表6 希金斯炭疽菌中糖基转移酶的分布情况
通过对希金斯炭疽菌中CAZymes进行分析,发现除含有典型的GHs、CBMs、AAs、CEs、GTs以外,还含有一些复合类别的蛋白,涉及GHs/CBMs、AAs/CBMs、CEs/CBMs等3类,尤以GHs/CBMs类别中的蛋白数量居多,为17个,所占比例高达68.00%(表7)。上述复合类别的蛋白所具有的具体类别呈现出多样性特点,推测它们在希金斯炭疽菌危害植物过程中发挥着多种功能。

表7 希金斯炭疽菌中复合类别蛋白的分布情况
3 讨论与结论
随着希金斯炭疽菌全基因组序列的释放[29],学术界对其致病因子开展了大量研究。本研究结果与前人报道[29]不仅在CAZymes总数量方面有所差别,而且在具体的酶类别数量方面也有一定的差别。究其原因,就总数量而言,由于本研究所依赖的蛋白属于前期通过生物信息学分析所获得的分泌蛋白;在数量统计方面,以CAZymes主要类别和复合类别分别进行的统计,使得归属类别方面存在一定的差异,若将复合类别中的CAZymes分别归属到主要类别中,那么总的数量为263个,当然该统计存在25个重复蛋白;此外,就不同类别中CAZymes数量而言,按照上述统计方法,本研究所获得GHs、GTs、CBMs、CEs、PLs以及AAs则分别含有75、4、48、30、23、33个。可以发现,本研究所获得GHs数量明显少于前人所报道的361个[29],原因尚不明确,有待于进一步研究。前人所开展的研究中CAZymes的具体类别有5个[29],而最新的分类类别有6个,造成具体不同类别分项数量方面的差异。
本研究基于前期通过全基因组序列预测所获得的分泌蛋白,利用现有在线工具所获得的CAZymes是否为希金斯炭疽菌中的全部,均有待于进一步开展试验进行验证。在数量方面,O′Connell等报道为689个[29],该报道的数量远远高于本研究结果,就每一类中的数量报道也有不同,原因也有待于进一步明确。
前期明确希金斯炭疽菌中含有658个分泌蛋白,本研究利用CAT在线预测程序,明确该菌中含有238个CAZymes,分为主要类别和复合类别2类,前者包括75个GHs、48个CBMs、33个AAs、30个CEs、23个PLs、4个GTs;后者则包括17个GHs/CBMs、4个AAs/CBMs、4个CEs/CBMs等。本研究为深入开展希金斯炭疽菌中CAZymes的功能研究以及进一步找寻新的药剂作用靶标打下了一定的理论基础。
[1]van den Brink J,de Vries R P.Fungal enzyme sets for plant polysaccharide degradation[J]. Applied Microbiology and Biotechnology,2011,91(6):1477-1492.
[2]陈相永,陈捷胤,肖红利,等. 植物病原真菌寄生性与分泌蛋白组CAZymes的比较分析[J]. 植物病理学报,2014,44(2):163-172.
[3]Lombard V,Ramulu H G,Drula E,et al.The carbohydrate-active enzymes database(CAZy) in 2013[J]. Nucleic Acids Research,2014,42:D490-D495.
[4]Bourne Y,Henrissat B.Glycoside hydrolases and glycosyltransferases: families and functional modules[J]. Current Opinion in Structural Biology,2001,11(5):593-600.
[5]Campbell J A,Davies G J,Bulone V,et al.A classification of nucleotide-diphospho-sugar glycosyltransferases based on amino acid sequence similarities[J]. Biochemical Journal,1998,329(3):939-941.
[6]Lombard V,Bernard T,Rancurel C,et al.A hierarchical classification of polysaccharide lyases for glycogenomics[J]. Biochemical Journal,2010,432(3):437-444.
[7]Levasseur A,Drula E,Lombard V,et al.Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes[J]. Biotechnology for Biofuels,2013,6(1):1-14.
[8]Boraston A B,Bolam D N,Gilbert H J,et al.Carbohydrate-binding modules: fine-tuning polysaccharide recognition[J]. Biochemical Journal,2004,382(3):769-81.
[9]沈瑞清,张 萍,郭成瑾,等. 宁夏炭疽菌属真菌资源研究[J]. 河南农业科学,2012,41(5):100-102.
[10]Hyde K D,Cai L,Cannon P F,et al.Colletotrichum-names in current use[J]. Fungal Diversity,2009,39(1):147-182.
[11]卢博彬,杨 暹. 菜心炭疽病研究进展[J]. 长江蔬菜,2009(24):1-5.
[12]况福元,吴小丽,吕风青,等. 菜心炭疽病菌拮抗细菌的筛选及鉴定[J]. 微生物学通报,2009,32(9):1350-1355.
[13]Huser A,Takahara H,Schmalenbach W,et al.Discovery of pathogenicity genes in the crucifer anthracnose fungusColletotrichumhigginsianum,using random insertional mutagenesis[J]. Molecular Plant-Microbe Interactions,2009,22(2):143-156.
[14]Ushimaru T,Terada H,Tsuboi K,et al.Development of an efficient gene targeting system inColletotrichumhigginsianumusing a non-homologous end-joining mutant andAgrobacteriumtumefaciens-mediated gene transfer[J]. Molecular Genetics and Genomics,2010,284(5):357-371.
[15]周 鹏. 希金斯刺盘孢T-DNA插入体库的构建、筛选及相关突变体基因的克隆[D]. 武汉:华中农业大学,2011.
[16]赵 典. 希金斯刺盘孢T-DNA插入体库的筛选及相关突变体基因的克隆[D]. 武汉:华中农业大学,2012.
[17]郭巨先,刘玉涛,杨 暹. 钾营养对菜薹(菜心)炭疽病发生和植株防御酶活性的影响[J]. 中国蔬菜,2012(14):86-89.
[18]林春花,蔡志英,黄贵修. 全基因组法绘制禾谷炭疽菌和希金斯炭疽菌中MAPK级联信号途径简图[J]. 热带作物学报,2012,33(4):674-680.
[19]韩长志. 希金斯炭疽菌RGS蛋白生物信息学分析[J]. 生物技术,2014,49(1):36-41.
[20]韩长志. 希金斯炭疽菌PITP生物信息学分析[J]. 湖北农业科学,2015,54(3):713-716.
[21]韩长志. 希金斯炭疽菌GPCR蛋白生物信息学分析[J]. 华中师范大学学报(自然科学版),2015(2):246-251.
[22]韩长志. 希金斯炭疽菌腺苷酸环化酶生物信息学分析[J]. 湖北农业科学,2014,53(20):4992-4996.
[23]韩长志. 希金斯炭疽菌腺苷酸环化酶相关蛋白生物信息学分析[J]. 生物技术,2014,53(6):4992-4996.
[24]韩长志. 希金斯炭疽菌14-3-3蛋白质生物信息学分析[J]. 湖北农业科学,2014,53(15):3669-3672.
[25]韩长志. 希金斯炭疽菌中5个典型Septin的生物信息学分析[J]. 河南农业科学,2014,43(8):91-96.
[26]韩长志. 希金斯炭疽菌磷酸二酯酶生物信息学分析[J]. 科学技术与工程,2014,14(27):163-167.
[27]韩长志. 全基因组预测希金斯炭疽菌的候选效应分子[J]. 生物技术,2015,25(6):546-551.
[28]Park B H,Karpinets T V,Syed M H,et al.CAZymes Analysis Toolkit (CAT): web service for searching and analyzing carbohydrate-active enzymes in a newly sequenced organism using CAZy database[J]. Glycobiology,2010,20(12):1574-1584.
[29]O’Connell R J,Thon M R,Hacquard S,et al.Lifestyle transitions in plant pathogenicColletotrichumfungi deciphered by genome and transcriptome analyses[J]. Nature Genetics,2012,44(9):1060-1065.
10.15889/j.issn.1002-1302.2017.02.006
2015-12-16
国家自然科学基金(编号:31560211);云南省森林灾害预警与控制重点实验室开放基金(编号:ZK150004);云南省优势特色重点学科生物学一级学科建设项目(编号:50097505);云南省高校林下生物资源保护及利用科技创新团队(编号:2014015)。
韩长志(1981—),男,河北石家庄人,博士,副教授,研究方向为经济林木病害生物防治与真菌分子生物学。E-mail:hanchangzhi2010@163.com。
S432.4+4
A
1002-1302(2017)02-0024-04
韩长志. 全基因组预测希金斯炭疽菌中碳水化合物酶类蛋白[J]. 江苏农业科学,2017,45(2):24-28.

