富集优势基因型的后备亲本筛选以及相关分子标记的遗传效应分析
张成锋苏胜彦朱 健石连玉
(1. 中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业与种质资源利用重点实验室, 无锡 214081; 2. 中国水产科学研究院黑龙江水产研究所, 农业部淡水水产生物技术与遗传育种重点实验室, 哈尔滨 150070)
富集优势基因型的后备亲本筛选以及相关分子标记的遗传效应分析
张成锋1苏胜彦1朱 健1石连玉2
(1. 中国水产科学研究院淡水渔业研究中心, 农业部淡水渔业与种质资源利用重点实验室, 无锡 214081; 2. 中国水产科学研究院黑龙江水产研究所, 农业部淡水水产生物技术与遗传育种重点实验室, 哈尔滨 150070)
选取黄河鲤新品系亲本和子代共450尾, 5对微卫星引物和4个基因区段扩增引物开展基因型检测, 并检测它们与体重的关联性以及分析相应的遗传效应。结果显示: Koi42和4个SNPs位点对体重有显著的影响, 获得具有超过富集2个优势基因型的候选亲本共13尾, 富集优势分子标记基因型的候选亲本生长性能优势明显。通过多元逐步回归分析, 利用AIC最佳模型筛选到COⅠ626、D-Loop253和Koi42共3个位点, 发现Koi42贡献率较大, 经Fisher精确性检验, 发现其与性别存在关联, 其加性效应接近显著水平(P<0.05)。检测的多个上位效应组分中, 仅有D-Loop253和Koi42的加性效应间的互作达到显著水平。对遗传方差组分进行剖分发现, 加性方差占11.4%, 两个位点的加性效应构成的上位效应占到77.5%, 因此这3个分子标记对体重的影响主要以上位效应为主, 而且是两两加性效应的占比较大, 可以推断出D-Loop 253和Koi42两个分子标记的上位效应起主要作用。综上所述结果提示Koi42及与其有互作的D-Loop253可用于黄河鲤新品系的标记辅助选择,可以开展多个分子标记的富集选择。
黄河鲤新品系; 基因型; 体重; 关联分析; 上位效应
鲤产业在我国水产养殖乃至世界淡水养殖中均占据举足轻重的地位, 因而运用现代育种技术提升我们鲤产业的科技水平, 发展鲤种业具有重要的意义, 这也是国家发展现代农业和农业产业升级的内在要求。分子标记辅助育种、分子育种、基因组育种、基因组关联选择、GBLUP (Genomic best linear unbiased prediction, GBLUP)选育等成为现代育种的重要内容和主要手段(Jonas and de Koning)[1]。目前, 在鲤育种方面主要还是基于数量遗传学的BLUP (Best linear unbiased prediction, BLUP)育种、分子辅助育种、分子育种。Vandeputte等[2]通过分子标记和全因子交叉设计, 成功的进行了基于分子标记的鲤配对繁殖技术, 并估计了体重和体长的遗传力, 均为0.33, 他们主要采用的是选育原始代亲本, 然后再从它们的后代中增加留选亲本的思路。使用同样的方法, Kocour等[3]在鲤加工和肉质性状上同样进行了以分子育种为基础的遗传力估计。随后, Vandeputte等[4]不仅估计了鲤实际分子育种的遗传力, 还对选择反应作出评估: 配对方式采用全因子交叉设计, 体重的遗传力范围为0.31—0.44, 体长的遗传力范围为0.21—0.33, 体重和体长的遗传相关为0.97, 选育第3代时的遗传进展比较大。2011年, Vandeputte等[5]从整个育种流程和理论上分析总结(主要涉及理论推导、数据模拟到真实育种3个层次), 基于分子标记的水产育种的育种效果, 证明了合适的标记对于实际育种很有意义,也证实了此种方法在鲤育种中的可行性。
在现有分子标记中, 微卫星因其长度表现为高度多态性, 已成为真核生物基因组作图中不可缺少的分子遗传标记。利用微卫星的结构特点和遗传特性, 绘制高精度遗传图谱, 进行个体、品种(系)鉴定是微卫星的用途之一。Cheng等[6]和Zhang等[7]分别通过微卫星构建了鲤遗传连锁图, 随后与BAC文库构建的物理图相结合, 产生了高密度图谱[8]。SNP是指在染色体基因组水平上单个核苷酸的变异引起的DNA序列多态性, 主要包括单碱基的转换、颠换、插入及缺失等形式。Xu等[9]从4个鲤品种的转录组测序结果中寻找SNPs位点。随后, 他们在2014年构建了第一个高通量的SNP芯片, 其包含25万个SNPs位点, 在检测的1072个样本中约74.06%具有多态性[10]。此后, Zhang等[11]再次通过分子标记构建了鲤连锁图并研究了与鲤肌纤维相关的QTL (Quantitative trait locus, QTL)。可以看出在鲤育种领域积累了大量的分子标记和有效的检测方法, 并做了相应的分子育种工作。
研究报道表明相比传统的BLUP选育, 基于高额成本的基因型检测的育种可获得较高的遗传进展[12]。如果遇到遗传进展不够大或难以支持如此高的基因型检测成本的情形, 结合传统的BLUP估计、候选个体的预选择和低密度SNP芯片的应用是克服这些困难的好的选择[1]。 因此, 基于实际情况本文通过筛选到的微卫星和SNP分子标记来研究它们富集后对鲤选育群体体重的影响。然而对于体重这种复杂的经济性状, 表型和基因型之间并不存在严格的一一对应关系, 而往往是参与表型形成的因素涉及同一位点上等位基因产生的加性效应和显性效应、不同位点上非等位基因相互作用产生的上位效应以及这些基因与环境的共同作用[13]。因此, 本文在获得与体重显著相关的分子标记后,尤其是多个分子标记后, 明确这些标记通过哪几种遗传效应影响该经济性状是充分利用这些标记进行育种的重要参考和依据。
1 材料与方法
1.1 实验材料
本实验选用的试验鱼均养殖于中国水产科学研究院淡水渔业研究中心无锡南泉养殖试验基地,随机选取450个黄河鲤选育候选个体, 其中亲本168尾, 子代282尾。这些试验鱼是中国水产科学研究院淡水渔业研究中心通过基于家系的BLUP选育和分子设计育种结合开展选育的黄河鲤核心群。具体养殖办法是根据BLUP选育原理和分子设计育种结合的选配方案挑选出亲鱼进行繁殖配对, 繁殖的条件是水温为18—20℃, 配对的雌鱼和雄鱼的近交系数低于2%; 雌鱼和雄鱼配对后放入置有棕榈片制成的鱼巢(水位高度为85—90 cm)1d后, 将鱼巢转入孵化网箱中进行孵化3—4d后, 得到子代鱼苗;要求将不同家系的鱼苗隔离在不同的网箱中进行早期培育, 温度控制在20—28℃内, 水位高度设置为70—80 cm; 要求每半月加注1次新水; 孵出鱼苗后的第2至第15天泼洒豆浆进行喂养, 每日泼洒2—3次, 之后用颗粒饲料喂养, 每日投喂2—3次, 投喂量为鱼苗体重的5%—6%; 当鱼苗长到7—15 g时,在每个家系中各选50尾鱼苗用注射器将无线射频标记注入鱼腹腔, 进行个体标记, 从而识别每尾鱼苗; 将标记好的鱼苗在室内水泥池暂养, 用颗粒饲料喂养, 每日投喂2—3次, 投喂量为鱼苗体重的3%—4%; 喂养5—7d后, 将标记后的鱼苗转入室外土池中进行常规养殖, 饲料为商用成鱼料, 养殖到第3年的3月份测生长性能数据并鉴别雌雄。
本实验中的子代来自于亲鱼群体, 亲鱼的平均体重为1747.23 g, 子代的平均体重为946.49 g。要求亲本对本文所使用引物(表 1)全部检测, 子代则要求进行Koi42微卫星多态性检测。在进行体重称重时, 采用丁香油与乙醇的混合液将黄河鲤麻醉后,采用电子天平称量体重(精确到1 g), 同时剪取的尾鳍立即于95%的酒精中固定, 采样完毕, 样品置于-20℃冰箱保存。按照TaKaRa DNA提取试剂盒说明书提取DNA。采样的同时记录采样个体的体重,亲本还需记录性别。
1.2 分子标记的多态性检测
分子标记的检测分为2种情况进行检测, 微卫星主要是通过聚丙烯酰胺凝胶电泳分离后统计位点的基因型, SNP通过PCR反应后直接测序, 获得的序列使用DNAMAN V6软件进行序列比对, 并通过Chromas2.22软件核查, 确定SNPs。
PCR反应体系为25 μL, 包括10×buffer 15 μL, Mg2+(25 mmol/L) 1 μL, dNTPs (各2 mmol/L) 1 μL,上下游引物(10 mmol/L)各1 μL, 模板DNA 1 μL, Taq DNA聚合酶(Promega)1 U, dd H2O; 扩增反应均在TaKaRa公司PCR仪上完成。PCR反应程序为: 94℃预变性3min; 94℃变性20s, 温度56—66℃退火20s, 72℃延伸30s, 33个循环; 72℃延伸10min。将反应后的PCR产物用8%非变性聚丙烯酰胺凝胶电泳结合goldview显色进行检测。如果是SNP位点,送到Introvigen上海生物技术有限公司测序。
1.3 数据的统计分析
数据录入到Office2010, 通过R3.1.14软件的线性模型筛选多态位点。采用Natural and Orthogonal InterActions(NOIA)模型计算已筛选位点的上位效应对体重的影响, 并对这些个位点影响体重的遗传方差进行剖分, 使用的是R软件包noia[18—20]。

表 1 用于分子标记筛选的基因及微卫星引物序列Tab. 1 Primers of different genes and microsatellite used to explore the candidate molecular locus
2 结果
2.1 本文所使用多态性位点的筛选结果
将线粒体基因COⅠ、D-loop以及IGF (Insulin like growth factor, IGF)家族基因IGF2R、IGF2a基因进行分段克隆、测序、寻找潜在的SNPs; 微卫星引物是通过PCR扩增、电泳分析并统计基因型, 然后将SNPs和微卫星基因型与生长性状进行关联统计, 获得对体重有显著影响的多态位点(图 1), 其描述的不同纯合基因型之间存在显著差异, 然后统计基因型频率(表 2)。而测序的SNPs位点必须经过测序峰图进行验证, 保证其准确性。
2.2 HLJ13、MFW4、MFW7、MFW11、Koi42、IGF2a4#、IGF2R第一内含子、COⅠ和D-Loop基因生长优势基因型的富集性个体筛选
对所有的具有生长优势的各个标记基因型进行亲本的富集性检测(表 3)。可以看出, 获得具有超过2个优势位点的候选亲本共13尾。然后对富集优势的候选亲本进行生长性能标记效果检测(图2)。可以看出, 富集优势分子标记基因型的候选亲本生长性能优势明显。
2.3 所筛选位点对体重的贡献率
为了研究这些个位点对体重的贡献率, 通过多元逐步回归分析, 利用AIC最佳模型筛选到COⅠ626、D-Loop 253和Koi42共3个分子标记。首先, 把性别和这3个分子标记作为自变量, 发现它们的贡献率从大到小分别是性别(9.153381e-01)、D-Loop 253 (8.481305e-04)、Koi42 (9.883973e-05)、COⅠ626 (7.514281e-05), 而且3个分子标记只有DLOOP253达到显著水平 。当去掉性别时, 发现3个位点的贡献率依大小顺序为: Koi42 (0.105199624)、D-Loop (0.022124762)、COⅠ626 (0.005664277)。综合这2个模型(有无性别作自变量)以及贡献率的大小可以推断Koi42位点可能与性别有一定的关联。经过Fisher精确性检验, 发现二者在所研究群体中确实存在关联。
2.4 所筛选分子标记的上位效应和遗传组分剖分
本文针对所筛选分子标记作遗传分析发现: 3个分子标记均存在加性效应, Koi42可以检测到显性效应, 检测到多个上位效应组分, 仅有D-Loop 253和Koi42的加性效应间的互作达到显著水平(表4)。对遗传方差组分进行剖分发现, 加性方差占11.4%, 两个位点的加性效应构成的上位效应占到77.5%, 3个位点加性效应构成的上位效应占到11.1%, 因此这3个分子标记对体重的影响主要以上位效应为主, 而且是两两加性效应的占比较大, 可以推断出D-Loop 253和Koi42两个分子标记的上位效应起主要作用。
3 讨论
3.1 富集分子标记亲本的选择
如前所述, 分子辅助育种在鲤中已经成功使用。对于富集SNPs的个体选择, 李红霞等[21]检测了900尾建鲤的鸟氨酸脱羧酶(Ornithine decarboxylase, ODC) jlODC1a基因上6个和jlODC1b基因上4个SNP位点, 发现了7个SNPs与建鲤增重显性相关, 富集4个的平均增重显著快于富集0—3的个体增重, 且比0标记的快约14%。这与本文中富集优势位点的亲本较未进行分子标记筛选的亲本具有较高的增重是一致的。

图 1 供筛选微卫星的引物和基因SNPs与体重的关联分析Fig. 1 Correlated analysis between candidate genotype of microsatellite and functional gene SNPs and body weightA. 表示COⅠ基因的第626位置出现的SNP位点不同基因型之间体重差异显著; B. 表示D-Loop基因的第253位置出现的SNP位点不同基因型之间体重差异显著; C. 表示IGF2a基因的设计的4#引物扩增出的SNP位点不同基因型之间体长体重比值差异显著; D. 表示IGF2R基因的第1内含子检测到的SNP位点不同基因型之间体重差异显著; 以上4个基因都是对亲本群体的检测效果, E. Koi42微卫星引物是对子代的基因型检测, 做分析时, 做了对数转换, 保证体重数据服从正态分布A. the significant body weight difference between two genotypes of 626thposition in COⅠ; B. the significant body weight difference between two genotypes of 253thposition in D-Loop; C. the significant body length/body weight difference between two genotypes of IGF2a4#primers amplified product; D. the significant body weight difference between two genotypes of IGF2R intron 1 amplified product; Such four genes SNPs were used to observe the genotype of parent individuals, while Koi42 in this graph is used to explore the genotype of offspring. Vertical line showed the logical bodyweight in order to conform the Gaussian distribution
为了弄清楚这些分子标记是通过什么样的遗传效应影响目标性状的, 在本研究中, 通过多元逐步回归进一步筛选SNPs位点, 得到3个多态位点, 这提示此3个位点可用于黄河鲤新品系的选育。深入分析发现Koi42位点加性效应接近显著水平, 贡献率最大, 因此该位点对于黄河鲤选育群体的育种值的计算有更为重要的作用, 可赋予较大的权重。有趣的是该位点与性别存在着一定的关联(Fisher精确检验), 这意味着对于性成熟较晚的鲤, 可用该位点进行早期选择(获取雌鱼信息)。尽管本文样本量已经达到450尾鱼, 但是具体的关联程度还需扩大样本量检测。
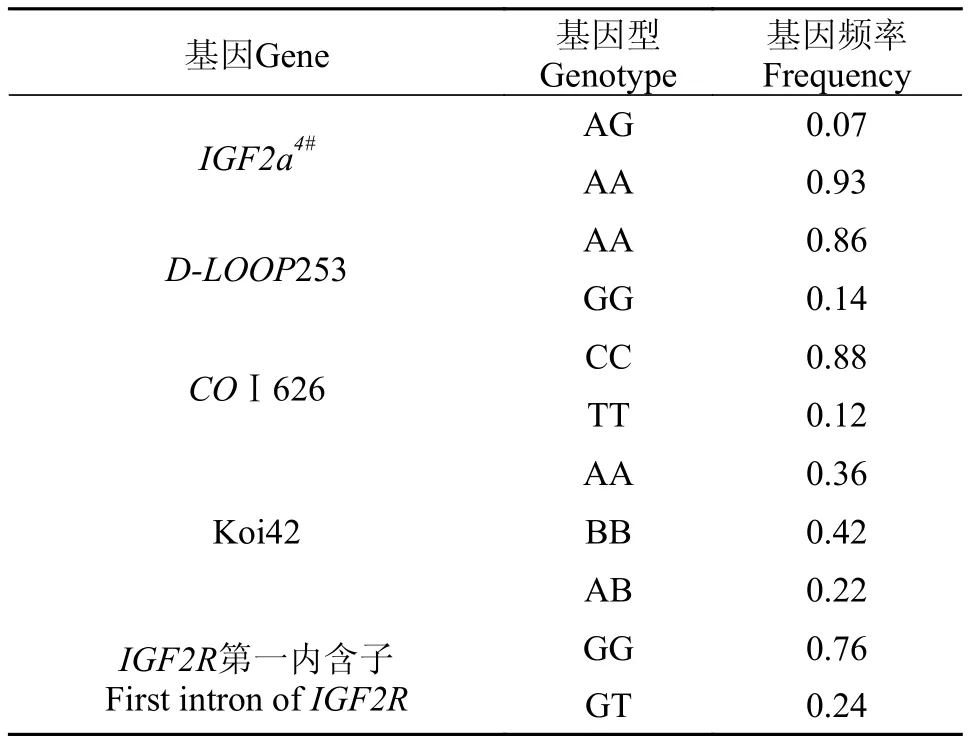
表 2 所筛选的与生长相关不同基因或微卫星引物所对应的基因型及频率Tab. 2 Genotypes and their frequency of different genes or microsatellite
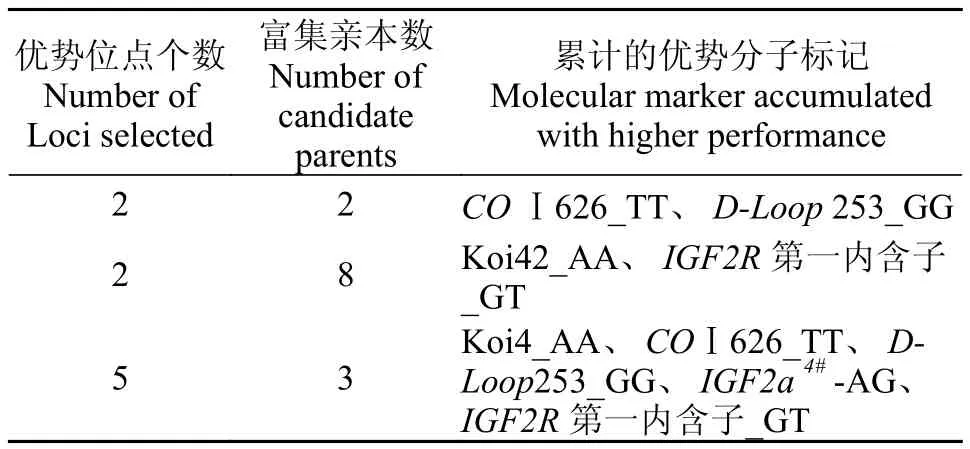
表 3 黄河鲤亲本的多态性位点富集情况Tab. 3 Polymorphism explored enrichment in the Huanghe carp candidate parents
3.2 不同位点间的上位效应
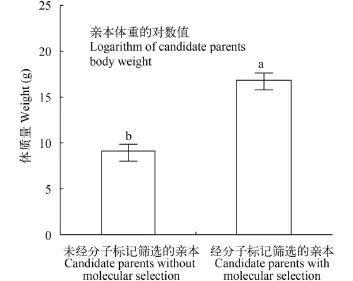
图 2 经过分子标记筛选到的亲本具有显著的体重差异(数据进行了正态分布检验, 做了对数转换)Fig. 2 Significant body weight difference was observed between candidate parents with selected loci and others without these loci (Vertical line showed the logical bodyweight in order to conform the Gaussian distribution)

表 4 所筛选3个位点的遗传效应分析Tab. 4 Genetic effect analysis of selected 3 loci
1918年Fisher从群体水平上提出上位效应是指不同位点上基因的相互作用, 其效应值是对单位点简单加性效应的偏离值[22]。随后的研究表明上位效应为功能基因间的表达调控网络[23]。本研究发现加性效应间、加性效应和显性效应间的多种上位效应, 但仅有D-Loop 253和Koi42的加性效应间的互作达到显著水平, 因此, 二者富集型个体的留种也是对该上位效应的选择, 也可能是黄河鲤新品系体重基因调控网络模式[24]之一。户国等[25]研究发现对肉鸡7周龄腹脂率有显著影响的载脂蛋白B基因T123G位点和解偶联蛋白基因C1197A位点存在基因间的上位效应组分, 并认为这种遗传互作模式可能是影响脂肪性状的重要因素。因此, 对目标性状有显著影响的多个分子标记的富集选择是一种有效的育种手段。
[1]Jonas E, de Koning D. Genomic selection needs to be carefully assessed to meet specific requirements in livestock breeding programs [J]. Frontiers in Genetics, 2015, 6: 49—56
[2]Vandeputte M, Kocour M, Mauger S, et al. Heritability estimates for growth-related traits using microsatell ite parentage assignment in juvenile common carp (Cyprinus carpio L.) [J]. Aquaculture, 2004, 235(1—4): 223—236
[3]Kocour M, Mauger S, Rodina M, et al. Heritability estimates for processing and quality traits in common carp (Cyprinus carpio L.) using a molecular pedigree [J]. Aquaculture, 2007, 270(1—4): 43—50
[4]Vandeputte M, Kocour M, Mauger S, et al. Genetic variation for growth at one and two summers of age in the common carp (Cyprinus carpio L.): Heritability estimates and response to selection [J]. Aquaculture, 2008, 277(1—2): 7—13
[5]Vandeputtea M, Rossignol M N, Pincent C. From theory to practice: Empirical evaluation of the assignment power of marker sets for pedigree analysis in fish breeding [J]. Aquaculture, 2011, 314(1—4): 80—86
[6]Cheng L, Liu L, Yu X, et al. A linkage map of common carp (Cyprinus carpio L.) based on AFLP and microsatellite markers [J]. Animal Genetics, 2010, 41(2): 191—198
[7]Zhang L, Zhang Y, Zheng X, et al. A consensus linkage map provides insights on genome character and evolution in common carp (Cyprinus carpio L.) [J]. Marine Biotechnology, 2013, 15(3): 275—312
[8]Zhao L, Zhang Y, Ji P, et al. A dense genetic linkage map for common carp and its integration with a BAC-based physical map [J]. PloS One, 2013, 8(5): e63928
[9]Xu J, Ji P, Zhao Z, et al. Genome-wide SNP discovery from transcriptome of four common carp strains [J]. PloS One, 2012, 7(10): e48140
[10]Xu J, Zhao Z, Zhang X, et al. Development and evaluation of the first hight-throughput SNP array for common carp (Cyprinus carpio L.)[J]. BMC Genomics, 2014, 15: 307
[11]Zhang Y, Xu P, Lu C, et al. Genetic linkage mapping and analysis of muscle fiber-related QTLs in common carp (Cyprinus carpio L.) [J]. Marine Biotechnology, 2011, 13(3): 376—392
[12]Sonesson A K, Meuwissen THE. Testing strategies for genomic selection in aquaculture breeding programs [J]. Genetics Selection Evolution, 2009, 41: 37
[13]Zhang W Y, Cheng J Q, Zhu J, et al. Epistasis and its application in genetics and breeding [J]. China Journal of Bioinformatics, 2004, 2(2): 39—41, 50 [张文英, 程君奇,朱军, 等. 上位性及其在遗传育种研究中的应用. 生物信息学, 2004, 2(2): 39—41, 50]
[14]David L, Rajasekaran P, Fang J, et al. Polymorphism in ornamental and common carp strains (Cyprinus carpio L.) as revealed by AFLP analysis and a new set of microsatellite markers [J]. Molecular Genetics & Genomics, 2001, 266(3): 353—362
[15]Dong Z J, Su S Y, Zhu W B, et al. Polymorphism analysis of the intron one of insulin-like growth factor 2 receptor gene in FFRC strain common carp [J]. Genetics and Molecular Research, 2015, 14(1): 407—418
[16]Crooijmans RPMA, Bierbooms VAF, Komen J, et al. Microsatellite markers in common carp (Cyprinus carpio L.) [J]. Animal Genetics, 1997, 28(2): 129—134
[17]Wei D W, Lou Y D, Sun X W, et al. Isolation of microsatellite markers in the common carp (Cyprinus carpio) [J]. Zoological Research, 2001, 22(3): 238—241 [魏东旺, 楼允东, 孙效文, 等. 鲤鱼微卫星分子标记的筛选.动物学研究, 2011, 22(3): 238—241]
[18]Alvarez-Castro J M, Carlorg O. A unified model for functional and statistical epistasis and its application in quantitative trait loci analysis [J]. Genetics, 2007, 176(2): 1151—1167
[19]Le Rouzic A, Alvarez-Castro JM. Estimation of genetic effects and genotype-phenotype maps [J]. Evolutionary Bioinformatics Online, 2008, 4: 225—235
[20]Alvarez-Castro J M, Le Rouzic A, Carlborg O. How to perform meaningful estimates of genetic effects [J]. PloS Genetics, 2008, 4(5): e1000062
[21]Li H X, Li J L, Tang Y K, et al. Correlation analysis between body weight gain and odc1 genotypes in Cyprinus carpio var. Jian [J]. Acta Hydrobiologica Sinica, 2014, 38(3): 414—421 [李红霞, 李健林, 唐永凯, 等.建鲤ODC1基因型与增重的相关性分析. 水生生物学报, 2014, 38(3): 414—421]
[22]Philips P C. Epistasis: the effestial role of gene interactions in the structure and evolution of genetic systems [J]. Nature Reviews Genetics, 2008, 9(11): 855—867
[23]Gjuvsland A B, Hayes B J, Omholt S W, et al. Statistical epistasis is a generic feature of gene regulatory networks [J]. Genetics, 2007, 175(1): 411—420
[24]Huang W, Richards S, Carbone M A, et al. Epistasis dominates the genetic architecture of Drosophila quantitative traits [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): 15553—15559
[25]Hu G, Wang S Z, Zhang S, et al. Genetic analysis of epistatic effects between ApoB and UCP on abdominal fat trait in chicken [J]. Hereditas, 2010, 32(1): 59—66 [户国,王守志, 张森, 等. ApoB与UCP基因间上位效应对鸡腹脂性状影响的遗传学分析. 遗传, 2009, 31(1): 1—8]
CANDIDATE PARENTS SELECTION CONTAINING MULTIPLE GENOTYPES WITH HIGHER PERFORMANCE AND CORRELATED MOLECULAR MARKERS’ GENETIC EFFECTS ANALYSIS
ZHANG Cheng-Feng1, SU Sheng-Yan1, ZHU Jian1and SHI Lian-Yu2
(1. Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Ministry of Agriculture, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China; 2. Key Laboratory of Freshwater Aquatic Biotechnology and Breeding, Ministry of Agriculture, Heilongjiang Fisheries Research Institute, Chinese Academy of Fishery Sciences, Harbin 150070, China)
Marker assistant breeding, molecular breeding and genome wide breeding can shorten the generation interval of germplasm improvement and creation, speed up the selection response. Contrast with traditional best linear unbiased prediction (BLUP), they can achieve the higher genetic gain with the higher cost. Practically, together with BLUP, pre-selection of candidate individuals, low density array, they will become an optional choice under the consideration of selection cost and efficiency. So, in the present article, we focus on the effect of limited markers enrichment on the candidate population growth performance.e.g. body weight. For the body weight phenotype can not only be determined by one genotype, but also by multiple loci additive effect, dominant effect and epitasis. Thus, multiple loci explored needed to do variance components analysis and identify the specific genetic effect of these loci. A total of 450 fish (parents and their offspring) were collected, which were from Nanquan farm in Freshwater Fisheries Research Center in Chinese Fishery Academy. A total of 5 microstallites primers and 4 primers used for functional gene regional amplification were selected to observe the corresponded genotypes of such individuals. Correlation analysis between effective molecular markers and body weight of Huanghe carp new strain and genetic effect analysis of such markers were examined too in the present paper. The result showed that, microstallite Koi42 and 4 SNPs from the functional genes have significant effect on the carp's body weight. 13 candidate parents which have over two genotypes with higher body weight were developed. These individuals have better growth performance compared to that without these perfect genotypes. Further analysis by multiple step-wise regressions was conducted in order to make sure which markers can be implicated in practical breeding together effectively. CO626, D-Loop 253 and Koi42 were left by both linear regression analysis and AIC criteria, where Koi42 has the highest contribution to the body weight variation among 3 markers. It is that Koi42 genotypes is related with sex by Fisher exact test. Genetic analysis supply the information Koi42 plays its role by additive effect (P<0.1). Among many epitasis components, significant effect between D-Loop 253 additive effect and Koi42 additive effect was found. Through variance components partition analysis, additive variance account for 11.4%, and effect of epitasis between two loci had the higher percentage (77.5%). This told us 3 mar-kers listed above contribute to the body weight mainly by epitasis. All of these results illustrated that Koi42 with its interaction partner D-Loop 253 could be used to marker assistant breeding or multiple markers based co-selection.
Huanghe carp new strain; Genotype; Body weight; Correlation analysis; Epitasis
Q347; S962.1
A
1000-3207(2017)01-0079-07
10.7541/2017.10
2016-01-24;
2016-04-11
农业部淡水水产生物技术与遗传育种重点实验室开放课题(FBB201401); “十二五”科技支撑计划“大宗淡水主养鱼类新品种选育”(2012BAD26B02); 中国水产科学研究院基本科研业务费(2016RC-LX03)资助 [Supported by the Open Project of Key Laboratory of Freshwater Aquatic Biotechnology and Breeding in Ministry of Agriculture (FBB201401); the Key Projects in the National Science & Technology Pillar Program during the Twelfth Five-Year Plan Period (2012BAD26B02); Special Scientific Research Funds for Central Non-profit Institutes, Chinese Academy of Fishery Sciences (2016RC-LX03)]
张成锋(1979—), 男, 山东德州人; 博士研究生; 研究方向为水产养殖。E-mail: zhangcf@ffrc.cn
石连玉(1960—), 男, 研究员; 研究方向为水产育种。Tel: 0451-84861311, E-mail: sly2552@aliyun.com

