豆科植物查尔酮合成酶基因密码子偏好性分析
张家琪,徐 悦,李晓冰,李思远,尹哲宁,周嘉裕,廖 海
(西南交通大学 生命科学与工程学院,四川 成都 610031)
豆科植物查尔酮合成酶基因密码子偏好性分析
张家琪,徐 悦,李晓冰,李思远,尹哲宁,周嘉裕,廖 海
(西南交通大学 生命科学与工程学院,四川 成都 610031)
查尔酮合成酶(Chalcone synthase,CHS)是参与合成植物苯丙烷类物质的主要酶类之一。研究豆科植物CHS基因密码子的偏好性,对提高豆科植物CHS基因的表达水平具有重要意义。为此,运用CHIPS、CUSP和CodonW程序分析豆科植物CHS基因密码子的偏好性,并用决明CHS基因对分析结果的可信度进行验证。结果表明,在豆科植物CHS基因的密码子中,以A或U结尾的密码子偏好性较强。基于CHS基因RSCU值的聚类结果显示,豆科植物与茄科植物(包括烟草)聚为一类,表明烟草作为研究豆科植物CHS基因功能的模式植物较为合适。通过对决明CHS基因密码子偏好性与大肠杆菌和酵母基因组密码子偏好性的比较,发现两者均存在差异,但酵母的差异低于大肠杆菌,表明酵母表达系统更加适合作为决明CHS基因的外源表达系统。然而,若要使决明CHS基因能够在酵母表达系统中高效表达,仍需对其密码子进行优化。
豆科植物;查尔酮合成酶;密码子偏好性;聚类分析
查尔酮合成酶(Chalcone synthase,CHS)是合成植物苯丙烷类物质的关键酶,能够催化丙二酰辅酶A和香豆酰辅酶A结合生成黄酮类化合物-查尔酮[1]。查尔酮是类黄酮物质的基本碳架结构,与类黄酮、异黄酮、花色素苷等物质的合成有关,参与植物多种生理活动,比如色素合成[2]、防紫外辐射[3]、花粉的正常发育[4]、抗病[5]等。豆科植物是类黄酮、异黄酮、花色素苷的重要来源,目前,已从苜蓿、大豆等多种豆科植物中克隆获得了CHS基因,这为以CHS基因为靶点的代谢调控研究奠定了基础。
自然界中所有的蛋白质都由氨基酸组成,而密码子的种类又直接影响到氨基酸的种类。在所有的密码子中用来编码氨基酸的有61种,但氨基酸的种类却只有20种,所以存在同义密码子(Synonymous codon),即不同密码子编码同一种氨基酸的现象[6-7]。基因对同义密码子的使用并不随机,而是具有偏好性[8]。研究外源基因密码子的偏好性,有利于外源基因对宿主表达系统的选择。如果外源基因密码子使用频率同宿主表达系统基因组密码子使用频率存在较大差异,会影响外源基因在宿主中的表达和翻译[9]。因此,研究密码子的偏好性对探究基因的表达有重要意义。
本试验以14种豆科植物的CHS基因编码序列为试材,借助CodonW和EMBOSS在线分析程序,分析其密码子的偏好性。利用SPSS 19.0软件进行聚类分析,以确定研究该基因的最佳模式植物,并比较豆科植物CHS基因密码子同大肠杆菌、酵母基因组密码子的偏好性,确定其最适宿主表达系统。
1 材料和方法
1.1 材料及分析软件
本试验所采用的15种豆科植物、3种茄科植物和4种十字花科植物的CHS基因序列(表1,2)均来自NCBI数据库(http://www.ncbi.nlm.nih.gov/)。大肠杆菌与酵母基因组的密码子偏好性数据来源于Codon Usage Database(http://www.kazusa.or.jp/codon/)。采用EMBOSS在线程序(http://emboss.toulouse.inra.fr/)中的CUSP(Create a codon usage table)和CHIPS(Codon heterozygosity in a protein coding sequence)以及CodonW程序分析密码子的偏好性,并通过SPSS 19.0软件对CHS基因密码子使用偏好性进行聚类分析。

表1 15种豆科植物CHS基因序列Tab.1 The CHS gene sequence from 15 species of leguminous plants

表2 茄科和十字花科植物的CHS基因序列Tab.2 The CHS gene sequence of solanacous and cruciferous plants
1.2 试验方法
1.2.1CHS基因同义密码子偏好性分析 首先,通过CodonW程序分析CHS基因的相对密码子使用度(Relative synonymous codon usage,RSCU)、密码子适应指数(Codon adapation index,CAI)、同义密码子第3位碱基的GC含量(GC3s)以及同义密码子GC含量。其次,根据所得到的CHS基因编码序列,利用CUSP和CHIPS在线分析程序,计算决明CHS基因密码子出现频率(以1/1000表示)以及有效密码子数(Effective number of codons,ENc)。
1.2.2 基于密码子偏好性的聚类分析 为确定研究豆科植物CHS基因的最佳模式植物,利用SPSS 19.0分析软件并基于密码子使用偏好性对21种植物(14种豆科植物、3种茄科、4种十字花科)的CHS基因进行聚类分析。在聚类过程中,将每一个被分析的物种作为聚类的对象,59种密码子(不考虑密码子AUG、UGG以及3个终止密码子)的RSCU值作为聚类的变量[10-11]。
1.2.3 决明CHS基因密码子偏好性分析 选取豆科植物决明CHS基因全长序列,利用CodonW和CUSP程序分析RSCU和密码子使用频率。并将上述结果同大肠杆菌和酵母基因组密码子的偏好性进行比较,以确定能使决明CHS基因高效表达的宿主表达系统。
2 结果与分析
2.1CHS基因的密码子偏好性分析
相对同义密码子使用度(RSCU)可作为判定密码子使用偏好性程度的标准,代表着编码对应氨基酸的同义密码子中某一密码子的相对使用概率[12]。若某一密码子的RSCU值为1,则表明该密码子的使用没有偏好性。当RSCU值大于1,则表明该密码子的使用频率相对较高。反之,当RSCU值小于1时,表示该密码子的使用频率相对较低[13]。
14种豆科植物CHS基因RSCU计算结果(表3)表明,有10个密码子偏好性较强,包括GCA、AGU、AUU、CUU、CCU、GUU(除台湾相思树外)、ACU(除苜蓿外)、CGU、UUG、GUG,其中有7个密码子以U结尾。可见豆科植物CHS基因密码子偏好于U结尾。除此之外,CGG、ACG、CCG等8个密码子的偏好性极低,RSCU均小于1。AUG和UGG的RSCU值均等于1,不具有偏好性。其中,CGU、UUG、GUG密码子的RSCU值均大于1,可推断这3个密码子是其最优密码子。

表3 14种豆科植物CHS基因的RSCUTab.3 The RSCU of CHS gene of 14 leguminous plant species

表3(续)
注:1.乌豆;2.大豆;3.胀果甘草;4.黄芪;5.台湾相思树;6.苜蓿;7.绿豆;8.红豆草;9.白羽扇豆;10.翅果铁刀木;11.白葛根;12.三野葛;13.乌拉甘草;14.蝶豆。大于1的RSCU值用下划线表示。表5同。
Note:1.Glycinesoja;2.Glycinemax;3.Glycyrrhizainflata;4.Astragalusmembranaceus;5.Acaciaconfuse;6.Medicagosativa;7.Vignaradiate;8.Onobrychisviciifolia;9.Lupinesalbus;10.Sennaalata;11.Puerariacandollei;12.Puerariamontana;13.Glycyrrhizauralensis;14.Clitoriaternatea.The data with underline mean that the value of RSCU>1.The same as Tab.5.
2.2 ENc、CAI、GC3s及GC含量分析
有效密码子数(Effective number of codons,ENc)能有效衡量密码子家族中同义密码子非均衡使用的偏好性程度。其值为20~61,值越接近20,表明密码子偏好性越强[14]。GC3s 则表示密码子的第3位碱基中G+C的含量与第3位碱基总量的比值。 CAI则指实际编码该蛋白的所有密码子对于完全使用最优密码子编码该蛋白情况下的适应性指数,常用来衡量基因表达水平的高低[15]。其取值为0~1,CAI值越接近0,表示密码子偏好程度越低[16]。
表4结果表明,14种豆科植物CHS基因的ENc值为42.76~54.52,平均值为49.97,标准偏差为2.809。根据Jiang等[17]得出的密码子偏好性强弱区分标准(ENC 取值35),可判定大部分豆科植物CHS基因密码子偏好性较低。14种豆科植物CHS基因的GC3s值为0.346~0.760,平均值为0.449,标准偏差为0.104,进一步表明同义密码子使用方式的均一性。从14种豆科植物CHS基因GC含量可看出,台湾相思树GC含量为0.584,表明其CHS基因编码区序列G+C含量多于A+T,并且密码子偏好于G/C结尾。其余13种豆科植物的GC含量均小于0.5,表明在CHS基因编码区序列中A+T含量大于G+C,且其密码子偏好以A/U结尾。
一般认为基因的表达水平与偏好性呈正相关,因而基因的密码子偏好程度常作为该基因表达水平的标志[18]。从表4的结果来看,CAI的变动为0.215~0.263,平均值为0.240,标准偏差为0.016,CAI值明显偏低,豆科植物CHS基因的密码子偏好性较低。由此,可以判定在豆科植物中CHS基因表达水平较低,查尔酮合成酶含量低,在一定程度上影响类黄酮等次生代谢产物的累积。
2.3 密码子偏好性聚类分析
在植物基因的表达与功能研究中,拟南芥和烟草是最为常见的模式植物。为确定CHS基因能够在何种模式植物中高效表达,有必要进行CHS基因密码子偏好性的聚类分析。选取14种豆科植物、3种茄科植物(包括烟草)和4种十字花科植物(包括拟南芥)进行聚类分析。从聚类结果可看出(图1),除乌拉甘草外,其他物种整体分为两大类。豆科植物除台湾相思树外,其余12种和茄科植物聚为1类。在一定意义上说明豆科植物CHS基因和茄科植物CHS基因的密码子使用偏好性相近。也就是说,烟草作为受体模式生物研究豆科植物CHS基因更具有优势。

表4 九个物种CHS基因的ENc、CAI、GC3s和GCTab.4 ENc values and CAI and GC3s and contents of GC for CHS gene in nine species

图1 21种植物CHS基因的聚类树状图Fig.1 Cluster dendrogram of CHS gene from 21 plant species
2.4 决明CHS基因密码子偏好性分析
决明CHS基因全长序列由西南交通大学中药资源与生物技术实验室克隆获得,分析其RSCU和密码子使用频率,从而验证密码子偏好性分析结果的可信度。
程序分析结果显示(表5),在决明CHS基因密码子中,有30个密码子的RSCU值大于1。其中,GCA、GCU、UGU、GAU、UUU、GGA、GGU、AUU、CUU、CCA、CCU、CAA、AGA、CGU、UCA、ACA、ACU共17个密码子以A/U结尾,且比例值也比较大,可认为是决明CHS基因的偏好性密码子。同时,应用CHIPS和CodonW分别计算决明CHS基因的ENc值、CAI值、GC含量和GC3s值,其结果分别为52.550,0.202,0.473和0.437,可见ENc值偏大,CAI值较小,GC含量和GC3s值都偏低,表明决明CHS基因密码子偏好以A/U结尾,但在总体上密码子偏好性较弱。此结果说明决明CHS基因表达水平低,需进行密码子的改造以提高基因表达水平。当然,这一结果也与密码子偏好性分析结果相一致,证明豆科植物CHS基因密码子偏好性分析具有一定的可信度。

表5 CUSP和CodonW程序分析决明CHS基因的密码子偏好性Tab.5 Codon bias of CHS gene of Senna tora analyzed by CUSP and CodonW program

表5(续)
注:*.终止密码子。表6同。
Note:*.Terminate codons.The same as Tab.6.
2.5 决明CHS基因组与酵母、大肠杆菌的基因组密码子偏好性比较
在基因的表达研究中,具有代表性的原核表达系统和真核表达系统分别是大肠杆菌和酵母。为确定决明CHS基因最适表达系统,实现CHS基因的高效表达,有必要将决明CHS基因密码子的偏好性与目标宿主基因组密码子偏好性进行比较。若密码子使用频率的比值为0.5~2.0表示密码子的偏好性接近,反之则意味密码子偏好性差别偏大[19]。如表6所示,决明CHS基因中分别有25,23个密码子的使用频率与大肠杆菌及酵母菌基因组密码子存在差异,表明酵母表达系统更加适合作为决明CHS基因的外源表达系统。但决明CHS基因密码子使用频率与酵母基因组仍有较大差别,若要使决明CHS基因能够高效表达,需要对其密码子进行优化。

表6 决明CHS基因与大肠杆菌基因组和酵母基因组的密码子出现频率Tab.6 Codon frequency of Senna tora CHS gene,and E.coli genome and yeast genome f/‰
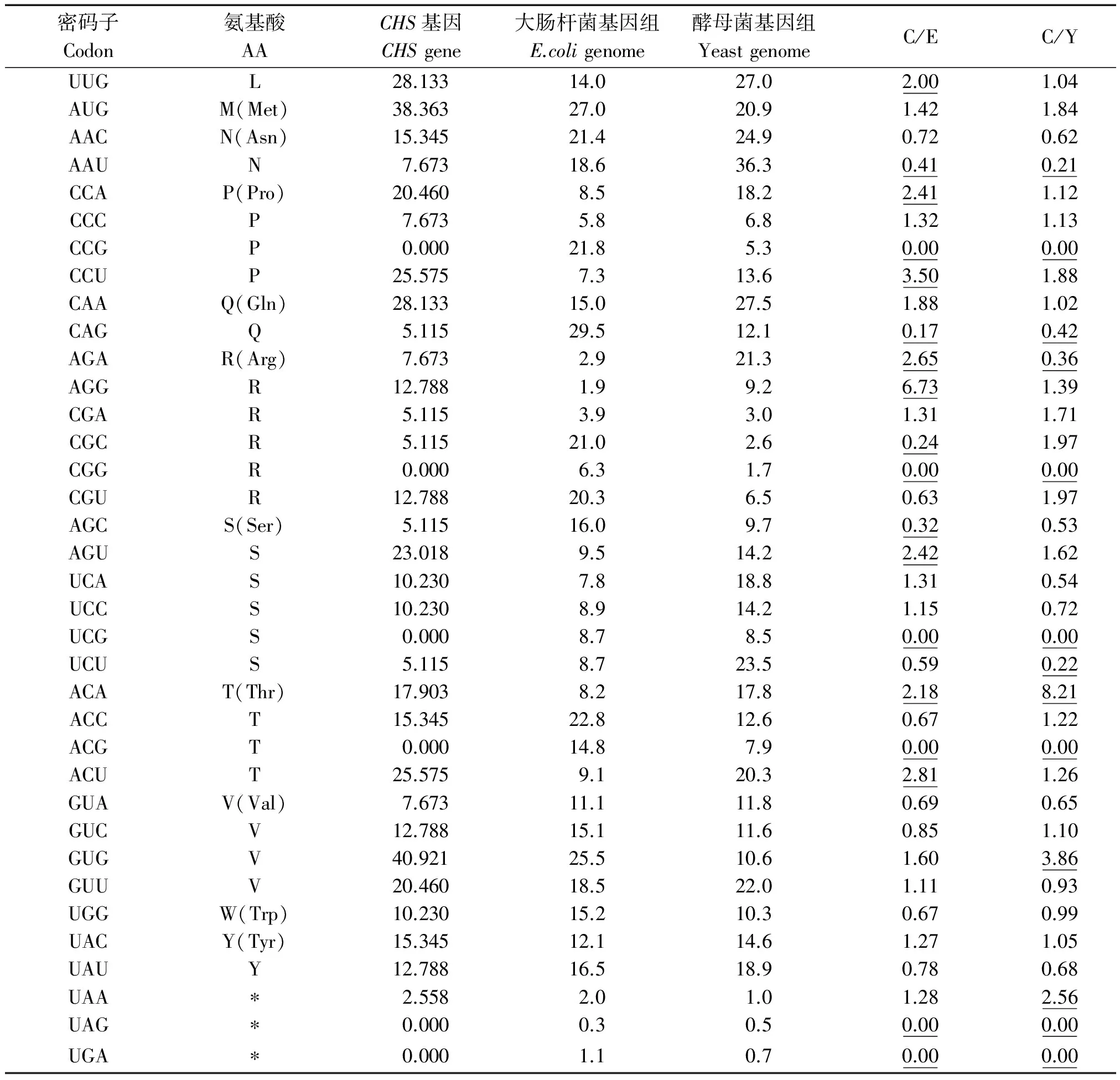
表6(续)
注:下划线表示2个物种密码子比较具有明显偏差(≤0.5,≥2)的分值。
Note:The data underlined meant there are obvious differences of values(≤0.5,≥2) between the codons of two species.
3 讨论
本试验使用相关在线分析软件和程序对豆科植物CHS基因密码子偏好性进行分析,分析结果显示,14种豆科植物CHS基因的ENc值、CAI和GC3s的平均值分别是49.97,0.240,0.449。从上述结果可看出,豆科植物CHS基因ENc值偏大,可判定其密码子偏好性较低;CAI值明显偏低,表明豆科植物CHS基因表达水平较低;GC3s小于0.5,表明了豆科植物CHS基因同义密码子使用方式的均一性;从GC含量来看除台湾相思外,其余豆科植物GC含量均小于0.5,说明大部分豆科植物CHS基因CDS序列中,A+T含量大于G+C含量。CondonW计算结果显示,14种豆科植物CHS基因中有10个密码子偏好性较强,其中有8个密码子以A/U结尾。可见豆科植物CHS基因密码子偏好以A/U结尾,特别是以U结尾。有研究表明,双子叶植物和单子叶植物在密码子偏好性方面差异明显[20]。一般认为,单子叶植物偏好以G/C结尾,而双子叶植物偏好以A/U结尾。豆科植物CHS基因密码子偏好以A/U结尾,表明该基因更适合导入双子叶植物。
为确定研究豆科植物CHS基因的最佳模式植物,依据CHS基因的RSCU值对豆科、茄科、十字花科植物进行聚类分析,结果表明,豆科植物和茄科植物的CHS基因密码子偏好性相近,可确定烟草是研究CHS基因表达和功能的最佳受体模式植物。这对以后CHS基因结构和功能的研究具有重要意义。除此之外,单独对决明CHS基因密码子偏好性进行分析,其结果与豆科植物CHS基因密码子偏好性相一致,表明对豆科植物CHS基因密码子偏好性的分析是可信的。
在基因表达与功能研究中,外源基因对宿主表达系统的选择至关重要。通常选择大肠杆菌为原核表达系统,而酵母则一般作为真核表达系统。宿主表达系统确定后,要实现目的基因的高效表达,对目的基因密码子进行优化是行之有效的方法。本试验发现对于决明CHS基因来说,其密码子偏好性同酵母基因组较为接近。这就表明相较于大肠杆菌,酵母更加适合作为决明CHS基因的表达系统。但决明CHS基因密码子与酵母基因组密码子的使用频率仍存在差异,若要使决明CHS基因能够在酵母表达系统中高效表达,需要对其密码子进行优化。
[1] Yu H N,Wang L,Sun B,et al.Functional characterization of a chalcone synthase from the liverwortPlagiochasmaappendiculatum[J].Plant Cell Reports,2015,34(2):233-245.
[2] Sun W,Meng X,Liang L,et al.Molecular and biochemical analysis of chalcone synthase from freesia hybrid in flavonoid biosynthetic pathway[J].PLoS One,2015,10(3):e0119054.
[3] Dao T,Linthorst H,Verpoorte R.Chalcone synthase and its functions in plant resistance[J].Phytochemistry Reviews,2011,10(3):397-412.
[4] 蒋 明,曹家树.查尔酮合成酶基因[J].细胞生物学杂志,2007,29(4):525-529.
[5] 王 燕,许 锋,程水源.植物查尔酮合成酶分子生物学研究进展[J].河南农业科学,2007(8):5-9.
[6] 苏 惠,李永光,谭文雍,等.大肠杆菌gdhA基因的密码子偏好性分析及优化[J].基因组学与应用生物学,2015,34(3):521-529.
[7] Grantham R,Gautier C,Gouy M.Codon frequencies in 119 individual genes confirm consistent choices of degenerate bases according to genome type[J].Nucleic Acids Research,1980,8(9):1893-1912.
[8] Yang X,Ma X,Luo X,et al.Codon usage bias and determining forces inTaeniasoliumgenome[J].The Korean Journal of Parasitology,2015,53(6):689-697.
[9] 杨春亮,王 良,武 斌,等.大豆GmRAV基因的密码子偏好性分析[J].东北农业大学学报,2012,43(7):34-41.
[10] 李娟娟,黄 宇,刘祖碧,等.Kunitz蛋白酶抑制剂基因的密码子偏好性分析[J].生物技术,2014,24(5):39-44.
[11] 孙 晶,何 涛,万闰兰,等.铁皮石斛尿苷二磷酸葡萄糖焦磷酸化酶基因(UGP)的密码子偏好性分析[J].应用与环境生物学报,2014,20(5):759-766.
[12] Xu Y,Jia R,Zhang Z,et al.Analysis of synonymous codon usage pattern inDuckcircovirus[J].Gene,2015,557(2):138-145.
[13] You E,Wang Y,Ding Z T,et al.Codon usage bias analysis for the spermidine synthase gene fromCamelliasinensis(L.) O.Kuntze[J].Genetics and Molecular Research,2015,14(3):7368-7376.
[14] 李 平,白云凤,冯瑞云,等.籽粒苋苹果酸酶(NAD-ME)基因密码子偏好性分析[J].应用与环境生物学报,2011,17(1):12-17.
[15] Sharp P M,Li W H.The codon adaptation Index-a measure of directional synonymous codon usage bias,and its potential applications[J].Nucleic Acids Research,1987,15(3):1281-1295.
[16] Ma Q P,Li C,Wang J,et al.Analysis of synonymous codon usage inFAD7 genes from different plant species[J].Genetics and Molecular Research,2015,14(1):1414-1422.
[17] Jiang Y,Deng F,Wang H,et al.An extensive analysis on the global codon usage pattern ofBaculoviruses[J].Archives of Virology,2008,153(12):2273-2282.
[18] 赵 薇,陈必链,王明兹,等.藻类△6脂肪酸脱饱和酶基因密码子偏好性分析[J].热带海洋学报,2010,29(3):126-134.
[19] 赵 耀,刘汉梅,顾 勇,等.玉米Waxy基因密码子偏好性分析[J].玉米科学,2008,16(2):16-21.
[20] Murray E E,Lotzer J,Eberle M.Codon usage in plant genes[J].Nucleic Acids Research,1989,17(2):477-498.
Analysis of Codon Bias of CHS Gene in Leguminous Plant
ZHANG Jiaqi,XU Yue,LI Xiaobing,LI Siyuan,YIN Zhening,ZHOU Jiayu,LIAO Hai
(School of Life Science and Engineering,Southwest Jiaotong University,Chengdu 610031,China)
Chalcone synthase(CHS) is one of the key enzymes in the phenylaprapanoid metabolism pathways.The research about codon bias fromCHSgenes of leguminous plants has great significance to increase the expression level ofCHSgene.Therefore,codon bias ofCHSgene from leguminous plants was analyzed by CodonW,CHIPS and CUSP.Furthermore,CHSgene fromSennatorawas used to verify the reliability of analysis results.According to the results,CHSgene of leguminous plants preferred to use the synonymous codons ended with A or U.The phylogenic analysis showed that Legumes and Solanaceae were attributed to one class,indicating tobacco was the optimum model plants forCHSgenes functional study.Compared withE.coliand yeast genome,the codon usage difference betweenCHSgene ofSennatoraandE.coligenome was more apparently than that betweenCHSgene ofSennatoraand yeast genome.Hence,the yeast may be more suitable for the expression ofCHSgene ofSennatora.In order to improve the expression level ofCHSgene in the yeast expression system,CHSgene codons should be optimized.
Leguminosae;Chalcone synthase;Codon bias;Phylogenic analysis
2016-07-16
国家自然科学基金项目(31371232;31500276);成都市科技技术研发项目(2015-HM01-00051-SF);西南交通大学校级大学生创新创业训练计划资助项目(151607)
张家琪(1994-),男,甘肃定西人,主要从事生物学研究。
廖 海(1974-),男,四川遂宁人,副教授,博士,主要从事生物技术研究。 周嘉裕(1976-),女,四川宜宾人,副教授,博士,主要从事生物技术研究。
Q78
A
1000-7091(2016)06-0012-09
10.7668/hbnxb.2016.06.003

