脊尾白虾血细胞ESTs的生物信息学与微卫星序列特征分析
段亚飞, 张 喆, 李吉涛, 李 健, 刘 萍
( 1.中国水产科学研究院 南海水产研究所,农业部南海渔业资源开发利用重点实验室,广东 广州 510300;2. 中国水产科学研究院 黄海水产研究所,农业部海洋渔业可持续发展重点实验室,山东 青岛 266071;3. 青岛海洋科学与技术国家实验室,海洋渔业科学与食物产出过程功能实验室,山东 青岛 266071 )
脊尾白虾血细胞ESTs的生物信息学与微卫星序列特征分析
段亚飞1, 张 喆1, 李吉涛2, 3, 李 健2, 3, 刘 萍2, 3
( 1.中国水产科学研究院 南海水产研究所,农业部南海渔业资源开发利用重点实验室,广东 广州 510300;2. 中国水产科学研究院 黄海水产研究所,农业部海洋渔业可持续发展重点实验室,山东 青岛 266071;3. 青岛海洋科学与技术国家实验室,海洋渔业科学与食物产出过程功能实验室,山东 青岛 266071 )
对前期测序得到的脊尾白虾血细胞2853条EST序列进行了生物信息学和微卫星序列特征分析。EST序列拼接得到1053条Unigenes,包括329条Contigs和724条Singlets。BLAST分析表明,593 (56.3%)条Uingenes与数据库中已知基因具有相似性。KEGG代谢途径分析表明,181条Unigenes映射到120条代谢途径。通过EST-SSR分析,共得到416条微卫星序列,检出率为14.58%。其中,两碱基重复序列374条,占89.90%,AG重复类型最多;三碱基35条,占8.41%,AAT重复类型最多;四碱基7条,占1.68%,AAGT重复类型最多。本研究可为脊尾白虾功能基因资源挖掘及分子标记筛选提供有效数据。
脊尾白虾;血细胞;EST;生物信息学;微卫星
脊尾白虾(Exopalaemoncarinicauda)是我国黄、渤海地区重要的经济虾类,具有繁殖能力强、生长速度快和环境适应性强等优点[1-2]。近年来,脊尾白虾养殖产业发展迅速,其遗传育种研究日益受到重视。脊尾白虾基因信息和分子遗传标记的发掘,可以为其功能基因筛选、遗传多样性分析、遗传连锁图谱构建及标记辅助育种等奠定基础。
表达序列标签(EST)是指从特定组织来源的cDNA文库中随机挑取单克隆进行单向测序而得到的部分cDNA序列,代表了生物体特定组织在某一时期的表达基因[3]。表达序列标签分析可以用于新基因发现、功能基因筛选、分子标记开发和基因时空表达状况分析等[3]。微卫星标记(SSR)是水产动物遗传育种重要的分子标记,具有多态性高、共显性、通用性好、数量丰富和在基因组上分布均匀等特点[4-5]。EST-SSR可以避免传统方法构建、筛选文库的繁琐步骤和人力物力的耗费,广泛应用于水生动物新基因的分离鉴定、比较基因组学、遗传图谱构建和基因芯片制备等研究[6-7]。
近年来,许多甲壳动物已获得EST-SSRs标记,如中国明对虾(Fenneropenaeuschinensis)[8]、凡纳滨对虾(Litopenaeusvannamei)[9]、斑节对虾(Penaeusmonodon)[10]、三疣梭子蟹(Portunustrituberculatus)[11]和拟穴青蟹(Scyllaparamamosain)[12]等。而目前脊尾白虾微卫星标记仅见通过构建基因组微卫星富集文库筛选获得[13]。本研究利用前期构建的脊尾白虾血细胞全长cDNA文库,对测序获得的EST序列进行生物信息学和微卫星序列特征分析,以期为脊尾白虾功能基因资源挖掘及分子标记筛选提供有效数据。
1 材料与方法
1.1脊尾白虾血细胞EST序列测定和分析
利用前期构建的脊尾白虾血细胞全长cDNA文库[3],对其进行批量测序,引物为5′pTriPLEx2 (5′-CTCCGAGATCTGGACGAGC-3′)。测得的EST序列,利用Cross-match软件进行引物序列、载体序列及低质量序列(<100 bp)去除;利用Phrap软件对高质量ESTs进行序列拼接,拼接结果为由Contigs和Singlets组成的Unigenes。
1.2 ESTs的相似性比对、分类及代谢途径分析
利用BLAST软件对拼接到的Unigenes与NCBI核苷酸和蛋白质数据库进行相似性比对(E-value < 1e-5)。将所有的Unigenes与日本京都大学的《京都基因和基因组百科全书》(KEGG)的PATHWAY子数据库进行比较和代谢途径分析,获得脊尾白虾血细胞的ESTs的部分代谢网络信息。
1.3 ESTs的微卫星序列特征分析
利用RepeatMasker软件从拼接的Unigenes中查找微卫星序列,最小重复参数设置为重复7次以上的两碱基序列,重复5次以上的三碱基序列,重复5次以上的四碱基序列。单核苷酸重复序列未进行筛选,因为一般认为其作为多态性标记的意义不大[18]。
2 结 果
2.1 ESTs的生物信息学分析
2.1.1 ESTs序列分析
对脊尾白虾血细胞全长cDNA文库批量测序获得的EST序列,去除载体序列、引物序列和低质量载体序列(<100 bp)后,共得到2853条高质量EST序列,平均长度为493 bp(表1)。利用Phrap软件进行序列拼接,共获得1053条Unigenes,包括329条(31.24%)Contigs和724条(68.76%)Singlets。对Unigenes中的ESTs数量分析可知,Contigs由2~284条ESTs拼接而成,其中大部分是由2~5条ESTs拼接而成,10条Contigs是由多于50条的ESTs拼接而成,只有1条Contigs是由284条ESTs拼接而成(图1)。

表1 脊尾白虾血细胞ESTs基本信息
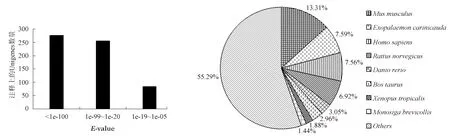
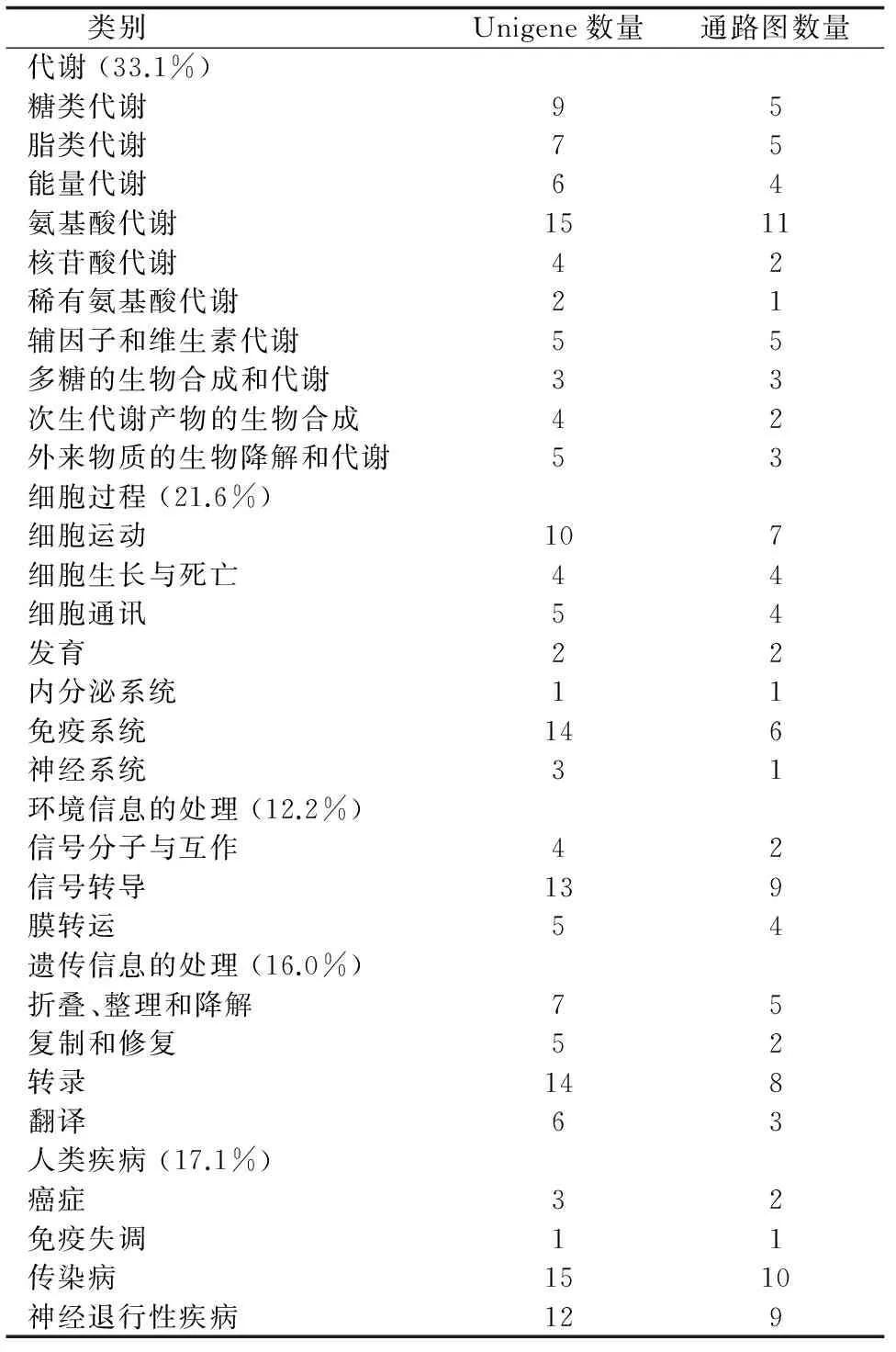
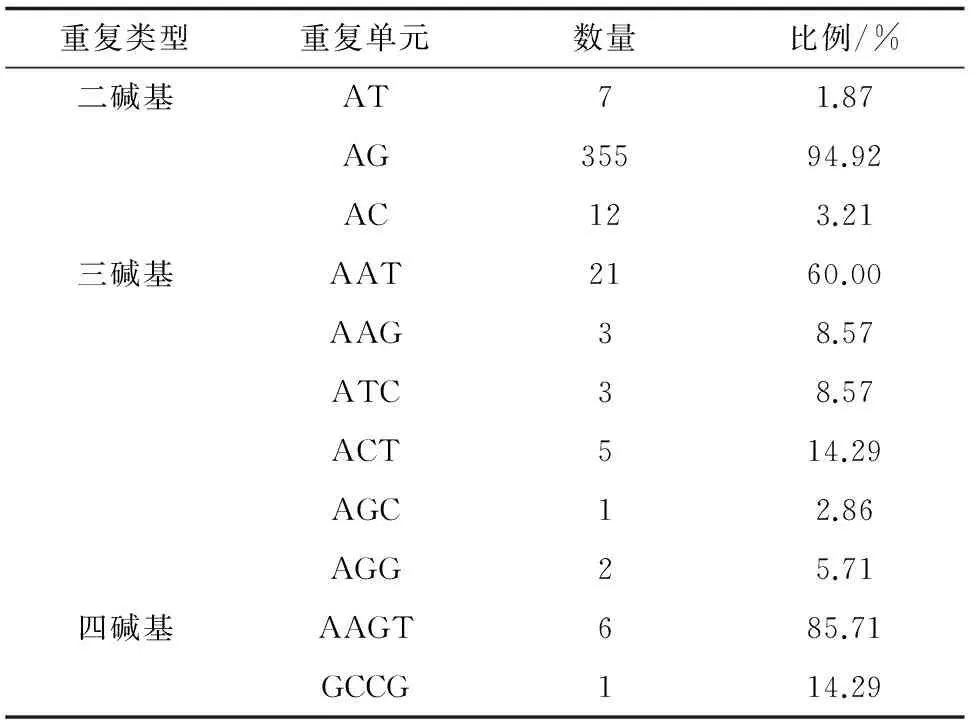
BLAST分析表明,593(56.3%)条Uingenes与数据库中已知基因具有相似性,22(2.1%)条Uingenes显示为未知基因,438(41.6%)条Uingenes没有任何相似性。ESTs相似比对的E-value分布表明,276(44.9%)条Uingenes具有高度相似性(E-value < 1e-100),256(41.6%)条Uingenes具有中度相似性(1e-99 图1 脊尾白虾血细胞ESTs与其所拼接形成的Unigene之间的关系 图2 脊尾白虾血细胞ESTs相似比对的E-value分布 图3 脊尾白虾血细胞ESTs匹配的相似物种分布 2.1.2 ESTs的代谢途径分析 脊尾白虾血细胞ESTs的代谢途径分析表明,共有181条Unigenes得到注释,相关酶类参与120条代谢途径,主要包括代谢、细胞过程、环境信息的加工、遗传信息的加工和人类疾病5类,其相关的基因序列分别占33.1%、21.6%、12.2%、16.0%和17.1%。其中,代谢途径最多的类别是氨基酸代谢(11%),其次为传染病(10%),第三为信号转导(9%)(表2)。 表2 脊尾白虾血细胞ESTs的代谢途径分析 从血细胞2853条ESTs序列中,共筛选到微卫星序列416个,占全部ESTs总数的14.58%。考虑到碱基互补配对和计数拷贝数起始碱基顺序的差异,本研究将同类重复兼并为一种重复类型代表,如AAG代表六种重复类型,分别为AAG、AGA、GAA、TTC、TCT和CTT。统计所有重复类型,以两碱基重复序列数量最多,为374条,占重复序列总数的89.90%;其次为三碱基,35个,占8.41%;再次是四碱基,7个,占1.68%(表3)。 表3 不同重复类型的重复序列数目及其百分比 同类型的SSR重复序列中,各重复拷贝类别所占的比例也各不相同。在两碱基重复类型中,AG重复拷贝类别最多,为355个,占94.92%;其次为AC和AT,分别为12(3.21%)和7(1.87%);未发现GC重复拷贝类别。三碱基重复类型中,共有6种重复拷贝类别,分别为AAT、AAG、ATC、ACT、AGC和AGG。其中,以AAT最多,为21个,占60%;其次为ACT,为5个,占14.29%;再次为AAG和ATC,这两种重复拷贝类别数量相同,为3个,占8.57%。四碱基重复类型中,共有2种重复拷贝类别,分别为AAGT和GCCG,其中以AAGT最多,为7个,占85.71%;其次为GCCG,为1个,占14.29%(表4)。 表4脊尾白虾血细胞ESTs筛选到的微卫星序列 重复类型重复单元数量比例/%二碱基AT71.87AG35594.92AC123.21三碱基AAT2160.00AAG38.57ATC38.57ACT514.29AGC12.86AGG25.71四碱基AAGT685.71GCCG114.29 通过对前期测序得到的脊尾白虾血细胞2853条ESTs进行序列拼接,共得到1053条Uingenes。BLAST分析表明,41.6%的Uingenes与美国国立生物技术信息中心数据库中的已知基因序列无相似性,与三疣梭子蟹[14]、中华绒螯蟹(Eriocheirsinensis)[15]等的研究一致。此类无相似性的ESTs可能为新基因序列,表明甲壳类尚有大量基因未被发掘;又可能为已知基因,由于虾类与其他脊椎动物的亲缘关系太远,而无法通过BLAST比对确定其相似关系。此外,Xu等[14]认为,ESTs是通过一次测序得到的序列,未必包含编码区,可能是ESTs未能注释的原因之一。因此,此类未得到注释的ESTs,有待进一步的深入研究。 KEGG数据库涵盖了物种的基因与基因组、酶促反应及其途径和各种生化物质的信息,在基因与基因组、代谢途径与代谢网络的相关研究中具有重要作用[16]。KEGG PATHWAY数据库不仅能够提供生化物质相互转化所有可能的代谢途径,还可以对催化各步反应的酶进行全面注解[16]。通过代谢途径分析,本研究共有181条Unigenes(17.2%)获得注释,发现酶类参与120条代谢途径。代谢途径分析在多个水产物种中已有相关报道。Meng等[17]对虾夷扇贝(Patinopectenyessoensis)肌肉EST序列进行KEGG代谢途径分析,共有11.3%的Unigenes得到注释,涉及103个代谢途径;其中以代谢方面的Unigenes比例最大,占30.5%,与本研究结果一致。 本研究通过对脊尾白虾血细胞ESTs进行SSR筛选,共得到416条(14.58%)SSR序列,表明脊尾白虾ESTs-SSR序列比例较高。研究表明,动物的ESTs-SSR以两碱基重复为主,大部分植物则以三碱基重复为主[18]。脊尾白虾血细胞ESTs-SSR共发现3种碱基重复类型,以两碱基重复类型最多,占89.90%;其次为三碱基重复类型(8.41%)和四碱基重复类型(1.68%)。本研究未发现五碱基和六碱基重复类型,可能与所分析的ESTs序列的组织差异有关。 两碱基重复类型中,共发现3种重复拷贝类别,其中以AG最多,其次是AC,与斑节对虾[19]、罗氏沼虾(Macrobrachiumrosenbergii)[20]和三疣梭子蟹[21]等一致。贾舒雯等[13]通过构建脊尾白虾微卫星富集文库对多态性标记进行了筛选,发现脊尾白虾基因组微卫星核心序列以AG重复最多,占88.94%;其次是AC重复,占5.03%。这与本研究两碱基重复类型结果(AG 94.92%,AC 3.21%)一致,由此说明AG重复拷贝类别在脊尾白虾基因组中含量丰富。但本研究并未发现GC重复,而GC两碱基重复在其他物种基因组中的含量均较少[22-23]。 三碱基重复类型中,共发现6种重复拷贝类别,其中以AAT最多,占60%。研究表明,中国明对虾[18]和红鳍东方鲀(Takifugurubripes)[24]中,以AAT重复最多。高焕等[18]通过分析中国明对虾基因组微卫星特征,发现三碱基重复类型中以AAT重复最多,占50.34%。四碱基重复类型中,共发现7种重复拷贝类别,总体数量较少。而本研究中未发现五碱基和六碱基重复类型,可能与所分析的ESTs序列相对较少、覆盖面较小有关。 目前脊尾白虾SSR的研究,主要是利用SSR核心序列进行探针杂交或以核心重复序列为引物进行PCR筛选,而从ESTs中筛选SSR序列的方法则更为简单,不仅能够节省人力和物力,而且由于ESTs-SSR标记位于基因编码区中,因此具有高度的物种间转移性[25]。本研究所筛选获得的SSR序列,可以用于脊尾白虾SSR标记筛选,对脊尾白虾遗传图谱、基因定位和遗传多样性分析等奠定基础。 [1] 梁俊平, 李健, 刘萍, 等. 脊尾白虾生物学特性与人工繁育的研究进展[J]. 中国农学通报, 2012,28(17):109-116. [2] 梁俊平, 李健, 李吉涛, 等. 氨氮对脊尾白虾幼虾和成虾的毒性试验[J]. 水产科学, 2012,31(9):526-529. [3] 段亚飞, 刘萍, 李吉涛, 等. 脊尾白虾血细胞全长cDNA文库的构建及EST序列分析[J]. 中国水产科学, 2013, 20(2):243-249. [4] 孙立元, 郭华阳, 朱彩艳, 等. 卵形鲳鲹育种群体遗传多样性分析[J]. 南方水产科学, 2014,10(2):67-71. [5] 杨兵, 林琳, 李纯厚, 等. 基于高通量测序的二长棘鲷微卫星标记开发与评价[J]. 南方水产科学, 2015,11(4):116-120. [6] Ji X S, Chen S L, Ma H Y, et al. Isolation and characterization of 19 EST-linked ploymorphic microsatellite loci for olive flounder (Paralichthysolivaceus)[J]. Aquaculture Research, 2009,40(8): 980-983. [7] Yu H, Li Q. Exploiting EST databases for the development and characterization of EST-SSRs in the Pacific oyster (Crassostreagigas)[J]. J Heredity,2008,99(2):208-214. [8] Wang H X, Li F H, Xiang J H. Polymorphic EST-SSR markers and their mode of inheritance inFenneropenaeuschinensis[J]. Aquaculture, 2005,249(1/4):107-114. [9] Perez F, Ortiz J,Zhinaula M, et al. Development of EST-SSR markers by data mining in three species of shrimp:Litopenaeusvannamei,Litopenaeusstylirostris, andTrachypenaeusbirdy[J]. Marine Biotechnol, 2005,7(5):554-569. [10] Maneeruttanarungroj C, Pongsomboon S, Wuthisuthimethavee S, et al. Development of polymorphic expressed sequence tag-derived microsatellites for the extension of the genetic linkage map of the black tiger shrimp (Penaeusmonodon) [J]. Animal Genetics, 2006,37(4):363-368. [11] 吕建建, 王渝, 高保全, 等. 三疣梭子蟹Ⅰ型微卫星标记的发掘及多态性分析[J]. 水产学报, 2013,37(6):816-822. [12] Zhou Z H, Zhang Z P, Wang Y L, et al. EST analysis on the gonad development related organs and microarray screen for differentially expressed genes in mature ovary and testis ofScyllaparamamosain[J]. Comp Biochem and Physiol D, 2011,6(2):150-157. [13] 贾舒雯, 刘萍, 韩智科, 等.脊尾白虾微卫星富集文库的构建与多态性标记的筛选[J]. 水产学报, 2011,35(12):1787-1794. [14] Xu Q H, Liu Y, Liu R L. Expressed sequence tags from cDNA library prepared from gills of the swimming crab,Portunustrituberculatus[J]. J Experimental Marine Biol Ecol, 2010,394(1/2):105-115. [15] Gai Y C, Wang L L, Zhao J M, et al. The construction of a cDNA library enriched for immune genes and the analysis of 7535 ESTs from Chinese mitten crabEriocheirsinensis[J]. Fish and Shellfish Immunol, 2009,27(6):684-694. [16] Priebe S, Linde J, Albrecht D, et al. FungiFun:a web-based application for functional categorization of fungal genes and proteins [J]. Fungal Genetics Biol, 2011,48(4):353-358. [17] Meng X Y, Chang Y Q, Qiu X M, et al. Generation and analysis of expressed sequence tags from adductor muscle of Japanese scallopMizuhopectenyessoensis[J]. Comp Biochem Physiol D, 2010,5(4):288-294. [18] 高焕, 刘萍, 孟宪红, 等. 中国对虾基因组微卫星特征分析[J]. 海洋与湖沼, 2004,35(5):424-431. [19] Xu Z,Dhar A K, Wyrzykowski J, et al. Identification of abundant and informative microsatellites from shrimp (Penaeusmonodon) genome [J]. Animal Genetics, 1999,30(2):150-156. [20] Charoentawee K, Poompuang S, Nakom U. Isolation and characterization of microsatellites in giant freshwater prawnMacrobrachiumrosenbergii[J]. Mol Ecol Resources, 2006,6(3):823-825. [21] 宋来鹏, 刘萍, 李健, 等. 三疣梭子蟹基因组微卫星特征分析[J]. 中国水产科学, 2008,15(5):738-743. [22] Zhang L,Bao Z, Cheng J, et al. Fosmid library construction and initial analysis of end sequences in Zhikong scallop (Chlamysfarreri) [J]. Marine Biotechnol, 2007,9(5):606-612. [23] Wang Y, Ren R, Yu Z, et al.Bioinformatic mining of EST-SSR loci in the Pacific oyster,Crassostreagigas[J]. Animal Genetics, 2008,39(3):287-289. [24] 崔建洲, 申雪艳, 杨官品, 等. 红鳍东方鲀基因组微卫星特征分析[J]. 中国海洋大学学报:自然科学版, 2006,36(2):249-254. [25] 吴文婷, 张磊, 宋志民, 等. 5个紫菜属物种丝状体的微卫星初步遗传分析[J]. 南方水产科学, 2012,8(4):29-36. BioinformaticsandMicrosatelliteSequencesAnalysisofESTSequenceinRidgeTailShrimpExopalaemoncarinicauda DUAN Yafei1, ZHANG Zhe1, LI Jitao2, 3, LI Jian2, 3, LIU Ping2, 3 ( 1. Key Laboratory of South China Sea Fishery Resources Exploitation & Utilization, Ministry of Agriculture, South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300, China; 2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China; 3. Function Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China ) The analysis of bioinformatics and microsatellite sequences characteristics of expressed sequence tag (EST) sequence were studied, basing on the hemocyte cDNA library of ridge tail shrimpExopalaemoncarinicaudaconstructed by our laboratory. The 1053 unique sequences were yielded from 2853 high quality ESTs, including 329 contigs and 724 singletons. BLAST analysis revealed 593 (56.3%) of the unique sequences as orthologs of genes from other organisms, 22 (2.1%) of which were hypothetical protein, and the remaining 438 (41.6%) had no homology to any sequences. Kyoto Encyclopedia of Genes and Genomes (KEGG) analysis indicated that 181 of the unique sequences mapped to 120 of metabolic pathways. A total of 416 (14.58%) of the microsatellite repeat sequences were found using EST-SSR analysis. In the 416 repeat sequences, most was dinucleotide with repeats of 374 (89.90%), followed by trinucleotide with repeats of 35 (8.41%), and the third tetranucleotide with repeats of 7 (1.68%). The most common repeat types were AG, AAT and AAGT repeat. The findings provide important information for screening the functional gene resource and molecular markers in ridge tail shrimp. Exopalaemoncarinicauda; hemocyte; EST; bioinformatics ; microsatellite 10.16378/j.cnki.1003-1111.2016.05.018 S917 A 1003-1111(2016)05-0562-06 2015-11-03; 2015-12-30. 国家虾产业技术体系项目(CARS-47);国家自然科学基金资助项目(31472275);山东省泰山产业领军人才工程项目(LNJY2015002);青岛海洋科学与技术国家实验室鳌山科技创新计划项目(2015ASKJ02). 段亚飞(1989—),男,助理研究员,硕士;研究方向: 虾类免疫学. E-mail: duanyafei89@163.com. 通讯作者:刘萍(1962—),女,研究员;研究方向: 海水养殖生物种质资源与遗传育种. E-mail: liuping@ysfri.ac.cn.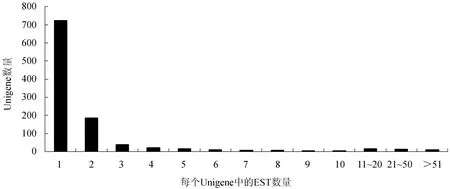
2.2 ESTs的微卫星序列分析

3 讨 论
3.1 ESTs生物信息学分析
3.2 ESTs的微卫星序列特征分析

