核壳型单磷酸腺苷分子印迹聚合物的制备
龚 霞,胡莹莹,尤祥宇,谷 晶,邓教宇,谢卫红
(1.湖北工业大学生物工程与食品学院,湖北 武汉 430068;
2.中国科学院武汉病毒研究所 中国科学院农业与环境微生物学重点实验室,湖北 武汉 430071)
核壳型单磷酸腺苷分子印迹聚合物的制备
龚 霞1,胡莹莹1,尤祥宇1,谷 晶2,邓教宇2,谢卫红1
(1.湖北工业大学生物工程与食品学院,湖北 武汉 430068;
2.中国科学院武汉病毒研究所 中国科学院农业与环境微生物学重点实验室,湖北 武汉 430071)
以聚苯乙烯微球(CP)为内核、单磷酸腺苷(AMP)为模板、丙烯酰胺(AM)为功能单体、乙二醇二甲基丙烯酸酯(EGDMA)为交联剂,制备了核壳型单磷酸腺苷分子印迹聚合物(AMP-MIP),讨论了不同洗脱方式、洗脱溶剂和聚合时间对AMP-MIP吸附性能的影响,通过扫描电镜、水中悬浮实验和吸附实验对AMP-MIP的形貌和性能进行了表征,得到最佳工艺条件:洗脱方式为超声、洗脱溶剂为甲醇-乙酸(4∶1,体积比)、聚合时间为30 min。分子印迹聚合物不仅对小分子模板有吸附作用,而且对大分子ADP-核糖基化的甘油醛-3-磷酸脱氢酶(gapA)具有识别作用,吸附量可达3.9μg·mg-1。
聚苯乙烯;单磷酸腺苷;核壳聚合;分子印迹技术;甘油醛-3-磷酸脱氢酶
ADP-核糖基化是一种蛋白质的翻译后修饰过程。研究发现,ADP-核糖基化蛋白质参与多种细胞活动[1-3],如DNA修复、转录、翻译、细胞信号传导和细胞凋亡,还与癌症、糖尿病、神经退行性疾病、心力衰竭等疾病[4-10]有关,是临床诊断的标志物。常规检测这类蛋白质主要是应用以酶或抗体为接受器的生物传感器,这类天然生物分子往往价格昂贵、处理复杂。而分子印迹聚合物(MIP)能够模拟天然生物分子的识别作用,是生物分子的潜在替代物。与天然生物分子相比,MIP在高或低的pH值、温度、压强时较稳定,制备简单,能在有机溶剂中操作,可抗生物腐败,储存时间更长,更稳定,可重复利用。
MIP的制备分为3步:(1)在合适的分散介质中,将模板分子与合适的功能单体混合,依靠官能团之间的共价键或非共价键结合形成复合物;(2)加入适当的致孔剂、交联剂和引发剂,在热或光的作用下,形成高度交联的聚合物,而在空间排列和空间定向上,功能基团被固定;(3)通过化学或物理方法,洗脱模板分子,这样就在MIP中留下一个在空间构象和功能基团上与模板分子相匹配的三维空穴。当与模板分子重新结合时,MIP对模板分子具有专一性、特异性的识别作用。分子印迹技术已在膜分离、仿生传感器[11-12]、模拟酶[13]、选择性催化、固相萃取和色谱分离等方面取得了极大的进步,并且应用于食品安全分析[14-16]、制药工业[17-20]、环境监测和疾病诊断上。
作者以带有双硫酯基团的聚苯乙烯微球(CP)为内核、单磷酸腺苷(AMP)为模板、丙烯酰胺(AM)为功能单体、乙二醇二甲基丙烯酸酯(EGDMA)为交联剂,采用可逆加成-断裂链转移自由基聚合法(RAFT)制备了核壳型单磷酸腺苷分子印迹聚合物AMP-MIP(图1),拟为大分子蛋白质的识别和富集提供一个新的策略。

图1 核壳型分子印迹聚合物的合成路线
1 实验
1.1 试剂与仪器
单磷酸腺苷(AMP)、偶氮二异丁腈(AIBN,纯度99%),上海源叶生物科技有限公司;乙二醇二甲基丙烯酸酯(EGDMA,纯度98%),Sigma公司;二乙基二硫代氨基甲酸苄酯(BDC),分析纯,武汉大学;丙烯酰胺(AM)、苯乙烯、二甲亚砜(DMSO)、乙腈、甲醇、乙酸均为分析纯,国药集团化学试剂有限公司。
JSM-6390LV型扫描电子显微镜,日本电子株式会社;UV-Lambda 35型紫外可见分光光度计,Perkin-Elmer公司;VP30型真空抽滤泵,北京莱伯泰科仪器公司;G:BOX Chemi XL1.4型凝胶成像系统,基因有限公司。
1.2 聚苯乙烯微球(CP)的制备
向圆底烧瓶中加入200 mL乙腈,向其中加入一定量的聚合单体苯乙烯、交联剂EGDMA、链转移剂BDC和引发剂AIBN,在磁力搅拌器上搅拌几分钟,再超声混匀。反应体系抽真空完毕后,置于70 ℃油浴锅中加热反应4 h后,停止加热,快速取出圆底烧瓶,浸入冰水浴冷却至室温。反应液真空抽滤,用50 mL甲醇洗涤白色粉末。反复洗涤5次后,于50 ℃真空干燥箱中干燥12 h,得带双硫酯基团的聚苯乙烯微球(CP)。
1.3 单磷酸腺苷分子印迹聚合物(AMP-MIP)的制备
1.3.1 模板洗脱方式的优化
称取模板AMP 604.2 mg、功能单体AM 123.7 mg、CP 180.0 mg置于样品瓶中,加入25 mL致孔剂DMSO,超声混匀。在30 ℃的水浴锅中搅拌30 min后,再加入交联剂EGDMA 1.310 mL、引发剂AIBN 4.2 mg、DMSO 25 mL,再次超声混匀。将上述混合液分装在4支小试管中,用真空抽滤泵抽真空12 min。将小试管置于65 ℃油浴锅中,反应30 min。浸入冰水浴冷却至室温,抽滤,收集聚合物,均分为3份,加入50 mL甲醇-乙酸(9∶1,体积比),第一份在磁力搅拌器上搅拌洗脱,第二份超声洗脱,第三份索氏提取洗脱。直至UV检测上清液在258 nm处无吸收峰,再用50 mL去离子水洗涤一次,最后用50 mL甲醇洗涤一次,真空干燥,得AMP-MIP。
非分子印迹聚合物(NIP)制备除了不加模板AMP外,其它步骤一样。
1.3.2 模板洗脱试剂的优化
将1.3.1所制备聚合物按表1所示洗脱试剂进行模板洗脱。
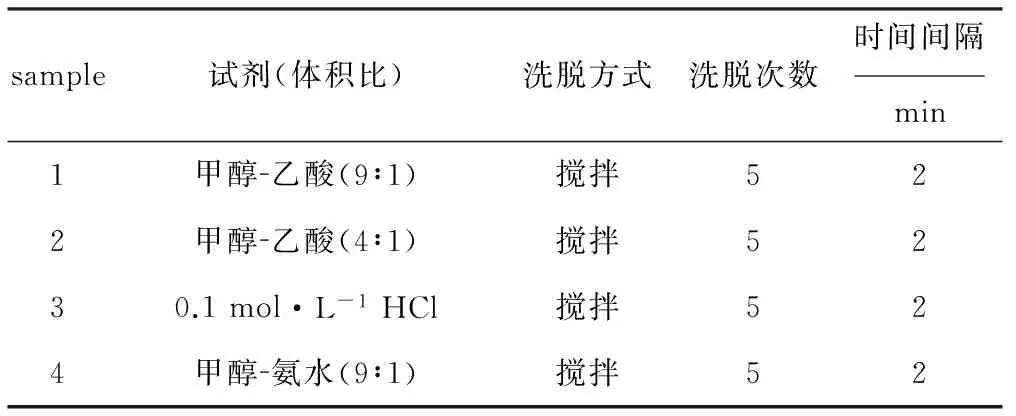
表1 模板洗脱试剂的优化
1.3.3 聚合时间的优化
按1.3.1步骤制备AMP-MIP和NIP,聚合时间分别为30 min、45 min和60 min,采用超声方式进行洗脱。
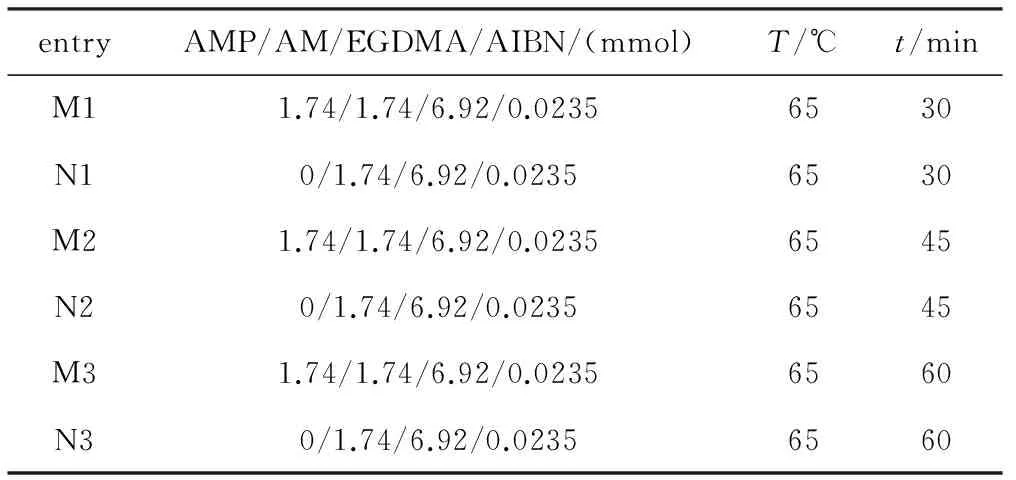
表2 聚合时间的优化
1.4 AMP-MIP的表征
1.4.1 不同洗脱条件下聚合物对AMP的吸附
用去离子水配制8μg·mL-1AMP溶液。称取不同洗脱条件得到的AMP-MIP、 NIP各20 mg于离心管中。加入1 mL 8μg·mL-1AMP溶液,超声,使其混合均匀。将离心管放置在恒温摇床中,温度设置为25 ℃,转速为200 r·min-1,振荡16 h后,12 000 r·min-1离心15 min。取上清液,0.45μm水相滤膜过滤。紫外可见分光光度计扫描,读取258 nm处吸光值,做平行实验3次。根据吸附前后溶液中AMP溶液浓度的变化,计算聚合物对模板分子AMP的吸附率和结合量:
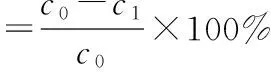

式中:c0为模板原始浓度,μg·mL-1;c1为上清液中模板浓度,μg·mL-1;V为溶液体积,mL;m为聚合物微球质量,mg。
1.4.2 不同聚合时间下聚合物的悬浮性能
分别用去离子水配制2 mL浓度为2 mg·mL-1AMP-MIP和NIP的悬浮液,超声,使聚合物充分分散悬浮。以黑板子为背景,按聚合时间顺序排列。室温下静置,分时间段进行拍照。
1.4.3 不同聚合时间下聚合物的形貌表征
取一定量的聚合物,先用乙醇溶解,超声混匀,将溶液滴在锡箔纸上,在空气中干燥后,用扫描电镜观察其形貌。
1.4.4 不同聚合时间下聚合物对gapA的吸附性能
称取1.3.3中所制备的3组AMP-MIP和NIP各10 mg于离心管中,用pH=7.5的50 mmol·L-1Tris-HCl对gapA进行稀释,向离心管中加入0.5 mL一定浓度的gapA溶液,混合均匀。孵育16 h后,离心收集上清液,进行SDS-PAGE。使用凝胶成像系统拍照,用Genetools半定量分析,以0.2 mg·mL-1BSA为标准量,定量上清液浓度。根据吸附前后溶液中gapA溶液浓度的变化,计算聚合物对蛋白质的结合量。
2 结果与讨论
2.1 AMP-MIP的洗脱条件优化
2.1.1 洗脱方式优化(图2)
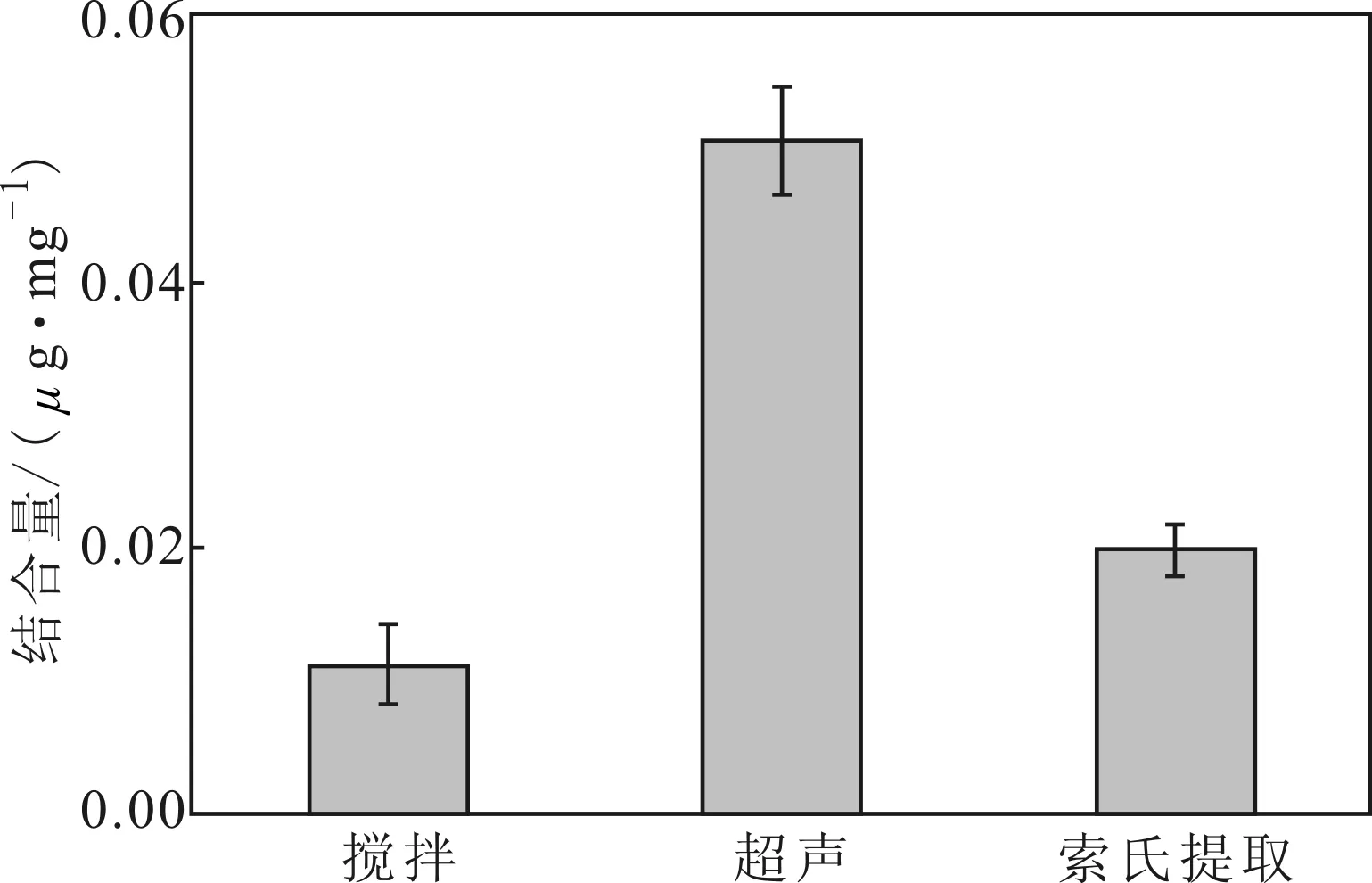
图2 不同洗脱方式下聚合物对AMP的结合量
从图2可以看出,超声洗脱方式所得聚合物对AMP的结合量最大,即超声洗脱方式的模板洗脱效果最好;索氏提取的次之,索氏提取比较节省溶剂,但是洗脱效率较低;搅拌洗脱方式的效果最差。因此,最佳洗脱方式为超声。
2.1.2 洗脱试剂优化(图3)
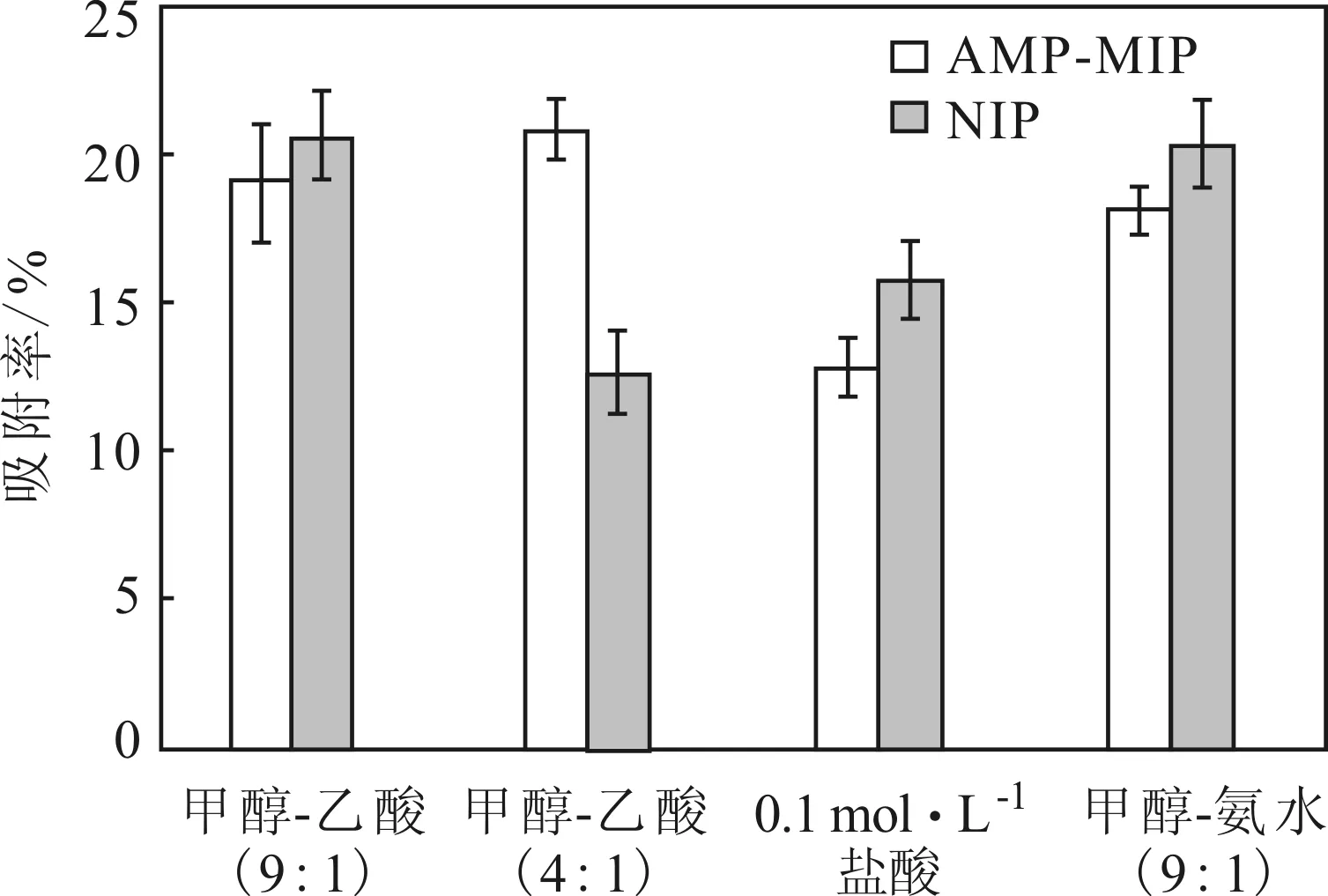
图3 不同洗脱试剂下聚合物对AMP的吸附率
模板分子与功能单体之间以非共价键相连,可以采用高极性洗脱溶剂反复萃取,将模板分子从印迹聚合物中除去。由图3可以看出,在洗脱溶剂为甲醇-乙酸(4∶1)时,AMP-MIP对AMP的吸附率最高;其它3种洗脱溶剂下,AMP-MIP的吸附率都比NIP要低。这可能是因为模板分子没有洗脱,也就是说在相同洗脱次数下,洗脱溶剂为甲醇-乙酸(4∶1)的洗脱效率最高,效果最好。
2.2 AMP-MIP的聚合时间优化
2.2.1 水中悬浮实验(图4)

样品从左到右依次为CP、M1、N1、M2、N2、M3、N3
从图4可知,所有的聚合物比CP内核沉淀的更快,说明印迹壳层已经修饰上去。随着聚合时间的延长,聚合物在水中的稳定性越来越差,亲水性越来越低。从沉降到瓶底聚合物的厚度可知,聚合物之间形成了一种支撑骨架。综上,当聚合时间为30 min时,聚合物在水中有更好的稳定性。
2.2.2 SEM分析(图5)

图5 不同聚合物的SEM照片
由图5可知,随着聚合时间的延长,壳层会越来越厚,聚合物越来越不均一。这主要是由于内核表面在进行RAFT反应,内核表面的双硫酯基团使聚合反应活性可控,反应相对较慢,同时游离在溶剂里的化合物也在进行不可控聚合反应,当溶液中的低聚物慢慢增多至形成一定大小交联度的颗粒,溶液稠度增大,游离的聚合物会将核壳型聚合物交联起来,形成粒径变大、不均一的颗粒。从聚合物M3、N3的扫描电镜照片可以看出,核壳型聚合物之间确实交联起来了,聚合物粒径过大,重力变大,也说明了水中聚合物稳定性变差的原因。
2.2.3 AMP-MIP对gapA的吸附
不同聚合时间下AMP-MIP、NIP对gapA吸附凝胶成像如图6所示。
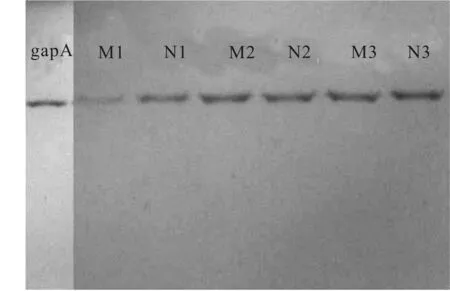
图6 不同聚合时间下AMP-MIP、NIP对gapA的吸附凝胶成像
从图6可知,随着聚合时间的延长,AMP-MIP和NIP对目标蛋白gapA的结合量越来越少,同时印迹指数α值也在慢慢变小。不同聚合时间下AMP-MIP、NIP对gapA的结合量见图7。
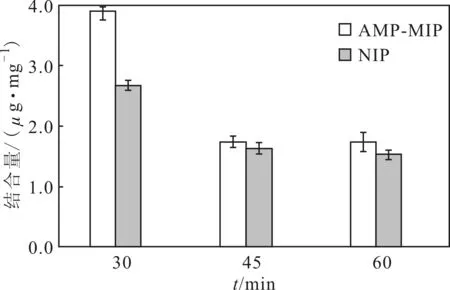
图7 不同聚合时间下AMP-MIP、NIP对gapA的结合量
由图7可知,当聚合时间为30 min时,AMP-MIP对ADP-核糖基化蛋白质的特异性结合效果最好,结合量达3.9μg·mL-1,聚合时间越长,结合效果越差。这主要是因为,聚合时间延长,核壳型微球相互交联,导致颗粒变大,影响了聚合物在水中的稳定性和亲水性能,而且聚合物比表面积减小,对蛋白质的特异性结合量也会变小。所以,选择聚合时间为30 min。
3 结论
以聚苯乙烯微球(CP)为内核、单磷酸腺苷(AMP)为模板、丙烯酰胺(AM)为功能单体、乙二醇二甲基丙烯酸酯(EGDMA)为交联剂,制备了核壳型单磷酸腺苷分子印迹聚合物(AMP-MIP)。通过扫描电镜、水中悬浮实验和吸附实验对AMP-MIP的形貌和性能进行了表征,得到最佳工艺条件:洗脱方式为超声、洗脱溶剂为甲醇-乙酸(4∶1)、聚合时间为30 min。分子印迹聚合物不仅对小分子模板有吸附作用,而且对大分子ADP-核糖基化的甘油醛-3-磷酸脱氢酶(gapA)具有识别作用,吸附量可达3.9 μg·mg-1。
[1] YANG X J,SETO E.Lysine acetylation:codified crosstalk with other posttranslational modifications[J].Molecular Cell,2008,31(4):449-461.
[2] SHAHAB U,FATIMA L,ANTONELLA S,et al.Role of Stat5 in type Ⅰ interferon-signaling and transcriptional regulation[J].Biochemical & Biophysical Research Communications,2003,308(2):325-330.
[3] LUDOVIC M,XENIA L,FARAJ T.Post-translational modifications of tau protein:implications for Alzheimer′s disease[J].Neurochemistry International,2011,58(4):458-471.
[4] GIANLUCA P,BARBARA F,PAOLO O,et al.Poly(ADP-ribose) polymerase inhibition in cancer therapy:are we close to maturity?[J].Expert Opinion on Therapeutic Patents,2009,19(19):1377-1400.
[5] MASUTANI M,NAKAGAMA H,SUGIMURA T.Poly(ADP-ribosyl)ation in relation to cancer and autoimmune disease[J].Clinical Nuclear Medicine,2005,37(11):1115-1116.
[6] MOTA R A,FRANCISCO S B,LUIS S,et al.Inhibition of poly(ADP-ribose) polymerase attenuates the severity of acute pancreatitis and associated lung injury[J].European Geriatric Medicine,2011,2(11):24-206.
[8] SHEVALYE H,STAVNIICHUK R,XU W,et al.Poly(ADP-ribose) polymerase (PARP) inhibition counteracts multiple manifestations of kidney disease in long-term streptozotocin-diabetic rat model[J].Biochemical Pharmacology,2010,79(7):1007-1014.
[9] TELLI M L.PARP inhibitors in cancer:moving beyond BRCA[J].Lancet Oncology,2011,12(12):827-828.
[10] UNDERHILL C,TOULMONDE M,BONNEFOI H.A review of PARP inhibitors:from bench to bedside[J].Annals of Oncology Official Journal of the European Society for Medical Oncology,2011,22(2):268-279.
[11] ABDIN M J,ALTINTAS Z,TOTHILL I E.In silico designed nanoMIP based optical sensor for endotoxins monitoring[J].Biosensors & Bioelectronics,2015,67:177-183.
[12] ALIZADEH T,HAMEDSOLTANI L.Graphene/graphite/molecularly imprinted polymer nanocomposite as the highly selective gas sensor for nitrobenzene vapor recognition[J].Journal of Environmental Chemical Engineering,2014,2(3):1514-1526.
[13] CAKIR P,CUTIVET A,RESMINI M,et al.Protein-size molecularly imprinted polymer nanogels as synthetic antibodies,by localized polymerization with multi-initiators[J].Advanced Materials,2013,25(7):1048-1051.
[14] NOIR M L,LEPEUPLE A S,GUIEYSSE B,et al.Selective removal of 17β-estradiol at trace concentration using a molecularly imprinted polymer[J].Water Research,2007,41(12):2825-2831.
[15] ALI W H,DERRIEN D,ALIX F,et al.Solid-phase extraction using molecularly imprinted polymers for selective extraction of a mycotoxin in cereals[J].Journal of Chromatography A,2010,1217(43):6668-6673.
[16] KHORRAMI A R,TAHERKHANI M.Synthesis and evaluation of a molecularly imprinted polymer for preconcentration of patulin from apple juice[J].Chromatographia,2011,73(73):151-156.
[17] ASADI E,AZODI-DEILAMI S,ABDOUSS M,et al.Cyproterone synthesis,recognition and controlled release by molecularly imprinted nanoparticle[J].Applied Biochemistry & Biotechnology,2012,167(7):2076-2087.
[18] SUKSUWAN A,LOMLIM L,RUNGROTMONGKOL T,et al.The composite nanomaterials containing (R)-thalidomide-molecularly imprinted polymers as a recognition system for enantioselective-controlled release and targeted drug delivery[J].Journal of Applied Polymer Science,2015,260(26):14368-14373.
[19] ESFANDYARI-MANESH M,JAVANBAKHT M,DINARVAND R,et al.Molecularly imprinted nanoparticles prepared by miniemulsion polymerization as selective receptors and new carriers for the sustained release of carbamazepine[J].Journal of Materials Science:Materials in Medicine,2012,23(4):963-972.
[20] ROSTAMIZADEH K,VAHEDPOUR M,BOZORGI S.Synthesis,characterization and evaluation of computationally designed nanoparticles of molecular imprinted polymers as drug delivery systems[J].Int J Pharm,2012,424(1/2):67-75.
Preparation of Core-Shell Adenosine-5′-Monophosphate Molecularly Imprinted Polymer
GONG Xia1,HU Ying-ying1,YOU Xiang-yu1,GU Jing2,DENG Jiao-yu2,XIE Wei-hong1
(1.InstituteofBioengineeringandFood,HubeiUniversityofTechnology,Wuhan430068,China;2.TheStateKeyLaboratoryofAgriculturalandEnvironmentalMicrobiologyofCAS,WuhanInstituteofVirology,CAS,Wuhan430071,China)
Usingpolystyrenemicrosphere(CP)asacore,adenosine-5′-monophosphate(AMP)asatemplate,acrylamide(AM)asafunctionalmonomerandethyleneglycoldimethacrylate(EGDMA)asacross-linker,whiledimethylsulfoxide(DMSO)waschosenasaporogenandazobisisobutyronitrile(AIBN)asaninitiator,core-shelladenosine-5′-monophosphatemolecularimprintedpolymer(AMP-MIP)wasprepared.Theeffectsofdifferentelutionmethods,solventsandpolymerizationtimeonthepropertiesofAMP-MIPrecognitionwerediscussed.ThemorphologyandpropertyofAMP-MIPwerecharacterizedbySEM,thesuspensioninaqueousmediumandthebindingexperiments.Theoptimalprocessconditionswereobtainedasfollows:theelutionmethodwasultrasound,theelutionsolventwasmethanol-aceticacid(4∶1)andpolymerizationtimewas30min.AMP-MIPshowedgoodselectivitiesnotonlyforAMPbutalsoforglyceraldehyde-3-phosphatedehydrogenase(gapA)whichwasADP-ribosylatedprotein.TheadsorptioncapacityofgapAcouldreach3.9 μg·mg-1.
polystyrene;adenosine-5′-monophosphate;core-shellpolymerization;molecularlyimprintingtechnology;glyceraldehydes-3-phosphatedehydrogenase
中国科学院病毒研究所国家重点实验室开放研究基金(20875024)
2016-06-06
龚霞(1990-),女,湖北荆门人,硕士研究生,研究方向:分子印迹材料,E-mail:gongxia824@126.com;通讯作者:谢卫红,博士,教授,E-mail:weihong.xie@mail.hbut.edu.cn。
10.3969/j.issn.1672-5425.2016.11.005
龚霞,胡莹莹,尤祥宇,等.核壳型单磷酸腺苷分子印迹聚合物的制备[J].化学与生物工程,2016,33(11):27-31.
TQ 317
A
1672-5425(2016)11-0027-05

