320)我国蜥蜴微卫星标记的开发及其应用*
赵 丹,赵文阁,刘 鹏
(哈尔滨师范大学)
320)我国蜥蜴微卫星标记的开发及其应用*
赵 丹,赵文阁,刘 鹏**
(哈尔滨师范大学)
微卫星属于短串联重复序列,微卫星标记是近年来分子生物学研究的重要课题之一.蜥蜴是爬行纲动物种类繁多的类群,在自然生态系统中具有重要的地位和作用.目前GenBank上公布我国共有15种蜥蜴开发出278个微卫星标记.通过分析和探讨这些微卫星标记的开发方式及应用前景,旨在为今后其他蜥蜴微卫星标记的开发和应用提供参考依据.
蜥蜴;微卫星标记;基因组;开发方式
0 引言
微卫星又称简单重复序列(Simple sequences repeats,SSRs)[1]或短串联重复(Short tandem repeats,STRs)[2],每个重复单位一般只有1~6个碱基.微卫星标记作为一种重要的分子遗传标记,具有探针序列可直接人工合成、多态性和杂合性较高、重组率低、遗传方式简单等优势,能较好地反映物种的遗传结构和遗传多样性变化,为种群生物学研究提供了丰富的遗传信息,因而比其他分子标记的应用更为广泛,在动物体中以双核苷酸(CA/GT)n最为常见[3].
微卫星标记主要有三种开发方法:第一种是从已发表的文献或公共数据库(如NCBI、EST等)中查找微卫星位点或引物,但该方法只限于已开发出微卫星引物的物种,局限性较大;第二种是跨物种扩增,即利用遗传距离相近物种的微卫星引物进行扩增,但成功率较低,盲目性较大;第三种是在基因组DNA中筛选微卫星位点并开发引物,这也是目前获取微卫星位点最好的途径[4].
1 已开发微卫星标记的我国蜥蜴种类
我国蜥蜴共有9科39属156种[5].截止到2016年9月11日,通过NCBI检索,已开发微卫星标记的我国蜥蜴有5科9属15种(GenBank上的公布时间从1999年7月6日至2016年8月25日),占我国蜥蜴总数的9.6%,包括蜥蜴科3属6种,即胎生蜥蜴(Lacertavivipara)、捷蜥蜴(Lacertaagilis)、密点麻蜥(Eremiasmultiocellata)、快步麻蜥(E.velox)、台湾草蜥(Takydromusformosanus)和白条草蜥(T.wolteri),壁虎科2属4种,即大壁虎(Gekkogecko)、多疣壁虎(G.japonicus)、无蹼壁虎(G.swinhonis)和哀磷趾虎(Lepidodactyluslugubris),鬣蜥科2属3种,分别为青海沙蜥(Phrynocephalusvlangalii)、荒漠沙蜥(P.przewalskii)、蜡皮蜥(Leiolepisreevesii),鳄蜥科和巨蜥科各1属1种,分别为鳄蜥(Shinisauruscrocodilurus)和圆鼻巨蜥(Varanussalvator)(见表1).这些蜥蜴共开发了278个微卫星标记,其中大壁虎开发的微卫星标记个数最多,为47个,白条草蜥开发的微卫星标记个数最少,为9个(见表1).
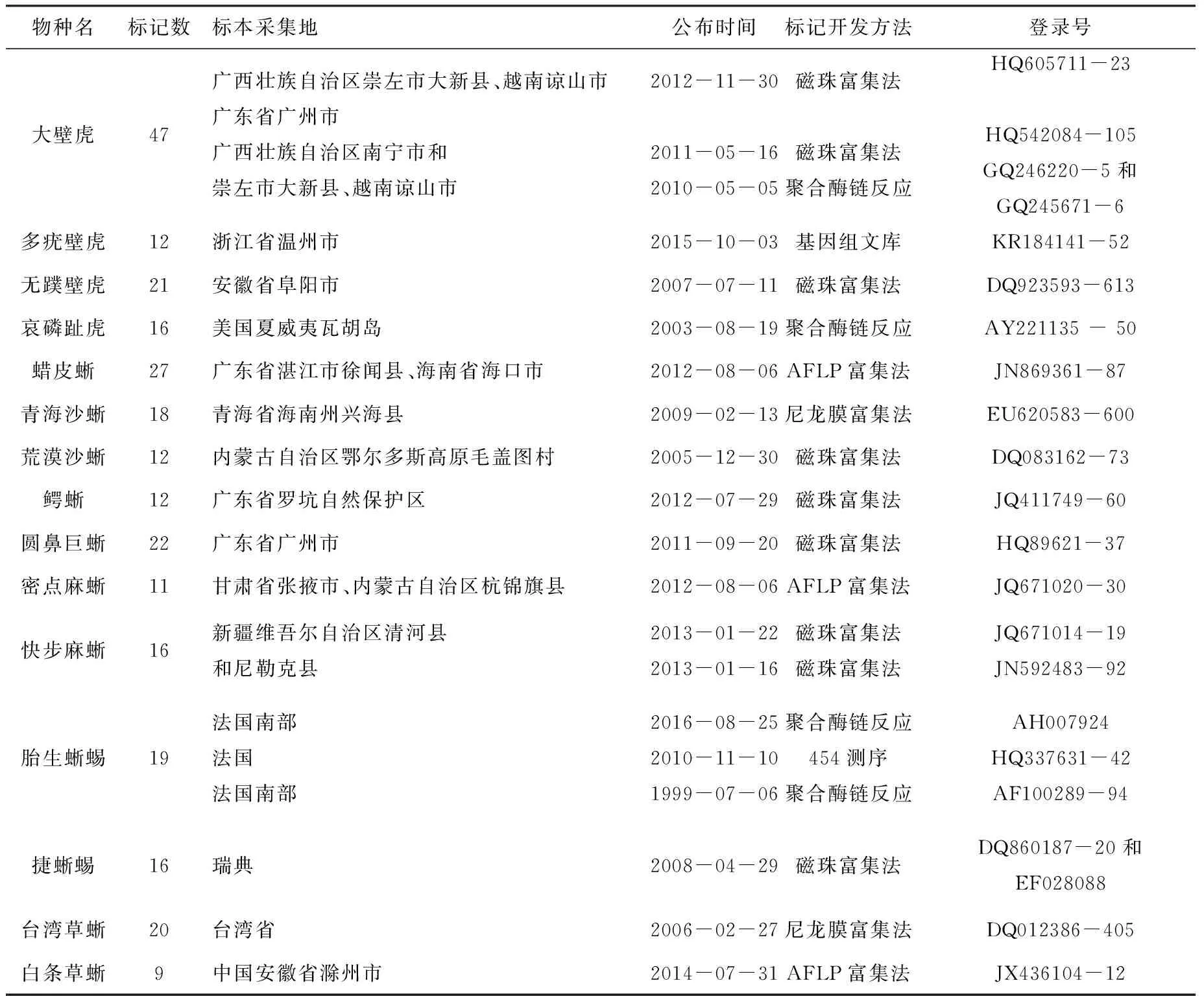
表1 GenBank上已经公布的我国蜥蜴微卫星标记
2 我国蜥蜴微卫星标记的开发方法
2.1 聚合酶链式反应
聚合酶链式反应是一种利用PCR技术扩增特定DNA片段来开发微卫星标记的技术[6],它是我国蜥蜴应用最早的开发微卫星标记的方法(1999年),首先通过 PAPD引物随机扩增获得DNA片段,然后选出其中带型集中大小适于克隆的扩增产物进行克隆.整个过程以PCR为基础,易于操作.我国蜥蜴类动物中的胎生蜥蜴[7]、哀磷趾虎[8]和大壁虎[9]使用该方法进行了微卫星标记的开发.但这种方法的应用前提是RAPD 产物必须与微卫星位点相关联,所以具有较大的局限性.
2.2 尼龙膜富集法
1993年,Karagyozov首次将微卫星探针固定在尼龙膜上并与基因组文库进行杂交,用于微卫星的富集[10].尼龙膜富集法的过程,首先用限制性内切酶酶切基因组DNA构建小片段基因文库,并与接头连接,随后接头序列进行PCR扩增,扩增片段需在沸水中热变性,最后变性DNA通过与吸附在尼龙膜上的许多微卫星探针杂交来富集基因组中的微卫星片段[11].在我国蜥蜴中,台湾草蜥[12]和青海沙蜥[13]使用尼龙膜富集法进行微卫星标记开发.
2.3 磁珠富集法
磁珠富集法是指用生物素标记的探针与基因组DNA片段杂交,再将亲和素或生物素与其杂交,两者相互结合附在磁性小珠上,经过后续的洗脱、变性等操作过程,完成对重复序列目的片段的富集,从而建立微卫星富集文库.磁珠富集法中探针与基因组 DNA 的杂交反应在液体介质中完成[14],与探针固定在尼龙膜上相比反应更加充分,因而,磁珠富集法与尼龙膜富集法相比分离效率较高.在我国蜥蜴中,大壁虎[15,16]、鳄蜥[17]、无蹼壁虎[18]、荒漠沙蜥[19]、圆鼻巨蜥[20]、快步麻蜥[21]和捷蜥蜴[22]使用磁珠富集法进行微卫星标记开发,是我国蜥蜴微卫星标记开发中运用最多的方法,也是当前应用于大规模开发微卫星标记最为常用的一种方法.
2.4 AFLP富集法
在我国蜥蜴中,蜡皮蜥[23]、密点麻蜥[24]和白条草蜥[25]使用AFLP富集法进行微卫星标记开发.在AFLP富集过程中基因组DNA被限制性内切酶随机酶切并与AFLP接头连接,连接产物适当稀释后用作PCR扩增模板,PCR扩增产物需纯化,最后转化到大肠杆菌感受态细胞,建成基因组微卫星富集文库[26,27].
2.5 454测序
454测序首先需要提取基因组DNA,送到可进行454测序的生物学公司测序,获得测序结果及其报告,然后进行数据分析和引物设计.该方法无需建库、克隆及筛选,操作过程简单,大大节省时间.但是,454测序技术所读基因组片段长度较短,微卫星没有足够的侧翼序列用于引物设计,且适合引物设计的微卫星序列较少,与其他开发方法相比,成本较高[28].在我国蜥蜴中,胎生蜥蜴[29]使用该方法进行了微卫星标记开发.
2.6 基因组文库法
基因组文库法是一种传统的方法,首先提取基因组DNA,用限制性内切酶将其切割成小片段,回收100~1000bp的片段,然后用标记探针进行杂交、克隆、测序,从而证实重复片段的存在,并对其进行引物设计,用PCR扩增进行检验,对小量样本进行预实验,最终挑选重复性好、多态性高的微卫星位点,从而进行微卫星标记的开发.近几年,应用该方法开发微卫星标记的物种中,大部分都是已有基因组文库或部分基因组文库发表,在此基础上进行微卫星标记的开发.在我国蜥蜴中,多疣壁虎的微卫星标记开发较晚(2015年),因此并未建立基因文库,而是在已有的部分基因组文库基础上,运用SSR Hunter微卫星位点鉴定软件[30],最终开发出12个微卫星标记[31].
3 我国蜥蜴微卫星标记的应用
目前,我国蜥蜴微卫星标记的开发主要用于不同物种、不同种群间的遗传多样性、遗传结构和亲子关系等遗传学分析[32],以及利用近缘物种的微卫星标记进行跨种扩增,对物种进行分类,从而作为确立有效物种的依据之一[33].另外,人们还利用微卫星标记对蜥蜴的洞穴分配、种群婚配制度及父权状况[34]、个体与社会互动所存在的潜在联系[35]以及生境隔离对种群的影响[36]等方面进行探讨.
我国蜥蜴科动物微卫标记的开发和报道工作还不够全面,开发更多物种的微卫星标记将对蜥蜴的遗传演化和系统发育具有重要意义.由于微卫星中存在无效等位基因[37],这些等位基因在PCR不能成功扩增,从而影响遗传学分析和亲权关系鉴定的准确性[38].但是随着分子生物学技术和生物信息学的飞速发展, 高通用性微卫星标记必将有着更为广阔的应用前景.
[1] Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers[J]. Nucleic Acids Res, 1989, 17(16): 6463-71.
[2] Edwards A, Civitello A, Hammond HA, et al. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats[J]. Am J Hum Genet, 1991, 49(4): 749-56.
[3] Maguire T L, Edwards KJ, Saenger P, et al. Characterization and analysis of microsatellite loci in mangrove species, Avicennia marina (Fork.) Vierh. (Avicenniaceae)[J]. Theor Appl Genet, 2000, 101(1): 279-85.
[4] 曾庆国, 陈艺燕.微卫星位点筛选方法综述[J]. 生态科学, 2005, 24(4): 368-372.
[5] 赵尔宓, 赵肯堂, 周开亚, 等. 中国动物志: 爬行纲有鳞目蜥蜴亚目[M]. 北京: 科学出版社, 1999.15-358.
[6] Lunt D H, Hutchinson W F, Carvalho GR. An efficient method for PCR-based isolation of microsatellite arrays (PIMA)[J]. Mol Ecol, 1999, 8(1): 891-894.
[7] Boudjemadi K, Martin O, Simon JC, et al. Development and cross-species comparison of microsatellite markers in two lizard species, Lacerta vivipara and Podarcis muralis[J]. Mol Ecol, 1999, 8(3): 518-520.
[8] Wilmhoff C D, Csepeggi C E, Petren K. Characterization of dinucleotide microsatellite markers in the parthenogenetic mourning gecko (Lepidodactylus lugubris)[J]. Molecular Ecology Notes, 2003,3(3):400-402.
[9] Peng Q K, Wang G C,Yang D,et al.Genetic variability of the tokay gecko based on microsatelliteanalysis[J].Biochemical Systematics and Ecology, 2010,38(1): 23-28.
[10] Karagyozov L, Kalcheva I D, Chapman VM. Construction of random smallinsert genomic libraries highly enriched for simple sequence repeats[J]. Nucleic Acids Research, 1993,21(16): 3911-3912.
[11] Armour J A, Neumann R, Gobert S, et al. Isolation of human simple repeat loci by hybridization selection[J]. Human Mol Genet, 1994, 3 (4): 599-605.
[12] Lin S M, Wang C J, Hsu Y, et al. Isolation and characterization of 12 tetrarepeated microsatellite loci from the Formosan grass lizard (Takydromus formosanus)[J]. Molecular ecology notes, 2006, 1(6): 57-59.
[13] Zhen A B, Fu J Z. Microsatellite DNA markers for three toad-headed lizard species (Phrynocephalus vlangalii, P. przewalskii and P. guttatus)[J]. Molecular ecology notes, 2000,9(2): 535-538.
[14] Kandpal R P, Kandpal G, Weissman SM. Construction of libraries enriched for sequence repeats and jumping clones, and hybridization selection for regionspecific markers[J]. Proc Natl Acad Sci USA, 1994, 91(1):88-92.
[15] Gao S F, Yu D M, Peng J J, et al. Isolation and characterization of novel microsatellitemarkers in Gekko gecko(Reptilia: Gekkonidae)[J]. Conservation Genetics Resources, 2011, 3(3): 46-48.
[16] Peng Q K, Wang G C, Tan S,et al. Isolation and characterization of thirteen polymorphic tetranucleotide microsatellite markers in the Tokay gecko(Gekkogecko)[J]. Conservation Genet Resour, 2011, 3(4):617-619.
[17] Bei R B, Liu H Y, Wu Z J, et al. Isolation and characterization of 12 microsatellite loci in the Chinese crocodile lizard (Shinisaurus crocodilurus)[J]. Conservation Genetics Resources, 2012, 4(3):743-745.
[18] Li J J, Zhou K Y. Isolation and characterization of microsatellite markers in the gecko Gekko swinhonis and cross-species amplification in other gekkonid species[J]. Molecular Ecology Notes, 2007, 7(4):674-677.
[19] Urquhart J, Bi K, Gozdzik A, et al. Isolation and characterization of microsatellite DNA loci in the toad-headed lizards, Phrynocephalus przewalskii complex[J]. Molecular Ecology Notes, 2005, 5:928-930.
[20] Fu M L, Yu D M, Peng J J, et al. Isolation and characterization of novel microsatellite markers in Water monitor (Varanus salvator)[J]. Conservation Genet Resour, 2011, 3(4):777-779.
[21] Li H, Zhou Z S, Guo J, et al. Polymorphic microsatellite loci in the rapid racerunner Eremias velox (Squamata: Lacertidae)[J]. GenetMol Res, 2012, 11(4):4707-4710.
[22] Schwartz T S, Olsson M. Microsatellite markers developed for a Swedish population of sand lizard (Lacerta agilis)[J].Conserv Genet, 2008, 9(3):715-717.
[23] Lei H, Mao L X, Chen C, et al. Isolation and characterization of microsatellite loci in the Reevese’s butterfly lizard Leiolepis reevesii (Agamidae)[J]. Conservation Genet Resour, 2012, 1(4): 791-794.
[24] Chen L, Guo L, Li H, et al. Microsatellite markers developed for the multiocellated racerunner, Eremias multiocellata (Lacertidae)[J]. Conservation Genetics Resources,2012, 4(3): 711-713.
[25] 罗来高,吴义莲,耿军,等. 白条草蜥多态性微卫星位点的分离与鉴定[J].动物学杂志, 2013, 48(3): 367-370.
[26] Zane L, Bargelloni L, Patarnello T. Strategies for microsatellite isolation: a review[J]. Molecular Ecology, 2002, 11(1): 1-16.
[27] 薛辉, 吴孝兵, 晏鹏. 微卫星标记在分子生态学中的应用及其位点的分离策略[J]. 应用生态学报, 2005, 16(2): 385-389.
[28] 程晓凤, 黄福江, 刘明典, 等. 454测序技术开发微卫星标记的研究进展[J]. 生物技术通报, 2011, 27(8): 82-90.
[29] Virginie MS, Murielle R, Colin B, et al. Twelve new polymorphic microsatellite loci for the common lizard, Zootoca vivipara. Molecular Ecology Resources, 2011, 11: 586-589.
[30] 李强, 万建民,Hunter S S R ,一个本地化的SSR位点搜索软件的开发[J]. 遗传, 2005, 27(5): 808-810.
[31] Wei L, Shao W W,Zhou H B, et al. Rapid microsatellite development in Gekko japonicus using sequenced restrictionsite associated DNA markers. Genetics and Molecular Research, 2015, 14(4): 14119-14122.
[32] 骆爽, 刘廼发.西藏高原两种沙蜥遗传结构及三种沙蜥亲子鉴定[D]. 甘肃: 兰州大学, 2009.
[33] Daniel W A Noble, Qi Y, Fu J Z. Species delineation using Bayesian model-based assignment tests: a case study using Chinese toad-headed agamas (genus Phrynocephalus)[J]. BMC Evol Biol, 2010, 10: 197.
[34] Qi Y, Daniel W A Noble, Fu J Z, et al. Spatial and social organization in a burrow-dwelling lizard (Phrynocephalus vlangalii) from China [J]. PLoS One, 2012, 7(7): e41130.
[35]Qi Y, Yang W Z, Lu B, et al. Genetic evidence for male-biased dispersal in the Qinghaitoad-headed agamid Phrynocephalus vlangalii and its potential link to individual social interactions[J]. Ecol Evol, 2013, 3(5): 1219-1230.
[36] Hu D, Fu J Z, Zou F D, et al. Impact of the Qinghai-Tibet railwayon population genetic structure of the toad-headed lizard,Phrynocephalus vlangalii[J]. Asian Herpetological Research, 2012, 3(4): 280-287.
[37] Oddou-Muratorio S, Vendramin G G, Buiteveld J, et al.Population estimators or progeny tests: what is the best method to assess null allele frequencies at SSR loci?[J]. ConservationGenetics, 2009, 10(5): 1343-1347.
[38] Wagner A P, Creel S, Kalinowski S T. Estimating relatedness and relationships using microsatellite loci with null alleles[J]. Heredity, 2006, 97(5): 336-345.Development and Application of Microsatellite Markers in Chinese Lizards
(责任编辑:季春阳)
Zhao Dan, Zhao Wenge, Liu Peng
(Harbin Normal University)
Microsatellites are known as Short Tandem Repeats (STR).Microsatellite markers become one of the important research topics in molecular biology. With abundant species in Reptilia, lizards have the important status and functions in the ecosystem. At present, there are 278 microsatellite markers developed in 15 species of Chinese lizards published on GenBank. The development methods and application prospect of microsatellite markers were also analyzed and discussed in this paper, aiming to provide reference for the future development and application of microsatellite markers in lizards.
Lizards; Microsatellite marker; Genome; Development method
2016-03-24
*黑龙江省自然科学基金项目(C2016035);国家自然科学基金项目(31172079)
Q959.6
A
1000-5617(2016)03-0083-04
**通讯作者:liupeng111111@163.com

