花生根瘤菌遗传多样性及高效固氮菌株的筛选
吴海龙,李 岩,陈明娜,刘 卫,任承钢,杨金保,4,解志红*,禹山林*
(1.中国科学院烟台海岸带研究所,山东 烟台 264003; 2.中国科学院大学,北京 100049;3.山东省花生研究所,山东 青岛 266100; 4.山西农业大学,山西 太谷 030801)
花生根瘤菌遗传多样性及高效固氮菌株的筛选
吴海龙1,2,李 岩1,陈明娜3,刘 卫1,任承钢1,杨金保1,4,解志红1*,禹山林3*
(1.中国科学院烟台海岸带研究所,山东 烟台 264003; 2.中国科学院大学,北京 100049;3.山东省花生研究所,山东 青岛 266100; 4.山西农业大学,山西 太谷 030801)
为揭示花生根瘤菌遗传多样性并筛选高效固氮菌株,本研究从山东省花生研究所莱西试验站的5个样点采集新鲜花生根瘤进行根瘤菌分离,对菌株的16S rRNA基因、持家基因(recA、atpD、glnII)序列进行系统发育分析以确定根瘤菌的系统发育地位,同时对共生基因nifH序列进行系统发育分析,并通过温室盆栽接种实验筛选高效共生固氮根瘤菌菌株。从5个采样点共分离到106株花生根瘤菌菌株,通过持家基因recA序列分析选定7株代表菌株。对代表菌株的16S rRNA基因与持家基因MLSA系统发育分析表明,所分离到的菌株分布于Bradyrhizobium的B.liaoningense、B.yuanmingense、Bradyrhizobiumsp.种群。同时,固氮基因nifH的系统发育分析结果表明,固氮基因高度保守,在系统进化树上分别与B.arachidisCCBAU 051107T、B.yuanmingenseCCBAU 10071T聚为两支。温室盆栽实验表明7株花生根瘤菌代表菌株均能与花生有效结瘤,其中代表菌株YIC61059共生固氮能力最强。结果分析表明,山东省莱西市花生根瘤菌遗传多样性较为丰富,其中B.liaoningense为优势种群。该地区的花生根瘤菌具有较好的结瘤和共生固氮能力,其中代表菌株YIC61059促生效果比较突出,具有良好的应用前景。
花生;根瘤菌;遗传多样性;高效菌株筛选
花生(ArachishypogaeaL.)为豆科一年生草本植物,是我国三大传统油料作物之一,在油料作物生产中具有极其重要的地位。花生与花生根瘤菌通过形成根瘤来固定空气中的氮气,为花生生长提供必需的氮素营养。花生—根瘤菌共生固氮体系可以满足花生生长所需氮量的50%左右,同时还可以改善土壤肥力,提高花生的产量和品质[1]。而我国花生种植所需氮素大部分依靠化学氮肥,不仅增加了种植成本,同时由于化肥的大量生产和施肥过量引起的水肥流失造成环境污染[2-3]。因此,花生根瘤菌高效菌株的筛选和菌剂的推广将有效降低种植成本,改善环境,促进花生种植产业的可持续发展。
莱西市是山东省花生种植的主要地区之一,花生的播种面积居山东省前列[4],具有优质和丰富的花生和花生根瘤菌种质资源。本研究通过对采集、分离自山东省花生研究所莱西试验站5个样点的106株根瘤菌实验分析,最终选取了7株花生根瘤菌代表菌株进行16S rRNA基因、持家基因(recA,atpD,glnII)及固氮基因nifH的系统发育分析研究,揭示山东莱西花生根瘤菌遗传多样性、分类地位及固氮基因的特性,为花生根瘤菌遗传多样性研究提供参考。花生温室盆栽实验筛选的高效共生固氮菌株,为研制根瘤菌剂提供了优质的种质资源,对提高花生产量,减少化肥施用,促进生态环境的可持续发展发挥积极作用。
1 材料和方法
1.1 供试菌株
采自山东省花生研究所莱西试验站花育33号的新鲜根瘤,分离纯化获得106株花生根瘤菌,置于20%甘油冻存管中-80℃保存[5]。
1.2 培养基
分离纯化及花生温室培养所用低氮培养液、YMA固体培养基、TY液体培养基成分和配制方法参照文献[6-7]。
1.3 DNA提取
供试菌株总DNA用TIANGEN细菌基因组DNA提取试剂盒提取,在含有EB 的0.8%的琼脂糖凝胶电泳,用Nano Drop 2000检测DNA的浓度和纯度,并于-20°C保存备用。
1.4 持家基因recA的扩增及代表菌株选取
PCR反应体系、引物及扩增条件参照文献[8]。PCR产物用1.0%琼脂糖凝胶电泳检测合格后送至北京奥科鼎盛生物科技有限公司测序。测序返回结果在NCBI上进行在线比对,然后用MEGA5.0软件采用邻接法(neighbor-joining method)同参比菌株序列进行比对构建进化树,自展值(bootstrap)为1000,将recA序列相似性100%的定为同一基因型并从中随机选取一株菌株作为代表菌株进行后续分析[9]。
1.5 16S rRNA基因、持家基因(recA,atpD,glnII)、固氮基因nifH系统发育分析
代表菌株的16S rRNA基因 PCR扩增引物和反应体系(50 μL)及条件参见文献[10]。持家基因(atpD,glnII)、固氮基因nifHPCR扩增引物,反应体系及条件参照文献[11]。将测序得到的基因序列,用MEGA5.1软件将测得的16S rRNA、nifH序列分别与其参比序列进行系统发育分析构建系统发育进化树。同时,将得到的代表菌株的recA、atpD、glnII的顺序依次进行剪切拼接处理,然后用多位点基因序列分析(MLSA)构建系统发育树进行菌株的分类鉴定[12-13]。
1.6 高效共生固氮根瘤菌的筛选
挑选大小均匀且表皮完整的花生种子,表面消毒后置于0.6%的无菌水琼脂板上避光萌发3d左右,待主根长到2 cm左右时种植到盛有灭菌蛭石的双层钵中。将待测菌株同时活化,用TY液体培养基28℃,180 rpm摇床震荡培养2~3d,测定菌液OD600=0.8~1.0。然后将1 mL的代表菌株菌液接种于幼苗根部,同时以不接菌液只接种1mL的TY培养基作为对照,每个处理设置5个重复。将所有处理全部随机置于温室内培养,温室温度为26℃,光照时间16 h/d,5~7d补充一次无菌水。培养至25d左右时用叶绿素计SPAD-502 Plus测量花生叶片叶绿素含量并做好记录。培养至35d收获,测量统计各处理花生植株的地上部株高、鲜重、根瘤重及根瘤固氮酶活性。采用乙炔还原法测定花生根瘤固氮酶活性,测出乙烯成分,经过固氮酶活性的换算公式,以每克根瘤产生的乙烯(mmol)量为固氮酶活性衡量指标[14]。
2 结果与分析
2.1recA基因型分群结果
将供试菌株recA测序结果同参比菌株序列进行聚类分析构建系统进化树并随机选取一株作为代表菌株进行后续实验。由于菌株数量较大,故将数据分析结果整理于表1中。如表1所示,分离得到106株供试菌株初步分为7个recA基因型,其中基因型I有90株根瘤菌,较其他类群菌株数量具有明显优势。

表1 供试菌株recA基因系统发育分析结果
2.2 代表菌株 16S rRNA 基因建树分析
将测序得到的7株代表菌株的16S rRNA序列在NCBI上BLAST进行序列比对并用Sequin 软件将序列提交至GenBank得到代表菌株的序列号。采用邻接法与参比菌株一起构建16S rRNA基因系统发育树(图1)。
由图1可以看出,7株代表菌株均属于慢生根瘤菌属(Bradyrhizobium)。其中YIC60756、YIC60933、YIC61000、YIC61059、YIC60767具有相同的序列,与参比菌株B.liaoningenseUSDA 3622T的16S rRNA基因序列相似性为100%。而代表菌株YIC60838与B.yuanmingenseCCBAU10071T亲缘关系最近,相似性为99.8%。在另一分支的代表菌株YIC60881的16SrRNA基因序列与参比菌株B.manausenseBR3351T聚在一起,相似性为99.9%。

图 1 7株代表菌株16S rRNA基因系统发育树Fig. 1 Neighbor-joining phylogenetic tree based on 16S rRNA genesequences showing diversity of the 7 representative isolates注:7株代表菌株为黑色加粗字体,菌株GenBank序列号在括号中给出。Note: Representative strains indicated with black bold. The accession numbers in GenBank areindicated after the bacterial names.
2.3 持家基因(recA、atpD、glnII)的系统发育分析结果
先前研究表明,持家基因的MLSA分析能有效确定根瘤菌的系统发育地位[15],因此本研究对7株代表菌株的持家基因(recA、atpD和glnII)序列依次拼接并构建系统发育树(图2)。以相似性97%作为定种标准[16],分析表明有5株代表菌株YIC61000、YIC61059、YIC60756、YIC60933、YIC60767相似性较高(97.3%~99.7%),在系统发育树上聚为一支,与参比菌株B.liaoningenseUSDA3622T相似性为97.9%~98.7%,故将这5株代表菌株归为B.liaoningense种群,为所分离菌株的优势种群。YIC60838与参比菌株B.yuanmingenseCCBAU 10071T相似性最高为98.5%,将其归于B.yuanmingense种群。YIC60881与其他参比菌株相似性均低于97%,故将其命名为Bradyrhizobiumsp.YIC60881。
对所有分离到的根瘤菌菌株统计分析表明,B.liaoningense为山东省花生研究所莱西试验站所分离菌株的绝对优势种群,占所分离菌株的98.1%,这与之前研究报道中B.liaoningense是山东省花生根瘤菌的优势菌群的结果一致[17]。

图 2 持家基因recA-atpD-glnⅡ拼接后的MLSA系统发育树Fig. 2 Phylogenetic tree of concatenated sequences of recA, atpD and glnII
2.4 固氮基因nifH的系统发育分析
固氮基因nifH对于根瘤菌与豆科植物的固氮至关重要,本实验通过对慢生根瘤菌属的7株代表菌株的nifH基因进行PCR扩增测序,然后采用邻接法构建系统发育树并分析计算与参比菌株的相似性。
如图3所示,7株代表菌株的nifH基因聚类形成2个不同分支,与16S rRNA、持家基因的系统发育结果存在一定差异。YIC60756、YIC60767、YIC60838、YIC61059、YIC61000与B.yuanmingenseCCBAU 10071T聚在一起,序列相似性为100%,说明nifH在同一根瘤菌种内不同基因型中高度保守。YIC60881与B.arachidisCCBAU 051107T聚在一起,序列相似性为100%,而YIC60933与参比菌株B.arachidisCCBAU 051107T亲缘关系最近,序列相似性为97.8%。
2.5 高效共生固氮菌株筛选
植物叶片叶绿素含量能够定量反映出叶片氮含量水平,由表2可以看出,与对照组相比,接种YIC60933的花生叶片叶绿素含量虽然低于对照组但无显著差异,而其他实验组叶片叶绿素含量均有不同幅度增长。除接种YIC60756的实验组株高低于对照组外,其余植株高度均高于对照组。植株地上部位鲜重和干重可以直观反映花生生长发育期间的氮素积累情况,本研究中不同接菌处理的花生鲜重和干重均有不同程度增加,其中YIC60756、YIC61000、YIC61059的地上鲜重增长最为明显,增幅均在38.7%以上。地上部干重的结果与鲜重基本一致,但差异不显著。
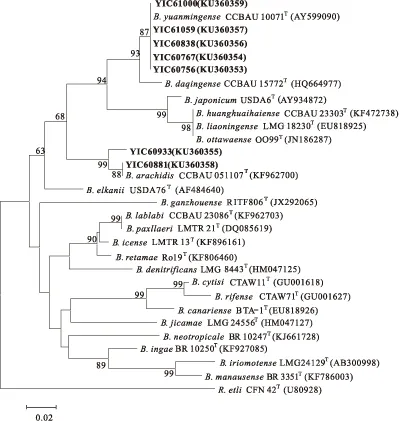
图 3 7株慢生根瘤菌代表菌株nifH基因系统发育分析Fig. 3 Phylogenetic tree of the 7 representative isolates and the reference strains based on nifH gene

菌株Strain叶绿素含量ChlorophyIIcontent株高Plantheight(cm)植株鲜重Freshshootweight(g)植株干重Dryshootweight(g)根瘤鲜重Nodulefreshweight(g)固氮酶活性ARA/TNFW(mmol·g-1·h-1)YIC6076746.28±2.8231.00±1.6312.47±2.54b*6.12±0.420.30±0.023.55±0.13YIC6075647.52±1.27a*26.67±3.4013.79±0.89b*6.15±0.780.23±0.034.43±0.21YIC6083846.82±2.15a*32.07±2.8212.97±3.47b*6.13±0.620.45±0.013.41±0.04YIC6105948.78±1.43a*29.20±3.5413.30±1.73b*6.01±0.090.24±0.026.95±0.15YIC6100043.42±0.8732.60±2.2413.72±1.32b*6.36±0.680.35±0.012.44±0.24YIC6088148.34±1.16a*32.67±1.8910.65±2.256.26±0.990.48±0.042.65±0.13YIC6093340.97±0.5833.60±2.4211.63±2.895.55±0.590.46±0.012.63±0.17CK43.30±1.4128.33±3.689.59±1.105.53±0.64 0 0
注:CK:对照;a*,b*:与对照组相比叶绿素含量和植株鲜重的T检验达到显著水平(p<0.05);ARA:乙炔还原法;TNFW:全部根瘤的鲜重;ARA/TNFW:固氮酶活性,以每克根瘤每小时产生的乙烯量(mmol)为衡量指标。*表示在0.05水平差异显著。
Note: CK, control; a*, b* indicate a significant increase in chlorophyll content plant and fresh shoot weight, respectively, according to t-test (p<0.05); ARA: Acetylene reduction assay; TNFW: the total nodule fresh weight; ARA/TNFW: It represents relative nitrogenase enzyme activity, mmol C2H4/(h·g nodule).
如表2所示,接种YIC60838、YIC60881和YIC60933的花生所结根瘤量最大。同时测定出固氮酶活性最高的3株接种菌株依次为YIC61059、YIC60756和YIC60767。对比发现,结瘤数最多的3株菌株并非固氮酶活性最高,因此,花生结瘤数量的多少不一定代表所结根瘤固氮能力的强弱。花生高效共生固氮根瘤菌的筛选应考虑花生生长指标和固氮能力各方面的因素,然后对菌株的促生效果做出综合评价。
3 讨 论
本研究采用持家基因recA聚类分析选取代表菌株、持家基因(atpD、recA和glnII)和固氮基因(nifH)系统发育分析等方法对分离自莱西地区的106株花生根瘤菌进行遗传多样性研究。持家基因recA聚类分析结果表明,莱西花生根瘤菌种群多样性较为丰富,106株待测菌株均分布于根瘤菌的Bradyrhizobium属中,共分为7个recA基因型,其中B.liaoningense分布范围最广、菌株数最多,为莱西市花生根瘤菌的优势种群,占分离菌株的98.1%。
结合16S rRNA基因序列和持家基因系统发育分析结果可以看出,7株代表菌株均属于Bradyrhizobium。其中代表菌株YIC60881的持家基因序列与亲缘关系最近的菌株相似性小于95%,故推测其有可能是新的种群,命名为Bradyrhizobiumsp.YIC60881。这还需要进一步的实验分析确认其分类地位。同时研究发现16S rRNA基因与持家基因的MLSA分析结果在属的水平上具有较好的一致性,但代表菌株YIC60881的聚类结果存在一些差异。YIC60881的16S rRNA基因序列与参比菌株B.cytisiCTAW11T聚群,相似性为99.6%,但在多位点序列分析结果中,其与参比菌株B.cytisiCTAW11T遗传距离较远,相似性仅90.9%。这种属内种间系统发育上的差异,表明属内种间存在广泛的基因交流。造成这种差异原因可能是由于16S rRNA基因序列高度保守不能有效区分不同种的根瘤菌,而MLSA在 97%相似性水平上能够有效确定根瘤菌的具体分类地位[18-19]。
固氮基因nifH编码固氮酶,它不仅与根瘤菌的固氮功能相关,同时也是植物与固氮微生物建立共生关系过程中必不可少的基因[20]。本研究发现相同菌株的16S rRNA基因与固氮基因在系统发育中存在明显差异的现象,如YIC60756、YIC61000、YIC61059、YIC60767与参比菌株B.liaoningenseUSDA 3622T的16S rRNA基因序列相似性为100%,而在固氮基因nifH的系统发育分析中这4株代表菌株则与B.yuanmingenseCCBAU 10071T亲缘关系最近并聚为一支,相似性为100%。虽然它们之间序列高度同源,但固氮基因nifH的系统发育表明花生根瘤菌种间可能存在水平基因转移。在先前的报道中就有关于固氮基因经过水平转移到其他的共生细菌中的研究,Barcellos等人研究表明B.japonicum的共生基因能够在宿主根瘤中转移给它的伴生菌[21]。Sullivan等人发现非共生根瘤菌也有可能通过接受共生基因而转化成共生菌株[22]。因此相同菌株的16S rRNA基因与共生基因在系统发育中的差异可能是根瘤菌为适应不同地理环境和宿主通过水平基因转移而产生了共生基因的遗传多样性。
根瘤菌与豆科植物形成的共生固氮体系,能固定空气中大量的氮气转化为氨供植物生长。高效共生固氮根瘤菌的应用能够降低花生种植成本,减轻化肥对环境和土壤的危害,对发展生态农业具有重要意义。本研究通过对分离自莱西地区的7株花生慢生根瘤菌代表菌株进行回接宿主和温室培养发现均能与原宿主花生有效结瘤,这与先前国内的研究结果一致[23-26]。其中代表菌株YIC61059表现出较好的固氮促生效果,叶绿素含量、地上部位干重分别提高12.66%、8.68%。本研究为高效花生根瘤菌剂的筛选提供了优质的种质资源,利用YIC61059生产成为可利用的高效花生根瘤菌剂还需要进一步小区及大田试验的应用效果研究。
[1] 李力,曹凤明,徐玲玫,等. 花生根瘤菌的抗逆性初步研究[J]. 微生物学通报,2000,27(1): 42-47.
[2] 孟瑶,徐凤花,孟庆有,等. 中国微生物肥料研究及应用进展[J]. 中国农学通报,2008,24(6): 276-282.
[3] Dobbelaere S, Vanderleyden J, Okon Y. Plant growth-promoting effects of diazotrophs in the rhizosphere[J]. Critical Reviews in Plant Sciences, 2003, 22(2): 107-149.
[4] 李士宁,孙明辉,周国,等. 莱西市花生生产现状和发展对策[J]. 花生科技,1999(增刊):109-110.
[5] Wang R, Chang Y L, Zheng W T, et al.Bradyrhizobiumarachidissp.nov.,isolated from effective nodules ofArachishypogaeagrown in China[J]. Systematic and Applied Microbiology, 2013, 36(2):101-105.
[6] Vincent J M. A manual for the practical study of the root-nodule bacteria[M]. Oxford: Blackwell Scientific,1970.
[7] 王蕊. 中国南方花生根瘤菌多样性及其与土壤因子相关性研究[D]. 北京:中国农业大学,2013.
[8] Yan J, Han X Z, Ji Z J, et al. Abundance and diversity of soybean-nodulating rhizobia in black soil are impacted by land use and crop management[J]. Applied and Environmental Microbiology, 2014, 80(17): 5394-5402.
[9] Zhang Y M, Tian C F, Sui X H, et al. Robust markers reflecting phylogeny and taxonomy of Rhizobia[J/OL]. PLoS ONE, 2012, 7(9): e44936 [2012-09-17]. http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0044936. doi:10.1371/journal.pone.0044936.
[10] Zhang C, Kong F. Isolation and identification of potassium-solubilizing bacteria from tobacco rhizospheric soil and their effect on tobacco plants[J]. Applied Soil Ecology, 2014, 82: 18-25.
[11] 窦雅静,陆俊锟,康丽华,等. 黑木相思根瘤菌遗传多样性[J]. 微生物学报,2012,52(12): 1439-1448.
[12] Islam M S, Kawasaki H, Muramatsu Y, et al.Bradyrhizobiumiriomotensesp.nov. , isolated from a tumor-like root of the legumeEntadakoshunensisfrom Iriomote Island in Japan[J]. Bioscience, biotechnology, and biochemistry, 2008,72 (6) :1416-1429.
[13] Maiden M C, Bygraves J A, Feil E. Multilocus sequence typing: A portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proc Natl Acad Sci,1998,95: 3140-3145.
[14] 孙杉杉,朱瑞艳,杜迎辉. 一株高效慢生型花生根瘤菌的筛选[J]. 安徽农业科学,2015,43(12):79-80,84.
[15] Cao Y, Wang E T, Zhao L, et al. Diversity and distribution of rhizobia nodulated withPhaseolusvulgarisin two ecoregions of China[J]. Soil Biology & Biochemistry, 2014, 7(78):128-137.
[16] Lopez-Guerrero M G, Ormeno-Orrillo E, Velazquez E, et al.Rhizobiumetlitaxonomy revised with novel genomic data and analyses[J]. Systematic and Applied Microbiology, 2012, 35(6): 353-358.
[17] 张丹. 中国北方花生主产区花生根瘤菌多样性和系统发育研究[D]. 北京:中国农业大学,2010.
[18] Haukka K, Lindstrom K, Peter J, et al. Diversity of partial 16S rRNA sequences among and within strains of African rhizobia isolated fromAcaciaandProsopis[J]. System. Appl Microbiol, 1996, 19: 352-359.
[19] Eardly B D, Wang F S, Van B P. Corresponding 16S rRNA gene segments onRhizobiaceaeandAeromonasyield discordant phylogenies[J]. Plant Soil, 1996, 186: 69-74.
[20] Laguerre G, Nour SM, Macheret V, et al. Classification of rhizobia based onnodCandnifHgene analysis reveals a close phylogenetic relationship amongPhaseolusvulgarissymbionts[J]. Microbiology-Uk, 2001, 147: 981-993.
[21] Barcellos F G, Menna P, da Silva Batista J S, et al. Evidence of horizontal transfer of symbiotic genes from aBradyrhizobiumelkaniiin a Brazilian savannah soil[J]. Appl Environ Microbiol, 2007, 73:2635-2643.
[22] Sullivan J T, Ronson C W. Evolution of rhizobia by acquisition of a 500-kb symbiosis island that integrates into a phe-tRNA gene[J]. Proceeding of the National Academy of Science of USA, 1998, 95(9) : 5145-5149.
[23] 张小平,陈强,李登煜,等. 花生根瘤菌在根瘤菌系统分类中的地位研究[J]. 微生物学报,1996(3): 227-233.
[24] 刘杰,汪玲玲,汪恩涛,等. 河北地区花生根瘤菌的系统发育多样性研究[J]. 中国农业科学,2006(2): 344-352.
[25] 常月立. 中国南方地区花生、扁豆根瘤菌的多相分类[D]. 北京:中国农业大学,2010.
[26] 陈文新,汪恩涛,陈文峰. 根瘤菌—豆科植物共生多样性与地理环境的关系[J]. 中国农业科学,2004(1):81-86.
Genetic Diversity of the Rhizobia and Screening of Effective Rhizobia Strains Isolated fromArachishypogaeaL. in Laixi County
WU Hai-long1,2, LI Yan1, CHEN Ming-na3, LIU Wei1, REN Cheng-gang1, YANG Jin-bao1,4, XIE Zhi-hong1*, YU Shan-lin3*
(1.YantaiInstituteofCoastalZoneResearch,ChineseAcademyofSciences,Yantai264003,China; 2.UniversityofChineseAcademyofSciences,Beijing100049,China; 3.ShandongPeanutResearchInstitute,Qingdao266100,China; 4.ShanxiAgriculturalUniversity,Taigu030801,China)
To reveal the genetic diversity of rhizobia isolated from breeding base of Shandong Peanut Research Institute (SPRI) and screen effective nitrogen-fixing bacteria strains, peanut rhizobia were collected and isolated from 5 separate samples in peanut breeding base, and all the isolates were selected to determine their phylogenetic relationship through analyses of the 16S rRNA gene, housekeeping genes (recA,atpDandglnII) and symbiotic genes (nifH). The growth-promoting efficiency was tested by plant inoculation assay on peanut in greenhouse. A total of 106 peanut rhizobium strains were isolated from 5 sampling sites. Through the phylogenetic analyses based onrecAsequences, all the isolates were classified to 7recAgenotypes, and 7 strains were selected as representative strains. The results of 16S rRNA and housekeeping gene MLSA phylogenetic analysis showed that all isolated strains belong toBradyrhizobium. Phylogeny analysis ofnifHshowed that the nitrogen-fixing genes is highly conserved. All the 7nifHsequences were subdivided to clade which related toB.arachidisCCBAU 051107TandB.yuanmingenseCCBAU 10071Trespectively, and thus indicated that horizontal gene transfer may existed among different peanut rhizobia species. Greenhouse experiments showed that 7 representative isolates were effective on symbiotic nitrogen fixation, among which YIC61059 showed the strongest symbiotic nitrogen-fixing capacity. It was found that severe genetic diversity existed in peanut rhizobia from Laixi Experimental Base of SPRI, whichBradyrhizobiumliaoningenseis the dominant species. Peanut rhizobia in this region showed perfect nodulation and symbiotic nitrogen fixation ability, one of the representative strains YIC61059 possessed prominent growth-promoting effect on peanut growth, and it has good prospects of application.
peanut; rhizobia; genetic diversity; high-efficient growth-promoting isolates
10.14001/j.issn.1002-4093.2016.02.001
2016-03-16
中科院重点部署项目(KZZD-EW-14);烟台市科技发展计划(2013JH021);中科院百人计划项目;国家自然科学基金(31370108,31570063);山东省自主创新及成果转化专项(2014ZZCX07303)
吴海龙(1990-),男,山东济宁人,硕士研究生,主要从事农业微生物方面研究。
*通讯作者:解志红(1976-),女,研究员,博士,主要从事根瘤菌与植物互作关系研究。E-mail: zhxie@yic.ac.cn
S565.2; Q939.11+4
A
禹山林(1956-),男,研究员,博士,主要从事花生遗传育种研究。E-mail: yshanlin1956@163.com
——固氮微生物

