柯萨奇病毒A6型VP1蛋白的生物信息学分析①
刘洪波 阳广菲 欧维琳 沈关心
(桂林医学院第二附属医院检验科,桂林541199)
柯萨奇病毒A6型VP1蛋白的生物信息学分析①
刘洪波 阳广菲②欧维琳③沈关心④
(桂林医学院第二附属医院检验科,桂林541199)
目的:预测柯萨奇病毒A组6型(Coxsackievirus A6,CVA6)的衣壳蛋白VP1的基本理化性质、结构功能及线性B细胞表位。方法:应用Bioedit软件、SubLoc、TargetP和生物信息学资源门户ExPASy中的多种在线工具对CVA6 VP1的氨基酸序列进行分析。结果:CVA6 VP1为一亲水性蛋白,其相对分子量为33.6 kD,等电点为7.92,含有24个可能的磷酸化位点,没有信号肽、跨膜区和可能的脂酰化位点;其二级结构中以无规则卷曲居多,有48.52%的氨基酸残基暴露于溶液界面;该分子内存在多个潜在的线性B细胞表位,其中的155~165位氨基酸残基区域的抗原指数最高。结论:成功预测到CVA6 VP1的基本理化性质、结构功能特征及可能的线性B细胞表位,为该蛋白的进一步研究及疫苗和免疫诊断试剂的研制打下基础。
柯萨奇病毒A组6型;VP1蛋白;生物学信息;B细胞表位;理化性质
手足口病(Hand,foot and mouth disease,HFMD)是儿童常见传染病,其病原包括柯萨奇病毒(Coxsackievirus,CV)、埃柯病毒和新型人肠道病毒(Human enterovirus,HEV)的各部分成员,EV71型和CV A组(CVA)16型为最常见[1,2]。然而,近年其他HEV感染病例有增多趋势[3]; CVA6型在多个国家和地区引起HFMD的暴发流行,也成为HFMD常见病原之一,并且可导致重症发生[4]。目前,鲜有针对该病原的基础性研究。
HEV的VP1具有与其血清型相对应的遗传多样性[5],是其中和表位存在的最主要区域[6],也是其毒力可能相关的区域之一[7],具有重要研究价值。本研究运用生物信息学方法,对CVA6代表株VP1的基本理化性质、结构功能特征和含有的线性B细胞表位进行预测分析,为该病原的生物学特性研究以及疫苗和诊断试剂的研发提供理论基础。
1 材料与方法
1.1 CVA6 VP1氨基酸序列和生物信息学工具 从NCBI数据库检索并下载CVA6的全长VP1氨基酸序列,选择Gdula株(protein ID为AAD17701.1)的VP1序列为代表进行分析。除Bioedit软件外,本研究主要利用SubLoc V1.0、TargetP 1.1和生物信息学资源门户网站ExPASy(http://www.expa-sy.org/)提供的多种在线工具对VP1进行分析。这些在线工具的网址链接、参考文献和选择的参数见表1。
1.2 CVA6 VP1的理化性质预测 利用Bioedit对VP1的氨基酸组成进行分析;利用ProtParam预测相关VP1的原子组成、等电点、消光系数、不稳定系数以及在哺乳动物网织红细胞、酵母和大肠杆菌中的半衰期等;利用ProtScale中Kyte和Doolittle方法预测共疏水性/亲水性。
1.3 CVA6 VP1的结构功能和B细胞表位等的预测 利用SignalP预测VP1蛋白的信号肽;利用Tmpred和TMHMM进行跨膜螺旋预测;利用NetPhos预测磷酸化位点;利用Net-NGlyc和MotifScan预测糖基化、酯酰化等修饰位点,并用Big-PI Predictor预测糖基磷脂酰肌醇(Glycosyl phosphatidyl inositol,GPI)修饰位点;利用SubLoc V1.0和TargetP 1.1对VP1蛋白的亚细胞定位进行预测,并利用NetNES进行核定位信号分析;利用InterProScan、SMART和PROSITE对VP1一级结构中包含的结构和功能域特征序列进行分析;利用SOPMA和Predictprotein预测蛋白质VP1的二级结构和拓扑结构等;运用Bepipred预测VP1中可能的B细胞线性表位。
2 结果
2.1 理化性质
2.1.1 氨基酸的组成 应用Bioedit软件对CVA6 VP1的氨基酸组成进行分析发现,由305个氨基酸组成的VP1中Ala(28个,9.2%)、Ser(28个,9.2%)、Thr(28个,9.2%)和Val(25个,8.2%)含量较多;所含碱性氨基酸和酸性氨基酸总数分别为26和25,分别占总氨基酸数的8.5%和8.2%(见图1)。
2.1.2 基本理化性质 利用ProtParam工具预测了VP1蛋白的基本理化参数。该蛋白的原子组成为C1478H2269N415O461S12 ,相对理论分子量为33.6 kD,等电点为7.92。假设全部胱氨酸残基以Cys形式形成二硫键,则其在水溶液中(A280nm)的摩尔消光系数为44 475 L/(mol·cm);假设所有二硫键打开,其在水溶液中(A280nm)的摩尔消光系数则为44 350 L/(mol·cm)。若VP1 N端为Asn,其在哺乳动物体外网织红细胞中的半衰期为1.4 h,在酵母体内为3 min,在大肠杆菌内大于10 h。其不稳定系数为43.50,属于不稳定蛋白。
表1 本文使用工具
Tab.1 Tools used in this paper

ToolsnameWebsiteReferenceBig-PIPredictorhttp://mendel.imp.ac.at/sat/gpi/gpi_server.htmlTrendsBiochemSci,2000,25(7):340-341[J].Bepipredhttp://tools.immuneepitope.org/bcell/ImmunomeRes,2006,2:2[J].InterProScanhttp://www.ebi.ac.uk/interpro/scan.html/Bioinformatics,2014,30(9):1236-1240[J].MotifScanhttp://myhits.isb-sib.ch/cgi-bin/motif_scanUnpublishedNetPhoshttp://www.cbs.dtu.dk/services/NetPhos/JMolBiol,1999,294(5):1351-1362[J].Net-NGlychttp://www.cbs.dtu.dk/services/NetNGlyc/UnpublishedNetNEShttp://www.cbs.dtu.dk/services/NetNESProteinEngDesSel,2004,17(6):527-536[J].ProtParamhttp://www.expasy.ch/tools/protparam.htmlTheProteomicsProtocolsHandbook,2005,571-607[M].ProtScalehttp://www.Expasy.org/cgi-bin/protscale.plTheProteomicsProtocolsHandbook,2005,571-607[M].Predictproteinhttps://www.predictprotein.org/homeNucleicAcidsRes,2014,42:W337-43[J].PROSITEhttp://au.expasy.org/prosite/NucleicAcidsRes,2006,34:W362-365[J].SOPMAhttp://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.htmlBMCBioinformatics,2006,7(1):255[J].SignalPV2.0.b2http://www.cbs.dtu.dk/services/SignalP-2.0/#submis-sionProcIntConfIntellSystMolBiol,1998,6:122-130[C].SubLochttp://www.bioinfo.tsinghua.edu.cn/SubLoc/Bioinformatics,2001,17(8):721-728[J].SMARThttp://smart.embl-heidelberg.de/NucleicAcidsRes,2015,43:D257-D260[J].Tmpredhttp://www.ch.embnet.org/software/TMPRED_form.htmlBiolChemHoppe-Seyler,1993,374:166[J].TMHMMhttp://www.cbs.dtu.dk/services/TMHMM-2.0/Bioinformatics,2001,17(7):646-653[J].TargetP1.1http://www.cbs.dtu.dk/services/TargetP/JMolBiol,2000,300(4):1005-1016[J].
Note:Tools are listed in alphabetical order;In this study,all tools were run in their default settings except MotifScan,which selected “mot_source:freq_pat” for application;The references for these tools were presented with some essential information for retrieval,but no publications are so far available for the two tools,MotifScan and Net-NGlyc.
2.1.3 疏水性分析 使用ProtParam预测VP1 氨基酸序列平均亲水性,其脂肪指数61.08,GRAVY值为-0.459(范围介于2和-2之间,正值表明为疏水性强,负值表明为亲水性强)。运用ProtScale 中Kyte和Doolittle法预测VP1氨基酸序列的疏水性,从结果(图2)可以看出,在整个肽链中,亲水性氨基酸均匀分布,远多于疏水性氨基酸;多肽链第161位的Arg分值-2.600最低(亲水性最强),第87位的Gly分值2.011最高(疏水性最强)。这些结果提示,VP1蛋白脂溶性差,亲水性好,为一可溶的亲水性蛋白。
2.2 功能相关序列/结构分析
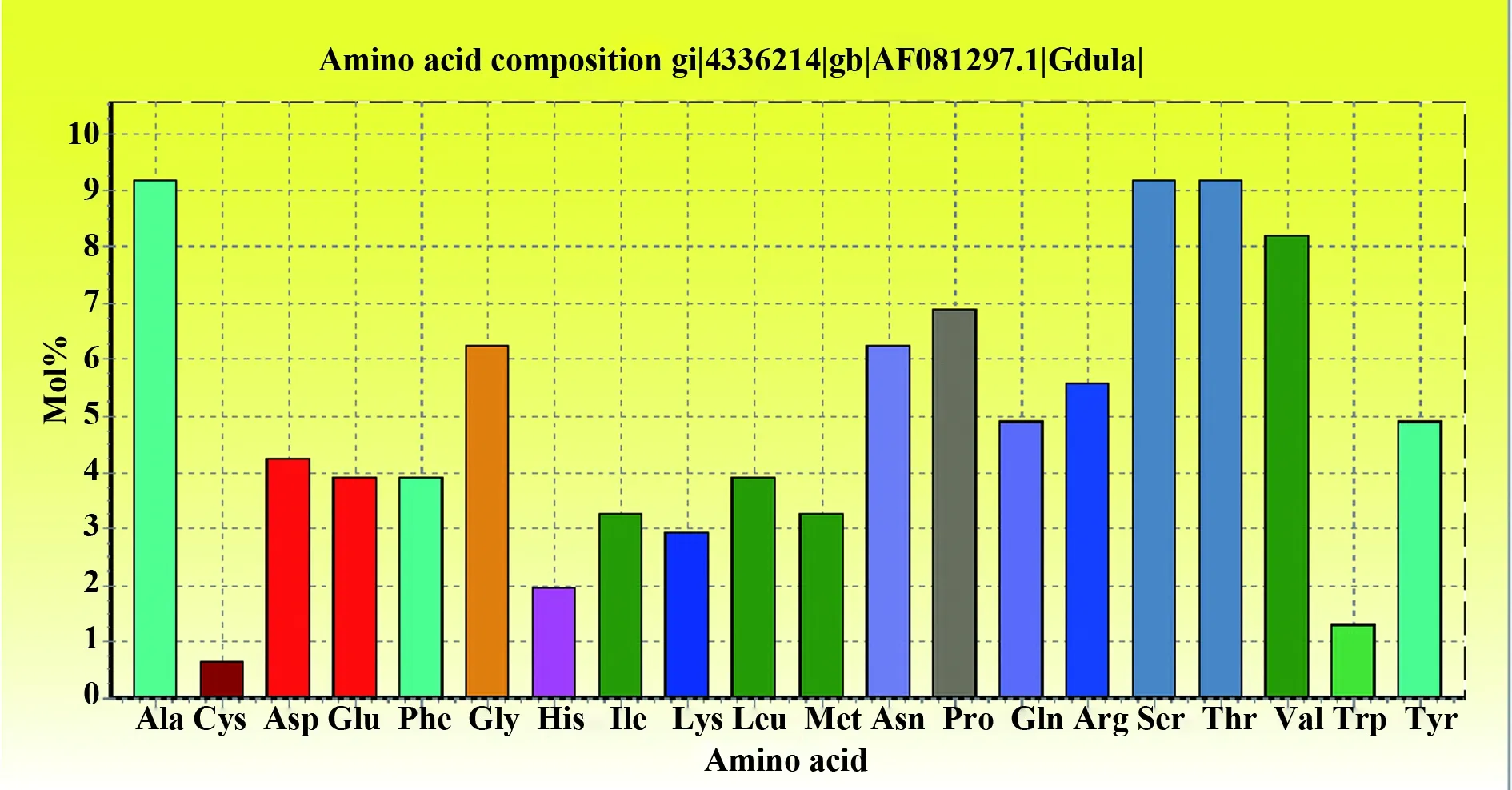
图1 CVA6 VP1的氨基酸组成分布图Fig.1 Amino acid composition of CVA6 VP1

图2 CVA6 VP1的疏水性分析Fig.2 Hydrophobicity analysis of CVA6 VP1
2.2.1 信号肽 应用Signal P对VP1蛋白进行信号肽分析,信预测值为0.002,远低于cutoff值0.5(图3),提示VP1为非分泌性蛋白。
2.2.2 跨膜螺旋 使用Tmpred预测,其分值小于0,没有发现可能的模型;用TMHMM预测,VP1整条肽链均处于膜外(图4)。据此推测,VP1不存在跨膜结构域。

图3 SignalP预测CVA6 VP1信号肽Fig.3 Prediction of CVA6 VP1 signal peptide by SignalP
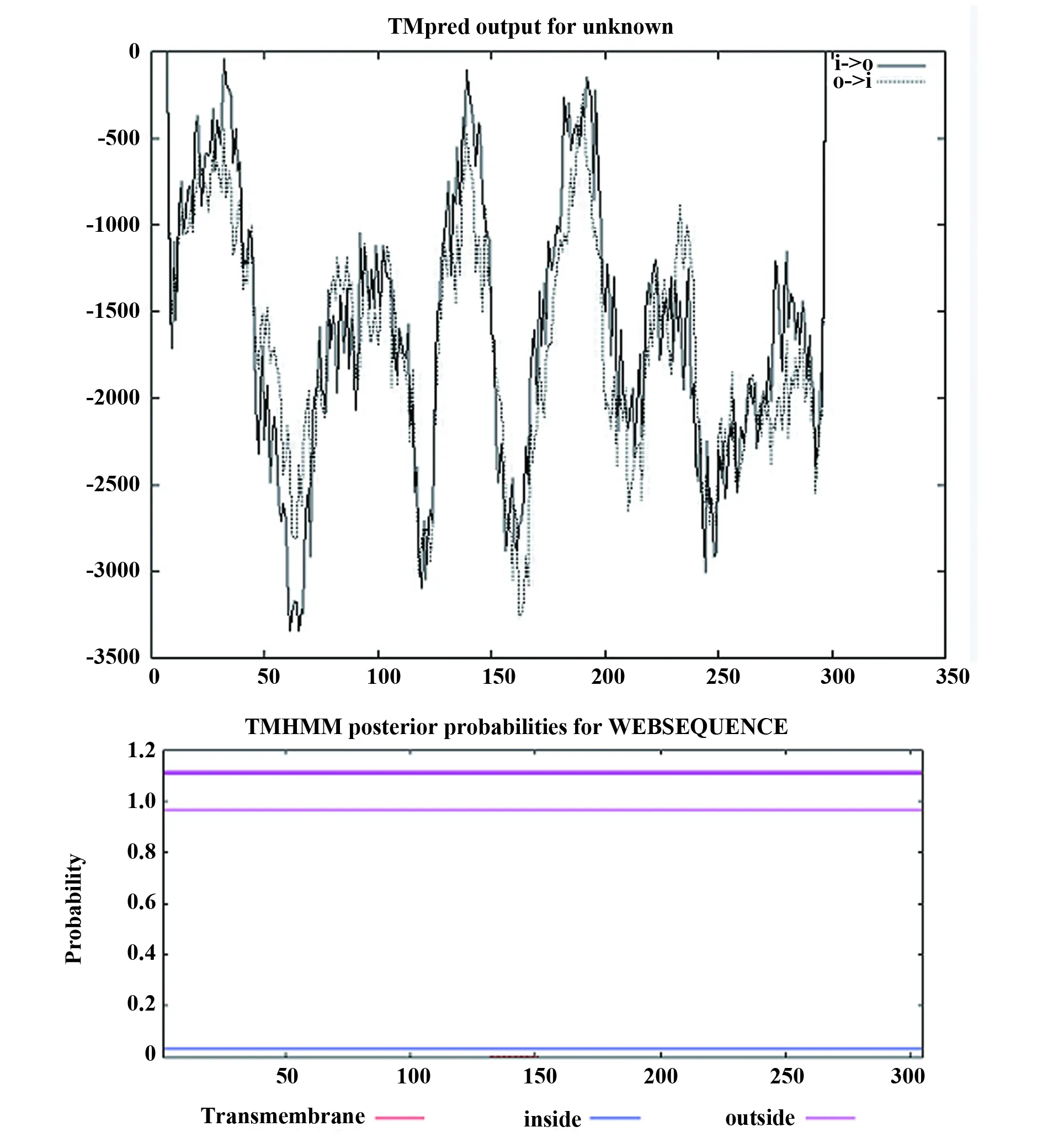
图4 CVA6 VP1的跨膜螺旋预测Fig.4 Prediction of transmembrane helices in CVA6 VP1
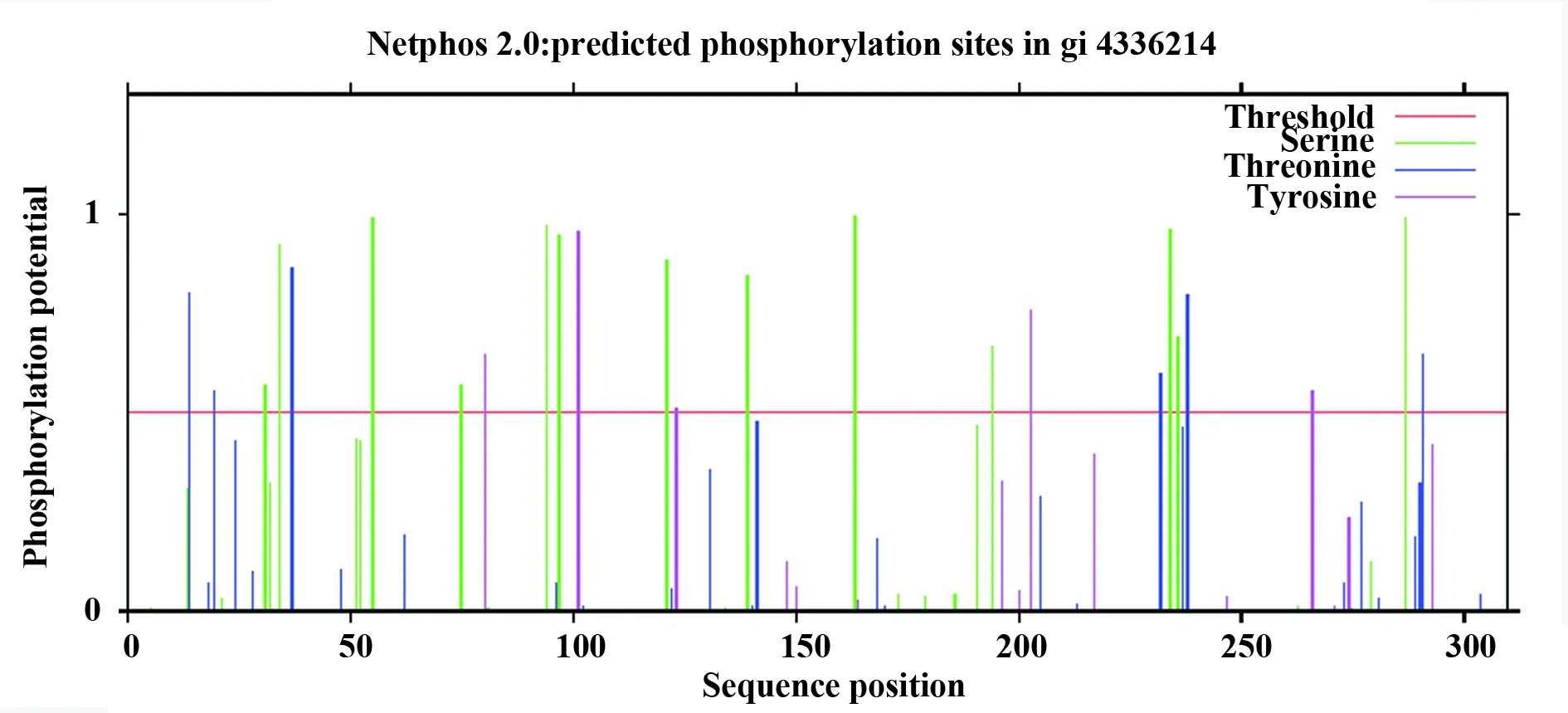
图5 CVA6 VP1的磷酸化位点预测Fig.5 Prediction of phosphorylation sites in CVA6 VP1

图6 CVA6 VP1的糖基化修饰位点预测Fig.6 Prediction of glycosylation sites in CVA6 VP1

图7 CVA6 VP1的核定位信号分析Fig.7 Analysis of nuclear localization signal in CVA6VP1
2.2.3 磷酸化位点 应用NetPhos对VP1进行分析的结果显示,VP1蛋白中共有24个可能的磷酸化位点,13个位于Ser,6个位于Thr,5个位于Tyr 残基(见图5)。
2.2.4 酯酰化和糖基化位点 用MotifScan预测,在VP1中没有找到可能的脂酰化修饰位点;以Big-PI预测也证实,VP1中无可能的GPI锚定位点。用Net-NGlyc预测,在VP1中发现可能的糖基化位点有3个,分别位于第53、137、171位氨基酸残基(见图6)。然而,由于VP1不含信号肽序列,因此几乎不可能产生糖基化修饰。
2.2.5 亚细胞定位 利用SubLoc预测,结果显示VP1可能定位在细胞核上。而利用TargetP预测,结果显示VP1 线粒体靶向肽(mTP)和分泌通路信号肽(SP)数值都不高,定位于其他任何位置均有可能。以NetNES进行核定位信号分析,各值均未超过阈值,提示该蛋白不含核定位信号(见图7)。各工具预测结果不同,可能是不同工具利用的数据库不同而产生的差异,说明针对病毒蛋白亚细胞定位的数据库或预测工具尚不成熟,其亚细胞定位需以实验验证。

图8 InterProScan预测CVA6 VP1蛋白一级结构中的保守功能域Fig.8 Prediction of conserved function domains in primary structure of CVA6 VP1 by InterProScan

图9 CVA6 VP1的拓扑结构、二级结构和亲水性特征Fig.9 Characteristics of topological structure,secondary structure and hydrophilicity of CVA6 VP1
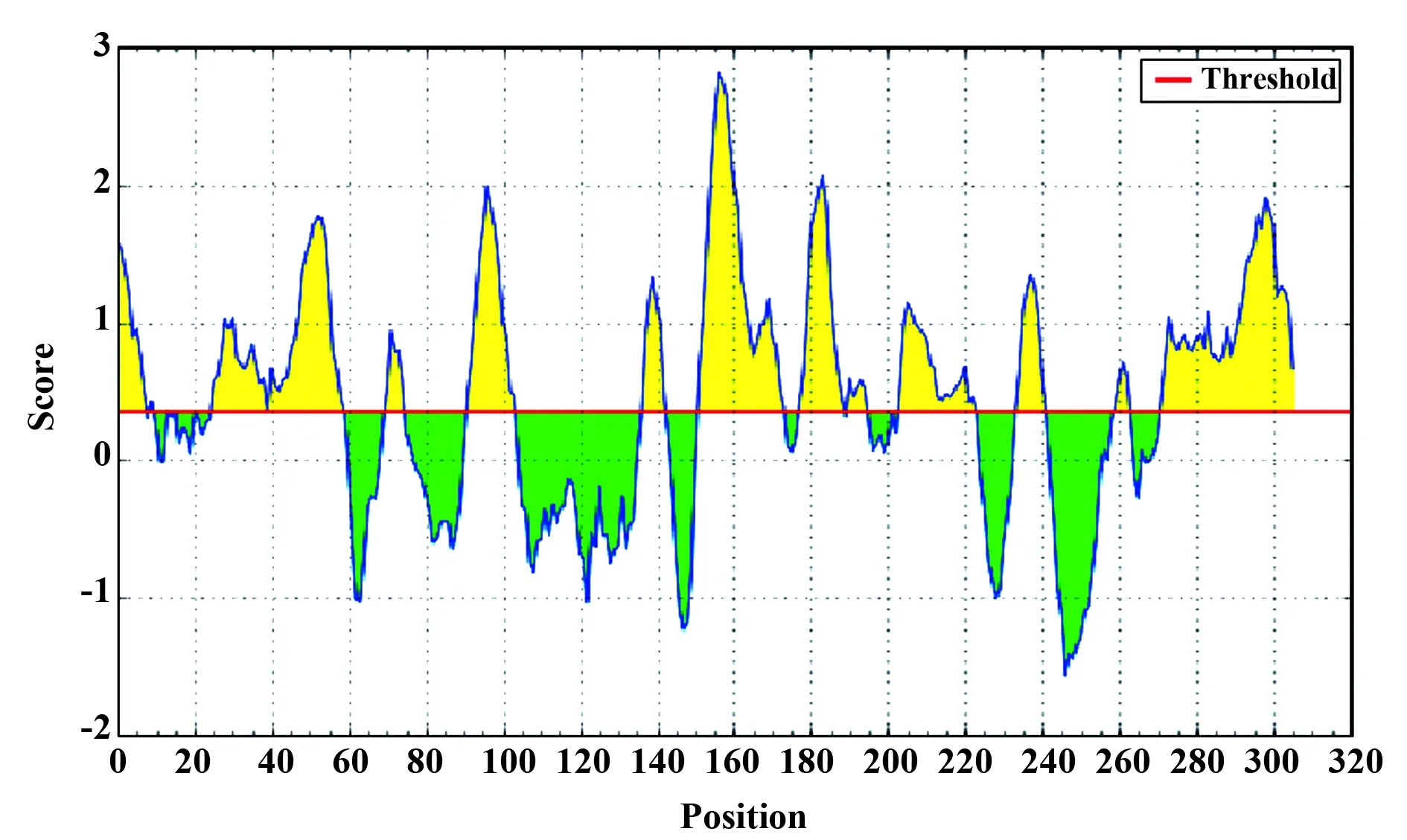
图10 Bepipred工具预测CVA6 VP1的线性B细胞表位Fig.10 Prediction of linear B-cell epitope of CVA6 VP1 by tool Bepipred
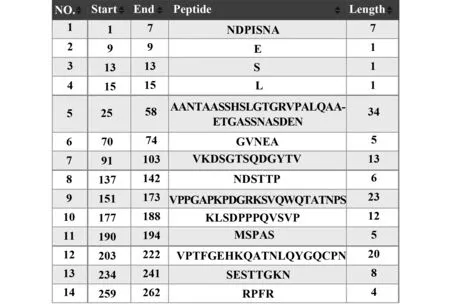
图11 CVA6 VP1 潜在的线性B细胞表位位点Fig.11 Potential linear B-cell epitope sites on CVA6 VP1
2.2.6 一级结构中所含结构和功能域特征序列分析 对VP1序列使用InterProScan分析的结果显示,VP1含有2个结构和功能域,一个是位于VP1中间与RhV具有同源性的小RNA病毒蛋白结构域,另一个是与DUF3724具有同源性的未知功能的RNA病毒蛋白结构域; 除VP1左端外的大部分序列属于SSF88633超家族,含有非整合信号(见图8)。以SMART预测发现,VP1序列第56~217位区域与Pfam数据库中RhV结构域相似,第262~284位与DUF3724结构域相似。而运用PROSITE预测,未发现存在特异性的结构和功能域。
2.3 二级结构特征 以SOPMA预测,VP1中α-螺旋比例为27.87%,β-折叠为24.92%,β-转角为8.52%,无规则卷曲为38.69%。以Predictprotein预测,α-螺旋比例为9.84%,β-折叠为25.90%,无规则卷曲为64.26%(见图9)。结合两种方法预测的结果可知,α-螺旋和β-折叠散布于整个VP1蛋白,成为蛋白的刚性支撑;而无规则卷曲为其中比例最高的结构元件,间杂少量的β-转角,为蛋白的柔性区域,是抗原表位存在的可能部位。Predictprotein预测也显示了VP1中每个氨基酸的溶剂可及性,其中48.52%的氨基酸残基暴露于溶液界面,44.26%的氨基酸残基包埋于蛋白质内部,其余7.21%的氨基酸处于中间态(见图9)。
2.4 线性B细胞表位 运用Bepipred 法预测B细胞线性表位,结果显示VP1 蛋白平均抗原性指数为0.442,分子内共有多个可能的抗原表位区域(图10)。其中,分值最高的表位区域为第151-173位氨基酸残基区段,分值达2.82(阈值0.350);其次为第177-188,91-103,203-222和25-58位氨基酸区段(按分值高低排列),其余位点见图11。
3 讨论
在基因组和蛋白质组学时代,运用生物信息学工具分析蛋白质序列已成为蛋白质研究的重要手段[8]。蛋白质理化性质预测可以用于蛋白质分离鉴定的实验设计等,如分子量、净电荷和亲和力数据为蛋白质色谱分离提供先验信息;蛋白质序列比对分析可以了解其进化和可能的结构和功能特征等。这些为蛋白质的进一步研究提供诸多信息,可大大提高蛋白质相关研究的工作效率。CVA6与EV71同属于HEV A组成员,亲缘关系很近,它们的VP1有着较高的序列一致性[9],因此已有的诸多EV71 VP1相关研究可为CVA6 VP1的生物信息学分析提供预示信息[6,10-12]。比较俞海洋等[10]的研究结果,我们发现,CVA6和EV71的VP1蛋白具有很多相同或相似的理化和结构特征,例如,两者均为可溶的亲水性蛋白,无信号肽和跨膜螺旋结构,均存在与RhV相似的结构域,约50%氨基酸残基暴露于溶液界面,二级结构以无规则卷曲为主等。这些相同或相似的理化和结构也预示它们VP1可能存在相似的功能。蛋白质的可逆磷酸化被认为是细胞调节和信号转导的最频繁和通用的机制[13]。病毒蛋白可以通过磷酸化/去磷酸化过程干扰宿主细胞的信号转导,也使自身的生物学功能发生改变[14,15]。有研究证实,EV71可通过VP1激活细胞钙调蛋白依赖的蛋白激酶II而对宿主细胞信号转导和EV71自身的复制产生影响[11]。本研究预测发现,与EV71 VP1相似[10],CVA6的VP1中也存在较多的磷酸化位点。因此我们推测,CVA6 VP1也是影响其自身复制循环和致病的重要分子。
运用生物信息学方法预测蛋白质的抗原优势表位,并通过体外合成肽段进行实验验证,已广泛应用于蛋白质抗原表位的鉴定[16]。通过该方法,已成功鉴定出包括EV71病毒在内的多种病原体抗原表位[6,16]。Bepipred方法是结合线性B细胞表位预测的一种经典算法和最佳预测模型-隐马尔科夫模型发展的一种改良的倾向量表法;以HIV数据集验证,其曲线下面积优于多种传统方法[17]。本研究利用该方法预测发现,CVA6 VP1分子的抗原性很强(抗原性指数为0.442),可能的线性B细胞表位分布于各个区段。与本文的其他预测结果非常相符,如较高的极性氨基酸比例、较强的亲水性和较多的无规则卷曲结构等。另外,本研究预测到CVA6的可能B细胞表位25-58和203-222区段与已经实验证实的EV71的SP12和SP70表位核心区域重叠且存在较多保守性氨基酸残基[12]。结果提示,该两肽段含B细胞表位的可能性较大,且EV71和CVA6之间可能存在交叉反应性;另一方面,这些表位中氨基酸残基变异亦可能是导致这些病原抗原性转变或其抗体亲合力下降的结构基础[18],这些值得进一步研究。
总之,本研究对CVA6的主要衣壳蛋白VP1的生物信息学分析,成功预测到其基本的理化性质、结构和功能特征以及潜在的线性B细胞表位,这将有助于对其VP1蛋白的深入研究以及为疫苗和免疫诊断试剂的研制奠定理论基础。
[1] 郑 媛,周步宇,关路媛,等.2009-2013年陕西省手足口病流行病学及病原学特征分析[J].中华疾病控制杂志,2015,19(2):135-137.
[2] 海 岩,李忠延,跃 华,等.内蒙古2008—2013年手足口病疫情分析[J].中国公共卫生,2015,31(1):29-31.
[3] 田树萍,何 菡,王文静,等.2011-2013年手足口病病原谱及临床特征分析[J].北京医学,2014,36(6):447-449.
[4] Bian L,Wang Y,Yao X,etal.Coxsackievirus A6:a new emerging pathogen causing hand,foot and mouth disease outbreaks worldwide[J].Expert Review of Anti-infective Therapy,2015,13(9):1061-1071.
[5] Brown BA,Oberste MS,Alexander JP,etal.Molecular epidemiology and evolution of enterovirus 71 strains isolated from 1970 to 1998[J].J Virol,1999,73(12):9969-9975.
[6] Kirk K,Poh CL,Fecondo J,etal.Cross-reactive neutralizing antibody epitopes against Enterovirus 71 identified by an in silico approach[J].Vaccine,2012,30(49):7105-7110.
[7] Yang E,Zhao H,Zhang Y,etal.A comparative study of the characteristics of two Coxsackie A virus type 16 strains(genotype B)[J].Sci China Life Sci,2012,55(4):336-342.
[8] Gupta PS,Banerjee S,Islam RN,etal.PHYSICO:An UNIX based Standalone Procedure for Computation of Individual and Group Properties of Protein Sequences[J].Bioinformation,2014,10(2):105-107.
[9] Oberste MS,Penaranda S,Maher K,etal.Complete genome sequences of all members of the species human enterovirus A[J].J Gen Virol,2004,85(Pt 6):1597-1607.
[10] 俞海洋,陈 伟,倪德群,等.肠道病毒71型六安地区分离株VP1基因的生物信息学分析及B细胞表位预测[J].安徽医科大学学报,2012,47(3):233-240.
[11] Haolong C,Du N,Hongchao T,etal.Enterovirus 71 VP1 activates calmodulin-dependent protein kinase II and results in the rearrangement of vimentin in human astrocyte cells[J].PLoS One,2013,8(9):e73900.
[12] Foo DG,Alonso S,Phoon MC,etal.Identification of neutralizing linear epitopes from the VP1 capsid protein of Enterovirus 71 using synthetic peptides[J].Virus Res,2007,125(1):61-68.
[13] Cesaro L,Pinna LA.The generation of phosphoserine stretches in phosphoproteins:mechanism and significance[J].Mol Biosyst,2015,11(10):2666-2679.
[14] Schulze zur Wiesch J,Schmitz H,Borowski E,etal.The proteins of the Hepatitis C virus:their features and interactions with intracellular protein phosphorylation[J].Archives Virol,2003,148(7):1247-1267.
[15] Ohman T,Soderholm S,Paidikondala M,etal.Phosphoproteome characterization reveals that Sendai virus infection activates mTOR signaling in human epithelial cells[J].Proteomics,2015,15(12):2087-2097.
[16] He L,Zhu J.Computational tools for epitope vaccine design and evaluation[J].Curr Opinion Virol,2015,11:103-112.
[17] Larsen JE,Lund O,Nielsen M.Improved method for predicting linear B-cell epitopes[J].Immunome Res,2006,2:2.
[18] Liu CC,Chou AH,Lien SP,etal.Identification and characterization of a cross-neutralization epitope of Enterovirus 71[J].Vaccine,2011,29(26):4362-4372.
[收稿2015-09-06 修回2015-09-15]
(编辑 许四平)
Bioinformatics analysis of VP1 protein of coxsackievirus A6
LIU Hong-Bo,YANG Guang-Fei,OU Wei-Lin,SHEN Guan-Xin.Department of Laboratory Medicine,the Second Affiliated Hospital of Guilin Medical University,Guilin 541199,China
Objective:To predict the basic physicochemical properties,structure and function,and linear B-cell epitopes of the capsid protein VP1 of coxsackievirus A6(CVA6).Methods: The amino acid sequence of the CVA6 VP1 was analyzed using Bioedit software and various online tools including SubLoc,TargetP and the others from ExPASy Bioinformatics Resource Portal.Results: The CVA6 VP1 protein was a hydrophilic protein with a relative molecular weight of 33.6 kD and an isoelectric point of 7.92.This protein containsed 24 phosphorylation sites,but no signal peptide,transmembrane domains and possible fatty acylation sites.Its secondary structure was characterized by the richest random coils,and 48.52 percent of its amino acid residues exposed at the solution interface.Epitope prediction by Bepipred showed a number of potential B cell epitopes in the protein,the highest antigenicity index among them located in the region of amino acids residue 155-165.Conclusion: The basic physicochemical properties,structure and function characteristics,and potential linear B-cell epitopes of CVA6 VP1 were successfully predicted,which laid foundations for the further study on the protein and the preparation of vaccines and immunological diagnostic reagents for CVA6 infection.
Coxsackievirus A6;VP1 protein;Bioinformatics;B-cell epitope;Physicochemical property
10.3969/j.issn.1000-484X.2016.04.019
①本文为国家自然科学基金项目(81460304;81360248)和广西自然科学基金(2015GXNSFDA139020)和广西壮族自治区卫生厅科研课题(Z2014298)。
刘洪波(1973年-),男,博士,副主任技师,硕士生导师,主要从事新型疫苗、分子流行病学和分子诊断技术研究,E-mail:hbliu@glmc.edu.cn。
R373.2+3
A
1000-484X(2016)04-0536-06
②并列第一作者,桂林医学院药学院临床药学, 桂林541004。
③桂林医学院附属医院儿科,桂林528403。
④华中科技大学同济医学院免疫学系,武汉430030。
指导教师:沈关心(1952年-),男,硕士,教授,博士生导师,主要从事分子免疫与免疫相关疾病研究。