新疆双峰驼纳米抗体噬菌体展示文库多样性的生物信息学分析
冯亚宁,姜志强,马晓玲,李江伟
(1.新疆大学生命科学与技术学院/新疆自治区生物资源基因工程重点实验室,乌鲁木齐 830046;2 .新疆生产建设兵团第十二师104团牧一场,乌鲁木齐 830076)
新疆双峰驼纳米抗体噬菌体展示文库多样性的生物信息学分析
冯亚宁1,姜志强2,马晓玲1,李江伟1
(1.新疆大学生命科学与技术学院/新疆自治区生物资源基因工程重点实验室,乌鲁木齐 830046;2 .新疆生产建设兵团第十二师104团牧一场,乌鲁木齐 830076)
【目的】骆驼免疫系统中只存在重链抗体,其可变区VHH片段是最小的天然抗体片段。通过对双峰驼VHH基因进行序列分析,了解双峰驼VHH基因特征,分析VHH文库多样性组成。【方法】采用新疆双峰驼VHH特异引物扩增全部抗体基因片段,连接到M13噬菌粒载体上,与pIII蛋白融合表达在噬菌体表面,构建噬菌体展示文库。基因测序和菌落PCR方法,分析文库大小和阳性克隆插入率。NCBI-IgGBLAST工具对抗体序列进行IMGT编号命名,WebLogo工具分析抗体保守序列分布频率,MEGA6软件对VHH序列进行同源性比对和系统进化树构建。【结果】利用免疫的新疆双峰驼淋巴细胞,构建了库容量为1.4×109的VHH抗体噬菌体展示文库。对91个随机挑选的文库TG1单菌落进行PCR。结果表明该文库VHH阳性插入率为97%,DNA测序表明文库多样性为97.8%。对91个克隆DNA测序结果进一步分析表明,这些基因中除2个为常规VH抗体外,其余均为重链VHH抗体。根据抗体序列中的特征氨基酸分布,将91个抗体序列划分为7个亚群。【结论】构建的新疆双峰驼VHH文库多样性较好,生物信息学分析揭示出文库中多样性克隆的分布和特征,有助于新疆双峰驼纳米抗体噬菌体展示文库的进一步构建。
双峰驼;单域抗体;噬菌体展示文库;VHH序列;生物信息学分析
0 引 言
【研究意义】骆驼科动物免疫系统中存在天然缺失轻链的重链抗体,为单一的折叠单元,保留了完整的抗原结合活性,由骆驼重链可变区结合抗原的单一结构域构成的抗体为单域重链抗体(variable domain of the heavy-chain of heavy-chain antibody,VHH),又称为纳米抗体,是目前天然来源分子最小的抗体[1]。鲨鱼体内也存在一种由重链同源二聚体组成的天然小分子抗体称作免疫球蛋白新抗原受体(immunoglobin new antigen receptor, IgNAR)[2]。近年来,VHH在疾病诊断、分子检测和治疗方面的应用受到了研究者的广泛关注[3]。当前,纳米抗体主要通过噬菌体展示技术获得。噬菌体展示抗体库是指利用B淋巴细胞抗体全套可变区基因,通过噬菌体展示技术,把抗体或抗体片段( Fab、Fv或scFv)表达在噬菌体表面的噬菌体群体构建成噬菌体抗体库,是制备大多数的基因工程抗体的主要方法[4]。通过亲和淘洗技术可以从该文库中筛选获得抗原特异的抗体。然而抗体库的质量,包括文库的大小、阳性克隆插入率和文库多样性等决定了获得的抗体的亲和力和活性[5]。因此,在对文库亲和筛选前,对文库质量的鉴定和评价尤为重要。【前人研究进展】对文库质量进行评价的主要方法是对文库获得的克隆进行PCR和DNA测序分析。主要的评价指标为库容量(library size)和多样性(diversity)[6]。尽管库容量对筛选获得抗体的高亲和力有很大影响,但文库的多样性决定了能否获得抗原特异的抗体,尤其是在非免疫文库中获得针对任何抗原的抗体。由于在文库构建过程中采用不同引物和不同的PCR扩增方法,导致构建文库的代表性和序列正确性都有很大不同。【本研究切入点】全面分析文库多样性对于文库质量的评估至关重要[7]。一个良好的抗体文库除具有高的多样性外,还需要多样性的CDR3长度分布和氨基酸组成,并且文库中能够涵盖的抗体亚群尽可能多。在进行文库筛选之前,利用生物信息学分析手段系统分析文库序列信息,有助于制定合理的文库筛选策略。【拟解决的关键问题】探讨新的文库多样性分析方法,研究以构建自非免疫骆驼的VHH抗体噬菌体展示文库为起始材料,采用序列测定结合生物信息学分析等方法对文库VHH序列进行系统分析,初步阐明新疆双峰驼VHH序列特征和文库的多样性。
1 材料与方法
1.1 材 料
采集5峰新疆双峰驼外周血约50 mL。利用淋巴细胞分离液分离淋巴细胞。Trizol试剂提取淋巴细胞总RNA。以Oligo(dT)为引物,反转录合成cDNA。分别采用两轮PCR扩增VHH基因片段。第一轮PCR引物为:Call01, 5’-GTCCTGGCTGCTCTTCTACAAGG-3’,Call02,5’-CTAGTGCGGCCGCTGAGGAGACGGTGACCTGGG T-3’;第二轮PCR引物为:VHH-back,5’-GGTACGTGCTGTTGAACTGTTCC-3’, 和 PMCF 5’-GATGTGCAGCTGCAGGAGTCTGGRGGAGG-3’。采用SfiⅠ分别酶切pCANTAB 5E质粒和基因片段,回收后进行连接反应。连接产物电击转化TG1感受态细胞,从而获得VHH抗体基因文库。VHH噬菌体展示文库的制备按文献[8]方法采用M13K07辅助噬菌体感染TG1文库细菌,分离和纯化噬菌体获得。
1.2 方 法
1.2.1 文库克隆的Sanger测序和菌落PCR
随机挑取91个文库TG1单菌落,采用含有VHH基因上游引物MP57(5’-ATGCTTCCGGCTCGTATG-3’),送华大基因公司进行Sanger测序。菌落PCR采用pCANTAB 5E载体上的两条引物MP57和GIII(5’-CCACAGACAGCCCTCATAG-3’),用牙签挑取微量100个菌落,在PCR管中直接进行PCR扩增。
1.2.2 测序结果的生物信息学分析
Sanger测序结果采用NCBI-IgGBLAST进行序列分析,以划分VHH抗体序列和进行氨基酸位点IMGT编号及CDR序列识别。VHH序列相似性采用MEGA6软件进行多重比对。VHH序列亚群的分类采用Harris[9]方法进行,以序列中决定抗体结构的关键氨基酸的种类和出现频率为分类依据。各亚群抗体保守序列分布频率采用WebLogo工具[10]进行分析和可视化显示。
1.2.3 文库多样性分析
文库克隆插入片段阳性率采用菌落PCR和DNA测序分析91个单个TG1菌落。文库多样性按下列公式计算:(独特序列的数量/测序序列数量)×100%。
2 结果与分析
2.1 新疆双峰驼重链抗体噬菌体展示文库的构建
采用PCR扩增得到两条带,结果表明,主带约 650~700 bp,另外在1 000 bp 处有弱带图1A。分别代表骆驼IgG常规抗体和重链抗体。回收上步 PCR 的 650~700 bp 的条带,作为后续 PCR 的模板,使用VHH-back 和 PMCF 引物扩增VHH,结果表明,其中450 bp 的 DNA 片段为VHH基因片段图1B。图1
采用SfiⅠ分别酶切pCANTAB 5E质粒和VHH片段,回收后进行连接反应。电转化感受态TG1细胞,系列梯度稀释后,计算平皿中菌落数量。其中 10-5平皿上得到克隆 465 个,10-6平皿上得到克隆 25 个,计算最终建库库容为(图2):
(465+25×10)/2×105×40=1.4×109.
2.2 文库VHH插入片段阳性率评估
对91个文库TG1单菌落进行菌落PCR鉴定,结果91个克隆中有88个扩增出VHH片段(约为450 bp),而其余3个克隆未扩增出450 bp的VHH片段。菌落PCR结果表明,文库VHH片段阳性率为97%。同时这91个克隆的DNA测序结果(结果见2.3)显示所有克隆含有VHH基因序列,阳性率为100%。图3
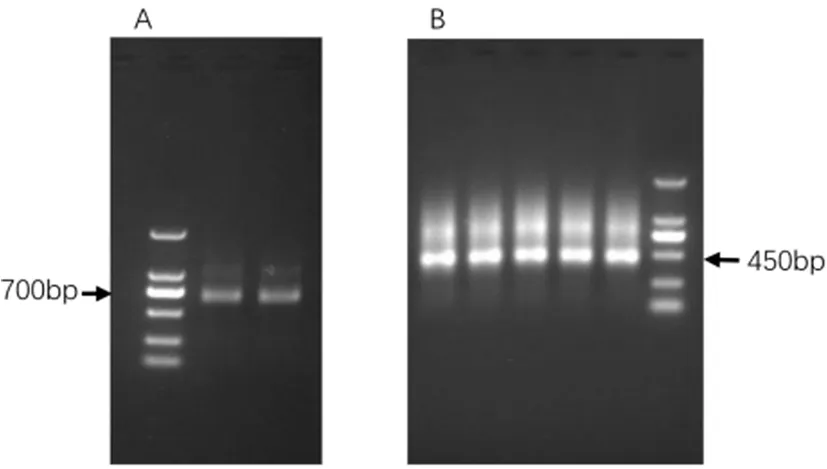
A.采用引物 Call01-leader 和 Call02-CH2 扩增VHH片段。B. 采用VHH-back 和 PMCF 引物扩增VHH片段
Call01-leader and Call02-CH2(A)andVHH-back and PMCF primer pairs(B)
图1 PCR扩增骆驼淋巴细胞cDNA中VHH基因片段
Fig.1 PCR amplification ofVHHgene fragments with
2.3 文库克隆Sanger测序
采用Sanger测序分析建库的 91个克隆,其中8个样品(2、9、12、15、21、26、61 和 91)出现套峰,可能的原因是这些克隆在平皿上未完全分离,或者是同一个宿主菌中转入了多个克隆。从测序结果明确的序列来看,有2个克隆出现内部终止密码子,L10和L50,L45和L28为重复序列,其他克隆测序结果良好,均为单个序列,DNA长度约在350~410 bp,序列阅读框架正确。根据独特序列的数量计算文库多样性为97.8%。分析抗体序列FR2特征氨基酸位点发现,除两个克隆为常规VH抗体特征(V42,G49,L50和W52)外,其余均为重链抗体VHH特征氨基酸(V42Y/V42F,G49E,L50R和W52X)。这些VHH序列CDR3长度分布介于9~30氨基酸之间,平均19氨基酸。丰度最高的CDR3长度为19个氨基酸,占整个测序序列的16%。文库中缺少长度为11,27,28和29氨基酸的CDR3序列。91个克隆中CDR3序列长度分布情况见图4,CDR3序列105-117位点的氨基酸组成和使用频率见图5。图4~5
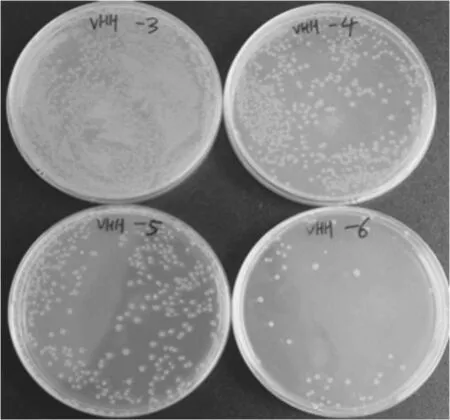
图2 系列稀释培养基中VHH基因转化TG1菌落数量(用于测定文库大小)Fig.2 The number of TG1 colonies at serial dilution transformed with VHH genes
2.4VHH序列特征氨基酸位点分析及亚群的分类
为评估构建文库的多样性,采用MEGA6软件对Sanger测序的91个VHH序列进行序列同源性比对并构建系统进化树(图6A)。聚类分析表明,91个VHH序列可以划分为7个聚类(cluster),其特征氨基酸在FR1序列中位于第12,15,24和26位点,在CDR1序列中位于第27和38位氨基酸,在FR2序列中位于第39,42,49,50,52和55位氨基酸,在CDR2序列中位于第56和66位氨基酸,在CDR3序列中位于第105,106和117位点,以及FR4序列中的118位点。这些特征氨基酸分布见WebLogo图(图6B),表明7个类群各具有特征氨基酸分布群。图6
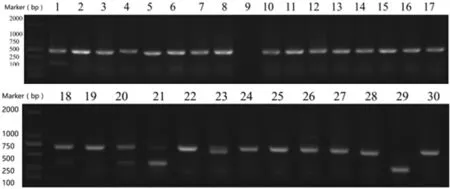
图3 菌落PCR扩展文库菌落VHH基因片段代表图Fig.3 Colony PCR amplification of VHH inserts in transformed TG1 E.coli

图4 VHH序列中CDR3长度分布频率(用于统计的总序列数为91)Fig.4 Frequency of the CDRH3 lengths and distribution within the VHH. CDRH3 lengths are expressed in amino acids. Total number ofVHH sequences subjected to statistical are 91

图5 文库中91个VHH克隆CDR3不同位点的氨基酸组成Fig.5 Amino acid composition at the different positions of CDR3 in the library

图6 91个序列聚类分析(A)和不同类群特征氨基酸的WebLogo图比较(B)Fig.6 Clustering analysis (A) and WebLogo plots (B) presenting sequence differences between diferent clusters
3 讨 论
噬菌体抗体表面展示文库技术是近年来发展较快制备单克隆抗体的有效手段,尤其可直接克隆抗体可变区基因,以利于进一步构建具有广阔应用前景的各种基因工程抗体。利用这种技术从HCAbs得到的VHHs和IgNAR单域抗体,与早期基因工程抗体Fab或scFv相比,水溶性更好,稳定性更高,并且扩大了对特殊抗原表位,如一些酶的活性中心位点的结合[11-13]。
能否从噬菌体抗体库筛选到有价值的抗体,与抗体库,尤其是非免疫文库的多样性有很大关系。文库的多样性不仅决定于文库大小,还决定于文库中多样性的VHH序列[14]。研究中从新疆双峰驼的淋巴细胞中构建了一个库容较大的非免疫VHH噬菌体展示文库。生物信息学分析了其中91个克隆的CDR3长度分布,特征氨基酸组成以及抗体序列分群,为今后评价文库多样性提供了新的指标。
在对文库克隆VHH插入片段阳性率分析中,DNA测序结果(100%)与菌落PCR(97%)不完全相符,可能原因是菌落PCR中由于菌落处理不当或数量过低导致的假阴性。这也表明DNA测序的结果更可靠和直接。
通过对VHH序列的比较分析,鉴定出新疆双峰驼免疫库中VHH抗体存在多个特征氨基酸位点(IMGT编号)15,40,42,49,50,52,87,96和106,常规抗体VH序列在这些位点的氨基酸往往不同[15]。这个结果与其他研究者相符。在分析的91个克隆中仅有2个代表了VH序列,VHH是所占比例高达98%。而多个研究者在VHH抗体文库中发现存在相当比例的VH序列[16-17]。研究构建的文库中VHH序列多的原因可能是新疆双峰驼中存在的VHH基因序列的丰度要远高于VH序列。根据VHH特征氨基酸的种类和分布频率,从91个VHH序列中划分出7个亚群,然而由于测序数量较少,还不足认定这些亚群是否稳定存在于抗体库中。进一步的分析依赖于对文库进行高通量的测序,可以一次测定107序列,这样就能够获得对抗体库多样性的全面分析和评估。
References)
[1]Hamerscasterman, C., Atarhouch, T., Muyldermans, S., Robinson, G., Hamers, C., & Songa, E. B., et al. (1993). Naturally occurring antibodies devoid of light chains.Nature, 363(6428): págs. 446-448.
[2] Andrew S. Greenberg, David Avila, Marianne Hughes, Hughes, A., E. Churchill McKinney, & Martin F. Flajnik. (1995). A new antigen receptor gene family that undergoes rearrangement and, extensive somatic diversification in sharks.Nature, 374(6518): 168-173.
[3] Peter Timmerman §, Susana G. Shochat ? §, Johan Desmet §, Rodrigo Barderas §, Jose Ignacio Casal §, & Rob H. Meloen §, et al. (2010). Binding of cdr-derived peptides is mechanistically different from that of high-affinity parental antibodies.JournalofMolecularRecognitionJmr, 23(6):559-568.
[4] Smith, G. P., & Petrenko, V. A. (1997). Phage display.ChemicalReviews, 97(97):391-410.
[5] Kretzschmar, T., & Rüden, T. V. (2002). Antibody discovery: phage display.CurrentOpinioninBiotechnology, 13(6):598-602.
[6] Bratkovic, T. (2010). Progress in phage display: evolution of the technique and its application.Cellular&MolecularLifeSciencesCmls, 67(5):749-767.
[7] Hust, M., & Dübel, S. (2004). Mating antibody phage display with proteomics.TrendsinBiotechnology, 22(1):8-14.
[8] Silacci, M., Brack, S., Schirru, G., M?rlind, J., Ettorre, A., & Merlo, A., et al. (2005). Design, construction, and characterization of a large synthetic human antibody phage display library.Proteomics, 5(9):2,340-2,350.
[9] Harris, L., & Bajorath, J. (2008). Profiles for the analysis of immunoglobulin sequences: comparison of v gene subgroups.ProteinScience, 4(2):306-310.
[10] Crooks, G. E., Hon, G., Chandonia, J. M., & Brenner, S. E. (2004). Weblogo: a sequence logo generator.GenomeResearch, 14(6):1,188-1,190.
[11] Liu, J., Anderson, G. J., Baumann, R., Hayhurst, A., & Goldman, E. (2007). Selection of cholera toxin specific ignar single-domain antibodies from a naive shark library.MolecularImmunology, 44(7):1,775-1,783.
[12] Genst, E. D., Silence, K., Decanniere, K., Conrath, K., Loris, R., & Kinne, J., et al. (2006). Molecular basis for the preferential cleft recognition by dromedary heavy-chain antibodies.ProceedingsoftheNationalAcademyofSciences, 103(12):4,586-4,591.
[13] Arbabi, G. M., Desmyter, A., Wyns, L., Hamers, R., & Muyldermans, S. (1997). Selection and identification of single domain antibody fragments from camel heavy-chain antibodies.FEBSLetters, 414(3):521-526.
[14] Pansri, P., Jaruseranee, N., Rangnoi, K., Kristensen, P., & Yamabhai, M. (2009). A compact phage display human scfv library for selection of antibodies to a wide variety of antigens.BMCBiotechnology, 9(1): 6.
[15] Harmsen, M. M., Ruuls, R. C., Nijman, I. J., Niewold, T. A., Frenken, L. G. J., & Geus, B. D. (2000). Llama heavy-chain v regions consist of at least four distinct subfamilies revealing novel sequence features.MolecularImmunology, 37(10):579-590.
[16] Saerens D; Pellis M; Loris R; Pardon E; Dumoulin M; Matagne A; Wyns L; Muyldermans S; Conrath K. (2005). Identification of a universal vhh framework to graft non-canonical antigen-binding loops of camel single-domain antibodies.JournalofMolecularBiology, 352(3):597-607.
[17] Desmyter, A., Transue, T. R., Ghahroudi, M. A., Thi, M. H., Poortmans, F., & Hamers, R., et al. (1996). Crystal structure of a camel single-domain VH antibody fragment in complex with lysozyme.NatureStructuralBiology, 3(9):803-811.
Fund project:Supported by National Natural Science Foundation of China ( 31570935) and Hi-tech research and development projects of Xinjiang, China, (201110102)
Analysis of the Diversity of VHH Library from Bactrian Camel (Camelusbactrianus) with Bioinformatics
FENG Ya-ning1, JIANG Zhi-qiang2, MA Xiao-ling1, LI Jiang-wei1
(1. College of Life Science and Technology, Xinjiang University, Urumqi 830046, China;2.No. 104AgriculturalandAnimalHusbandryRegimentalFarmofNo.12AgriculturalProductionDivisionofXinjiangProductionandConstructionCorps,Urumqi830076,China)
【Objective】 Heavy chain antibodies exist only in the immune system of the camel and theVHHfragment of the variable region is the smallest natural antibody fragment. Through the sequence analysis ofVHHgene to understand the genetic characteristics of Bactrian camel, camelVHH, and analyze the diversity ofVHHLibrary are the purpose of this project.【Method】TheVHHlibrary was constructed by PCR amplification of theVHHsequences from Xinjiang Bactrian camel (Camelusbactrianus) peripheral blood lymphocytes. Sanger DNA sequencing and colony PCR were used to determine the size andVHHinserts of the library. A comprehensive analysis ofVHHgene including the sequence homologous, CDR3 length distribution, sequence clustering were conducted via bioinformatics.【Result】The size of theVHHlibrary was 1.4×109. The positiveVHHinserts in the 91 randomly selected clones was 97% in colony PCR detection. The result of DNA sequencing revealed the diversity of the library was 97.8%. Bioinformatics analysis showed almost all sequences wereVHHderived except that from two clones. According to the sequence similarity, 91 sequences were classified into 7 clusters.【Conclusion】The constructed library has relatively large diversity and it will be helpful for analyzing the diversity of library by using bioinformatics tools.
Bactrian camel;nanobody; phage display library;VHHsequence;bioinformatics analysis
10.6048/j.issn.1001-4330.2016.08.023
2016-03-23
国家自然科学基金面上项目(31570935);新疆维吾尔自治区高技术研究发展项目(201110102)
冯亚宁(1990-),女,新疆乌鲁木齐人,硕士,研究方向为生物化学与分子生物学,(E-mail)fengyaning81@163.com
李江伟(1967-),男,河南人,教授,博士,研究方向为基因工程抗体,(E-mail)jwli67@sina.com
S182
A
1001-4330(2016)08-1533-06

