吡虫啉与人血清白蛋白相互作用热力学行为
郭清莲潘凌立杨立云何 欢张业中刘 义,*
(1武汉大学中南医院,武汉 430071;2黄石市中心医院,湖北 黄石 435002;3武汉大学化学与分子科学学院化学系,武汉 430072)
吡虫啉与人血清白蛋白相互作用热力学行为
郭清莲1潘凌立2杨立云3何 欢3张业中3刘 义3,*
(1武汉大学中南医院,武汉 430071;2黄石市中心医院,湖北 黄石 435002;3武汉大学化学与分子科学学院化学系,武汉 430072)
在模拟人体生理条件下,综合利用荧光光谱、紫外吸收光谱、圆二色谱和分子模拟等方法,研究了吡虫啉(IMI)和人血清白蛋白(HSA)相互作用的热力学行为。荧光光谱和紫外吸收光谱的分析表明:吡虫啉能有效猝灭HSA的内源荧光,猝灭机制为静态猝灭;通过所获取的相互作用热力学参数,可知两者之间的相互作用是一个吉布斯自由能降低的自发过程,且二者之间的主要作用力为氢键和范德华力。位点竞争实验和分子模拟的结果表明:吡虫啉在HSA的主要结合位置为位点I。圆二色谱、同步荧光光谱和三维荧光的分析发现:吡虫啉引起HSA的构象发生改变,其α-螺旋含量降低,无规卷曲含量升高,肽链结构在吡虫啉的作用下有所伸展。
吡虫啉; 人血清白蛋白; 相互作用; 热力学参数
1 引 言
蛋白质是生物体内具有重要生理功能的大分子,对一切生命过程都起着至关重要的作用,也是生命科学研究的重要对象1,2。血清白蛋白是血液中最丰富的载体蛋白,它具有贮运内源和外源化合物的作用3,4。许多生物活性物质,如代谢物、医药及其他有机物,与血清白蛋白的亲和性,影响它们在体内的分布和新陈代谢5–8。研究白蛋白与药物分子的相互作用,可以了解药物在体内的运输过程以及作用机制,对药物化学和药物分子设计具有重要的意义。
吡虫啉(imidacloprid) [1-(6-氯-3-吡啶甲基)-N-硝基咪唑烷-2-基胺]作为一种新型氯代烟碱类杀虫剂,可以选择性地抑制烟酸乙酰胆碱酯酶的受体,进而阻断昆虫神经系统的正常传导,起到杀虫作用9–11。一般认为吡虫啉对哺乳动物低毒,但越来越多的证据显示其严重的毒副作用,如可以产生基因毒性和免疫毒性12,13。随着吡虫啉农药的长期大量使用,势必会影响人体的健康,吡虫啉对人类的毒性作用,可以导致定向障碍、嗜睡、头晕、心悸和呕吐,严重的会造成心动过缓和心肺骤停14。吡虫啉还会破坏成年人的内分泌,干扰性腺类固醇性能,从而影响成年人的生殖系统15。研究吡虫啉与蛋白质的结合,有助于了解吡虫啉在体内的分布和代谢情况,对于阐明吡虫啉对人的毒副作用机制具有重要意义。近年来,吡虫啉与蛋白质作用引起了国内外许多研究人员的关注。Kazuhiko等16通过电化学与计算模拟的方法,研究了吡虫啉与α4β2烟碱乙酰胆碱受体的相互作用,发现吡虫啉主要结合在α4β2烟碱乙酰胆碱受体的loop C和loop D区域。Sun等14通过荧光光谱、紫外吸收光谱等手段,研究了吡虫啉与血红蛋白的相互作用。Ding和Peng17运用多种光谱手段和分子模拟的方法,研究了吡虫啉对多种人体球蛋白的抑制作用。
人血清白蛋白(HSA)是血浆中最丰富的蛋白质,由585个氨基酸残基组成。血清白蛋白是循环系统中含量最多的可溶性蛋白质,约占总蛋白的55%,在血浆中的含量约为0.05 g·mL–1, 维持血液循环系统的渗透压。其生理功能主要表现为:维持体内生理环境相对稳定;可以结合多种药物和生物活性小分子,便于其在血液中的运输18。人血清白蛋白对药物在生物体内的运输有着重要的生理作用,无论从药物动力学还是从临床药理学角度,研究血清蛋白与药物的作用有助于了解药物的体内运输性质和作用机制。当然,从当今药物研发的过程和经验来看,新药发现始于化学,但离不开药理学的指导和临床医学的检验。
本文选择人血清白蛋白作为模型蛋白,利用多种光谱手段和分子模拟的方法,研究了血清白蛋白与吡虫啉的相互作用机制,获取了两者相互作用的系列热力学参数、结合位点和作用力类型等,并研究了吡虫啉对人血清白蛋白构象的影响。
2 实验部分
2.1 仪器和试剂
带恒温系统的 Perkin Elmer LS55 荧光光度计(美国Perkin Elmer公司),TU-1901紫外-可见分光光度计(北京普析通用仪器有限公司),圆二色(CD)光谱仪(英国应用光物理公司),BS110S电子分析天平(北京赛多利斯仪器系统有限公司),Milli-Q Advantage A10超纯水系统(美国Millipore公司)。
采用Sybyl 8.1中Surflex-Dock模块进行计算模拟,软件购自Tripos公司,运行环境为CentOS系统。
人血清白蛋白(HSA)(电泳纯试剂)购自Sigma公司,吡虫啉(IMI)购自沙隆达农药公司。HSA溶液均用浓度为0.05 mol·L–1,pH = 7.4的Tris-HCl缓冲溶液配成,保存于4 °C的避光处。三羟甲基氨基甲烷(Tris, 2-amino-2-(hydroxymethyl)-1,3-propanediol)纯度不低于99.5%,NaCl、HCl等均为分析纯。吡虫啉溶液用无水乙醇配成,在整个实验中的水均为Millipore纯水。
2.2 实验方法
2.2.1 荧光光谱
荧光猝灭光谱:设定激发波长为λex= 280nm,激发狭缝宽度为15 nm,发射狭缝宽度为4.0 nm。在模拟人体生理条件下,测定加入不同量的IMI时HSA的荧光发射光谱,记录温度为292、298、304、310 K,波长在300–450 nm范围内的荧光发射光谱。为了避免滤波效应,在实验中使用极稀溶液(HSA 2.0 × 10–6mol·L–1, IMI 的浓度范围为0–1.6 × 10–5mol·L–1)。
位点竞争实验:选择华法林(warfarin)和布洛芬(ibuprofen)两种荧光探针作为位点标记剂来研究IMI和HSA之间的结合位点。HSA的浓度固定为2 × 10–6mol·L–1,然后在HSA-warfarin或HSA-ibuprofen的混合物中,逐渐滴加IMI。设定激发波长为λex= 280 nm,记录波长为300–475 nm范围内的荧光发射光谱。
同步荧光光谱:固定Δλ = 60 nm,记录IMI与HSA相互作用的同步荧光光谱。
三维荧光光谱:初始激发波长为200 nm,发射波长为200–500 nm,扫描次数为31,增量为5 nm,其它参数与HSA的荧光猝灭光谱一致。
2.2.2 紫外-可见吸收光谱
紫外-可见吸收光谱均用TU-1901紫外-可见分光光度计测量,波长范围500–200 nm。
2.2.3 圆二色谱
记录持续氮气流条件下,室温时波长范围为200–260 nm内样品的CD光谱。比色皿的光路长为0.1 mm,扫描速度200 nm·min–1。在相同实验条件下,缓冲溶液作为空白,从样品光谱图中扣除,实验的结果由SELCON3.0软件处理。
2.2.4 分子对接实验
HSA的晶体结构来自于数据库Protein Data Bank (PDB),本实验选择了蛋白编号为1h9z的晶体结构数据19,原始晶体结构数据经去水、加氢、加电荷处理。小分子三维结构由软件Sybyl8.1构建,并用最小能量法简单优化。应用选择HSA中配体的方法定义对接原型分子,配体选择为1h9z中配体WRR2001。
3 结果与讨论
3.1 荧光猝灭机制及猝灭常数
任何能够通过相互作用(如激发态反应,分子重排,能量转移,形成基态复合物及碰撞猝灭等)使荧光物质荧光强度下降的现象,都可称为荧光猝灭作用20。图1给出了pH = 7.4时在不同浓度IMI作用下的HSA的荧光猝灭光谱。可以看出,随着IMI的不断加入,其浓度增大,同时HSA的荧光强度逐渐降低,但最大发射波长并没有明显的移动,表明IMI可以有效猝灭HSA的内源荧光。

图1 加入不同浓度IMI之后HSA的荧光发射光谱Fig.1 Fluorescence emission spectra of HSA in the presence of various concentrations of IMI
通常,荧光猝灭过程被认为是猝灭剂和荧光团之间的碰撞过程或者是二者之间形成了复合物,即动态猝灭或者是静态猝灭21,22。不同的猝灭机制可以根据温度对结合常数和粘度的影响及测定荧光寿命的方法来加以区分。一般情况下,对于静态猝灭,猝灭常数KSV随着温度的升高而减小;相反地,对于动态猝灭,猝灭常数KSV随着温度的升高而增大23,24。

表1 pH = 7.4时不同温度下IMI与HSA相互作用的猝灭常数Table 1 Quenching constants for the interaction of IMI with HSA at different temperatures and pH = 7.4
为了判断上述体系的猝灭机制,对猝灭过程的荧光实验结果按照Stern-Volmer方程25进行处理:

式(1)中F0和F分别表示不存在和存在猝灭剂时荧光物质的荧光强度;KSV表示Stern-Volmer猝灭常数;[Q]表示猝灭剂的浓度;kq表示生物大分子的猝灭速率常数;τ0表示不存在猝灭剂时生物大分子的平均荧光寿命。很明显:

生物大分子的荧光平均寿命为10–8s,kq可以通过式(2)计算得到。不同温度下KSV和kq的计算结果列在表1中。由上述的分析结果可知:KSV的值随着温度的升高而减小,且kq值远大于生物大分子的最大碰撞猝灭常数(2.0 × 1010Lmol–1s–1)26,可以判断上述实验中的荧光猝灭是由于形成了复合物的静态猝灭过程。
为进一步证实IMI与HSA相互作用的猝灭机制,测定了室温条件下HSA、IMI 和HSA-IMI的紫外-可见吸收光谱(如图2所示)。对于动态猝灭,只涉及荧光分子的激发态,因而猝灭剂对荧光物质的吸收光谱一般不产生影响;而静态猝灭,由于基态复合物的形成,该过程一般会改变荧光物质的吸收光谱27。显然,HSA的吸收光谱在加入IMI前后发生了改变,进一步证明了IMI对HSA的猝灭是静态猝灭过程。

图2 IMI与HSA相互作用的紫外-可见吸收光谱Fig.2 UV-Vis absorption spectra of interaction of HSA and IMI
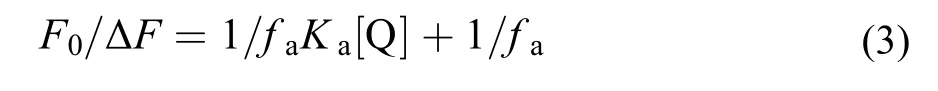
对于静态猝灭过程,可以使用修正的Stern-Volmer方程,进一步分析荧光猝灭数据28:式中,ΔF表示体系在无猝灭剂与猝灭剂浓度为[Q]时的荧光强度的差值;fa表示荧光团可接近猝灭剂的部分;Ka表示有效猝灭常数,此值相当于IMIHSA作用体系的结合常数。图3显示了F0/ΔF与1/[Q]呈线性关系,且计算得到的相应的Ka值列于表2中。从表2可以看出:随着温度上升,Ka值下降,表明尽管IMI和HSA发生了结合作用,但当温度升高时其作用力减弱。
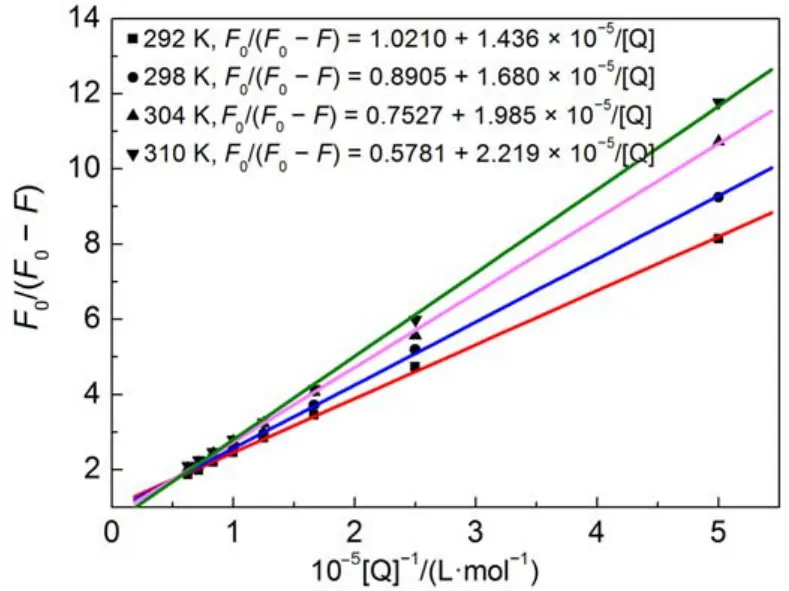
图3 pH = 7.4时四个不同温度下IMI猝灭人血清白蛋白荧光的修正Stern-Volmer曲线Fig.3 Modified Stern-Volmer plots for the quenching of HAS by IMI at four different temperatures and pH 7.4

表2 不同温度下IMI与HSA相互作用的热力学参数Table 2 Thermodynamic parameters of the interaction of HSA and IMI at different temperatures
3.2 IMI与HSA间的作用模式
通常,药物等活性小分子与蛋白质等生物大分子之间的相互作用力可分为四类:疏水作用力、氢键、范德华力和静电作用力29。为了说明HSA-IMI体系的作用模式,计算了四个不同温度下的热力学参数(ΔH、ΔS、ΔG),根据热力学参数的符号和大小可以得出相互作用的主要的作用力类型。利用van't Hoff方程30,可以计算反应的焓变ΔH和熵变ΔS:

其中,Ka为相应温度下的有效猝灭常数,R是气体常数。吉布斯自由能变(ΔG)由下式计算:


图4 HSA与IMI相互作用的范特霍夫曲线Fig.4 Van′t Hoff plot for the interaction of HSA and IMI
如图4所示,以lnKa对1/T作图,由斜率和截距,可以分别计算出焓变ΔH和熵变ΔS,进而可以得到不同温度下的自由能变ΔG,计算结果列于表2。负的ΔG值,表明结合过程是自发的。此外,ΔH
和ΔS均小于0,根据Ross理论31可以推测:在结合过程中,氢键和范德华力是主要作用力。
3.3 IMI在HSA上结合部位的确定
HSA由三个主要的结构域(I、II、III)组成,每个区域又包含两个亚域(A和B)。两个结合位点site I和siteII,分别位于亚域IIA和亚域IIIA的疏水腔内32。我们选择华法林(warfarin)和布洛芬(ibuprofen)分别作为site I和site II位点标记物,进行位点竞争实验,希望得到IMI在HSA上的结合部位的信息。
如图5(B)所示,华法林加入后,HSA的荧光强度明显下降,继续向warfarin-HSA体系中滴加IMI,HSA的荧光强度逐渐稳定降低,但是与图1相比,其荧光强度远低于不存在华法林时的荧光强度。而加入布洛芬后(如图5(A)),体系的荧光猝灭变化趋势与未加之前相似,因此可以推断华法林占据了部分与HSA结合的IMI的位置,布洛芬并没有影响IMI在HSA上的结合,说明IMI在HAS上的结合位点与华法林的结合位点相同33,34。
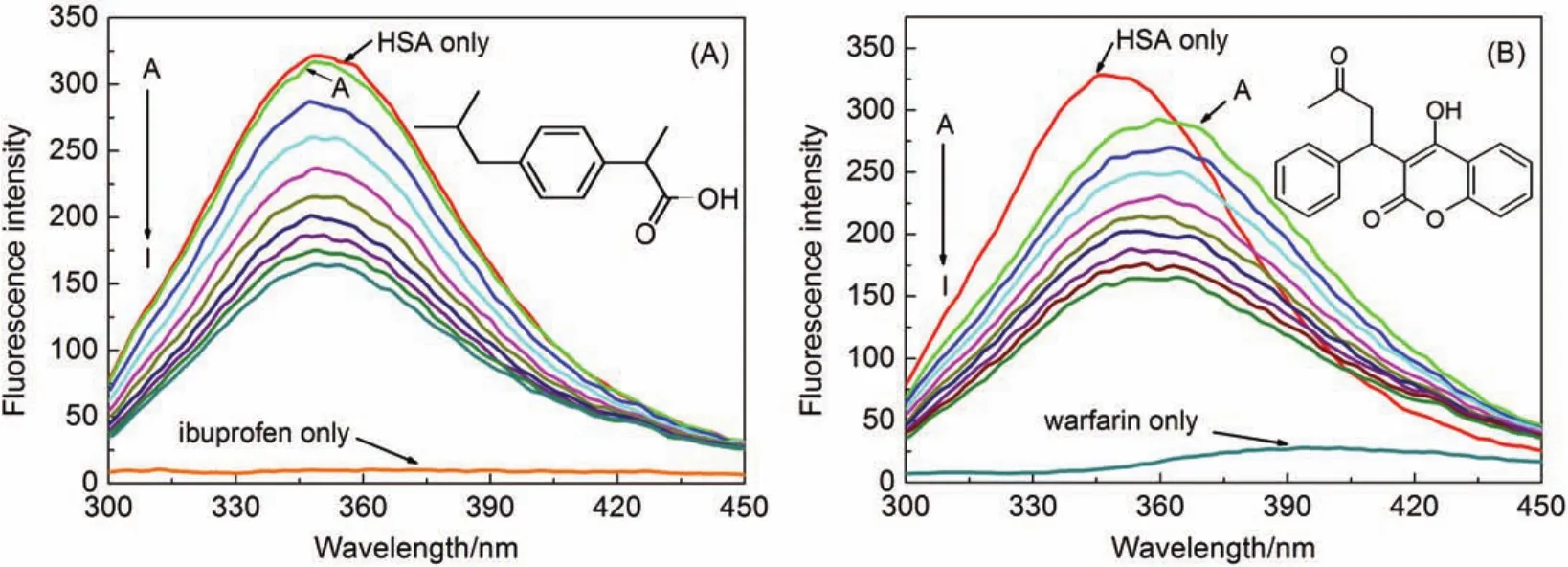
图5 位点探针对IMI-HSA体系的影响Fig.5 Effect of site markers on IMI-HSA system
此外,为了便于比较说明华法林和布洛芬对体系的影响,采用修正的Stern-Volmer方程,对相应的实验数据进行了处理(如图6所示),结果列于表3中。显然,加入华法林标记物后,Ka值是未加时的66%;而加入布洛芬后与未加时的结合常数相近。说明华法林对IMI与HSA的结合作用会产生很大的影响,而布洛芬几乎不产生影响。综上,华法林与IMI竞争同一个位点,IMI与HSA的结合主要发生在site I位点上。

图6 位点竞争实验的修正Stern-Volmer曲线Fig.6 Modified Stern-Volmer plots of site marker competitive experiments

Site marker · 10–4Ka/(Lmol–1) R S.D. blank 5.301 0.9998 0.05 ibuprofen 4.873 0.9999 0.04 warfarin 3.021 0.9998 0.08
3.4 IMI对HSA构象的影响
3.4.1 同步荧光
同步荧光光谱可以提供关于蛋白质构象改变的信息,因为其最大发射波长的偏移与蛋白质氨基酸残基周围的微环境有关。当激发波长和发射波长的波长差固定为60 nm时,可以得到色氨酸残基周围微环境改变的信息35。如图7所示,随着IMI的加入,HSA-IMI体系的同步荧光光谱发生了明显的红移,表明IMI的加入使HSA的构象发生了一定的变化,致使色氨酸残基所处的环境极性增强,疏水性有所减弱。

图7 IMI与HSA相互作用的同步荧光光谱Fig.7 Synchronous fluorescence spectra of HAS in the presence of IMI
3.4.2 圆二色谱
室温条件下,使用圆二色谱,研究HSA构象的改变,更加科学合理。如图8所示,在208和222 nm的远紫外区,出现两个负的特征肩峰谱带,这是蛋白质α-螺旋结构的特征峰36。当加入IMI后,HSA的峰强度明显减小,说明IMI对HSA的二级结构产生了影响。
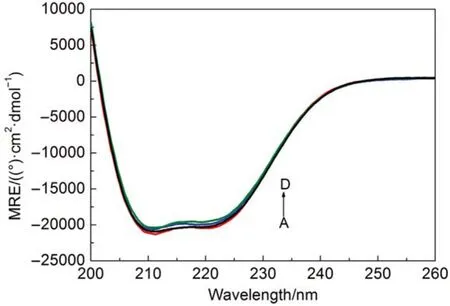
图8 HSA-IMI体系的圆二色谱图Fig.8 CD spectra of IMI-HSA system

表4 由SELCON3软件分析得到的二级结构的含量Table 4 Fractions of different secondary structures determined by SELCON3 software

图9 HSA (A)和IMI-HSA复合物(B)的三维荧光光谱Fig.9 Three-dimensional fluorescence spectra of HSA (A)and HSA-IMI complex (B)
由程序SELCON3,可以方便地计算出蛋白质各二级结构的百分比。不同摩尔比时的各二级结构的计算结果,列于表4中。当IMI和HSA的摩尔比达到12 : 1时,α-螺旋的含量从59.4%下降到57.6%,表明HSA分子的肽链结构在IMI的作用下有所伸展。进一步也说明了IMI与蛋白质主要多肽链的氨基酸残基结合,并且破坏了蛋白质的氢键结构37,38。当存在和不存在IMI时,HSA的CD光谱形状相似,表明在发生结合反应后,HSA的结构仍然主要是以α-螺旋为主。因此,IMI与HSA发生相互作用时,在一定程度上使HSA的构象发生了一定的变化。
3.4.3 三维荧光光谱
三维荧光光谱不仅可以全面展现待测样品的荧光信息,而且可以使蛋白质特征构象变化的研究更具有科学性和可靠性。它也是另外一种证实HSA构象和微环境是否改变的方法。图9显示了HSA和HAS-IMI复合物的三维荧光光谱,相应的特征参数列于表5中。
图9中,峰a为瑞利散射峰(λem= λex),且随着IMI的加入,峰a的荧光强度降低,可能原因是IMI在血清白蛋白表面结合后,破坏了蛋白质表面的保护水层,使原来较为分散的蛋白质更加分散,即所谓的解聚作用,引起蛋白质粒径减小,荧光强度减弱。Peak 1 (λex= 280 nm, λem= 351.0 nm)为蛋白质荧光猝灭的荧光峰,主要显示TRP和TYR的荧光光谱行为。另外,在peak 1旁边有一个很强的新的荧光峰peak 2 (λex= 230.0 nm, λem= 352.0 nm),主要展现的是蛋白质肽链结构的特征荧光行为39。在加入IMI后,peak 2的荧光强度轻微降低,说明蛋白质肽链结构发生了改变,这也正与CD光谱中α-螺旋结构含量的降低相吻合。表5主要反映HSA和HSA-IMI的三维荧光图谱特征。从峰的强度看,加入IMI后,HSA的两荧光峰强度明显减弱,且减弱的程度有所不同。说明蛋白质的多肽有一部分解旋,在HSA水溶液中加入IMI后,IMI在改变HSA所处微环境的同时也改变了HSA的构象。

表5 HSA和IMI-HSA复合物的三维荧光光谱Table 5 Three-dimensional fluorescence spectral characteristics of HSA and IMI-HSA system

图10 (a) IMI与HSA分子对接模型, (b) IMI周围0.5 nm范围内的氨基酸残基, (c) IMI与HIS288和ARG257形成的氢键Fig.10 (a) Molecular docking model of IMI and HSA, (b) amino acid residues around IMI within 0.5 nm,(c) hydrogen bonds between IMI and ARG222
3.5 分子模拟
研究表明,大多数药物分子与HSA的作用位置位于由亚结构域IIA和亚结构域IIIA的疏水腔内,即site I和site II40。IMI和HSA分子对接结果的最佳构象见图10(a),可以看出:IMI和HSA的作用区域更趋于位于site I,即华法林位点,这与位点竞争实验的结果一致。在0.5 nm范围内,IMI周围的氨基酸残基主要有12个,如图10(b)所示:TYR150, PHE157, SER192, LYS195, GLN196, ARG197, HIS212, ARG257, SER287, HIS288, LYS289, LEU292。此外,IMI分子上的氧原子与HIS288、ARG257的氢原子形成了氢键(图10(c)黑色虚线),这有助于IMI在HSA疏水腔中稳定存在,这也与前面热力学数据分析的结果相符合。
4 结 论
在模拟人生理条件下,研究了IMI与HSA相互作用的热力学行为。由变温滴定实验可知,Stern-Volmer猝灭常数KSV和有效猝灭常数Ka均随着温度的升高而减小,HSA的吸收光谱也在IMI加入之后发生变化,说明IMI对HSA的猝灭是生成了复合物的静态猝灭过程。在获取的热力学参数中,焓变和熵变均为负值,说明IMI与HSA之间相互作用力类型主要是氢键和范德华力。位点竞争实验和分子模拟的结果表明IMI在HSA上的结合位点位于site I。同步荧光光谱、CD光谱及三维荧光光谱的结果表明IMI与HSA的结合会引起HSA构象的改变。这些研究结果为获得蛋白质分子信息,了解吡虫啉在体内的作用机理,提供了一些具有重要理论意义的信息。
(1)Wang, N.; Liu, Z. Y.; Hu, X. L.; Bo, F. Q.; Zhao, X. Z. Chem. J. Chin. Univ. 2011, 32 (2), 241. [王 宁, 刘忠英, 胡秀丽, 卜凤泉, 赵学忠. 高等学校化学学报, 2011, 32 (2), 241.]
(2)Ding, F.; Diao, J. X.; Sun, Y.; Sun, Y. J. Agric. Food Chem. 2012, 60, 7218. doi: 10.1021/jf300424w
(3)Bekale, L.; Agudelo, D.; Tajmir-Riahi, H. A. Colloid Surf. BBiointerfaces 2015, 130, 141. doi: 10.1016/j.colsurfb. 2015.03.045
(4)Luo, Y.; Chen, T. F.; Huang, X. C.; Wang, Y.; Huang, Y. C.;Zheng, W. J. Acta Chim. Sin. 2012, 70 (11), 1295. [罗 懿, 陈填峰, 黄晓纯, 王 弋, 黄荫成, 郑文杰. 化学学报, 2012, 70 (11), 1295.] doi: 10.6023/A1202031
(5)Andrasi, M.; Lehoczki, G.; Nagy, Z.; Gyemant, G.; Pungor, A.;Gaspar, A. Electrophoresis 2015, 36, 1274. doi: 10.1002/elps. v36.11-12
(6)Khan, A. B.; Khan, J. M.; Ali, M. S.; Khan, R. H.; Din, K. U. Colloid Surf. B-Biointerfaces 2011, 87 (2), 447. doi: 10.1016/j.colsurfb.2011.06.007
(7)Chi, Z.; Liu, R.; Teng, Y.; Fang, X.; Gao, C. J. Agric. Food Chem. 2010, 58, 10262.
(8)Peng, Y. L.; Wang, S. J.; Fu, L.; Zhang, C. G.; Liu, X. G. Acta Phys. -Chim. Sin. 2012, 28 (5), 1054. [彭玉苓. 王树军. 傅 丽,张成根, 刘新刚. 物理化学学报, 2012, 28 (5), 1054.] doi: 10.3866/PKU.WHXB201202222
(9)Uhl, P.; Bucher, R.; Schafer, R. B.; Entling, M. H.; Ling, E. Chemosphers 2015, 132, 152. doi: 10.1016/j.chemosphere. 2015.03.027
(10)Zhao, J.; Wang, M. L.; Dong, B. L.; Feng, Q.; Xu, C. X. Org. Process Res. Dev. 2013, 17, 375. doi: 10.1021/op300320a
(11)Ji, R. D.; Zhao, Z. M.; Zhang, L.; Ji, L.; Zhang, J. H.; Shen, L. B.; Lan, X. F. Spectrosc. Spect. Anal. 2013, 33 (3), 668. [季仁冬,赵志敏, 张 林, 季 雷, 张吉华, 沈令斌, 兰秀风. 光谱学与光谱分析, 2013, 33 (3), 668.]
(12)Costa, C.; Silvari, V.; Melchini, A.; Catania, S.; Heffron, J. J.;Trovato, A.; De Pasquale, R. Mutat. Res. Genet. Toxicol. Environ. Mutagen. 2009, 672 (1), 40. doi: 10.1016/j.mrgentox. 2008.09.018
(13)Gawade, L.; Dadarkar, S. S.; Husain, R.; Gatne, M. Food Chem. Toxicol. 2013, 51, 61. doi: 10.1016/j.fct.2012.09.009
(14)Ding, F.; Han, B. Y.; Liu, W.; Zhang, L.; Sun, Y. J. Fluoresc. 2010, 20, 753. doi: 10.1007/s10895-010-0618-0
(15)Ding, F.; Peng, W.; Diao, J. X.; Zhang, L.; Sun, Y. J. Agric. Food Chem. 2013, 61, 4497. doi: 10.1021/jf3048065
(16)Kayoko, T.; Satoshi, K.; Satoshi, K.; Atsushi, Y.; Miki, A.;David, B. S.; Kazuhiko, M. Neuropharmacology 2009, 56, 264. doi: 10.1016/j.neuropharm.2008.08.022
(17)Ding, F.; Peng, W. J. Photochem. Photobiol. B-Biol. 2015, 147, 24. doi: 10.1016/j.jphotobiol.2015.03.010
(18)Wang, J.; Li, S.; Peng, X.; Yu, Q.; Bian, H.; Huang, F.; Liang, H. J. Luminesc. 2013, 136, 422. doi: 10.1016/j.jlumin.2012.12.004
(19)Petitpas, I.; Bhattacharya, A. A.; Twine, S.; East, M.; Curry, S. J. Biol. Chem. 2013, 276, 22804.
(20)Hemmateenejad, B.; Yousefinejad, S. J. Mol. Struct. 2013, 1037, 317. doi: 10.1016/j.molstruc.2013.01.009
(21)Dangkoob, F.; Housaindokht, M. R.; Asoodeh, A.; Rajabi, O.;Zaeri, Z. R.; Doghaei, A. V. Spectroc. Acta Pt. A-Molec. Biomolec. Spectr. 2015, 137, 1106. doi: 10.1016/j.saa. 2014.08.149
(22)Song, W.; Zhang, D.; Pan, X.; Lee, D. J. J. Luminesc. 2013, 136, 80. doi: 10.1016/j.jlumin.2012.11.008
(23)Li, D. W.; He, H.; Lin, B. B.; Xu, Z. Q.; Jiang, F. L.; Liu, Y. RSC Adv. 2014, 4, 3913. doi: 10.1039/C3RA46172F
(24)Li, J. H.; Wang, S. M. J. Chem. Thermodynamics 2013, 58, 206. doi: 10.1016/j.jct.2012.11.009
(25)Zaidi, N.; Ahmad, E.; Rehan, M.; Rabbani, G.; Ajmal, M. R.;Zaidi, Y.; Subbarao, N.; Khan, R. H. J. Phys. Chem. B 2013, 117, 2595. doi: 10.1021/jp3069877
(26)Gong, Q. L.; Hu, X. G.; Fang, G. Y.; Li, X. H. J. Mol. Model. 2012, 18, 493. doi: 10.1007/s00894-011-1069-5
(27)Hu, Y. J.; Yue, H. L.; Li, X. L.; Zhang, S. S.; Tang, E.; Zhang, L. P. J. Photochem. Photobiol. B-Biol. 2012, 112, 16. doi: 10.1016/j.jphotobiol.2012.04.001
(28)Markarian, S. A.; Aznauryan, M. G. Mol. Biol. Rep. 2012, 39 (7), 7559. doi: 10.1007/s11033-012-1590-3
(29)Banerjee, M.; Chakrabarti, A.; Basu, S. Dyes Pigment. 2013, 97,446. doi: 10.1016/j.dyepig.2013.01.005
(30)Han, X. L.; Tian, F. F.; Ge, Y. S.; Jiang, F. L.; Lai, L.; Li, D. W.;Yu, Q. L. Y.; Wang, J.; Lin, C.; Liu, Y. J. Photochem. Photobiol. B-Biol. 2012, 109, 1. doi: 10.1016/j.jphotobiol.2011.12.010
(31)Ross, P. D.; Subramanian, S. Biochemistry 1981, 20 (11), 3096. doi: 10.1021/bi00514a017
(32)Wu, X.; Liu, J.; Huang, H.; Xue, W.; Yao, X.; Jin, J. Int. J. Biol. Macromol. 2011, 49 (3), 343. doi: 10.1016/j.ijbiomac. 2011.05.010
(33)Wang, Q.; He, J. W.; Wu, D.; Wang, J.; Yan, J.; Li, H. J. Luminesc. 2015, 164, 81. doi: 10.1016/j.jlumin.2015.03.025
(34)Zhang, Y. Z.; Dai, J.; Xiang, X.; Li, W. W.; Liu, Y. Mol. Biol. Rep. 2010, 37, 1541. doi: 10.1007/s11033-009-9555-x
(35)Azimi, O.; Emami, Z.; Salari, H.; Chamani, J. Molecules 2010, 16 (12), 9792.
(36)Zhang, G. W.; Ma, Y. D.; Wang, L.; Zhang, Y. P.; Zhou, J. Food Chem. 2012, 133, 264. doi: 10.1016/j.foodchem.2012.01.014
(37)Chen, T. T.; Zhu, X. T.; Chen, Q.; Ge, M.; Jia, X. P.; Wang, X.;Ge, C. W. Food Chem. 2015, 186, 292. doi: 10.1016/j.foodchem. 2014.11.041
(38)Zhang, Y. Z.; Zhou, B.; Liu, Y. X.; Zhou, C. X.; Ding, X. L.;Liu, Y. J. Fluoresc. 2008, 18, 109. doi: 10.1007/s10895-007-0247-4
(39)Punith, R.; Seetharamappa, J. Spectroc. Acta Pt. A-Molec. Biomolec. Spectr. 2012, 92, 37. doi: 10.1016/j.saa.2012.02.038
(40)Tian, F. F.; Jiang, F. L.; Han, X. L.; Xiang, C.; Ge, Y. S.; Li, J. H.; Zhang, Y.; Li, R.; Ding, X. L.; Liu, Y. J. Phys. Chem. B 2010, 114, 14842. doi: 10.1021/jp105766n
Thermodynamics of the Interaction of Imidacloprid with Human Serum Albumin
GUO Qing-Lian1PAN Ling-Li2YANG Li-Yun3HE Huan3ZHANG Ye-Zhong3LIU Yi3,*
(1Zhongnan Hospital, Wuhan University, Wuhan 430071, P. R. China;2Center Hospital of Huangshi City, Huangshi 435002, Hubei Province, P. R. China;3Department of Chemistry, College of Chemistry and Molecular Sciences, Wuhan University, Wuhan 430072, P. R. China)
The thermodynamics of the interaction between human serum albumin (HSA) and imidacloprid(IMI) was inνestigated using fluorescence, UV-Vis absorbance, and circular dichroism spectroscopy, in addition to molecular modeling under physiological conditions. The fluorescence quenching of HSA by IMI was a static process, which was confirmed by the UV-Vis absorption spectra. The calculated enthalpy (ΔH) and entropy (ΔS) changes implied that hydrogen bonds and νan der Waals forces played a predominant role in the binding process. Site marker competitiνe experiments along with molecular docking indicated that the binding of IMI to HSA took place primarily in site I. The circular dichroism and synchronous fluorescence spectroscopy demonstrated that the secondary structure of HSA changed after its interaction with IMI, causing the α-helix content to decrease with an increase in anunordered structure. The peptide structure extended after binding with IMI.
Imidacloprid; Human serum albumin; Interaction; Thermodynamics parameter
O642
10.3866/PKU.WHXB201511021
Received: September 21, 2015; Revised: November 2, 2015; Published on Web: November 2, 2015.
*Corresponding author. Email: yiliuchem@whu.edu.cn; Tel: +86-27-68756667.
The project was supported by the Key Project of Health and Family Planning Commission of Hubei Province, China (WJ2015MB097) and Wuhan Yellow Crane Talents Program for Science and Technology, China (2014[10]).
湖北省卫生计生委重点项目(WJ2015MB097)和武汉黄鹤英才(科技)计划(2014[10])资助
©Editorial office of Acta Physico-Chimica Sinica

