与CRY1相互作用的转录因子蛋白的筛选
何志敏, 冯盼盼, 荣朵艳, 赵小英, 刘选明,2
(1. 湖南大学 生物学院 化学生物传感与计量学国家重点实验室,长沙410082;2. 湖南大学 生物学院 植物功能基因组学与发育调控湖南省重点实验室,长沙410082)
与CRY1相互作用的转录因子蛋白的筛选
何志敏1, 冯盼盼1, 荣朵艳1, 赵小英1, 刘选明1,2
(1. 湖南大学 生物学院 化学生物传感与计量学国家重点实验室,长沙410082;2. 湖南大学 生物学院 植物功能基因组学与发育调控湖南省重点实验室,长沙410082)
拟南芥CRY1(cryptochrome1)通过蛋白相互作用启动蓝光信号的传导,从而促进植物光形态建成。为深入研究CRY1介导蓝光信号传导的机制,以CRY1为诱饵蛋白,通过酵母双杂交系统的营养缺陷型试验、液体显色试验、分段相互作用试验方法,从拟南芥转录因子酵母库中筛选与其相互作用的转录因子蛋白。结果表明,通过营养缺陷型筛选试验以及以ONPG为底物的液体显色试验初步获得与CRY1相互作用的41个转录因子蛋白;选取与CRY1相互作用蓝光依赖程度较高或酶活性高的转录因子以CPRG为底物的液体显色试验进行进一步验证,发现选取的转录因子蛋白与CRY1的相互作用具有蓝光依赖性且随蓝光处理时间的延长相互作用强度增强;CRY1各分段与转录因子蛋白相互作用试验结果表明转录因子SPL3和IDD4主要与CRY1的N端相互作用,与C端的相互作用比较微弱,但是SPL3与CRY1 C端的相互作用具有蓝光特异性而IDD4与CRY1 C端的蓝光特异性相互作用不明显。
CRY1;酵母双杂交;转录因子;拟南芥
光通过多种光受体调节植物的生长发育,这些光受体包含光敏色素与隐花色素。隐花色素CRY1自1993年被发现以来,其在接收和传递蓝光信号影响植物生长发育方面的功能得到了很大程度的研究,尤其是在植物光形态建成方面的功能已经很清楚,这部分的功能与光敏色素在光信号传导上的作用是冗余的[1-2]。光形态建成包括抑制下胚轴的伸长,促进子叶的张开,促进幼苗的去黄化,诱导基因表达的变化和叶绿体的发育等[3-8]。
CRY1通过与下游的信号蛋白相互作用传递蓝光信号,从而引起一系列的光形态建成反应以实现其对植物生长发育的调节。目前,从遗传学和生理学上报道了许多与CRY1相互作用的蛋白(图1)。CRY1与这些蛋白的相互作用大致可分为以下几类:1)CRY1/COP1/SPA1之间的相互作用;2)CRY1与光敏色素(PHYA和PHYB)的相互作用;3)CRY1与ZTL/ADO1/LKP1相互作用;4)CRY1与PHOT1和PHOT2的相互作用。COP1能分别与CRY1、SPA1直接相互作用,这三者之间的关系:黑暗下,SPA1与COP1相互作用并激活COP1的E3泛素化连接酶活性,促进COP1对下游底物如HY5和CO等蛋白的降解。蓝光下,激活的CRY1构象打开,并通过C端的CCE结构域与SPA1的WD结构域相互作用,CRY1-SPA1光下的相互作用竞争性抑制了SPA1-COP1的相互作用,进而COP1的活性受到抑制,最终COP1的底物得到了积累并引起光诱导基因表达的变化[9]。PHYA作为光激活蛋白激酶通过与CRY1的C端结构域CCR相互作用并磷酸化CRY1,实现光激活CRY1的过程,但是这一磷酸化过程是没有光特异性的,PHYA在红光和蓝光下均能磷酸化CRY1,但是磷酸化的CRY1只能在蓝光下发挥功能,所以PHYA磷酸化CRY1的反应并不是CRY1功能的信号传导机制[10-11]。CRY1与PHYB的相互作用目前仅在体外试验得到验证[12]。ZTL/ADO1/FKF1是一类含LOV/F-BOX/Kelch的蓝光受体,在体外酵母系统里和pulldown assay试验中证实了ADO1与CRY1、PHYB的相互作用。ADO1与CRY1的C端结构域和PHYB的羧基末端有很强的相互作用[13]。向光素PHOT1和PHOT2是一类与隐花色素不相关的黄素单核苷酸FMN绑定蛋白,但是在生理生化证据上,目前没有明确的实验证据证明向光素PHOT1和PHOT2与隐花色素有直接的相互作用。虽然以上蛋白被证明在植物或者体外系统中与CRY1存在直接或间接的相互作用,但是没有一个是属于转录因子蛋白的。所以筛选与CRY1相互作用的转录因子蛋白,探明转录因子参与CRY1信号通路途径的作用机制具有重要的科学理论意义。
本研究利用酵母双杂交系统,以CRY1为诱饵蛋白从拟南芥转录因子酵母库中筛选与CRY1相互作用的转录因子蛋白,验证了蓝光对其相互作用的影响,分析了与转录因子相互作用的CRY1结构域,以及筛选出来的转录因子的mRNA组织特异性表达以初步判断CRY1可能参与的植物生长发育调节。
1 材料和方法
1.1材料
1.1.1菌种、质粒、转录因子酵母库
含有全长cry1的质粒pBridge-cry1由本实验构建;大肠杆菌DH5α由本实验室保存;酿酒酵母AH109菌株、酿酒酵母Y187菌株来自北京农科院;pDEST22、 pDEST32由本实验室提供;转录因子酵母文库由加州大学林辰涛教授实验室提供[14]。
1.1.2 培养基和试剂
YPDA酵母培养基、-Leu、-Trp、-Leu/-Trp、-Leu/-Trp/-His 选择性培养基以及细菌培养基为自行配制。高保真酶primerstar购自大连宝生物;ONPG购自上海生工;3-AT购自sigma;CPRG购自罗氏;其他试剂为国产分析纯。
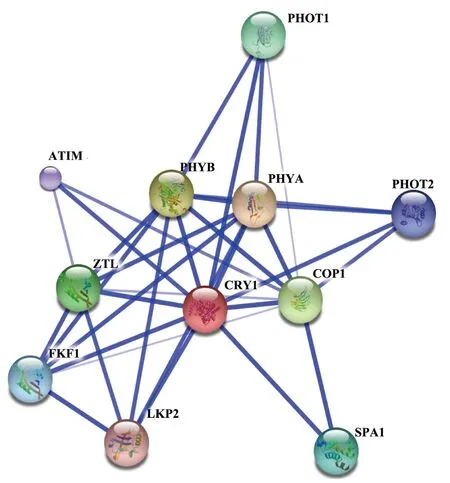
图1 与CRY1相互作用的已知蛋白(http://www.string.com)Fig 1 Known proteins which interact with CRY1 (http://www.string.com)
1.2 方法
1.2.1 诱饵载体pDEST32-cry1的构建
根据cry1的序列设计引物cry1F(5′-ATGTCTGGTTCTGTATCTGGTTG-3′)和cry1R(5′-TTACCCGGTTTGTGAAAGC-3′),加上gateway的接头attBF (5′-CAAAA AAGCAGGCTTC-3′),attBR(5′-CAAGAAAGCTGGGTC-3′),以pBridge-cry1质粒为模板,用连了接头的cry1全长引物进行第一轮PCR克隆,以第一轮PCR产物为模板,用attB接头引物扩增出含有接头的cry1全长的目的片段。回收纯化后cry1全长与入门载体pDONER-ZEO在BP酶的作用下构建重组质粒,转入DH5α检验后经测序验证重组质粒pDONER-ZEO-cry1的正确性。测序结果返回后,重组质粒pDONER-ZEO-cry1与诱饵空载体pDEST32在LR酶的作用下构建目的克隆载体pDEST32-cry1。
1.2.2 酵母融合
本方法参照Clontech酵母杂交手册采用酵母双杂交融合筛库法,具体如下:将构建的诱饵重组载体pDEST32-cry1和BD空载pDEST32分别转化入酵母菌株AH109;AD空载pDEST22 转化入酵母菌Y187。分别在单缺选择性培养基-Leu、-Trp的平板上挑选阳性克隆以进行后续的酵母融合。将表达了pDEST32-CRY1和pDEST32的AH109重组菌株分别与转录因子酵母库中的表达了单个转录因子的Y187酵母融合。融合后的酵母细胞涂布于-Leu/-Trp的双缺培养基,30℃培养3~5 d。点板至-Leu/-Trp/-His三缺选择性培养基上,分别放于蓝光和黑暗下,以期初步筛选到在蓝光下有相互作用的阳性克隆。
1.2.3 酵母双杂liquid assay 验证
用ONPG或者CPRG为底物的液体显色试验(liquid assay)来测定酵母中lacZ酶活性以进一步验证经酵母融合营养缺陷型培养基初步筛选出来的转录因子与CRY1蛋白在蓝光下的相互作用以及相互作用的强度。具体方法可参考Clontech的酵母双杂交手册。
1.2.4 CRY1结构域与转录因子的相互作用
将CRY1全长、CRY1N493(氨基酸序列1~493)、CRY1N505(氨基酸序列1~505)、CRY1N515(氨基酸序列1~515)、CRY1M251-545(氨基酸序列251~545)、CRY1C301(氨基酸序列382~682)的CDS序列克隆后构建到BD载体pDEST32,并转入AH109酵母菌株中。经-Leu单缺营养缺陷型培养基筛选阳性克隆后,分别与酵母库中表达了SPL3与IDD4转录因子蛋白的Y187酵母菌株融合。融合后的菌株经-Leu/-Trp筛选阳性克隆。经-Leu/-Trp/-His营养缺陷型和liquid assay(CPRG为底物)检验CRY1各片段与SPL3与IDD4的相互作用。
1.2.5 mRNA组织表达分析
使用GE公司的RNA提取试剂盒提取各组织的RNA。采用Superscript first-strand cDNA合成系统(Invitrogen)取1 μg总RNA合成cDNA的第一链。Platinum SYBR Green qPCR Supermix-UDG (Invitrogen)进行定量PCR反应。简单如下:将合成的cDNA稀释50倍,取2 μL稀释的cDNA于10 μL的qPCR反应体系中作模板,95℃预变性5 min;95℃变性10 s,56℃退火15 s, 72℃延伸30 s,循环40次。以ACTIN2的mRNA水平作为内参。定量PCR的结果为3次生物学重复结果。所使用的定量引物分别为qspl3F(AAGCAAAGC GGAAGGGAAG),qspl3R(TCTCGACCTGACAAACTCCACT);qIdd4F(TCGACGTTCAATTTGGAGAC A),qIdd4R(GAAACTCCGCCACGAGCA)。
2 结果
2.1 酵母双杂交筛选结果
CRY1具有明显的自激活,黑暗下pDEST32-CRY1在100 mmol/L 3-AT浓度下自激活被明显抑制,但是蓝光下的自激活没有被完全抑制,说明pDEST32-CRY1在蓝光下还具有一定程度的蓝光自激活[15]。当3-AT的浓度高达200 mmol/L时,pDEST32-CRY1的自激活背景完全消除。在后续试验中选用含有100 mmol/L 3-AT的-Leu/-Trp/-His的营养缺陷型平板对pDEST32-CRY1与各转录因子融合的阳性克隆进行相互作用的筛选,并分别放于50 μmol/m2s的蓝光和黑暗下,生长3 d,初步筛选到41个阳性克隆。将筛选到的候选阳性克隆以ONPG为底物并以pDEST32-CRY1 / pDEST22为阴性对照,在蓝光与黑暗下做进一步的liquid assay试验以进行验证,并计算蓝光/黑暗下的β-半乳糖苷酶活性的倍数大小,结果如图2,初步获得3个与CRY1相互作用酶活性高,蓝光/黑暗的酶活性倍数相对较大的转录因子。将获得的3个酵母阳性克隆提取DNA后进行测序以确定试验过程中无操作污染。这3个与CRY1相互作用的转录因子分别为3#A9(spl3 At2g33810)、3#E2(tcp2 At4g18390)和7#A12(idd4 At2g02080)。spl3是一个与开花相关的转录因子;tcp2是一个与细胞分化、叶片发育和形态相关的转录因子;idd4是一个定位于叶绿体的转录因子。
2.2 蓝光对CRY1与各转录因子相互作用的影响
为进一步确定CRY1与筛选出的转录因子相互作用的蓝光依赖性,将筛选出来的阳性克隆点在-Leu/-Trp/-His/+200 mmol/L 3-AT的营养缺陷型平板上,并在50 μmol/m2s蓝光和黑暗下生长3 d。由于200 mmol/L的3-AT能消除pDEST32-CRY1在蓝光和黑暗下的自激活背景,所以更能真实地反映CRY1与转录因子的相互作用。结果如图3A,CRY1能分别与筛选出的3个转录因子(SPL3、TCP2和IDD4)在营养缺陷型平板上蓝光依赖地相互作用。以比ONPG灵敏度更高的CPRG为底物进行liquid assay显色复验证试验,也同样证明了CRY1与这3个转录因子蛋白在蓝光下的相互作用更强(图3B)。通过测定不同蓝光处理时间下CRY1蛋白与转录因子相互作用的酶活性,进一步确认了蓝光对其相互作用的影响,随着蓝光时间的延长,其相互作用的酶活性也随着增强(图3C)。而从图3C中各转录因子与CRY1相互作用酶活性随蓝光处理时间的变化曲线可知,3#E2(TCP2)与CRY1的相互作用随蓝光处理时间的延长增长速度最快,而3#A9(SPL3)与CRY1的相互作用不论是在黑暗还是蓝光下,相较于其他两个转录因子,其酶活性都是最强的。

图2 ONPG液体显色试验筛选结果
Fig 2 The screening results of ONPG liquid assay
A、C:蓝光和黑暗下初步筛选出来的转录因子蛋白与CRY1蛋白相互作用的β-半乳糖苷酶活性;B、D:对应图A和图C中所选转录因子蛋白与CRY1蛋白相互作用蓝光和黑暗下β-半乳糖苷酶活性的比值
2.4CRY1各结构域与转录因子蛋白相互作用的分析
CRY1的结构域主要包括两个部分,绑定发色团黄素腺嘌呤二核苷酸FAD的N端PHR结构域和C端功能结构域CCE。但是鉴于CRY1在蓝光下有自激活活性,所以本研究想通过更细的分段,在研究结构域与转录因子蛋白相互作用的同时能找到CRY1蛋白不会自激活的结构域,所以本研究对CRY1的实际分段如方法1.2.4中所示。
由于3#E2(TCP2)与CRY1相互作用的较强的蓝光依赖性,本研究已将其单独进行了更深入的研究[16]。在以His为报告基因的营养缺陷型试验中,在-Leu/-Trp/-His缺陷型培养基中加入10 mmol/L 3-AT,在蓝光和黑暗下生长3 d的酵母显示CRY1全长以及CRY1N493都具有很强的自激活,其相互作用的结果无法正确显示,而CRY1N505与CRY1N515在10 mmol/L 3-AT的抑制下,His表达泄漏得到抑制,但是这两个片段分别与3#A9和7#A12的相互作用的蓝光依赖程度减弱了,蓝光和黑暗下都能相互作用,CRY1M251~545与这两个转录因子在蓝光和黑暗下均无相互作用,而CRY1C301与3#A9在蓝光下有较弱的相互作用,与7#A12在蓝光和黑暗下均有较弱的相互作用,黑暗下相对较强(图4A、B)。在以LacZ为报告基因的liquid assay显色试验中,由于CRY1的自激活,导致背景很高,但是依旧可以判断出与营养缺陷型试验相似的试验结果(图4C、D)。

图3 蓝光对CRY1与转录因子相互作用的影响分析
Fig 3 Analysis of blue light influence to the interactions of CRY1 and transcription factors
A: 酵母双杂交营养缺陷型试验结果显示CRY1与各转录因子在蓝光与黑暗下的相互作用,组氨酸缺陷型试验中,将酵母点在含有200 mmol/L 3-AT浓度的-Leu/-Trp/-His的平板上,并分别放置于50 μmol m2/s蓝光与黑暗下生长3 d;B: β-半乳糖苷酶活性液体显色试验分析CRY1与各转录因子在50 μmol/m2s蓝光与黑暗下的相互作用,各P值均小于0.05;C: β-半乳糖苷酶活性液体显色试验分析50 μmol/m2s蓝光处理时间对CRY1与各转录因子相互作用影响的分析
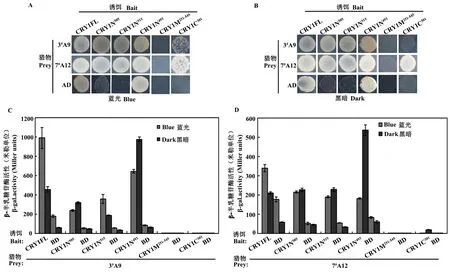
图4 CRY1各结构域与筛选出的转录因子的相互作用分析Fig 4 Interactions anlaysis of CRY1 domains with transcription factors
A、B:酵母营养缺陷型试验结果显示CRY1结构域与转录因子在蓝光和黑暗下的相互作用,酵母细胞生长条件与图3A一致;C、D:β-半乳糖苷酶活性试验显示与各转录因子相互作用需要的CRY1结构域,酵母细胞在蓝光或黑暗下处理4 h,3#A9(SPL3)和7#A12(IDD4)与CRY1、 CRY1N505、 CRY1N515、CRY1N493在蓝光与黑暗下的P值均小于0.05。
2.5spl3与idd4 mRNA组织特异性表达分析
基因在各组织器官中的表达量,从侧面可以反映其对该组织器官生长发育的影响。通过分析spl3和idd4在拟南芥不同组织器官中的表达谱,发现spl3基因是一个广谱表达的基因,在大部分的组织器官中都有表达,但是主要是在茎和花中表达积累,尤其是在花中表达量高(图5A),而idd4主要是在茎生叶和果荚中表达(图5B)。
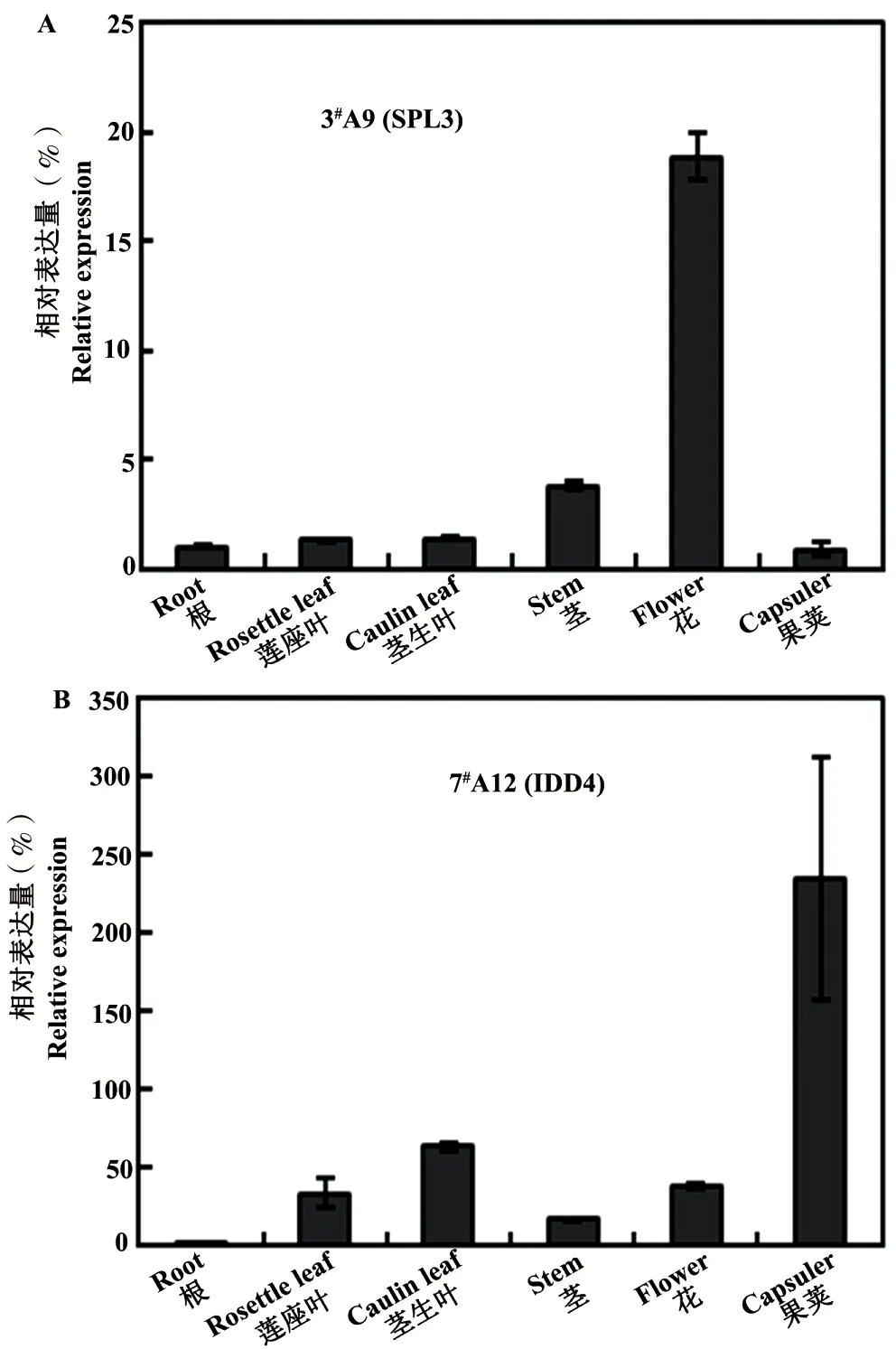
图5 SPL3(A)和IDD4(B)在拟南芥不同组织中的表达Fig 5 SPL3(A) and IDD4(B) expressions in different tissues of Arabidopsis
3 讨论
本研究的最初是为了筛选参与CRY1蓝光信号传导途径的转录因子蛋白,所以在筛选时以在蓝光下具有较强相互作用或具有较强的蓝光依赖性为筛选宗旨。通过多次复筛并提高3-AT的浓度和液体显色试验筛选验证,最终确定SPL3(3#A9)、TCP2(3#E2)和IDD4(7#A12) 3个转录因子蛋白能与CRY1相互作用。筛选出来的3个转录因子中TCP2 (3#E2)因其与CRY1的较强蓝光依赖的相互作用而进行了单独深入的研究[16],SPL3(3#A9)和IDD4(7#A12)与CRY1的相互作用虽然在蓝光下的相互作用比在黑暗下的强,但是它们的相互作用蓝光依赖性比较弱,尤其是SPL3(3#A9),其与CRY1在黑暗下的相互作用也很强(图3B和C)。暗示着第三方一个或者多个蛋白有可能参与它们之间的相互作用而影响CRY1与SPL3相互作用的蓝光依赖性,而这一猜测是否真实需要在植物体内验证其蛋白相互作用的蓝光依赖性。
在CRY1结构域与转录因子蛋白相互作用的分析中发现,CRY1N505和CRYN515的自激活活性较弱,这两个结构域与各转录因子相互作用的蓝光依赖性相比CRY1全长有所减弱,能为以后研究CRY1蛋白与其他蛋白在酵母细胞中的相互作用,提供了可参考的结构域分析以提高相互作用的可靠性。CRY1C301与SPL3(3#A9)的相互作用具有蓝光依赖性,但是强度较弱(图4A),在液体显色试验中,其相互作用β-半乳糖苷酶活性相对于具有自激活活性的其他结构域很小。CRY1C301与IDD4(7#A12)的相互作用没有明显蓝光依赖性,且其黑暗下的相互作用比蓝光下的强(图4B和D)。以上结果初步说明了CRY1与各转录因子蓝光依赖的相互作用所依赖的结构域,但是需要进一步研究以确定CRY1C301是影响CRY1与SPL3(3#A9)相互作用蓝光依赖性的结构域,找出影响CRY1与IDD4(7#A12)蓝光依赖相互作用的具体的结构域。
spl3是一个具有SBP-box的转录因子,是leafy(lfy)、fruitfull(ful)和apetala1(ap1)的上游激活因子,受miRNA156的调节参与植物从营养生长到生殖生长的转换以及顶端组织发育[17-19]。IDD4 (indeterminate domain 4)含有C2H2锌指结构域,与DELLA蛋白相互作用并绑定DELLA蛋白的下游目标基因的启动子如scl3,且DELLA与scl3竞争idd4以调节GA信号通路[20〗。本研究中对筛选出的转录因子的组织表达特异性分析结果与其功能一致,其与cry1的相互作用间接地说明了cry1有可能通过spl3调节植物的开花;通过idd4介导DELLA蛋白调控GA信号通路。
后期我们将通过BiFC、Co-IP以及免疫共定位等手段对CRY1与SPL3和IDD4的相互作用进行进一步的验证,对这两个转录因子在蓝光下的转基因或者突变体表型进行分析以及其他遗传学分析,以确定cry1是否会通过spl3介导植物的开花,通过idd4参与DELLA蛋白对GA信号通路的调控。
[1]MOCKLER T C, GUO H, YANG H, et al. Antagonistic actions of Arabidopsis cryptochromes and phytochrome B in the regulation of floral induction[J]. Development, 1999, 126(10): 2073-2082.
[2]MAZZELLA M A, CERDAN P D, STANELONI R J, et al. Hierarchical coupling of phytochromes and cryptochromes reconciles stability and light modulation of Arabidopsis development[J]. Development, 2001, 128(12): 2291-2299.
[3]AHMAD M, CASHMORE A R. HY4 gene ofA.thalianaencodes a protein with characteristics of a blue-light photoreceptor[J]. Nature, 1993, 366(6451): 162-166.
[4]BLUM D E, NEFF M M, VAN VOLKENBURGH E. Light-stimulated cotyledon expansion in the blu3 and hy4 mutants ofArabidopsisthaliana[J]. Plant Physiology, 1994, 105(4): 1433-1436.[5]LIN C, AHMAD M, CASHMORE A R. Arabidopsis cryptochrome 1 is a soluble protein mediating blue light-dependent regulation of plant growth and development [J]. Plant Journal, 1996, 10(5): 893-902.
[6]AHMAD M, Lin C, CASHMORE A R. Mutations throughout an Arabidopsis blue-light photoreceptor impair blue-light-responsive anthocyanin accumulation and inhibition of hypocotyl elongation[J]. Plant Journal, 1995, 8(5): 653-658.
[7]FUGLEVAND G, JACKSON J A, JENKINS G I. UV-B, UV-A, and blue light signal transduction pathways interact synergistically to regulate chalcone synthase gene expression in Arabidopsis[J]. Plant Cell, 1996, 8(12): 2347-2357.
[8]THUM K E, KIM M, CHRISTOPHER D A, et al. Cryptochrome 1, cryptochrome 2, and phytochrome a co-activate the chloroplast psbD blue light-responsive promoter[J]. Plant Cell, 2001, 13(12): 2747-2760.
[9]LIU B, ZUO Z, LIU H, et al. Arabidopsis cryptochrome 1 interacts with SPA1 to suppress COP1 activity in response to blue light[J]. Genes Development, 2011, 25(10): 1029-1034.
[10]ELICH T D, CHORY J. Phytochrome: if it looks and smells like a histidine kinase, is it a histidine kinase? [J]. Cell, 1997, 91(6): 713-716.
[11]AHMAD M, JARILLO J A, SMIRNOVA O, et al. The CRY1 blue light photoreceptor of Arabidopsis interacts with phytochrome A in vitro[J]. Mol Cell, 1998, 1(7): 939-948.
[12]HUGHES R M, VRANA J D, SONG J, et al. Light-dependent, dark-promoted interaction between Arabidopsis cryptochrome 1 and phytochrome B proteins[J]. The Journal of Biological Chemistry, 2012, 287(26): 22165-22172.
[13]JARILLO J A, CAPEL J, TANG R H, et al. An Arabidopsis circadian clock component interacts with both CRY1 and phyB[J]. Nature, 2001, 410(6827): 487-490.
[14]PRUNEDA-PAZ J L, BRETON G, NAGEL D H, et al. A genome-scale resource for the functional characterization of Arabidopsis transcription factors[J]. Cell Report, 2014, 8(2): 622-632.
[15]曾 婷, 何志敏, 段桂芳,等. 拟南芥转录因子HB22与CRY1相互作用研究[J].生命科学研究, 2013, 17(3): 223-229.
[16]HE Z, ZHAO X, KONG F, et al. TCP2 positively regulates HY5/HYH and photomorphogenesis in Arabidopsis[J]. Journal of Experimental Botany, 2015, 67(3):775-785.
[17]WU G, POETHIG R S. Temporal regulation of shoot development inArabidopsisthalianaby miR156 and its target SPL3[J]. Development, 2006, 133(18): 3539-3347.
[18]YAMAGUCHI A, WU M F, YANG L, et al. The MicroRNA-Regulated SBP-box transcription factor SPL3 is a direct upstream activator of LEAFY, FRUITFULL, and APETALA1[J]. Developmental Cell, 2009, 17(2): 268-278.
[19]CARDON G H, HÖHMANN S, NETTESHEIM K, et al. Functional analysis of theArabidopsisthalianaSBP-box gene SPL3: a novel gene involved in the floral transition[J]. Plant Journal, 1997, 12(2): 367-377.
[20]YOSHIDA H, UEGUCHI-TANAKA M. DELLA and SCL3 balance gibberellin feedback regulation by utilizing indeterminate domain proteins as transcriptional scaffolds[J]. Plant Signaling & Behavior, 2014, 9(9): e29726.
Identification of CRY1-interactive transcription factor proteins
HE Zhi-min1, FENG Pan-pan1, RONG Duo-yan1, ZHAO Xiao-ying1, LIU Xuan-ming1,2
(1. State Key Laboratory of Chemo/Biosensing and Chemometrics, College of Biological Sciences;2. Hunan Province Key Laboratory of Plant Functional Genomics and Developmental Regulation,College of Biological Sciences, Hunan University, Changsha 410082, China )
Arabidopsis CRY1 (cryptochrome 1) protein initiates blue light signal transduction in a protein interaction manner, consequently causes the photomorphogenesis in plants. To further study the mechanism of CRY1 mediating blue light signal transduction, CRY1 was used as a bait, the methods of yeast two hybrid system, such as preliminary auxotrophy, liquid assays and domain interactions analyses in yeast cells were employed to screen the transcription factor proteins from the Arabidopsis transcription factor yeast library. The results showed that 41 transcription factor proteins were screened out by the preliminary auxotrophy and the liquid assays with ONPG as substrate; the transcription factor proteins which interacted with CRY1 with high blue light dependent or strong enzyme activity were picked out for a further validation using liquid assay with CPRG as substrate, and found that their interactions with CRY1 showed blue light dependent and the activity of interactions would increase with the time of blue light treatment; the interactions analyses results of CRY1 domains and transcription factor protein showed that the transcription factor proteins SPL3 and IDD4 interacted with the N terminus of CRY1 primarily, and relatively weak with the C terminus, however, the blue light dependent existed between the interaction of the C terminus of CRY1 and SPL3, but not so obvious with IDD4.
CRY1; yeast two-hybrid system; transcription factors;Arabidopsisthaliana
2016-01-06;
2016-01-14
国家自然科学基金资助项目(31171176);湖南省自然科学基金资助项目(11JJA002);湖南省生物发育工程及新产品研发协同创新中心(20134486)
何志敏,博士,主要从事植物生化分析与分子生物学研究,E-mail:zhiminhe318@163.com;冯盼盼,硕士,主要从事植物生物化学与分子生物学研究,E-mail:13787180145@163.com;何志敏与冯盼盼同为第一作者
刘选明,教授,博士生导师,主要从事植物生化分析与分子生物学研究,E-mail:xml05@hnu.edu.cn;赵小英,教授,博士生导师,主要从事植物生物化学与分子生物学研究,E-mail:zxy_mm@163.com;刘选明与赵小英同为通信作者
Q71;Q943.2
A
2095-1736(2016)05-0047-06
doi∶10.3969/j.issn.2095-1736.2016.05.047

