ssc-miR-148a启动子克隆及其特征分析
王 萍,郝文艳,曹丽华,沈开元,代小丽,李海艳,梁贤威,石德顺*,李湘萍*
(1.广西大学,亚热带农业生物资源保护与利用国家重点实验室,南宁 530005;2.中国农业科学院广西水牛研究所,南宁 530001)
ssc-miR-148a启动子克隆及其特征分析
王萍1,郝文艳1,曹丽华1,沈开元1,代小丽1,李海艳1,梁贤威2,石德顺1*,李湘萍1*
(1.广西大学,亚热带农业生物资源保护与利用国家重点实验室,南宁 530005;2.中国农业科学院广西水牛研究所,南宁 530001)
为研究猪miR-148a(ssc-miR-148a)的转录调控机制,对其启动子进行了克隆及分析。本试验首先设计特异性PCR扩增引物,分别得到ssc-miR-148a前体上游3个片段,并将其连接到荧光素酶报告载体pGL3-Basic上。通过生物信息学方法,在线分析ssc-miR-148a启动子大概区域、甲基化部位和转录因子结合部位。将重组报告质粒转染293T细胞,分析启动子活性。采用不同浓度碱性成纤维生长因子(bFGF)处理猪成纤维细胞和转染有重组报告质粒的猪成纤维细胞,检测ssc-miR-148a和DNA甲基化转移酶1(DNMT1)的表达,及其对启动子活性的影响。结果显示,克隆得到的ssc-miR-148a启动子区2 043 bp片段具有启动子活性,该序列存在5个CpG岛、Sp1及AP2等转录因子结合位点。 0、5和10 ng·mL-1浓度bFGF处理猪成纤维细胞和转染重组报告质粒的猪成纤维细胞后,ssc-miR-148a表达均显著下降(P<0.05),DNMT1 mRNA显著增加(P<0.05)。启动子活性均显著下降(P<0.05),5和10 ng·mL-1浓度间无显著差异(P>0.05)。结果表明,ssc-miR-148a启动子位于前体上游2 043 bp片段内,启动子区域有转录因子SP1结合位点,其表达受bFGF的调控。
ssc-miR-148a;启动子;克隆;bFGF;DNMT1
miRNA是一类调控真核生物基因表达的内源性非编码小RNA分子,长约22个碱基。miRNA的功能是参与转录后基因表达调控,主要通过与目的基因的mRNA转录物反向互补结合,进而抑制靶基因的表达[1];或是通过加速靶标mRNA 3′端poly(A)的脱腺苷酸化,导致整个mRNA迅速降解[2]。根据在染色体上的位置不同,miRNA可分为3类:基因间miRNA、内含子miRNA和位于反义链miRNA。基因间和反义链miRNA具有独立的启动子,基因内miRNA和宿主基因共用启动子[3-4]。现认为miRNA由RNA聚合酶II和RNA聚合酶III转录而来,且大部分由RNA聚合酶II转录[5-6]。研究发现一些miRNA的表达受增强子和激素的调控[7-8]。miRNA自身的转录调控是研究其调控网络的第一步,它对了解miRNA的生物学功能具有重要作用。
miR-148/152家族包括3个成员miR-148a、miR-148b和miR-152。其中miR-148a属于基因间miRNA,具有独立的启动子。miR-148/152家族在许多肿瘤发生和正常器官的生长、发育和胚胎发育过程中都有表达,说明其在这些过程中具有重要作用[9]。miR-148a/152家族参与调控的靶基因涉及增殖(DNMT1[10-11]、P27[12]、CDC25B[13])、分化(ACVR1[14])和凋亡(Bcl2[15])。其中,DNMT1受miR-148/152的调控在人类的一些疾病中已被证实,如系统性红斑狼疮[10]、胆管癌[11]、肝癌[16]、急性淋巴细胞白血病[17]、子宫内膜癌[18]等。DNA甲基化的机制是依赖DNMTs使CpG岛双核苷酸胞嘧啶的5号碳原子发生甲基化,DNMT1是维持性DNA甲基化转移酶,作用是对新生的DNA链进行甲基化。在多种肿瘤细胞中发现miR-148a表达异常,主要是由于其启动子区异常的高甲基化引起的。A.K.Zhu等证实,胃癌细胞中miR-148a启动子区DNA高甲基化导致其表达下调。沉默miR-148a后,降低其对DNMT1的抑制,进而导致DNMT1的过表达[19]。这些研究表明可能存在一个新的miR-148/152-DNMT1循环调控网络。研究表明,miR-148a的表达受激素调控,S.F.Tao等试验证实,胃癌细胞中E2通过GPER抑制miR-148a的表达,可能是通过GPER/EGFR/ERK信号通路影响核转录因子EGR-1的表达,进而下调miR-148a的表达[20]。大家畜的miR-148a启动子区是否含有高CpG位点,其表达是否受激素调控,调控miR-148a的表达是否影响DNMT1的表达等问题尚不清楚。
本试验以猪卵巢为材料扩增ssc-miR-148a启动子,并通过生物信息学方法分析其启动子区内的甲基化位点和转录因子结合位点,同时分析了bFGF对miR-148a表达和启动子活性的影响,以及对DNMT1表达的影响。研究结果为进一步阐述ssc-miR-148a的表达模式及其调控机制奠定了基础。
1 材料与方法
1.1材料
1.1.1试验材料启动子克隆材料为三元杂种猪卵巢基因组(来自南宁鲁班路屠宰场)。pMD18-T克隆试剂盒来自TaKaRa公司;pGL3-Basic、pGL3-promoter、pRL-TK克隆载体为Promega公司产品。293T细胞和猪胎儿成纤维细胞为实验室自存。
1.1.2酶与试剂Taq酶、dNTPs、Supercoiled DNA ladder Marker、DNA Marker、T4 DNA ligase、反转录酶(AMV)均购自大连TaKaRa公司;限制性内切酶XhoⅠ、Hind Ⅲ购自Fermentas公司;琼脂糖(BIOWEST AGAROSE)为进口分装产品;普通质粒小提试剂盒、普通琼脂糖凝胶回收试剂盒购自北京天根公司;去内毒素提取试剂盒购自OMEGA公司;脂质体LipofectamineTM2000购自Invitrogen公司。
1.2方法
1.2.1启动子克隆取约20 mg猪卵巢组织,通过酚/氯仿抽提的方法提取总DNA。从UCSC数据库中下载得到ssc-miR-148a前体上游3 000 bp序列,采用oligo 6设计特异性扩增引物,扩增3段不同长度的启动子片段。PCR反应体系(20 μL):模板1.0 μL、LA Taq 0.25 μL、GC Buffer I 10 μL、dNTPs 2 μL、上下游引物(表1)各0.5 μL、ddH2O 5.75 μL。反应条件:95 ℃预变性5 min;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸2 min,共40个循环;72 ℃延伸7 min。
PCR产物经琼脂糖凝胶电泳分析,并对目的条带胶回收,使其与pMD-18T载体连接,然后转化大肠杆菌DH5α,挑菌后进行扩大培养。Cracking法快速鉴定重组质粒,并用天根普通质粒小提试剂盒提取重组质粒,将含目的片段的重组质粒寄送华大公司测序。
表1ssc-miR-148a启动子克隆及qRT-PCR引物
Table 1Oligonucleotides primers used in ssc-miR-148a promotor clone and qRT-PCR

引物名称Primername(ForR)正向和反向引物序列(5'-3')Primersequence退火温度/℃Annealingtemperature用途ApplicationmiR-148a-0.6KFCTCGAGGTGGAGCGGAGAGAAGAGG60启动子克隆miR-148a-1KFCTCGAGGGAACCTGCTGACTTGACAC60启动子克隆miR-148a-2KFCTCGAGGCCTTTCGGTTAGTCCTTGC60启动子克隆miR-148aRAAGCTTGACCAAAGTGGCTGTCTTCC60启动子克隆Loop-RT-miR-148aGTCGTATCCAGTGCAGGGTCCGAGGTAT-TCGCACTGGATACGACACAAAG-反转录RT-U6CGCTTCACGAATTTGCGTGTCAT-反转录qRT-UGTGCAGGGTCCGAGGT55RT-PCRU6FCTCGCTTCGGCAGCACA55RT-PCRU6RAACGCTTCACGAATTTGCGT55RT-PCRmiR-148a-upGGTCAGTGCACTACAGAACTTT55RT-PCRDNMT1FCAGACAAGTGGTGAGGAC60RT-PCRDNMT1RTGTGAAATGAGATGTGATGG60RT-PCRB-ACTINFGGACCTGACCGACTACCTCA60RT-PCRB-ACTINRCCATCTCCTGCTCGAAGTCC60RT-PCR
下划线为引入酶切位点,Loop-RT-miR-148a反转录特异性引物为茎环引物,反转录特异性引物退火温度根据试剂盒说明确定The underline is the introducing restriction enzyme cutting sites.Loop-RT-miR-148a reverse transcription primer is the specific stem-loop RT primer.Annealing temperature of reverse transcription specificity primer is determined according to kit instructions
1.2.2启动子生物信息学分析将克隆得到的最长片段导入到甲基化分析软件MethPrimer,在线预测甲基化CpG岛。并利用启动子在线分析软件Promoter SCAN、TSSW、TFSEARCH预测ssc-miR-148a启动子区和可能的转录因子结合部位。
1.2.3启动子活性检测载体构建将pMD-18T-148a-0.6K、pMD-18T-148a-1K、pMD-18T-148a-2K和pGL3-Basic重组质粒分别经XhoⅠ和Hind Ⅲ双酶切,琼脂糖凝胶电泳回收酶切片段,用T4 DNA连接酶将回收片段与pGL3-Basic载体连接,重组载体分别命名为pGL3-148a-0.6K、pGL3-148a-1K、pGL3-148a-2K。转化大肠杆菌DH5α,挑菌后进行扩大培养,经双酶切验证的阳性质粒,用去内毒素提取试剂盒提取,-20 ℃保存用于转染细胞。1.2.4启动子活性检测采用含10%胎牛血清的DMEM培养293T细胞,转染前1 d接种1×104个细胞于24孔板,第2天将pGL3-Basic、pGL3-148a-0.6K、pGL3-148a-1K、pGL3-148a-2K、pGL3-promoter及内参照质粒pRL-TK按50∶1的比例用脂质体进行共转染,72 h后收集细胞。按照Dual Luciferase Reporter Assay System(Promega)说明书操作分析启动子活性。
1.2.5bFGF处理猪成纤维细胞将1×105个猪成纤维细胞接种于35 cm培养皿中,分别用含0、5和10 ng·mL-1bFGF的DMEM培养猪成纤维细胞,72 h后收获细胞,TRIzol法提取总RNA,通过紫外分光光度计检测其纯度和完整性。分别对提取的RNA用全式金快速反转试剂盒进行反转录。得到的cDNA经紫外分光光度计检测其浓度,稀释到100 ng·μL-1用于RT-PCR反应,qRT-PCR所用引物见表1。
以相同的bFGF处理条件处理共转pGL3-148a-2K及内参照质粒pRL-TK的成纤维细胞,转染质粒的比例为10∶1,72 h后收获细胞检测双荧光素酶活性。
1.2.6数据分析数据以“mean±SE”表示,各组之间的差异采用t检验分析,P<0.05表示差异有统计学意义。
2 结 果
2.1ssc-miR-148a启动子的克隆及分析
应用特异的PCR引物扩增得到ssc-miR-148a前体上游3个特异性条带,片段大小与预期一致(图1),将PCR片段分别与pMD-18T载体连接,筛选得到阳性重组质粒,并命名为pMD-18T-148a-0.6K、pMD-18T-148a-1K、pMD-18T-148a-2K。经华大公司测序,分析PCR片段大小分别为646、1 355、2 043 bp。利用MethPrimer软件在线分析克隆得到的2 043 bp序列CpG岛情况,发现该序列存在5个CpG岛(图2)。利用启动子在线分析软件Promoter SCAN、TSSW、TFSEARCH,推测克隆得到的2 043 bp片段存在2个启动子区,转录因子以Sp1、AP2为主(图3)。
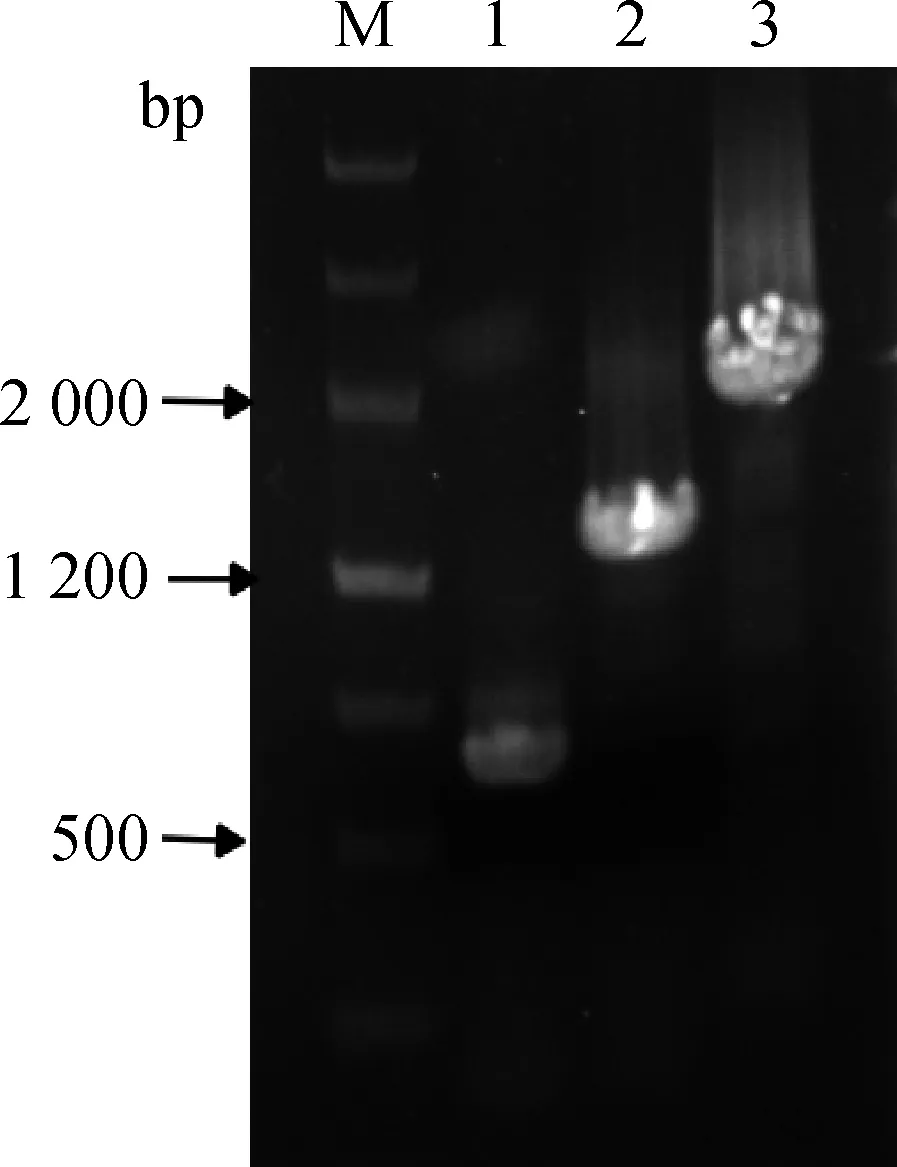
M.Marker III;1.miR-148a-0.6K;2.miR-148a-1K;3.miR-148a-2K图1 ssc-miR-148a启动子PCR扩增结果Fig.1 PCR amplification result of ssc-miR-148a promoter

图2 ssc-miR-148a启动子序列甲基化预测图谱Fig.2 The methylation prediction map of ssc-miR-148a promoter
2.2双荧光素表达载体的构建
将测序正确的重组质粒pMD-18T-148a-0.6K、pMD-18T-148a-1K、pMD-18T-148a-2K和pGL3-Basic分别经XhoⅠ和Hind Ⅲ双酶切,而后连接得到3个具有不同启动子片段长度的双荧光素报告载体pGL3-148a-0.6K、pGL3-148a-1K和pGL3-148a-2K。重组质粒经XhoⅠ和Hind Ⅲ双酶切后,均得到1条预期大小片段(图4),说明重组质粒均为阳性克隆。
2.3启动子表达活性
将含启动子的双荧光素酶报告质粒pGL3-148a-0.6K、pGL3-148a-1K、pGL3-148a-2K、pGL3-promoter分别与内参照质粒pRL-TK共转染293T细胞,以转染质粒pGL3-Basic的细胞为对照。转染72 h后检查荧光素酶的表达水平,计算Fluc/Rluc的比值。与对照质粒相比,转染pGL3-148a-0.6K、pGL3-148a-1K质粒组细胞的荧光素酶比值无显著变化(P>0.05),转染pGL3-148a-2K和pGL3-promoter质粒组细胞的荧光素酶比值显著增加(P<0.05),说明pGL3-148a-2K具有启动子效率(图5)。
2.4bFGF对相关基因表达及启动子活性的影响
采用0、5和10 ng·mL-1的bFGF分别处理猪成纤维细胞及共转含启动子的双荧光素酶报告质粒pGL3-148a-2K和内参照质粒pRL-TK的猪成纤维细胞,72 h后收集细胞提取RNA进行分析。RT-PCR分析结果显示,5和10 ng·mL-1bFGF处理后,miR-148a表达均显著下降(P<0.05),DNMT1 mRNA显著增加(P<0.05)(图6)。双荧光素酶表达水平均显著下降(P<0.05),5和10 ng·mL-1浓度间无显著差异(P>0.05)(图7)。以上结果说明bFGF可通过影响ssc-miR-148a启动子活性,进而调控相关基因表达。

图3 ssc-miR-148a启动子调控区序列Fig.3 Nucleotide sequence of the regulatory region of the ssc-miR-148a
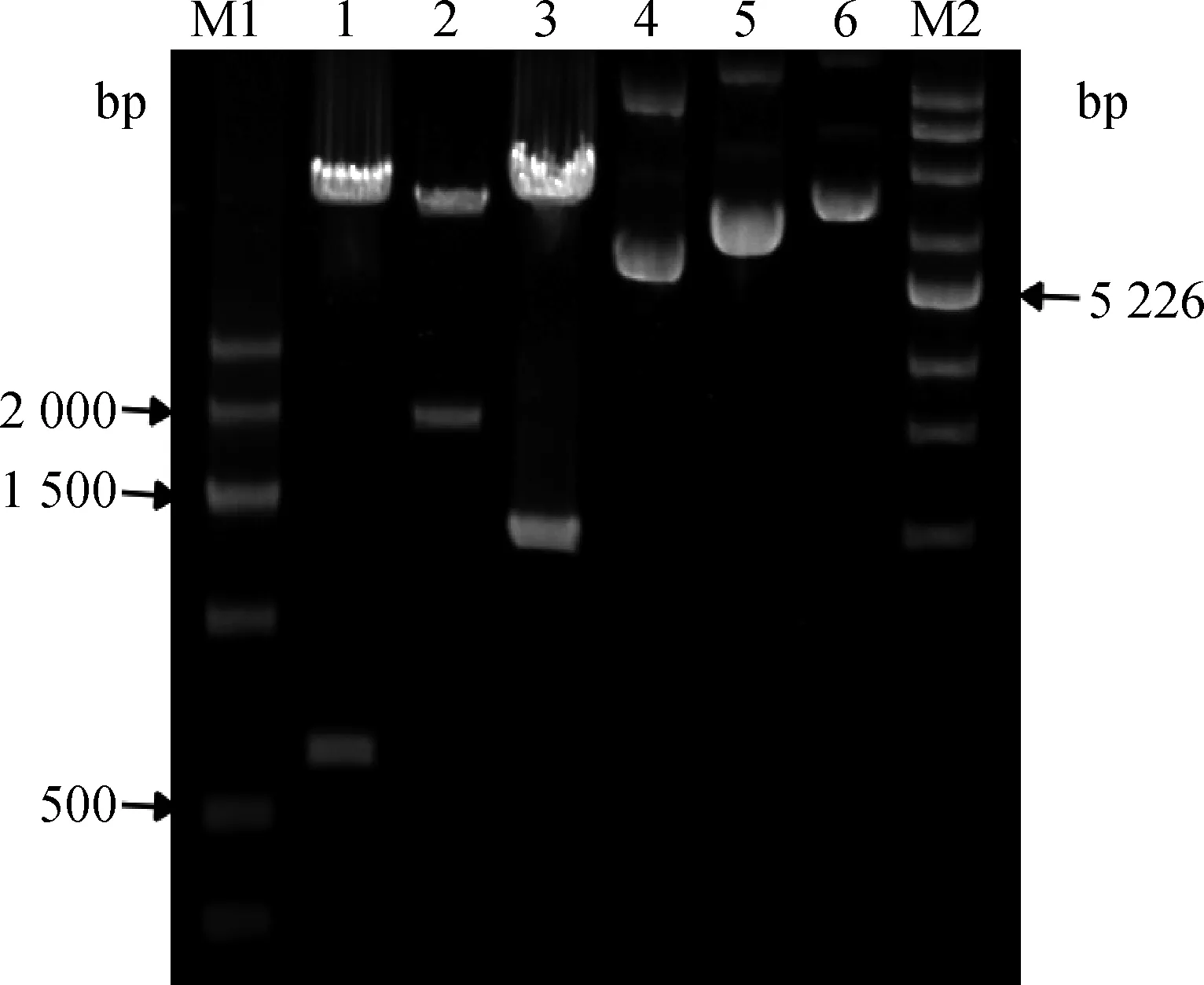
M1.Marker VII;1.miR-148a-0.6K;2.miR-148a-2K;3.miR-148a-1K;4.pGL3-148a-0.6K;5.pGL3-148a-1K;6.pGL3-148a-2K;M2.Supercoiled DNA ladder marker图4 重组质粒双酶切鉴定结果Fig.4 The enzyme digestion results of recombinant plasmid
3 讨 论
H.K.Saini等利用生物信息学方法分析了人类基因间miRNA启动子转录起始位点(Transcriptional start site,TSS)的位置,发现大部分基因间miRNA TSS位于pre-miRNA上游2 kb内[21]。因此,本试验克隆了ssc-miR-148a前体上游最长2 043 bp片段并对其进行了分析。X.F.Zhou等对4种模式生物的miRNA启动子进行分析发现,80%的miRNA核心启动子位于转录起始位点上游-500 bp内,has-mir-148a启动子区域位于pre-miR-148a上游-395 bp处[22]。A.Lujambio等通过5′RACE方法得到has-miR-148a的TSS位于pre-miR-148a上游-518 bp处[23]。M.Bhattacharyya等通过分析人pri-miRNA 5′UTR区CAGE tags、TSS Seqs、H3K4me3预测miRNA转录起始位点,得到hsa-miR-148a TSS位于pre-miR-148a上游-1 097 bp处[24]。将人和猪miR-148a上游-3 000 bp序列进行比对,同源性为83%,推测人和猪可能具有相似的转录起始位点、启动子和转录因子结合位点。将克隆得到的ssc-miR-148a前体上游2 kb序列进行预测分析,存在两个启动子区。双荧光素酶报告分析结果显示,pre-ssc-miR-148a上游2 043 bp具有启动子活性,646和1 355 bp不具有启动子活性,说明ssc-miR-148a启动子位于-1 355~-2 043 bp。这与软件预测的启动子区结果接近,同时与A.Lujambio等[23]得到的人miR-148a TSS位点在上游500 bp接近,但与X.F.Zhou等[22]预测的启动子区位于-395 bp不同,可能是由于miR-148a具有多个启动子区,其在不同组织中的表达由不同的启动子启动。
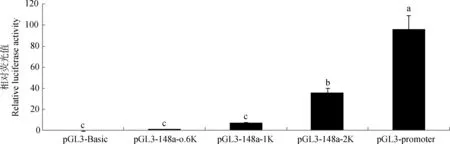
图中不同字母表示差异显著(P<0.05)。下同Different letters means significant difference (P<0.05).The same as below图5 启动子活性检测结果Fig.5 Detection of promoter activity
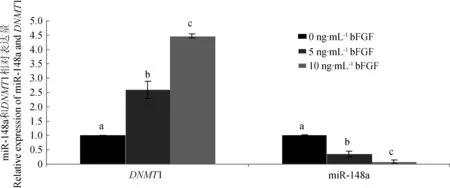
图6 bFGF对ssc-miR-148a和DNMT1表达的影响Fig.6 The effect of bFGF on the expression of ssc-miR-148a and DNMT1

图7 bFGF对启动子活性的影响Fig.7 The effect of bFGF on promoter activity
碱性成纤维生长因子(bFGF)是一种多效能生长因子,诱导的信号转导通路包括促分裂原活化蛋白激酶(MAPK)途径、Src家族的酪氨酸激酶、磷酸肌醇3激酶(PI3K)和磷脂酶(PLC)途径[25]。其中,MAPK作用的底物有ERK、p38和JNK,这些激酶活化后转移到细胞核内调节转录因子的活性。转录因子SP1被ERK磷酸化并激活,磷酸化的SP1可以上调或下调目的基因。M.Wang等研究表明bFGF通过ERK/Sp1通路下调TSP50的表达[26]。S.Liu等研究KIT过表达引起的急性髓性白血病,Sp1与NFκB/HDAC形成复合体,结合到miR-29b启动子区抑制miR-29b的表达[27]。H.Liu等研究睾丸间质细胞发现,bFGF可以通过调节miRNA的表达,抑制LH诱导的雄激素生成过程,在前体睾丸间质细胞中miR-29a、miR-29c、miR-142-3p表达下调;miR-451、miR-335表达上调[28]。T.Numakawa研究发现,bFGF处理皮质神经细胞促进miR-132的表达,处理星形胶质细胞使miR-134表达上调,可能是通过ERK1/2的激活引起的[29-30]。本研究通过生物信息学预测,ssc-miR-148a启动子区含有大量Sp1结合位点,bFGF处理猪成纤维细胞影响了miR-148a启动子活性,使miR-148a表达受到抑制。S.F.Tao等试验证实,胃癌细胞中E2通过GPER抑制miR-148a的表达,可能是通过GPER/EGFR/ERK通路影响核转录因子EGR-1的表达,进而下调miR-148a的表达,这与bFGF影响miR-148a的表达通路相似[20]。bFGF和E2是否通过MAPK-ERK-Sp1/EGR-1调控miR-148a表达的还需要后续试验验证。
在许多肿瘤细胞中发现miR-148/152家族基因DNA高甲基化。N.Hanoun等研究胰腺导管腺癌发现,miR-148a因DNA编码区高甲基化而表达降低[31]。A.Lujambio等比较了正常组织与癌细胞中miR-148a CpG岛甲基化情况,发现癌细胞miR-148a CpG岛高甲基化,并用DNA去甲基化药物(5-aza-2′-deoxycytidine)处理癌细胞,miR-148a的表达上调[23]。DNMT1作为miR-148a的一个靶基因在许多肿瘤细胞中都已有报道。Q.Xu等发现DNMT1过表达会引起miR-148a启动子高甲基化[32]。因此,miR-148a 启动子区CpG岛的甲基化可能降低miR-148a的表达,DNMT1是miR-148a的一个靶基因,相反,也可以影响miR-148a的表达,在肿瘤细胞中存在miR-148a-DNMT1调控通路。本研究克隆得到的miR-148a启动子,预测含有5个CpG岛。bFGF处理成纤维细胞,miR-148a表达下调,DNMT1表达上调,可能是由于miR-148a的降低,解除了对DNMT1的抑制作用。本研究为体细胞核移植中miR-148a的表达及作用奠定了基础。
[1]KIM V N,HAN J,SIOMI M C.Biogenesis of small RNAs in animals[J].NatRevMolCellBiol,2009,10(2):126-139.
[2]WU L,ZHAO Q,ZHU X,et al.A novel function of microRNA Let-7d in regulation of galectin-3 expression in attention deficit hyperactivity disorder rat brain[J].BrainPathol,2010,20(6):1042-1054.
[3]GRIFFITHS-J S,SAINI H K,DONGEN S V,et al.miRBase:tools for microRNA genomics[J].NucleicAcidsRes,2007,36:D154-D158.
[4]RODRIGUEZ A,GRIFFITHS-J S,ASHURST J,et al.Identification of mammalian microRNA host genes and transcription units[J].GenomeRes,2004,14(10A):1902-1910.
[5]LEE Y,KIM M,HAN J,et al.microRNA genes are transcribed by RNA polymerase II[J].EMBOJ,2004,23:4051-4060.
[6]BORCHERT G M,LANIER W,DAVIDSON B L.RNA polymerase III transcribes human microRNAs[J].NatStructMolBiol,2006,13(12):1097-1101.
[7]BRENNECKE J,HIPFNER D H,STARK A,et al.Bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila[J].Cell,2003,113(1):25-36.
[8]SEMPERE L F,SOKOL N S,DUBROVSKY E B,et al.Temporal regulation of microRNA expression in Drosophila melanogaster mediated by hormonal signals and Broad-Complex gene activity[J].DevBiol,2003,259(1):9-18.
[9]CHEN Y,SONG Y X,WANG Z N.The microRNA-148/152 family:multi-faceted players[J].MolCancer,2013,12:43.
[10]BRACONI C,HUANG N,PATE T.microRNA-dependent regulation of DNA methyltransferase-1 and tumor suppressor gene expression by interleukin-6 in human malignant cholangiocytes[J].Hepatology,2010,51(3):881-890.
[11]PAN W,ZHU S,YUAN M,et al.microRNA-21 and microRNA-148a contribute to DNA hypomethylat in lupus CD4+T cells by directly and indirectly targeting DNA methlytransferase 1[J].JImmunol,2010,184:6773-6781.
[12]GUO S L,PENG Z,YANG X,et al.miR-148a promoted cell proliferation by targeting p27 in gastric cancer cells[J].IntJBiolSci,2011,7(5):567-574.
[13]LIFFERS S T,MUNDING J B,VOGT M,et al.microRNA-148a is down-regulated in human pancreatic ductal adenocarcinomas and regulates cell survival by targeting CDC25B[J].LabInvest,2011,91:1472-1479.
[14]SONG H,WANG Q,WEN J G,et al.ACVR1,a therapeutic target of fibrodysplasia ossificans progressiva,is negatively regulated by miR-148a[J].IntJMolSci,2012,13(2):2063-2077.
[15]ZHANG H,LI Y,HUANG Q,et al.miR-148a promotes apoptosis by targeting Bcl-2 in colorectal cancer[J].CellDeathDiffer,2011,18:1702-1710.
[16]HUANG J F,WANG Y,GUO Y J,et al.Down-regulated microRNA-152 induces aberrant DNA methylation in hepatitis B virus-related hepatocellular carcinoma by targeting DNA methyltransferase 1[J].Hepatology,2010,52(1):60-70.
[17]STUMPEL D J,SCHOTTE D,LANGE-T E A,et al.Hypermethylation of specific microRNA genes in MLL-rearranged infant acute lymphoblastic leukemia:major matters at a micro scale[J].Leukemia,2010,25(3):429-439.
[18]TSURUTA T,KOZAKI K,UESUGI A,et al.miR-152 is a tumor suppressor microRNA that is silenced by DNA hypermethylation in endometrial cancer[J].CancerRes,2011,71(20):6450-6462.
[19]ZHU A K,XIA J Z,ZUO J B,et al.microRNA-148a is silenced by hypermethylation and interacts with DNA methyltransferase 1 in gastric cancer[J].MedOncol,2012,29(4):2701-2709.
[20]TAO S F,HE H F,CHEN Q.Estradiol induces HOTAIR levels via GPER-mediated miR-148a inhibition in breast cancer[J].JTranslMed,2015,13:131.
[21]SAINI H K,GRIFFITHS-J S,ENRIGHT A J,et al.Genomic analysis of human microRNA transcripts[J].ProcNatlAcadSciUSA,2007,104(45):17719-17724.
[22]ZHOU X F,RUAN J H,WANG G D,et al.Characterization and identification of microRNA promoters in four model species[J].PLoSComputBiol,2007,3(3):e37.
[23]LUJAMBIO A,CALIN G A,LILLANUEVA A,et al.A microRNA DNA methylation signature for human cancer metastasis[J].ProcNatlAcadSciUSA,2008,105(36):13556-13561.
[24]BHATTACHARYYA M,DAS M,BANDYOPADHYAY S.miRT:A database of validated transcription start sites of human microRNAs[J].GenomicsProteomicsBioinformatics,2012,10(5):310-316.
[25]MAKINO T,JINNIN M,MUCHEMWA F C,et al.Basic fibroblast growth factor stimulates the proliferation of human dermal fibroblasts via the ERK1/2 and JNK pathways[J].BrJDermatol,2010,162(4):717-723.
[26]WANG M,BAO Y L,WU L,et al.Basic FGF downregulates TSP50 expression via the ERK/Sp1 pathway[J].JCellBiochem,2010,111(1):75-81.
[27]LIU S,WU L C,PANG J,et al.Sp1/NFκB/HDAC/miR-29b regulatory network in KIT-Driven myeloid leukemia[J].CancerCell,2010,17(4):333-347.
[28]LIU H,YANG Y,ZHANG L,et al.Basic fibroblast growth factor promotes stem Leydig cell development and inhibits LH-stimulated androgen production by regulating microRNA expression[J].JSteroidBiochemMolBiol,2014,144:483-491.
[29]NUMAKAWA T,YAMAMOTO N,AHIBA S,et al.Growth factors stimulate expression of neuronal and glial miR-132[J].NeurosciLett,2011,505(3):242-247.
[30]NUMAKAWA T,NAKAJIMA S,YAMAMOTO N,et al.Basic fibroblast growth factor induces miR-134 upregulation in astrocyte for cell maturation[J].BiochemBiophysResCommun,2015,456(1):465-470.
[31]HANOUN N,DELPU Y,SURIAWINATA A A,et al.The silencing of microRNA 148a production by DNA hypermethylation is an early event in pancreatic carcinogenesis[J].ClinChem,2010,56(7):1107-1118.
[32]XU Q,JIANG Y,YINY,et al.A regulatory circuit of miR-148a/152 and DNMT1 in modulating cell transformation and tumor angiogenesis through IGF-IR and IRS1[J].JMolCellBiol,2013,5(1):3-13.
(编辑郭云雁)
Cloning and Analysis of ssc-miR-148a Promoter
WANG Ping1,HAO Wen-yan1,CAO Li-hua1,SHEN Kai-yuan1,DAI Xiao-li1,LI Hai-yan1,LIANG Xian-wei2,SHI De-shun1*,LI Xiang-ping1*
(1.StateKeyLaboratoryforConservationandUtilizationofSubtropicalAgro-bioresource,GuangxiUniversity,Nanning530005,China;2.BuffaloResearchInstitute,ChineseAcademyofAgriculturalSciences,Nanning530001,China)
In order to understand the transcriptional regulatory mechanism of ssc-miR-148a,we have cloned and analyzed its promoter.Firstly,we designed specific PCR primers to amplify 3 upstream fragments of ssc-miR-148a precursors,then inserted them into pGL3-Basic expression vector.Based on the amplified fragments,ssc-miR-148a promoter region,methylation sites and transcription factor binding sites were predicted by bioinformatics.The plasmid was transfected into 293T cells to analyze the promoter activity.Expression of ssc-miR-148a and DNA methylation transferase 1 (DNMT1) in treated porcine fibroblasts by Basic Fibroblast Growth Factor(bFGF)with different concentrations was detected.The results showed that the cloned 2 043 bp sequence had promoter activity,which had 5 CpGs and transcriptional factor binding sites,such as Sp1,AP2.After treated porcine fibroblasts with 0,5 and 10 ng·mL-1bFGF,the expression of ssc-miR-148a was decreased significantly(P<0.05),DNMT1 mRNA level increased significantly (P<0.05).The promoter activity significantly reduced (P<0.05),but no significant difference between 5 and 10 ng·mL-1concentrations (P>0.05).The results indicate that the promoter region of ssc-miR-148a locate at -2 043 bp,which has Sp1 transcription factor binding sites.The expression of ssc-miR-148a is regulated by the bFGF.
ssc-miR-148a;promoter;cloning;bFGF;DNMT1
10.11843/j.issn.0366-6964.2016.10.004
2015-11-19
广西壮族自治区研究生教育创新计划项目(YCBZ2014013);国家自然科学基金(31560632);广西自然科学基金项目(2014GXNSFAA118084);广西水牛遗传繁育重点实验室开放课题项目(SNKF-2014-03)
王萍(1987-),女,山东济宁人,博士,主要从事动物遗传育种与繁殖研究,E-mail:wangping.online@163.com
李湘萍,研究员,博士生导师,E-mail:xiangpingli@163.com;石德顺,研究员,博士生导师,E-mail:ardsshi@gxu.edu.cn
S828;S813.3
A
0366-6964(2016)10-1969-08

