CRISPR/Cas9介导RB1基因敲除及其在鸡前脂肪细胞分化、增殖中的功能研究
张 琦,黄娇娇,杨彩侠,王宇祥,张 慧,赵建国,李 辉*
(1.东北农业大学动物科学技术学院,哈尔滨 150030; 2.农业部鸡遗传育种重点实验室,哈尔滨150030;3.中国科学院动物研究所 干细胞与生殖生物学国家重点实验室,北京 100101)
CRISPR/Cas9介导RB1基因敲除及其在鸡前脂肪细胞分化、增殖中的功能研究
张琦1,2,黄娇娇3,杨彩侠1,王宇祥1,2,张慧1,2,赵建国3,李辉1,2*
(1.东北农业大学动物科学技术学院,哈尔滨 150030; 2.农业部鸡遗传育种重点实验室,哈尔滨150030;3.中国科学院动物研究所 干细胞与生殖生物学国家重点实验室,北京 100101)
旨在研究RB1基因在鸡前脂肪细胞分化和增殖过程中的功能,本研究利用CRISPR/Cas9系统在鸡前脂肪细胞中敲除RB1基因,检测鸡前脂肪细胞的分化和增殖能力及相关标志基因的表达水平。结果表明,CRISPR/Cas9系统能够有效介导RB1基因的敲除并引起RB1基因翻译的提前终止,敲除效率可以达到10%。油红O提取比色和CCK-8的结果显示,敲除RB1基因后,鸡前脂肪细胞的分化能力减弱、增殖能力增强;qRT-PCR结果显示,RB1基因敲除之后脂肪细胞分化标志基因G0S2、FAS、A-FABP的mRNA表达水平下降,尤其是G0S2基因的表达水平在前脂肪细胞分化的各个阶段都表现为显著的下降。同时RB1基因敲除还引起细胞周期相关基因CyclinD1、E2F1、Ki67、PCNA的mRNA表达水平升高。初步推断,RB1基因可能是调控鸡前脂肪细胞分化和增殖的关键调节因子。
CRISPR/Cas9;RB1;鸡;脂肪沉积;分化;增殖
肉鸡是全世界广泛饲养并具有重要经济价值的禽类。由于高强度选择,肉鸡的生长速度和肉产量得以明显提高。然而肉鸡体内脂肪(尤其是腹脂)蓄积过多一直是困扰世界范围内肉鸡育种工作者的重大问题。脂肪组织中脂肪细胞的过度增殖和分化会造成脂肪形成加剧从而引发机体脂肪的过度蓄积。因此,研究脂肪沉积的分子遗传机制对防止动物体内脂肪过度沉积,提高肉鸡的饲料转化效率具有重要的意义。脂肪细胞的分化和增殖是一个由众多因子共同调控的复杂过程,本课题组前期对鸡1号染色体上影响腹脂性状的QTL(数量性状基因座)进行了初步定位,在鸡1号染色体上发现了多个影响鸡腹脂重和腹脂率的QTL;进一步通过加大标记密度和扩大群体规模的方法将影响鸡腹脂率的QTL精细定位在鸡1号染色体上3.7 Mb的区域内;对该区域内的基因进行分析,结果鉴定出了RB1(视网膜母细胞瘤基因)等与鸡体脂性状相关的重要基因[1-3]。因此,本课题组选择RB1基因作为影响鸡腹脂沉积的重要基因进行后续研究。在哺乳动物上针对RB1基因的研究较多,它参与机体内许多生长发育调节过程比如细胞周期调控、细胞凋亡和细胞衰老[3],但在家禽上尤其是对鸡脂肪细胞分化和增殖的影响的研究比较少。
自2013年CRISPR/Cas9系统的出现为基因组编辑提供了很好的解决方案。CRISPR系统是许多细菌的一种免疫防御机制,可用来抵抗外源核酸(如病毒或质粒)的入侵[4]。CRISPR/Cas9系统利用人工设计的向导RNA(Single-guide RNA,sgRNA)介导外源表达的Cas9蛋白与基因组的靶点特异性地结合,从而实现对基因组DNA特异性地切割。该系统已经在多种模式生物、细胞以及家畜中得到大量研究与应用,具有非常广阔的应用前景[5-7]。但是迄今为止,Cas9介导的基因组编辑在家禽上尚未见报道。
本研究的主要目的有两个:一是探讨CRISPR/Cas9技术对鸡细胞基因组编辑的可行性;二是以鸡前脂肪细胞为模型,利用CRISPR/Cas9系统构建RB1基因敲除的鸡前脂肪细胞,检测敲除RB1基因后细胞的分化和增殖能力,研究RB1对鸡脂肪细胞分化和增殖的作用机制。
1 材料与方法
1.1材料
试验所用的细胞为实验室前期构建的鸡前脂肪细胞系(命名为ICPA-II),Cas9载体为pX330-U6-Chimeric_BB-CBh-hSpCas9 载体。
1.2方法
1.2.1gRNA的设计和载体构建利用MIT张峰实验室提供的在线工具(http://crispr.mit.edu/)进行gRNA的设计。根据鸡RB1基因的序列(GenBank登录号:NM_204419),针对RB1基因的第5外显子和第6外显子共设计了3个gRNAs,构建gRNA的寡核苷酸序列分别为(下划线标记的碱基为构建gRNA的寡核苷酸上引入的BbsI酶切位点):RB1-5E-gRNA1-F:5′-CACCGCAAAGAGGTGGATGTTAACA-3′,RB1-5E-gRNA1-R:5′-A-AACTGTTAACATCCACCTCTTTGC-3′;RB1-6E-gRNA1-F:5′-CACCGGACTTGTGGCCTTATTT-ATC-3′,RB1-6E-gRNA1-R:5′-AAACGATAAAT-AAGGCCACAAGTCC-3′;RB1-6E-gRNA2-F:5′-CACCGTATTTATCTGGAGCAACCCA-3′,RB1-6E-gRNA2-R:5′-AAACTGGGTTGCTCCAGAT-AAATAC-3′。
利用限制性内切酶片段长度多态性(RFLP)技术对上述3条gRNAs打靶后细胞的基因型进行鉴定。RB1-5E-gRNA1目标打靶区域存在HpaI酶切位点,RB1-6E-gRNA1和RB1-6E-gRNA2目标打靶区域存在HaeIII酶切位点。打靶成功可能造成潜在打靶位置附近基因组碱基的突变、缺失或者插入,从而使扩增的包含打靶片段的PCR产物不能被HpaI或HaeIII内切酶切割开。根据限制性内切酶的切割与否可以初步断定RB1是否发生了基因打靶并分析打靶效率。筛选为阳性的细胞克隆再通过测序确定RB1基因的突变类型。
针对RB1基因的gRNA-CRISPR/Cas9打靶载体的构建:1)pX330空载体的酶切:用BbsI酶(NEB,R0539)线性化pX330质粒,并进行切胶回收。2)gRNA寡核苷酸双链的合成:将合成的两条寡核苷酸以100 μmol·L-1的形式稀释,取等量的上游链和下游链混合。反应程序:95 ℃ 210 s,降温至室温。3)gRNA寡核苷酸双链与线性化的pX330载体连接:连接体系:线性化的pX330载体35 ng、退火形成的gRNA寡核苷酸双链2 μL、T4 buffer 1 μL、T4连接酶1 μL、补水至10 μL。连接条件:室温5 min。随后将连接产物进行转化,并挑取单克隆菌落进行测序,将测序结果正确的菌液进行质粒大提。构建的3个载体的名称分别为RB1-g4、RB1-g5、RB1-g6。
1.2.2gRNA的体外转录及体外切割活性检测根据InvitroTranscription T7 Kit(TaKaRa,6140)试剂盒的说明书进行gRNA的体外转录。将体外转录获得的gRNA与包含潜在打靶区域的DNA片段按照Cas9酶(NEB,M0386)的说明书进行酶切。酶切体系:包含潜在打靶区域的DNA片段150 ng、gRNA 100 ng、Cas9酶1 μL、10×buffer 2 μL、补水至20 μL。反应条件:37 ℃ 酶切1 h。酶切后的产物利用1.5%琼脂糖凝胶电泳进行检测,根据电泳结果分析3个gRNAs的体外切割活性。
1.2.3细胞转染细胞培养条件:10%血清的DMEM(Gibco,12800017)无抗生素培养基,37 ℃ 5% CO2中培养。野生型的鸡前脂肪细胞按照6×104个·孔-1的浓度传到6孔板中,然后按照LipofectamineTM2000 Transfection Reagent(Invitrogen,11668019)操作说明书进行转染。将前期构建的RB1-g4、RB1-g5、RB1-g6质粒和GFP质粒共转。转染后24 h利用流式细胞仪进行分选,将带有绿色荧光的细胞分选到96孔板中,每孔1个细胞。
1.2.4CRISPR/Cas9介导的细胞打靶效率和突变模式检测将分选好的细胞进行培养,适时传代。当细胞传到24孔板时,每个孔取一半细胞继续培养,剩下的一半细胞提取基因组进行打靶效率鉴定和突变模式检测。对提取的细胞基因组的打靶区域进行PCR扩增,PCR扩增引物:RB1-5e-F:5′-AGCCAAGGTATCAGTGTTGGG-3′,RB1-5e-R:5′-AAACGGGCACTTCTCAGACA-3′。
对扩增的PCR产物进行酶切鉴定,不能被HpaⅠ酶切开的产物连接到pMD18-T载体上进行测序,进一步分析鸡RB1基因的突变模式。
1.2.5前脂肪细胞诱导分化和油红O染色及提取比色利用油酸诱导前脂肪细胞分化,然后利用油红O染色和提取比色的方法检测细胞的脂滴沉积情况,从表观上观察细胞的分化情况。具体步骤:向贴壁细胞中加入油酸(终浓度320 μmol·L-1)诱导细胞分化,持续诱导24、48、72和96 h后,弃去培养基,用PBS洗3遍,10%甲醛固定30 min,PBS洗2遍。油红O工作液染10 min,弃去多余的染色液,60%异丙醇分色10~20 s,蒸馏水冲洗3次,室温干燥,100%异丙醇溶解被染细胞中的油红O 15 min。用100%异丙醇作为空白对照,用酶标仪测定 510 nm波长的吸光度值,每组内设置3个重复。
1.2.6CCK-8法检测细胞增殖将敲除组和野生型组的细胞分别计数。然后将细胞传入96孔板中,每个孔放入4 800个细胞。分别在细胞增殖0、24、48、72和96 h时向细胞培养基中加入培养基体积10%的CCK-8溶液,将细胞放入培养箱中培养1 h,然后用酶标仪测定450 nm波长的吸光度值。每组内设置3个重复。
1.2.7基因表达检测用qRT-PCR方法进行相关基因表达水平的定量分析。qRT-PCR反应按FastStart Universal SYBR Green Master(ROX)(Roche)说明书操作。反应体系:FastStart Universal SYBR Green Master(ROX)(2×) 5 μL,上游引物(10 μmol·L-1) 0.2 μL,下游引物(10 μmol·L-1) 0.2 μL,cDNA模板 1 μL,ddH2O 3.6 μL。反应条件:95 ℃预变性 10 min,95 ℃变性 15 s,60 ℃复性延伸 60 s,共40个循环。熔解曲线:95 ℃ 15 s,60 ℃ 10 min,95 ℃ 15 s,每个样品设3孔重复。以β-actin基因为内参,利用2-△Ct方法将原始Ct值转换为相对的基因表达量。所用引物序列见表1。
2 结 果
2.1鸡RB1基因打靶载体的构建以及gRNA打靶效率的体外酶切检测
将设计好的3个gRNA载体RB1-g4、RB1-g5、RB1-g6进行体外转录,获得mRNA(图1A),将上述得到的mRNA、包含潜在打靶区域的DNA片段和Cas9蛋白共孵育,通过比较DNA片段被酶切的比例来估计相应gRNA的打靶效率(酶切率=切开条带/检测条带的吸光度值)。结果显示,RB1-g4组的DNA片段全部被酶切,体外切割效率为100%,而RB1-g5和RB1-g6的体外切割效率分别为70%和60%(图1B)。所以后续选取RB1-g4进行细胞转染等试验。
表1qRT-PCR的引物
Table 1Primers used for qRT-PCR

基因名称NameGenBank登录号GenBankaccessionNo.引物序列(5'-3')Primersequence产物长度/bpSize退火温度/℃AnnealingtemperatureRB1NM_204419GCCTTATTTATCTGGAGCAACAAGACGCACAGCAACAACT16060CyclinD1NM_205381AGAAGTGCGAAGAGGAAGTTGATGGAGTTGTCGGTGTA18660PCNANM_204170GTGCTGGGACCTGGGTTCGTATCCGCATTGTCTTCT15960Ki67NM_205505AGGTCCGTTCCCTCGTTCATTGTCGTCTGGGTCATC27060G0S2NM_001190924CGGGGCGAAAGAGCTGAGAGCACGTACAGCTTCACCAT17160A-FABPAY675941ATGTGCGACCAGTTTGTTCACCATTGATGCTGATAG14360E2F1NM_205219GGAATGGGTGCTGTGGGAGATAGCCAGGGAGGAGGAAACAAAC25360FASNM_205155AAGGAGGAAGTCAACGGTTGATGGTGAGGAGTCG19660β-actinNM_205518TCTTGGGTATGGAGTCCTGTAGAAGCATTTGCGGTGG33160
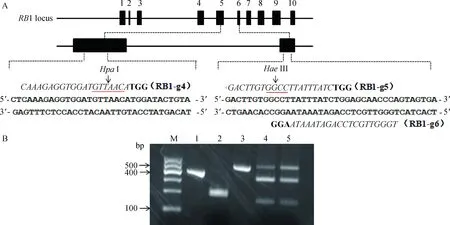
A.RB1基因上gRNA的靶位点示意图:黑色方框为外显子,斜体字母是gRNA的打靶序列,加粗字母是PAM序列,带有下划线的部分是打靶区域的酶切位点。B.各个gRNA打靶效率的体外酶切检测结果:1.包含RB1-g4潜在打靶区域的DNA片段,长度为403 bp;2.RB1-g4打靶效率的体外酶切检测结果,将DNA片段切成213和190 bp;3.包含RB1-g5和RB1-g6潜在打靶区域的DNA片段,长度为499 bp;4、5.分别是RB1-g5和RB1-g6打靶效率的体外酶切检测结果,将DNA片段切割成了346和153 bpA.gRNA target sites in RB1:Exons are shown as black boxes,the gRNA-targeting sequences are the labeled in italic letters,and the protospacer-adjacent motif (PAM) sequences are the labeled in bold letters,the restriction sites at the target regions are underlined.B.In vitro cleavage activity assay of gRNA:The 1st (403 bp) and 3rd lanes (499 bp) are positive control;The 2nd lane is the RFLP result of RB1-g4 targeting activity in vitro detection,which separates the amplification fragment into 213 and 190 bp;The 4th and 5th lanes indicate the results of RB1-g5 and RB1-g6 targeting activity in vitro detection,which separate the amplification fragment into 346 and 153 bp图1 gRNA的设计和打靶效率检测Fig.1 Design and in vitro cleavage activity assay of gRNA
2.2CRISPR/Cas9介导的鸡前脂肪细胞中RB1基因的敲除
将RB1-g4质粒和GFP质粒共转染ICPA-Ⅱ细胞。对筛选到的细胞提取DNA进行PCR扩增,然后用HpaⅠ内切酶对PCR产物进行切割。共鉴定了10个细胞克隆,其中有1个克隆(命名为RB1-KO-1-4)即第3泳道的PCR产物不能被HpaⅠ酶切开(图2A),初步推算CRISPR/Cas9系统在鸡前脂肪细胞系中RB1基因的敲除效率为10%(1/10)。RB1-KO-1-4细胞克隆的PCR产物测序结果显示,这个细胞克隆的两条同源染色体的打靶类型分别为插入1个碱基和缺失5个碱基,造成了氨基酸的移码突变(图2B),使氨基酸翻译提前终止(图2C)。对敲除RB1基因的鸡前脂肪细胞和野生型的鸡前脂肪细胞进行了qRT-PCR分析,结果发现敲除RB1基因的鸡前脂肪细胞的RB1基因表达量极显著(P<0.01)的低于野生型鸡前脂肪细胞的RB1基因表达量(图2D)。由于目前针对鸡这个物种的Western blot的抗体较少,之前的试验中也尝试了一些商业化的针对哺乳动物RB1基因的抗体,但是都没有特异性反应。所以本试验没有利用Western blot的方法验证RB1基因的敲除。

A.HpaⅠ酶切鉴定;B.RB1敲除细胞的基因组类型;C.基因突变和氨基酸突变结果,1个碱基的插入和5个碱基的缺失导致了氨基酸翻译提前终止;D.鸡RB1基因在野生型的鸡前脂肪细胞和敲除RB1基因的鸡前脂肪细胞中的表达情况。*.P<0.05,**.P<0.01,下同A.gRNA mutagenesis efficiencies were assessed by HpaⅠ;B.Sequence results confirmed RB1 deletion;C.1 bp insertion and 5 bp deletion in RB1 gene results in premature translation termination condon;D.RB1 transcriptional expression was significantly reduced in RB1 knockout chicken preadipocyte when compared with wild-type cells.*.P<0.05,**.P<0.01,the same as below图2 CRISPR/Cas9介导RB1基因在鸡脂肪前体细胞中的敲除Fig.2 Identification of RB1 gene knockout mediated by CRISPR/Cas9 in chicken preadipocyte
2.3RB1基因促进鸡前脂肪细胞的分化
为了检测RB1基因对鸡前脂肪细胞分化的影响,分别将敲除RB1基因的鸡前脂肪细胞和野生型的鸡前脂肪细胞以6×104个·孔-1的密度传到6孔板中,待细胞稳定贴壁后,加入油酸(终浓度为320 μmol·L-1)诱导细胞分化,此时记为试验0 h。持续诱导24、48、72和96 h后进行油红O染色(图3A)并进行提取比色和细胞数目的校正。结果如图3B所示,在诱导分化24、48、72和96 h时,敲除组的油红O提取比色的OD值均极显著低于野生型组(P<0.01)。敲除组RB1基因的表达量极显著或显著(P<0.01,P<0.05)低于野生型组RB1的表达量(图3C)。

A.油红O染色(100×);B.油红O提取比色表明RB1基因敲除阻碍鸡前脂肪细胞分化过程中的脂滴形成;C.RB1基因在鸡脂肪前体细胞分化过程中表达呈上升趋势,同时RB1基因在敲除组细胞中显著低于在野生型鸡前脂肪细胞的表达;D.敲除RB1基因影响了脂肪细胞分化标志基因(G0S2、FAS、A-FABP)的表达A.Oil red O staining(100×);B.Oil red O extraction results showed that RB1 knockout impairs lipid formation;C.Expression of RB1 was increased during the differentiation of chicken preadipocyte and decreased in the RB1 knockout cells when compared with wild type cells;D.RB1 knockout decreased expression of differentiation marker genes such as G0S2,FAS,A-FABP图3 敲除RB1基因对鸡前脂肪细胞分化的影响Fig.3 Effects of RB1 knockout on chicken preadipocyte differentiation
为了研究RB1基因对鸡前脂肪细胞分化过程中成熟脂肪细胞标志基因表达的影响,在进行油红O染色的同时收集了5个时间点的细胞,利用qRT-PCR的方法检测鸡前脂肪细胞分化过程中成熟脂肪细胞标志基因(G0S2、FAS、A-FABP基因)的表达水平。结果显示,诱导分化0、24、48、72和96 h时,敲除组G0S2的表达量极显著低于野生型组(P<0.01);诱导分化48 h时,敲除组FAS的表达量显著低于野生型组(P<0.05);诱导分化48 h时,敲除组A-FABP的表达量极显著低于野生型组(P<0.01),诱导分化72 h时,敲除组A-FABP的表达量显著低于野生型组(P<0.05)(图3D)。
2.4RB1基因抑制鸡前脂肪细胞的增殖
为了检测RB1基因对鸡前脂肪细胞增殖的影响,对敲除RB1基因的鸡前脂肪细胞和野生型的细胞进行了CCK-8检测,结果显示在细胞增殖24和48 h时,敲除RB1基因的鸡前脂肪细胞的增殖速度要极显著高于野生型的鸡前脂肪细胞的增殖速度(P<0.01)(图4A)。CCK-8试验证明RB1基因抑制细胞增殖,为了探究此抑制作用是否是由细胞周期变化引起的,随后利用流式细胞仪进行了细胞周期检测,结果如图4B所示。敲除RB1基因引起鸡前脂肪细胞中G1期细胞比例显著降低(P<0.05),G2期细胞比例显著升高(P<0.05)。由此结果可以推测,鸡RB1基因对鸡前脂肪细胞的细胞周期进程具有一定的抑制作用。
为了在mRNA水平上研究RB1基因对鸡前脂肪细胞增殖相关基因的影响,分别将敲除RB1基因的鸡前脂肪细胞和野生型的鸡前脂肪细胞以每孔3×104个·孔-1的密度传到6孔板中,待细胞稳定贴壁后记为0 h,然后分别在细胞增殖24、48、72和96 h收集细胞,以β-actin为内参,利用qRT-PCR的方法分析RB1基因敲除对鸡前脂肪细胞增殖过程中重要基因(CyclinD1、E2F1、Ki67、PCNA)表达情况的影响。
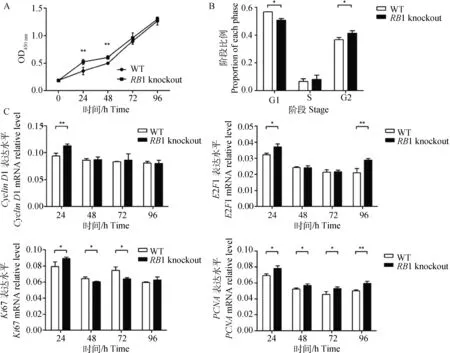
A.敲除RB1基因促进鸡前脂肪细胞数量的增加;B.敲除RB1基因降低鸡前脂肪细胞细胞G1期细胞的比例,增加了G2期细胞的比例;C.敲除RB1基因影响了鸡前脂肪细胞增殖过程中重要基因(Cyclin D1、E2F1、Ki67 and PCNA)的表达A.RB1 knockout increased chicken preadipocyte proliferation;B.RB1 knockout decreased the ratio of cells at G1 stage but increased the ratio of cells at G2 stage;C.RB1 knockout increased expression of cell proliferation associated genes such as Cyclin D1,E2F1,Ki67 and PCNA图4 敲除RB1基因对鸡前脂肪细胞增殖的影响Fig.4 Effects of RB1 knockout on chicken preadipocyte proliferation
如图4C所示,增殖24 h时,敲除组CyclinD1的表达量极显著高于野生型组(P<0.01);增殖24 h时,敲除组E2F1的表达量显著高于野生型组(P<0.05),在96 h时达到极显著水平(P<0.01);增殖24 h时,敲除组Ki67的表达量显著高于野生型组(P<0.05);增殖24、48和72 h时,敲除组PCNA的表达量显著高于野生型组(P<0.05),在96 h时达到极显著水平(P<0.01)。
3 讨 论
基因编辑技术是研究基因功能的重要工具,早期的同源重组技术虽然能对目的基因进行有效编辑,但效率极低。人工核酸酶技术的出现极大地提高了基因组编辑的效率。目前比较常见的人工核酸酶系统主要有ZFNs、TALENs和CRISPR/Cas9 3种。但ZFNs和TALENs系统在质粒构建时费时费力[8-9],而CRISPR/Cas9系统对靶点的识别依赖于gRNA与靶标DNA之间的碱基互补配对,使得质粒的构建更为简单、高效[10]。因此,本研究选用CRISPR/Cas9系统对鸡前脂肪细胞进行基因打靶。结果显示,CRISPR/Cas9系统对鸡前脂肪细胞的敲除效率为10%。这与CRISPR/Cas9系统在其他物种细胞上的敲除效率相比要低一些[11],造成打靶效率低的原因可能是由于这个系统在鸡上的应用还不够完善,还需要进一步优化。
RB1 基因已经被证明参与了许多生长发育调节过程。对人和小鼠的研究表明,RB1促进肌细胞[12]、成骨细胞分化[13]以及红细胞生成[14]。但是RB1基因对鸡前脂肪细胞分化和增殖的调控机理仍不明确。本研究利用CRISPR/Cas9系统成功构建了敲除RB1基因的鸡前脂肪细胞,为研究RB1基因的作用机制提供了有效的模型。为了确定鸡RB1基因对鸡前脂肪细胞分化的作用,本研究将RB1基因在鸡前脂肪细胞中进行了敲除,并结合油红O染色和提取比色的方法比较了敲除组和野生型组细胞的分化能力。结果显示,敲除RB1基因之后,鸡前脂肪细胞脂滴沉积能力减弱。先前有研究显示,在体外培养条件下,RB1基因所编码的蛋白pRb可促进小鼠胚胎成纤维细胞转分化成脂肪细胞[15],造成上述结果是因为在小鼠胚胎成纤维细胞中pRb能激活C/EBPs家族介导的转录途径,进而诱发脂肪细胞分化。M.Classon等[16]也发现,在3T3成纤维细胞中加入pRb,随着pRb浓度的增加,C/EBPα介导的转录被激活,使细胞进行分化,反之缺失pRb时3T3成纤维细胞表现为分化受阻。这与本试验的研究结果是一致的。本研究利用qRT-PCR的方法检测了在诱导分化的过程中脂肪细胞分化标志基因的表达情况。结果显示,诱导分化24、48、72和96 h时,敲除组G0S2基因的表达量极显著低于野生型组(P<0.01);诱导分化48 h时,敲除组的FAS基因的表达量显著低于野生型组(P<0.05);诱导分化48和72 h敲除组的A-FABP基因的表达量显著低于野生型组(P<0.05,P<0.01)。T.Ma等[17]研究表明,在小鼠上敲除G0S2基因,小鼠的脂肪沉积能力要显著的低于野生型的小鼠。H.X.Cui等[18]研究表明,FAS基因的表达决定着脂肪酸的合成能力,而脂肪酸又是脂肪的主要储存形式甘油三酯合成所必须的原料。K.Motojima[19]研究表明,A-FABP基因在哺乳动物的脂肪酸运输和脂肪细胞分化过程中发挥着重要的作用。因此A-FABP、G0S2、FAS对于脂肪的合成以及脂肪的沉积起着重要的作用,并且这3个基因也是前脂肪细胞分化成成熟脂肪细胞的重要标志基因。本研究结果显示,在鸡前脂肪细胞上敲除RB1基因之后,G0S2、FAS、A-FABP基因的表达量都呈现下降的趋势。由此可以得出结论,在鸡前脂肪细胞敲除RB1基因减弱了前脂肪细胞分化成脂肪细胞的能力,即RB1基因促进鸡前脂肪细胞的分化。
为了检测RB1基因对鸡前脂肪细胞增殖的影响,通过CCK-8的方法发现敲除RB1基因促进鸡前脂肪细胞增殖,这与对鼠坐骨神经膜细胞的研究中发现干扰RB促进细胞增长的结果[20]是一致的。在哺乳动物中,RB1主要通过与E2F作用,调控细胞周期进程,从而调节细胞的增殖[21]。由此猜测RB1基因对鸡前脂肪细胞增殖的抑制作用可能与其调节细胞周期的进程有关。本研究对细胞周期的检测结果表明,敲除RB1基因引起鸡前脂肪细胞中G1期细胞比例显著降低(P<0.05),G2期细胞比例显著升高(P<0.05)。由此可以推测,鸡RB1基因对鸡前脂肪细胞的细胞周期进程具有一定的抑制作用。利用qRT-PCR的方法检测了细胞周期相关基因的表达情况。结果显示,增殖24 h时,敲除组的CyclinD1的表达量极显著高于野生型组(P<0.01);增殖24 h时,敲除组的E2F1的表达量显著高于野生型组(P<0.05),在96 h时达到极显著水平(P<0.01);增殖24、48 和72 h时,敲除组的Ki67的表达量显著高于野生型组(P<0.05);增殖24、48和72 h时,敲除组的PCNA的表达量显著高于野生型组(P<0.05),在96 h时达到极显著水平(P<0.01)。CyclinD1为细胞周期蛋白,是细胞G1期到S期的重要调控因子[22],E2F转录因子在调控细胞周期中也具有重要角色[23-24],CyclinD1与E2F共同参与以pRb 为主的细胞周期调控网络[25]。Ki67和PCNA都是反映细胞增殖状态的良好指标[26-27]。在鸡前脂肪细胞上敲除RB1基因之后,CyclinD1、E2F1、Ki67、PCNA基因的表达量都呈现上升的趋势。由此可以得出结论,鸡RB1基因主要是通过抑制鸡前脂肪细胞的细胞周期进程,进而抑制鸡前脂肪细胞的增殖。
综上所述,本研究首次验证了CRISPR/Cas9系统在鸡这一物种上应用的可行性,即CRISPR/Cas9系统能够有效介导鸡细胞的基因敲除。另外,本研究发现RB1基因对鸡前脂肪细胞的分化具有促进作用,而对鸡前脂肪细胞的增殖具有抑制作用。本研究结果验证了笔者之前的QTL定位分析结果,同时为更加深入的研究RB1基因在鸡前脂肪细胞分化、增殖过程中所发挥的作用奠定了基础。
[1]LIU X,LI H,WANG S,et al.Mapping quantitative trait loci affecting body weight and abdominal fat weight on chicken chromosome one[J].PoultSci,2007,86(6):1084-1089.
[2]LIU X,ZHANG H,LI H,et al.Fine-mapping quantitative trait loci for body weight and abdominal fat traits:effects of marker density and sample size[J].PoultSci,2008,87(7):1314-1319.
[3]ZHANG H,ZHANG Y D,WANG S Z,et al.Detection and fine mapping of quantitative trait loci for bone traits on chicken chromosome one[J].JAnimBreedGenet,2010,127(6):462-468.
[4]HWANG W Y,FU Y,REYON D,et al.Efficient genome editing in zebrafish using a CRISPR-Cas system[J].NatBiotechnol,2013,31(3):227-229.
[5]JINEK M,EAST A,CHENG A,et al.RNA-programmed genome editing in human cells[J].Elife,2013,2:e471.
[6]BARRANGOU R,FREMAUX C,DEVEAU H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709-1712.
[7]RAN F A,HSU P D,LIN C Y,et al.Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J].Cell,2013,154(6):1380-1389.
[8]GEURTS A M,COST G J,FREYVERT Y,et al.Knockout rats via embryo microinjection of zinc-finger nucleases[J].Science,2009,325(5939):433.
[9]CERMAK T,DOYLE E L,CHRISTIAN M,et al.Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J].NucleicAcidsRes,2011,39(12):e82.
[10]CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339(6121):819-823.
[11]WANG X,ZHOU J,CAO C,et al.Efficient CRISPR/Cas9-mediated biallelic gene disruption and site-specific knockin after rapid selection of highly active sgRNAs in pigs[J].SciRep,2015,5:13348.
[12]GU W,SCHNEIDER J W,CONDORELLI G,et al.Interaction of myogenic factors and the retinoblastoma protein mediates muscle cell commitment and differentiation[J].Cell,1993,72(3):309-324.
[13]BERMAN S D,YUAN T L,MILLER E S,et al.The retinoblastoma protein tumor suppressor is important for appropriate osteoblast differentiation and bone development[J].MolCancerRes, 2008,6(9):1440-1451.
[14]SANKARAN V G,ORKIN S H,WALKLEY C R.Rb intrinsically promotes erythropoiesis by coupling cell cycle exit with mitochondrial biogenesis[J].GenesDev, 2008,22(4):463-475.
[15]CHEN P L,RILEY D J,CHEN Y,et al.Retinoblastoma protein positively regulates terminal adipocyte differentiation through direct interaction with C/EBPs[J].GenesDev, 1996,10(21):2794-2804.
[16]CLASSON M,KENNEDY B K,MULLOY R,et al.Opposing roles of pRB and p107 in adipocyte differentiation[J].ProcNatlAcadSciUSA, 2000,97(20):10826-10831.
[17]MA T,LOPEZ-AGUIAR A G,LI A,et al.Mice lacking G0S2 are lean and cold-tolerant[J].CancerBiolTher, 2014,15(5):643-650.
[18]CUI H X,ZHENG M Q,LIU R R,et al.Liver dominant expression of fatty acid synthase (FAS) gene in two chicken breeds during intramuscular-fat development[J].MolBiolRep, 2012,39(4):3479-3484.
[19]MOTOJIMA K.Differential effects of PPAR alpha activators on induction of ectopic expression of tissue-specific fatty acid binding protein genes in the mouse liver[J].IntJBiochemCellBiol, 2000,32(10):1085-1092.
[20]COLLINS M J,NAPOLI I,RIBEIRO S,et al.Loss of Rb cooperates with Ras to drive oncogenic growth in mammalian cells[J].CurrBiol, 2012,22(19):1765-1773.
[21]KREK W,EWEN M E,SHIRODKAR S,et al.Negative regulation of the growth-promoting transcription factor E2F-1 by a stably bound cyclin A-dependent protein kinase[J].Cell, 1994,78(1):161-172.
[22]COATS S,FLANAGAN W M,Nourse J,et al.Requirement of p27Kip1 for restriction point control of the fibroblast cell cycle[J].Science, 1996,272(5263):877-880.
[23]DU W,POGORILER J.Retinoblastoma family genes[J].Oncogene,2006,25(38):5190-5200.
[24]SHERR C J.Cancer cell cycles[J].Science,1996,274(5293):1672-1677.
[25]LI M,LOCKWOOD W,ZIELENSKA M,et al.Multiple CDK/CYCLIND genes are amplified in medulloblastoma and supratentorial primitive neuroectodermal brain tumor[J].CancerGenet, 2012,205(5):220-231.
[26]SANCHEZ-MUNOZ A,PLATA-FERNANDEZ Y,JAEN A,et al.Proliferation determined by ki67 marker and pCR in locally advanced breast cancer patients treated with neo-adjuvant chemotherapy[J].BreastJ, 2013,19(6):685-686.
[27]BROICH G,LAVEZZI A M,BIONDO B,et al.PCNA——a cell proliferation marker in vocal cord cancer.Part II:Recurrence in malignant laryngeal lesions[J].Invivo, 1996,10(2):175-178.
(编辑郭云雁)
CRISPR/Cas9 MediatedRB1 Knockout and Its Impact on Chicken Preadipocytes Differentiation and Proliferation
ZHANG Qi1,2,HUANG Jiao-jiao3,YANG Cai-xia1,WANG Yu-xiang1,2,ZHANG Hui1,2,ZHAO Jian-guo3,LI Hui1,2*
(1.CollegeofAnimalScienceandTechnology,NortheastAgriculturalUniversity,Harbin150030,China;2.KeyLaboratoryofChickenGeneticsandBreeding,MinistryofAgriculture,Harbin150030,China;3.StateKeyLaboratoryofStemCellandReproductiveBiology,InstituteofZoology,ChineseAcademyofSciences,Beijing100101,China)
In order to study the role ofRB1 gene on chicken preadipocyte differentiation and proliferation.CRISPR/Cas9 system was established and conducted to knockout theRB1 gene in the chicken preadipocyte.The proliferation and differentiation of chicken preadipocytes and the expression of related marker genes were detected.Results showed that the CRISPR/Cas9 system could effectively mediateRB1 knockout and cause truncated translation in chicken at the rate of 10%.Oil Red O and CCK-8 were used to evaluate the ability of differentiation and proliferation in chicken preadipocyte respectively.Knockout ofRB1 in chicken preadipocyte resulted in enhanced proliferation capability and decreased differentiation capability in chicken preadipocyte.RB1 knockout decreased the transcriptional expression of adipocyte differentiation associated genes such asG0S2,FASandA-FABP.Especially,the expression ofG0S2 was significantly decreased at all stages of preadipocyte differentiation.Further,RB1 knockout increased the expression of cell proliferation associated genes such asCyclinD1,E2F1,Ki67 andPCNA, which was corresponded to the enhanced proliferation capability in theRB1 knockout cells.In summary,RB1 may be a key regulator on differentiation and proliferation of chicken preadipocyte.
CRISPR/Cas9;RB1;chicken;fat deposition;differentiation;proliferation
10.11843/j.issn.0366-6964.2016.09.005
2016-03-22
国家自然科学基金(青年科学基金)(31301960);国家863项目(2013AA102501);现代农业产业技术体系建设专项(CARS-42)
张琦(1990-),女,黑龙江齐齐哈尔人,硕士生,主要从事家禽遗传育种研究,E-mail:zhangxiaoqi0405@163.com
李辉,教授,E-mail:lihui@neau.edu.cn
S831.2
A
0366-6964(2016)09-1775-10

