microRNA-212对胃癌细胞系SGC7901迁移和侵袭能力的影响
应江辉,蒋佩佩,金灿灿,庞文洋,蔡一奇,朱冠保
(温州医科大学附属第一医院,浙江 温州 325015,1.烧伤科;2.胃肠外科)
·论著·
microRNA-212对胃癌细胞系SGC7901迁移和侵袭能力的影响
应江辉1,蒋佩佩2,金灿灿2,庞文洋2,蔡一奇2,朱冠保2
(温州医科大学附属第一医院,浙江温州325015,1.烧伤科;2.胃肠外科)
目的:研究人microRNA-212(miR-212)对胃癌细胞系SGC7901迁移和侵袭能力的影响。方法:通过化学方法合成人成熟型miR-212抑制基因i-miR-212,分别以25、50、75、100 nmol/L浓度通过脂质体转染方法转染SGC7901细胞,同时设空白组和无序列对照组。采用Real-time PCR、细胞计数和Transwell小室等实验方法,分别检测各组细胞miR-212的表达、miR-212对细胞增殖的影响及miR-212对细胞迁移和侵袭能力的影响。结果:各浓度i-miR-212转染SGC7901细胞后,miR-212表达均降低,以50 nmol/L i-miR-212转染组最佳,较无序列对照组降低约94.60%;50 nmol/L i-miR-212转染组与无序列对照组在细胞计数上差异有统计学意义(P<0.01)。在细胞侵袭实验中50 nmol/L i-miR-212转染组透膜细胞明显多于无序列对照组(P<0.01),在迁移实验中i-miR-212转染组(50 nmol/L)的透膜细胞明显多于无序列对照组(P<0.01)。结论:转染成熟型人i-miR-212能使胃癌细胞系SGC7901细胞中miR-212的表达明显下降,并能显著促进胃癌细胞系SGC7901的增殖、迁移及侵袭能力,表明miR-212与胃癌肿瘤细胞的转移具有相关性。
胃癌;microRNA-212;迁移;侵袭;增殖
胃癌是导致人类死亡的第二大病因[1],以手术为主的综合治疗仍是胃癌目前的主要治疗方式,但是进展期胃癌术后的5年生存率仍不理想,而转移和复发是影响胃癌预后的关键因素。目前,已发现与胃癌相关的基因有数千条,如p53、MUC1、CEA、E-cadherin、p16、CD44等[2],随着基因芯片技术在胃癌分子生物学和预后分级中的广泛应用研究[3-4],microRNA(miRNA)与肿瘤发生发展机制的相关性研究已成热点。
miRNA是一种非编码蛋白的小分子RNA,它通过与靶基因3’非编码区(3’UTR)互补配对,进而促进目的基因的降解或抑制,从而参与细胞增殖、分化、凋亡、基因调控及肿瘤的发生发展[5]。随着对miRNA深入研究,miRNAs与肿瘤的关系也成为肿瘤学研究的新热点[6]。研究发现多种恶性肿瘤与正常组织之间存在miRNA的差异性表达[7-11],这些差异表达不仅见于肿瘤与正常组织之间,也见于肿瘤的不同分期之间,且与肿瘤的生物学特性及预后密切相关[12-13]。本实验室通过对胃癌组织筛选实验发现人microRNA-212(miR-212)在胃癌转移淋巴结间存在差异性表达,淋巴结转移组中miR-212的表达较无淋巴结转移组降低2~3倍[8]。
本课题通过化学方法合成成熟型的人miR-212抑制基因以瞬时转染的方法转染胃癌细胞系SGC7901细胞,并通过细胞计数试剂盒-8(cell counting kit-8,CCK-8)和Transwell小室等系列实验方法检测肿瘤细胞的增殖和侵袭能力,来比较转染组与空白组及无序列对照组之间是否存在差异,并为进一步实验明确相关生物分子学机制提供理论依据。
1 材料和方法
1.1 材料 胃癌细胞系SGC7901购自中科院上海细胞库;DMEM购自美国Gibco公司;胎牛血清(FBS)购自北京元亨圣马生物科技公司;Trizol购自美国Invitrogen公司;CCK-8试剂盒购自日本同仁化学所;人成熟型miR-212抑制基因(i-miR-212)由上海吉玛生物公司合成;转染试剂脂质体Lipofectamin 2000(lip-2000)购自美国Invitrogen公司;Matrigel购自美国BD Bioscience公司;Transwell小室购自澳洲Costar公司;PCR Marker(100 bp DNA lad-der)购自杭州吉诺生物医药技术有限公司;制冰机(AF100,SCOTSMAN,意大利);Eppendorf 5415R超速低温离心机购自德国Eppendorf公司;细胞培养箱购自德国Binder公司;分光光度仪购自德国Eppendorf公司;Thermo Multiskan MK3酶标仪购自美国Thermo Fisher Scientific公司。
1.2 细胞培养与转染及RNA提取 SGC7901细胞接种于6孔培养板中,置于5% CO2、37 ℃培养6 h至单层后进行转染。脂质体转染方法按lip-2000试剂盒说明书进行。设置i-miR-212剂量分别为25、50、75、100 nmol/L。并设置无序列对照组及空白组。采用Trizol法提取RNA。提取的总RNA使用紫外分光光度计测量获得260 nm和280 nm波长下的吸光光度值(A值)。A260/A280>1.8为RNA质量合格,用于下一步实验。
1.3 Real-time PCR检测转染细胞的miR-212表达
分别提取i-miR-212转染各浓度组、空白组和无序列对照组细胞总RNA,进行Real-time PCR测定,每样品测3次。包括2个步骤:①RNA反转录合成反应:将1 μg总RNA反转录合成cDNA,10 μL反转录反应体系包括2 μL RT Buffer、0.5 μL RT Enzyme Mix、0.5 μL Primer Mix,并根据总RNA加入量调解去RNA酶的双蒸水至10 μL。内参miR-let7也同时进行了反转录。反应条件为:16 ℃ 30 min,42 ℃ 30 min,75 ℃ 15 min,将反应产物对倍稀释至20 μL后置于-20 ℃保存。②参考文献[14]中的PCR反应方法:以hsa-miR-let7a作为内参,检测miRNA的表达量。利用miRNA特异的正向、反向引物和染料分子标记探针对茎环RT-PCR反应产物进行定量检测,反应条件如下:95 ℃ 2 min,95 ℃ 15 s,60 ℃ 1 min,40循环。同时加做溶解曲线以验证每个PCR反应产物的特异性。该方法将目的基因与内参基因的CT值比较(△CT)后,再将实验组与空白组比较(△△CT)。故△△CT=样品的[(hsa-miR-212)CT-(let7a)CT]-空白组第1次检测的[(hsa-miR-212)CT-(let7a)CT]。
1.4 肿瘤细胞的增殖能力检测 参照CCK-8试剂盒说明书进行:①将转染24 h的SGC7901细胞以及对照组分别按每孔7×103个接种于96孔培养板(每孔100 μL)中。每组分别接种7孔以作为重复对照。②24 h后,待细胞贴壁后加入100 μL DMEM完全培养液、10 μL CCK-8试剂,置于5% CO2、37 ℃培养箱中培养1 h。③酶标仪于490 nm处测OD值。
1.5 肿瘤细胞的侵袭能力检测 按Transwell说明书操作:①冰上将matrigel与无血清培养液1∶1稀释混匀。②将marigel膜加入Transwell小室,37 ℃下凝固成固态后备用。③SGC7901细胞经胰酶消化后用无血清培养液洗2次并计数。④向Transwell小室上室内加入细胞悬液(含10万个细胞),加入无血清DMEM稀释至400 μL,各个浓度组设3个复孔。⑤下室加入600 μL含10% FBS的DMEM完全培养液。造成上下室之间的浓度梯度,趋化细胞向下室侵袭。⑥37 ℃、5% CO2培养24 h后吸去嵌室内的液体,用棉棒将上室表面上的细胞擦去。⑦将嵌室浸入50%甲醇中固定15 min,PBS洗3遍。结晶紫染色30 min,风干。⑧荧光显微镜下每个培养孔随机选5个视野拍照并计算各视野细胞数,各个浓度组设3个复孔。细胞计数方法:每个小室显微镜下随机选取5个视野,计算出染色细胞数量,每组3个重复实验。
1.6 统计学处理方法 采用SPSS16.0统计学软件进行数据分析。采用t检验。P<0.05为差异有统计学意义。
2 结果
2.1 i-miR-212转染后SGC7901细胞中miR-212的表达 i-miR-212转染组的2-△△CT值在25、50、75、100 nmol/L时分别为0.1544±0.0011、0.0549± 0.0010、0.0479±0.0016、0.0288±0.0006,空白组及无序列对照组2-△△CT值分别为1.0177±0.0514、0.9807±0.0448;转染i-miR-212 25、50、75、100 nmol/L后SGC7901细胞miR-212表达量明显下降,分别下降84.82%、94.60%、95.53%及97.17%。考虑到转染剂量过大对细胞毒性作用,遂采用50 nmol/L作为正式转染浓度。见图1。
2.2 i-miR-212转染后对SGC7901细胞增殖能力的影响 i-miR-212转染24 h后SGC7901细胞的OD值明显高于空白组和无序列对照组,差异有统计学意义(P<0.01),无序列对照组和空白组之间差异无统计学意义(P>0.05)。CCK-8实验表明抑制miR-212能明显提高SGC7901细胞的增殖能力。见表1。
2.3 i-miR-212转染后对SGC7901细胞侵袭能力的影响 i-miR-212转染24 h后,采用Transwell小室侵袭实验比较各组之间的侵袭力差别。结果显示i-miR-212转染SGC7901细胞后穿膜数明显高于空白组和无序列对照组,差异有统计学意义(P<0.01)。无序列对照组和空白组之间差异无统计学意义(P>0.05)。实验表明,抑制miR-212的表达可明显提高细胞的侵袭力。见表2和图2。

图1 Real-time PCR下不同组别之间的miR-212表达量
表1 各组SGC7901细胞增殖能力比较(±s)

表1 各组SGC7901细胞增殖能力比较(±s)
与无序列对照组比:aP<0.01;与空白组比:bP<0.01
组别样本量(孔)OD值i-miR-212转染组71.380±0.070ab无序列对照组71.180±0.038空白组71.167±0.081
表2 各组SGC7901细胞侵袭能力比较(±s)

表2 各组SGC7901细胞侵袭能力比较(±s)
与无序列对照组比:aP<0.01;与空白组比:bP<0.01
组别样本量(孔)透膜细胞数i-miR-212组15152.933±3.283ab无序列对照组15 46.800±3.877空白组15 44.600±4.068
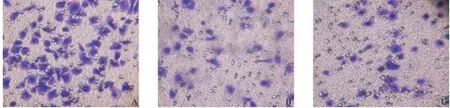
图2 显微镜下各组SGC7901细胞侵袭能力结果显示(×200)
2.4 i-miR-212转染后对SGC7901细胞迁移能力的影响 i-miR-212转染24 h后,采用Transwell小室迁移实验对比各组细胞之间的迁移能力。结果显示转染i-miR-212后SGC7901细胞跨膜细胞数明显多于空白组和无序列对照组,差异有统计学意义(P<0.01)。无序列对照组和空白组差异无统计学意义(P>0.05)。实验表明抑制miR-212的表达明显提高细胞的迁移力。见表3和图3。
表3 各组SGC7901细胞迁移能力比较(±s)

表3 各组SGC7901细胞迁移能力比较(±s)
与无序列对照组比:aP<0.01;与空白组比:bP<0.01
组别样本量(孔)透膜细胞数i-miR-212组15243.600±7.029ab无序列对照组15 75.333±3.994空白组15 76.000±3.066
3 讨论
miRNA作为癌基因和抑制癌基因调控肿瘤的发生发展过程已被学者广泛认同,不断有研究报道miRNA与肿瘤的侵袭、转移等生物学效应相关。有研究[15]报道在B细胞淋巴瘤中miRNA-17-92可通过抑制蛋白E2F1(具有诱导凋亡作用)表达,促进肿瘤的发生。miRNA-21通过调控抑癌基因PTEN的表达,影响金属蛋白酶和磷酸激酶表达,促进肝癌的增殖、迁移和侵袭[16]。miR-196a-2在胰腺癌中表达提示预后较差[17-18]。在胃癌中有学者已经发现miR-10A表达上调能增加胃癌细胞的侵袭能力[19],miR-29表达下调能显著抑制胃癌细胞的侵袭、迁移及增殖能力[20]。本研究前期筛选实验发现miR-212、miR-125a-5p、miR-143、miR-15b、miR-199a-3p、miR-199a-5在胃癌中均有差异性表达[8]。

图3 显微镜下各组SGC7901细胞迁移能力结果显示(×200)
本研究通过转染i-miR-212从而使胃癌细胞系SGC7901中miR-212表达下降,并通过CCK-8及Transwell小室等实验监测i-miR-212转染后SGC7901细胞的增殖、迁移及侵袭能力,实验结果证明转染imiR-212后SGC7901细胞增殖、迁移及侵袭能力显著提高,与前期筛选实验相互印证,并为后期分子生物学研究提供理论基础。miR-212作用于胃癌的机制目前尚不明确,有研究[21]报道miR-212可能是通过直接结合MeCP2的3’-UTR区域,从而靶向调节甲基化CpG结合蛋白MeCP2,影响细胞甲基化过程。MeCP2是甲基化结合蛋白(MBDs)家族的重要一员,是一类与甲基化CpG二核苷酸结合的核蛋白。它对DNA的甲基化过程相当重要,因为它直接参与调节表观遗传。它有2个功能区域,85个氨基酸的甲基化蛋白直接结合5-甲基胞嘧啶,104个氨基酸的转录抑制域影响组蛋白去乙酰化酶[22]。miR-212还可能通过作用于凋亡蛋白PED而增加肿瘤对肿瘤凋亡坏死因子相关凋亡诱导配体的敏感性[23-25]。miR-212是否还存在其他作用机制目前仍不清楚,有待进一步分子生物学研究来明确。
以上实验证实了miR-212在胃癌侵袭、迁移中的作用,并且为miRNA作为基因靶向治疗提供了新证据,相信随着基因技术的不断发展及模式物种cDNA文库的不断完善,miRNA不仅可以作为新的生物学标准为肿瘤的早期发现及预后提供预测,并且可以作为新的靶向药物应用于临床中。
[1] BRENNER B, HOSHEN M B, PURIM O, et al. MicroRNAs as a potential prognostic factor in gastric cancer[J]. World J Gas Troenterol, 2011, 17(35): 3976-3985.
[2] CHEN C N, LIN J J, CHEN J J, et al. Gene expression profile predicts patient survival of gastric cancer after surgical resection[J]. J Clin Oncol, 2005, 23(29): 7286-7295.
[3] LUO H, ZHANG H, ZHANG Z, et al. Down-regulated miR-9 and miR-433 in human gastric carcinoma[J]. J Exp Clin Cancer Res, 2009, 28: 82.
[4] KATADA T, ISHIGURO H, KUWABARA Y, et al. microRNA expression profile in undifferentiated gastric cancer[J]. Int J Oncol, 2009, 34(2): 537-542.
[5] MALLORY A C, VAUCHERET H. Functions of microRNAs and related small RNAs in plants[J]. Nat Genet, 2006, 38(Suppl): S31-S36.
[6] 季菊玲, 张锦生. microRNA与肿瘤[J]. 中华病理学杂志, 2006, 35(10): 628-630.
[7] GUO J, MIAO Y, XIAO B, et al. Differential expression of microRNA species in human gastric cancer versus non-tumorous tissues[J]. J Gastroenterol Hepatol, 2009, 24(4):652-657.
[8] WU W Y, XUE X Y, CHEN Z J, et al. Potentially predictive microRNAs of gastric cancer with metastasis to lymph node[J]. World J Gastroenterol, 2011, 17(31): 3645-3650.
[9] LIU L, TANG H, LANG Y, et al. MicroRNA-27a functions as an oncogene in gastric adenocarcinoma by targeting prohibitin[J]. Cancer Lett, 2009, 273(2): 233-242
[10] DU Y, XU Y, DING L, et al. Down-regulation of miR-141 in gastric cancer and its involvement in cell growth[J]. Gastroenterol, 2009, 44(6): 556-561
[11] BANDRES E, BITARTE N, ARIAS F, et al. microRNA-451 regulates macrophage migration inhibitory factor production and proliferation of gastrointestinal cancer cells[J]. Clin Cancer Res, 2009, 15(7): 2281-2290.
[12] LEE E J, GUSEV Y, JIANG J, et al. Expression profilingidentifies microRNA signature in pancreatic cancer[J]. Int J Cancer, 2007, 120(5): 1046-1054.
[13] NEGRINI M, CALIN G A. Breast cancer metastasis: a microRNA story[J]. Breast Cancer Res, 2008, 10(2): 203.
[14] INOMATA M, TAGAWA H, GUO Y M, et al. MicroRNA-17-92 down-regulates expression of distinct targets in different B-cell lymphoma subtypes[J]. Blood, 2009, 113(2): 396-402.
[15] HAUCK L, VONHARSDORF R. E2F transcription factors and pRb pocket proteins in cell cycle progression[J]. Methods Mol Biol, 2005, 296: 239-245.
[16] MENG F, HENSON R, WEHBE-JANEK H, et al. MicroRNA-21 regulates expression of the PTEN tumor suppressor gene in human hepatocellular cancer [J]. Gastroenterology,2007, 133(2):647-658.
[17] BLOOMSTON M, FRANKEL W L, PETROCCA F, et al. MicroRNA expression patterns to differentiate pancreatic adenocarcinoma from normal pancreas and chronic pancreatitis[J]. JAMA, 2007, 297(17): 1901-1908.
[18] 韩玉萍, 秦玉花, 杨红亚, 等. miRNA与肿瘤[J]. 生命的化学, 2009, 29(2): 252-256.
[19] 彭亮, 潘健, 胡海, 等. microRNA-10a对胃癌细胞系BGC823迁移和侵袭能力的影响[J]. 中国肿瘤生物学杂志, 2008, 15(5): 417-421.
[20] LANG N, LIU M, TANG Q L, et al. Effects of microRNA-29 family members on proliferation and invasion of gastric cancer celllines[J], Chin J Cancer, 2010, 29(6): 603-610.
[21] WADA R, AKIYAMA Y, HASHIMOTO Y, et al. miR-212 is downregulated and suppresses methyl-CpG-binding protein MeCP2 in human gastric cancer[J]. Int Journal of Cancer,2010, 127(5): 1106-1114.
[22] SANSOM O J, MADDISON K, CLATKE A R. Mechanisms of disease: methyl-binding domain proteins as potential therapeutic targets in cancer[J]. Nat Clin Pract Oncol, 2007, 4(5): 305-315.
[23] INCORONATO M, GAROFALO M, URSO L, et al. miR-212 increases tumor necrosis factor-related apoptosis-inducing ligand sensitivity in non-small cell lung cancer by targeting the antiapoptotic protein PED[J]. Cancer Res, 2010,70(9): 3638-3646.
[24] PARK J K, HENRY J C, JIANG J M, et al. miR-132 and miR-212 are increased in pancreatic cancer and target the retinoblastoma tumor suppressor[J]. Biochem Biophys Res Commun, 2011, 406, 518-523.
[25] REMENYI J, HUNTER C J, COLE C, et al. Regulation of the miR-212/132 locus by MSK1 and CREB in response to neurotrophins[J]. Biochem J, 2010, 428(2): 281-291.
(本文编辑:吴昔昔)
Effects of microRNA-212 on migration and invasion of gastric cancer cell lines SGC7901
YING Jianghui1,JIANG Peipei2, JIN Cancan2, PANG Wenyang2, CAI Yiqi2, ZHU Guanbao2. 1.Department of Burn Surgery, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou, 325015; 2.Department of Gastroenterological Surgery, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou, 325015
Objective: To investigate the effect of human microRNA212 (miR-212) on the migration and invasion of gastric cancer cell line SGC7901. Methods: The i-miR-212 as the suppressor gene of miR-212 was transfected into SGC7901 cells according to different concentration (25, 50, 75, 100 nmol/L) via lipofectamin 2000. Empty vectors and unrelated fragment were also applied as controls. The expression of mature type miR-212 was detected by real-time PCR. Cell Counting Kit-8 was used to evaluate the effect of miR-212 on the proliferation of SGC-7901 cells, meanwhile Transwell assay for migration and invasion of SGC7901 cells. Results: Real-time PCR showed that cells Transfected with i-miR-212 had significantly low expression of miR-212, with the optimal concentration of miR-212 being 50 nmol/L; the expression of miR-212 was 18 times lower than empty group (P<0.01). The Cell Counting Kit-8 showed that the proliferation of cancer cells with i-miR-212(50 nmol/L) was higher than that of the control group (P<0.01). The Transwells showed the migration and invasion of cancer cells with i-miR-212 (50 nmol/L) was higher than that of the control group (P<0.01), respectively. Conclusion: The i-miR-212 can effectively reduce miR-212 expression and promote the migration and invasion of SGC7901 cells. miR-212 may be a tumor suppressor gene in gastric cancer tumor cells.
gastric cancer; microRNA-212; migration; invasion; proliferation
R73-37;R735.2
A
10.3969/j.issn.2095-9400.2016.09.005
2015-10-20
浙江省自然科学基金资助项目(Y2100660)。
应江辉(1986-),男,浙江温州人,住院医师,硕士。
朱冠保,主任医师,教授,Email:zgbwmc@126.com。

